DOI: https://doi.org/10.1016/j.ccell.2023.12.021
PMID: https://pubmed.ncbi.nlm.nih.gov/38242124
تاريخ النشر: 2024-01-18
أنماط الخلايا الليفية المرتبطة بالسرطان مرتبطة بنتائج المرضى في سرطان الرئة غير صغير الخلايا
مقالة
أنماط الخلايا الليفية المرتبطة بالسرطان مرتبطة بنتائج المرضى في سرطان الرئة غير صغير الخلايا
الملخص
الملخص على الرغم من التقدم في العلاج، تظل معدلات البقاء على قيد الحياة في سرطان الرئة منخفضة. سيساعد فهم أفضل للتنوع الخلوي والتفاعل بين الخلايا الليفية المرتبطة بالسرطان (CAFs) داخل بيئة الورم الدقيقة في تطوير العلاجات الشخصية. نحن نقدم تحليل تصوير الخلايا الفردية بدقة مكانية (IMC) للخلايا الليفية المرتبطة بالسرطان في مجموعة سرطان الرئة غير صغير الخلايا تضم 1,070 مريضًا. نحدد أربع مجموعات مرضى تنبؤية بناءً على 11 نمطًا من الخلايا الليفية المرتبطة بالسرطان مع توزيعات مكانية مميزة ونظهر أن الخلايا الليفية المرتبطة بالسرطان هي عوامل تنبؤية مستقلة لبقاء المرضى. وجود خلايا ليفية شبيهة بالورم مرتبط ارتباطًا وثيقًا بتشخيص سيء. بالمقابل، ترتبط الخلايا الليفية الالتهابية وخلايا CAFs المستجيبة للإنترفيرون ببيئات ورمية ملتهبة وزيادة بقاء المرضى. الكثافة العالية من خلايا CAFs المصفوفة مرتبطة بانخفاض في التسلل المناعي وترتبط سلبًا ببقاء المرضى. باختصار، تحدد بياناتنا الميزات النمطية والمكانية لخلايا CAFs المرتبطة بنتائج المرضى في NSCLC.
المقدمة
تعتبر حجرة الستروما أيضًا مهمة لتقدم السرطان ونجاح العلاج ولكنها ليست مفهومة جيدًا.
النتائج
تحليل IMC لعينات سرطان الرئة غير صغير الخلايا
أنماط CAF في سرطان الرئة غير صغير الخلايا
خلية سرطانية
مقالة
سيل برس
| الجدول 1. وصف المجموعة السريرية | |
| جنس | |
| ذكر | 710 (66.4%) |
| أنثى | 348 (32.5%) |
| مفقود | 12 (1.1%) |
| عمر | |
| المتوسط (الانحراف المعياري) | 64.0 (9.96) |
| الوسيط [الحد الأدنى، الحد الأقصى] | 64.5 [18.0, 87.0] |
| مفقود | 12 (1.1%) |
| نوع الورم | |
| سرطان الغدد | 618 (57.8%) |
| سرطان الخلايا الحرشفية | 401 (37.5%) |
| سرطان الخلايا الحرشفية الغدي | 32 (3.0%) |
| سرطان الخلايا الكبيرة | 9 (0.8%) |
| سرطان الرئة غير صغير الخلايا | 2 (0.2%) |
| درجة | |
| 1 | 62 (5.8%) |
| 2 | 481 (45.0%) |
| ٣ | 515 (48.1%) |
| مفقود | 12 (1.1%) |
| الحجم [سم] | |
| المتوسط (الانحراف المعياري) | ٤.٠٤ (٢.٢٦) |
| الوسيط [الحد الأدنى، الحد الأقصى] | 3.50 [0.300, 16.0] |
| مفقود | 12 (1.1%) |
| ت | |
| 1 | 131 (12.2%) |
| 2 | 129 (12.1%) |
| ٣ | 381 (35.6%) |
| ٤ | 118 (11.0%) |
| ٥ | 215 (20.1%) |
| ٦ | 84 (7.9%) |
| مفقود | 12 (1.1%) |
| ن | |
| 0 | 555 (51.9%) |
| 1 | 268 (25.0%) |
| 2 | 220 (20.6%) |
| ٣ | 14 (1.3%) |
| مفقود | 13 (1.2%) |
| M | |
| 0 | 967 (90.4%) |
| 1 | 71 (6.6%) |
| 2 | 19 (1.8%) |
| مفقود | 13 (1.2%) |
| مرحلة | |
| 1 (IA) | 182 (17.0%) |
| 2 (IB) | 191 (17.9%) |
| 3 (IIA) | 191 (17.9%) |
| 4 (IIB) | 113 (10.6%) |
| 5 (IIIأ) | 289 (27.0%) |
| 6 (IIIB) | 32 (3.0%) |
| 7 (الرابع) | 60 (5.6%) |
| الجدول 1. متابعة | |
| مفقود | 12 (1.1%) |
| العلاج الكيميائي (المساعد قبل الجراحة) | |
| لا علاج كيميائي مسبق | 961 (89.8%) |
| العلاج الكيميائي المساعد قبل الجراحة | 78 (7.3%) |
| مفقود | 31 (2.9%) |
| العلاج الإشعاعي (ما قبل الجراحة) | |
| لا علاج إشعاعي مسبق | ١٠٣٥ (٩٦.٧٪) |
| العلاج الإشعاعي المساعد | 4 (0.4%) |
| مفقود | 31 (2.9%) |
| انتكاسة | |
| لا انتكاسة | 555 (51.9%) |
| انتكاسة | 480 (44.9%) |
| مفقود | 35 (3.3%) |
| البقاء العام | |
| المتوسط (الانحراف المعياري) | 1490 (1190) |
| الوسيط [الحد الأدنى، الحد الأقصى] | 1230 [0, 5730] |
| مفقود | 2 (0.2%) |
| البقاء على قيد الحياة خالٍ من المرض | |
| المتوسط (الانحراف المعياري) | 1430 (1220) |
| الوسيط [الحد الأدنى، الحد الأقصى] | 1060 [0, 5100] |
| مفقود | 138 (12.9%) |
| حالة التدخين | |
| لا | 86 (8.0%) |
| حالياً | 730 (68.2%) |
| سابق | ٢٦ (٢.٤٪) |
| غير معروف | 197 (18.4%) |
| مفقود | 31 (2.9%) |
| العلاج الكيميائي (المساعد) | |
| نعم | 138 |
| لا | 901 |
| مفقود | 31 |
كان هناك وفرة أكبر من tCAFs في LUAD، بينما كانت tCAFs المهيجة، وPDPN CAFs، وmCAFs متزايدة في LUSC (الشكل S3A). وجدنا أيضًا اختلافات طفيفة بين أنماط النمو المختلفة لـ LUAD، حيث كانت mCAFs وcollagen CAFs متزايدة في الأورام الصلبة والميكروبابيلية، مما يتماشى مع المحتوى النسجي النسبي العالي لهذه الأنواع من الأورام (الشكل S3B). تم تجميع المرضى في أربع مجموعات بناءً على نسب أنواع CAF (الأشكال 3E، 3F، وS3C)، حيث كانت المجموعة 1 غنية بـ SMA CAFs، والمجموعة 2 غنية بـ matrix وcollagen CAFs، والمجموعة 3 تحتوي على عدة أنواع من CAF بما في ذلك tCAFs، وhypoxic CAFs، وhypoxic tCAFs وvCAFs، والمجموعة 4 تحتوي على iCAFs وifnCAFs.
أنماط CAF هي عامل تنبؤ مستقل في سرطان الرئة غير صغير الخلايا
خلية سرطانية
مقالة

| علامة CAF | علامة CAF | علامة مناعية | البطانة |
| PDPN | p75 | CD45RA/R0 (المناعة) | VWF/ CD31 (الدم) |
| IDO (إذا كان CAF) | CXCL13 | CD3 (خلايا T) | PNAd (HEV) |
| VCAM | CD146 (vCAF) | خلايا CD4 (T) | LYVE-1 (ليمفاوي) |
| CAIX (CAF ناقص الأكسجين) | PDGFR-b | خلايا CD8 (T) | VCAM-1 (وعاء) |
| CXCL12 | CCL21 (rCAF) | فوكس بي 3 (الخلايا التائية التنظيمية) | CCL21 (لمفاوي) |
| CD10 (tCAF) | CD73 (tCAF) | PD-1 (تنشيط خلايا T) | علامة ورمية |
| كاديرين-11 | MMP11 (mCAF) | TCF1/7 (خلايا T) | بان سايتوكراتين |
| فاب | CD248 (iCAF) | IDO (استجابة IFN) | CAIX (نقص الأكسجين) |
| SMA | CAIX (نقص الأكسجين) | CD68 (البلاعم) | |
| FSP-1 | IDO (استجابة IFN) | MPO + MMP9 (N.) | |
| فيمنتين | CD20 (خلايا B) | علامة عامة | |
| كي-67 (dCAF) | CD11b (الخلية النخاعية) | هيستون H3 (DNA) | |
| فيبرونيكتين + كولاجين 1 | CD15 (العدلات) | كي-67 (الانقسام) | |
| CD34 (iCAF) | HLA-DR (نخاعي) | كافولين-1 |
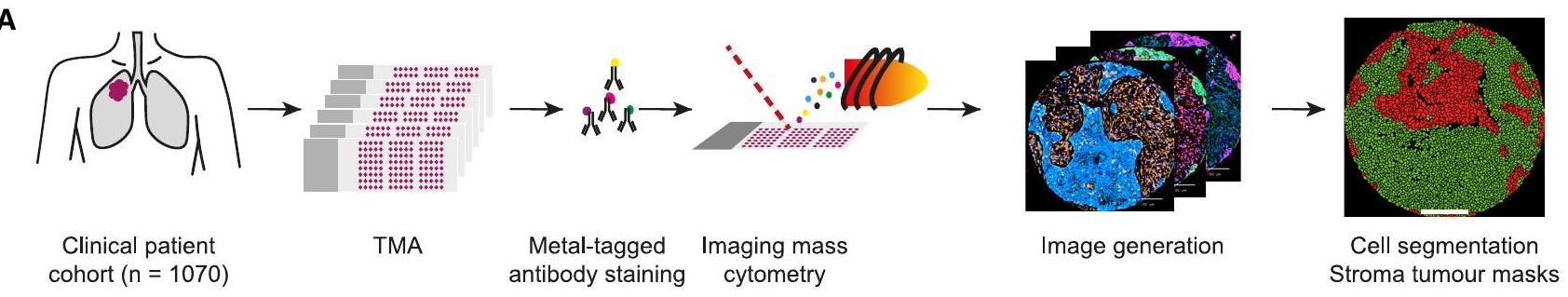
(أ) إعداد التجربة. تم صبغ عينات سرطان الرئة غير صغير الخلايا (2070 نواة، 1070 مريض) والشواهد باستخدام أجسام مضادة موسومة بالمعادن وتم تصويرها باستخدام IMC. تم إنشاء أقنعة للخلايا والورم-الستروما.
(ب) الإعداد التحليلي. تم استخدام بيانات الخلايا الفردية لتحديد أنماط الخلايا، ولتقييم الارتباطات بين أنواع الخلايا والبيانات السريرية. تم استخدام تصنيفات المرضى المعتمدة على خلايا CAF لتحليل البقاء وتركيب TMA. أخيرًا، تم تحليل الخصائص المكانية لخلايا CAF وأنواع الخلايا المناعية ودمجها مع البيانات السريرية.
(ج) علامات في لوحة الأجسام المضادة.
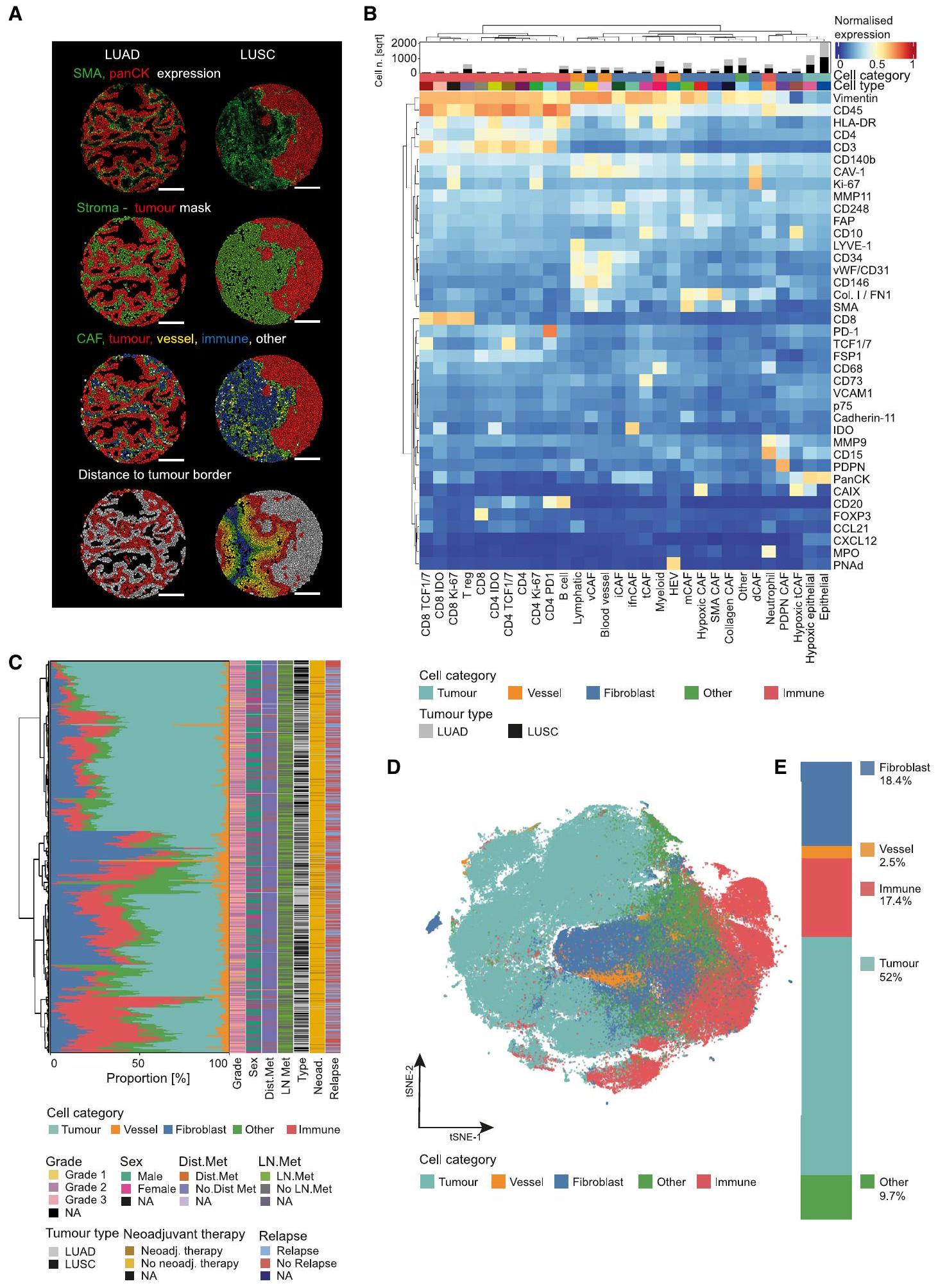
(أ) أنوية LUAD النموذجية (يسار) و LUSC (يمين) بعد تقسيم الخلايا بناءً على panCK كعلامة ورمية وSMA كعلامة دعامية. من الأعلى إلى الأسفل، الخلايا ملونة حسب متوسط تعبير SMA وpanCK؛ تعيين الخلايا بناءً على الأقنعة الناتجة عن الورم والدعامات؛ تعيين فئة الخلايا؛ والمسافة المجمعة لجميع الخلايا غير الورمية إلى أقرب حدود ورم-دعامي.
التشخيص (tCAFs، tCAFs الناجمة عن نقص الأكسجين، CAF الكولاجين، وmCAFs) أو التنبؤ الأفضل (ifnCAFs، iCAFs، وSMA CAFs) في سرطان الرئة غير صغير الخلايا.
ترتبط أنواع CAF بمقاومة العلاج الكيميائي والانتقال.
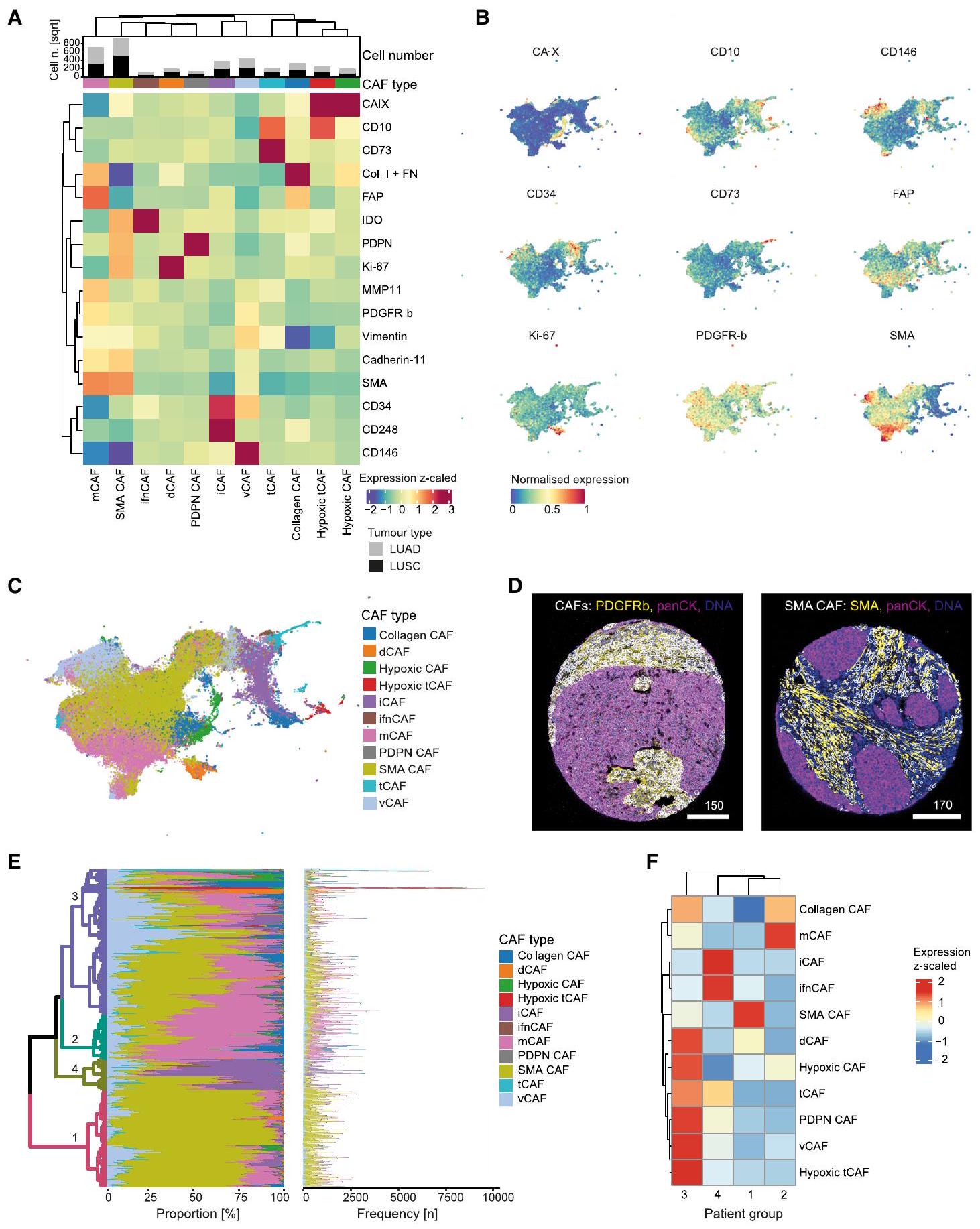
(أ) خريطة حرارية للتعبير المتوسط للعلامات (مقيسة من -2 إلى 3) لجميع أنماط CAF التي تم تحديدها بعد تجميع FLOWSOM. يوضح الرسم البياني الشريطي فوق الخريطة الحرارية العدد (الجذر التربيعي للتصور) من CAFs لكل نوع ورم لكل مجموعة.
(ب) UMAP لمجموعة فرعية عشوائية (
(ج) UMAP لمجموعة فرعية عشوائية (
(د) صور تظهر جميع CAFs (يسار) و CAFs SMA (يمين)؛ يتم عرض CAFs مع أقنعة بيضاء مغطاة على العلامات المحددة.
يتم عرض النسب المتوسطة (الرسم البياني الأيسر) والأرقام المطلقة (الرسم البياني الأيمن) لكل نوع من أنواع CAF لكل مريض. تشير شريط الألوان (اليمين) إلى أنواع CAF. يتم تلوين شجرة التجميع الهرمي (بمقياس ألوان مختلف عن أنواع CAF) بواسطة أربع مجموعات مرضى متجمعة (1، 2، 3، 4).
خريطة حرارية (تعبير مقاس z) تظهر إثراء كل نوع من أنواع CAF لكل مجموعة مرضى كما هو محدد في E.
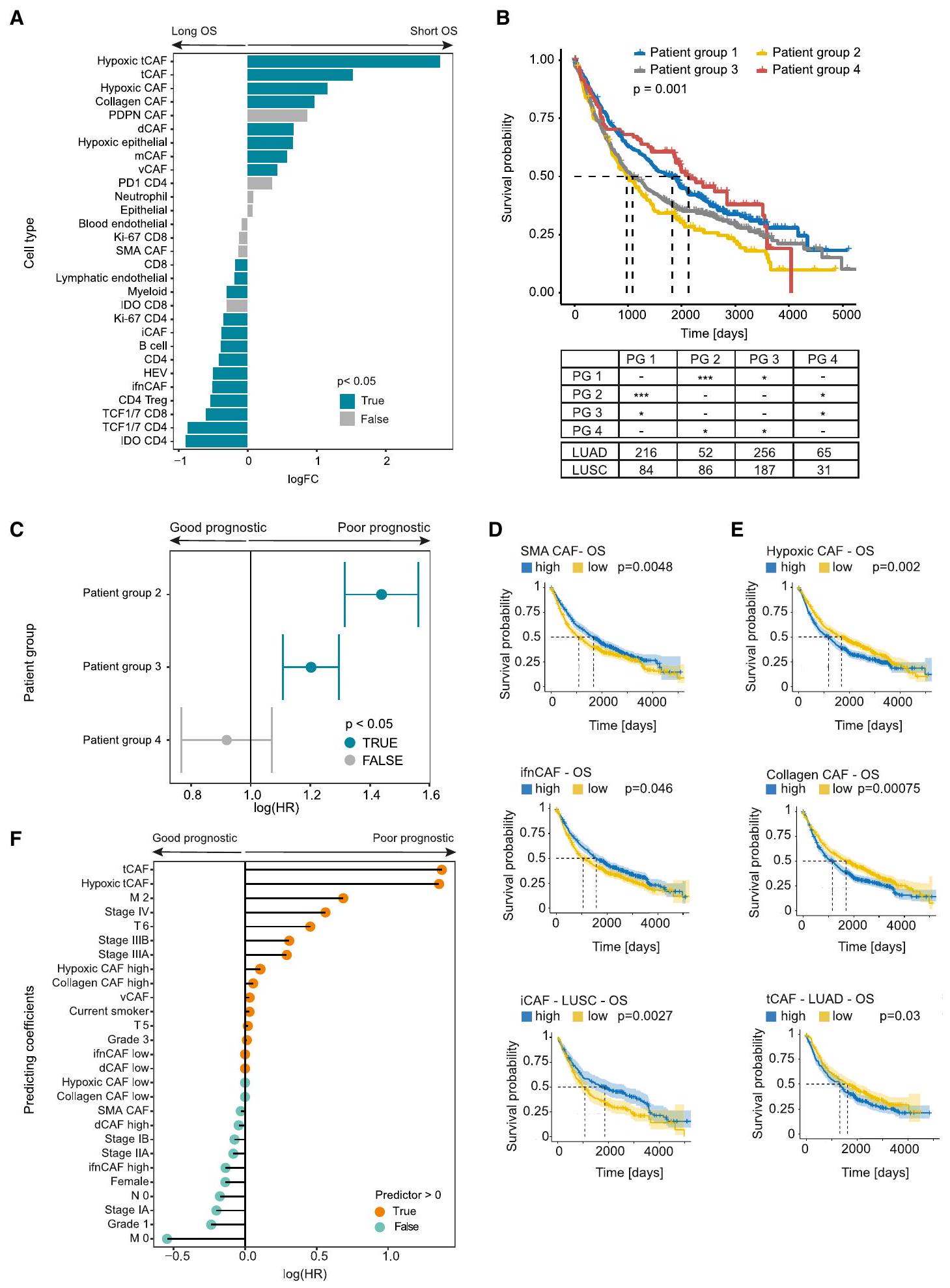
(أ) اختبار وفرة الاختلاف لجميع أنواع الخلايا بين مجموعات المرضى المحددة من خلال البقاء على قيد الحياة الطويل (يسار) والقصير (يمين)، مقسومًا حسب الوسيط. قيم p
(ب) منحنيات بقاء كابلان-ماير العامة لمجموعات المرضى (PG) المحددة بناءً على تركيبة CAF (PG 1-4). الجدول يوضح أعداد المرضى وفقًا لنوع الورم وقيم p للمقارنة بين المجموعات.
أنواع CAF مرتبطة بتراكيب TME مميزة
iCAFs كانت مرتبطة عكسيًا بشكل كبير، وإن كان ضعيفًا، مع جميع الخلايا المهيجة (LUAD
توزيعات مكانية محددة لـ CAFs داخل TME
خلايا السرطان
المقالة
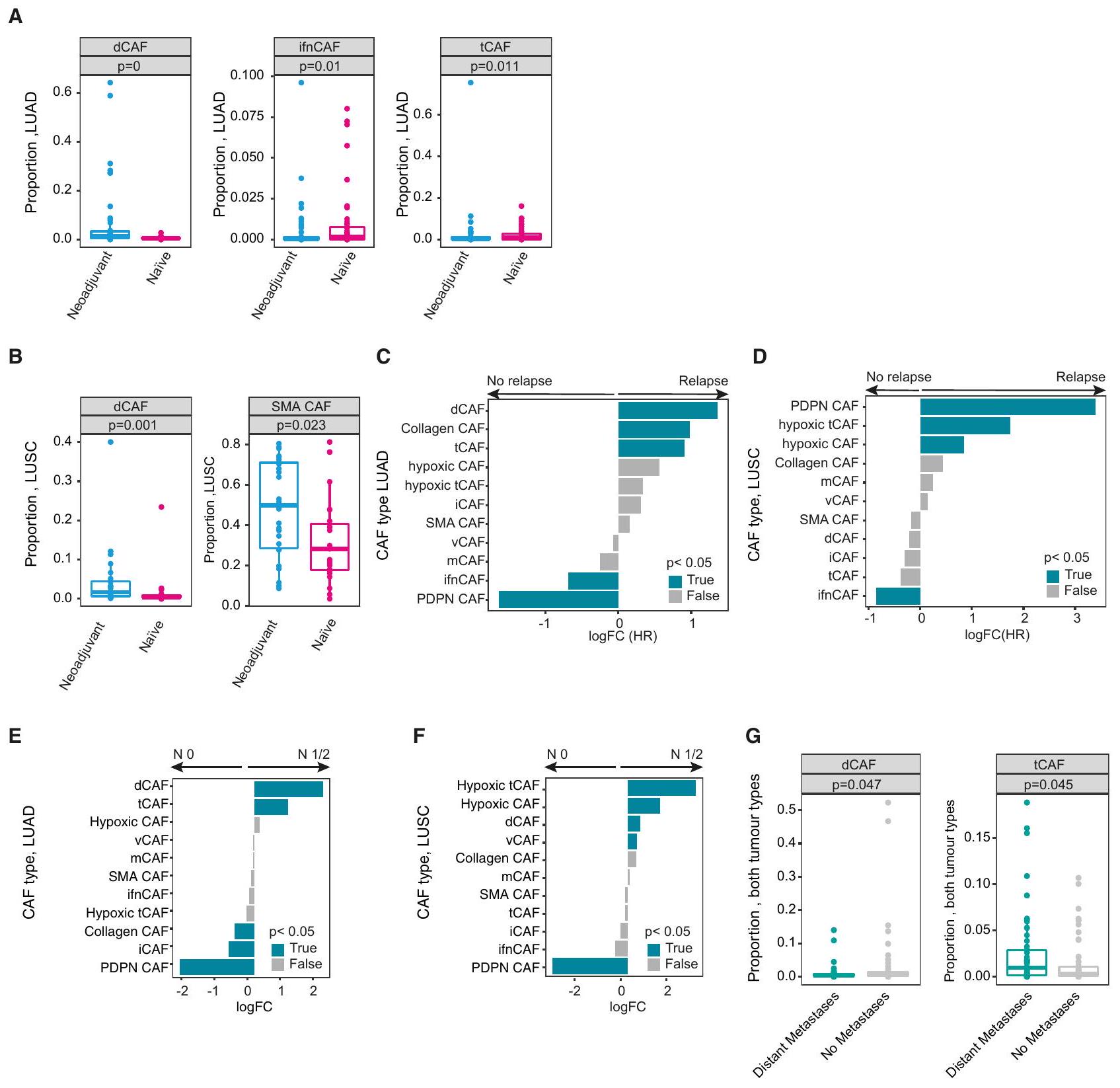
(أ و ب) مخططات صندوقية تظهر متوسط نسب المرضى لأنواع CAF المحددة مقارنة بالمرضى الذين تلقوا علاجًا مسبقًا مقابل مجموعة علاج مطابقة غير معالجة لـ LUAD (أ) و LUSC (ب). القيم الدقيقة لـ p موضحة.
(ج و د) اختبار وفرة تفاضلية لجميع أنواع CAF مقارنة بالمرضى الذين يتلقون العلاج الكيميائي المساعد الذين انتكسوا أو لم ينتكسوا، في LUAD (ج) و LUSC (د). القيم p < 0.05 موضحة باللون الأزرق، القيم p > 0.05 باللون الرمادي.
(هـ و و) اختبار وفرة تفاضلية لجميع أنواع CAF مقارنة بالمرضى الذين لديهم (N1/2) وبدون (N0) عقد لمفاوية عند التشخيص، في LUAD (هـ) وفي LUSC (و). القيم p < 0.05 موضحة باللون الأزرق، القيم p > 0.05 باللون الرمادي.
(ز) مخطط صندوقي يظهر متوسط النسب لكل مريض من أنواع CAF المحددة في مجموعات المرضى المطابقة مع وبدون نقائل بعيدة عند التشخيص. القيم الدقيقة لـ p موضحة.
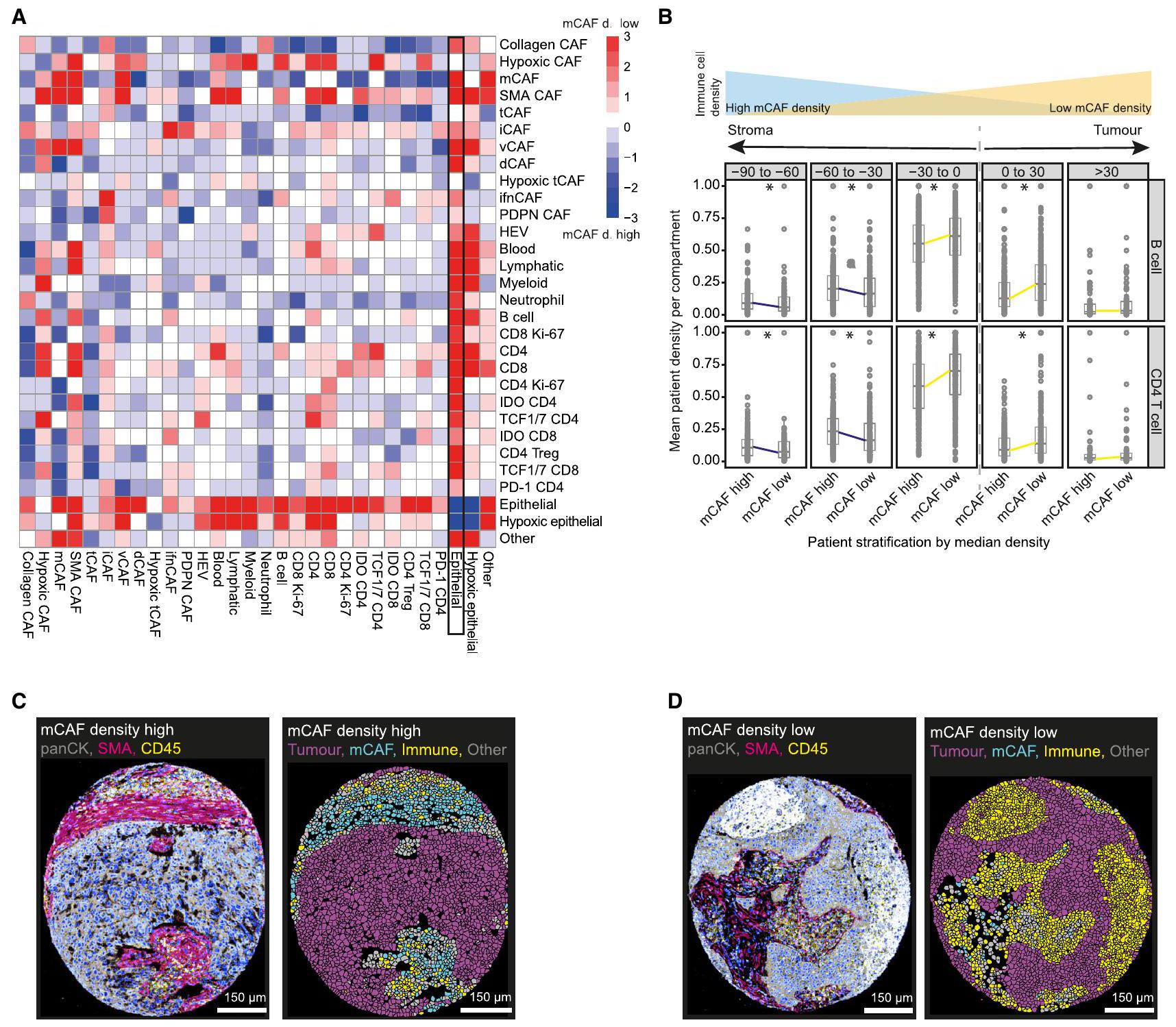
ككثافة mCAF منخفضة (الشكل 7A). علاوة على ذلك، وُجدت جميع خلايا المناعة، بما في ذلك
(أ) خريطة حرارية تظهر غنى أو نقص أنواع الخلايا بين مجموعات المرضى المحددة بواسطة تركيبة CAF (
(ب) منحنيات الكثافة تظهر المسافة لكل خلية من أنواع CAF المحددة إلى أقرب حدود ورم-ستروما لـ LUAD (يسار) و LUSC (يمين).
(ج) خريطة حرارية لتحليل الجوار تظهر أقرب 15 جارة لكل خلية ضمن نصف قطر من
(د) صور مع تراكبات قناع (بيضاء) لـ SMA CAFs (SMA
خلية سرطانية
مقالة
(أ) تحليل SpicyR لزوج الخلايا في المرضى المصنفين حسب كثافة المCAF المتوسطة. تشير الدرجات الإيجابية (الحمراء) إلى تفاعلات خلوية أعلى بين أزواج الخلايا في المرضى ذوي كثافة المCAF المنخفضة، بينما تشير الدرجات السلبية (الزرقاء) إلى تفاعلات خلوية أعلى في المرضى ذوي كثافة المCAF العالية؛ بينما يشير اللون الأبيض إلى عدم وجود فرق.
(ب) نسب خلايا B (الأعلى) و CD4
(C و D) صور تظهر العلامات المحددة لصورة كثافة المCAF العالية (C) وصورة كثافة المCAF المنخفضة (D)، مع الأقنعة المقابلة الملونة بخلايا الورم (أرجواني)، خلايا المناعة (أصفر) وmCAFs (سماوي). شريط القياس،
بواسطة بيئات ميكروية ناقصة الأكسجين ومثبطة للمناعة. على النقيض من ذلك، كانت iCAFs التي تعبر عن CD34 وCD248 بالإضافة إلى ifnCAFs مرتبطة ببيئات ميكروية للورم ملتهبة وبمعدل بقاء عام أعلى للمرضى.
نقاش
مجموعة كاملة وفي مجموعة فرعية من المرضى الذين تلقوا العلاج الكيميائي بعد الجراحة، مما يشير إلى دور مثبط للورم لـ ifnCAFs من خلال إشارات الإنترفيرون.
IDO المثبط للمناعة المحتمل
قيود الدراسة
تشير إلى التغيرات التي تحدث عند حواف الخلايا. ثالثًا، تم تصوير نواة واحدة أو اثنتين فقط لكل مريض، وتم أخذ جميع النوى من الجبهة الغازية. وبالتالي، فإن ميزات CAF والتنوع الذي نصفه قد تكون فريدة من نوعها في هذه المنطقة من أورام NSCLC. أخيرًا، دراستنا وصفية وحققت في الارتباط بدلاً من السببية، وبالتالي، فإن الفرضيات المستخلصة لدينا هي فرضيات تخمينية. ومع ذلك، فإن هذه الدراسة هي أكبر تحليل تصوير متعدد الأبعاد لأورام NSCLC والألياف حتى الآن ويجب أن تكون مرجعًا وموارد للعمل المستقبلي.
طرق النجوم
- جدول الموارد الرئيسية
- توافر الموارد
- جهة الاتصال الرئيسية
- توفر المواد
- توفر البيانات والشيفرة
- تفاصيل النموذج التجريبي وشارك الدراسة
- تفاصيل الطريقة
- لوحة الأجسام المضادة، التلوين، والتصوير
- خط أنابيب التقسيم
- تجميع نمط الخلية
- التكميم والتحليل الإحصائي
- استبعاد البيانات وتحديد المجموعات
- تحليل المجموعات المتطابقة
- التحليلات الإحصائية
- التحليل المكاني
- تحليل كشف بقع نقص الأكسجين
معلومات إضافية
شكر وتقدير
مساهمات المؤلفين
خلية سرطانية
مقالة
إعلان المصالح
تمت المراجعة: 2 نوفمبر 2023
تم القبول: 21 ديسمبر 2023
نُشر: 18 يناير 2024
REFERENCES
- Sung, H., Ferlay, J., Siegel, R.L., Laversanne, M., Soerjomataram, I., Jemal, A., and Bray, F. (2021). Global cancer statistics 2020: GLOBOCAN estimates of incidence and mortality worldwide for 36 cancers in 185 countries. CA. Cancer J. Clin. 71, 209-249.
- Schabath, M.B., and Cote, M.L. (2019). Cancer progress and priorities: Lung cancer. Cancer Epidemiol. Biomarkers Prev. 28, 1563-1579.
- Pikor, L.A., Ramnarine, V.R., Lam, S., and Lam, W.L. (2013). Genetic alterations defining NSCLC subtypes and their therapeutic implications. Lung Cancer 82, 179-189.
- Garon, E.B., Hellmann, M.D., Rizvi, N.A., Carcereny, E., Leighl, N.B., Ahn, M.J., Eder, J.P., Balmanoukian, A.S., Aggarwal, C., Horn, L., et al. (2019). Five-year overall survival for patients with advanced non-small-cell lung cancer treated with pembrolizumab: results from the phase I KEYNOTE001 study. J. Clin. Oncol. 37, 2518-2527.
- Robert, C. (2020). A decade of immune-checkpoint inhibitors in cancer therapy. Nat. Commun. 11, 3801.
- Sahai, E., Astsaturov, I., Cukierman, E., DeNardo, D.G., Egeblad, M., Evans, R.M., Fearon, D., Greten, F.R., Hingorani, S.R., Hunter, T., et al. (2020). A framework for advancing our understanding of cancer-associated fibroblasts. Nat. Rev. Cancer 20, 174-186.
- Hanahan, D., and Weinberg, R.A. (2011). Hallmarks of cancer: the next generation. Cell 144, 646-674.
- Turley, S.J., Cremasco, V., and Astarita, J.L. (2015). Immunological hallmarks of stromal cells in the tumour microenvironment. Nat. Rev. Immunol. 15, 669-682.
- Smyth, M.J., Ngiow, S.F., Ribas, A., and Teng, M.W.L. (2016). Combination cancer immunotherapies tailored to the tumour microenvironment. Nat. Rev. Clin. Oncol. 13, 143-158.
- Altorki, N.K., Markowitz, G.J., Gao, D., Port, J.L., Saxena, A., Stiles, B., McGraw, T., and Mittal, V. (2019). The lung microenvironment: an important regulator of tumour growth and metastasis. Nat. Rev. Cancer 19, 9-31.
- Finak, G., Bertos, N., Pepin, F., Sadekova, S., Souleimanova, M., Zhao, H., Chen, H., Omeroglu, G., Meterissian, S., Omeroglu, A., et al. (2008). Stromal gene expression predicts clinical outcome in breast cancer. Nat. Med. 14, 518-527.
- Bruni, D., Angell, H.K., and Galon, J. (2020). The immune contexture and Immunoscore in cancer prognosis and therapeutic efficacy. Nat. Rev. Cancer 20, 662-680.
- Su, S., Chen, J., Yao, H., Liu, J., Yu, S., Lao, L., Wang, M., Luo, M., Xing, Y., Chen, F., et al. (2018). CD10+GPR77+ Cancer-Associated Fibroblasts Promote Cancer Formation and Chemoresistance by Sustaining Cancer Stemness. Cell 172, 841-856.e16.
- Krishnamurty, A.T., Shyer, J.A., Thai, M., Gandham, V., Buechler, M.B., Yang, Y.A., Pradhan, R.N., Wang, A.W., Sanchez, P.L., Qu, Y., et al. (2022). LRRC15+ myofibroblasts dictate the stromal setpoint to suppress tumour immunity. Nature 611, 148-154.
- Genova, C., Dellepiane, C., Carrega, P., Sommariva, S., Ferlazzo, G., Pronzato, P., Gangemi, R., Filaci, G., Coco, S., and Croce, M. (2021). Therapeutic Implications of Tumor Microenvironment in Lung Cancer: Focus on Immune Checkpoint Blockade. Front. Immunol. 12, 799455.
- Caligiuri, G., and Tuveson, D.A. (2023). Activated fibroblasts in cancer: Perspectives and challenges. Cancer Cell 41, 434-449.
- Grout, J.A., Sirven, P., Leader, A.M., Maskey, S., Hector, E., Puisieux, I., Steffan, F., Cheng, E., Tung, N., Maurin, M., et al. (2022). Spatial Positioning and Matrix Programs of Cancer-Associated Fibroblasts Promote T-cell Exclusion in Human Lung Tumors. Cancer Discov. 12, 2606-2625.
- Ogawa, Y., Masugi, Y., Abe, T., Yamazaki, K., Ueno, A., Fujii-Nishimura, Y., Hori, S., Yagi, H., Abe, Y., Kitago, M., and Sakamoto, M. (2021). Three distinct stroma types in human pancreatic cancer identified by image analysis of fibroblast subpopulations and collagen. Clin. Cancer Res. 27, 107-119.
- Özdemir, B.C., Pentcheva-Hoang, T., Carstens, J.L., Zheng, X., Wu, C.C., Simpson, T.R., Laklai, H., Sugimoto, H., Kahlert, C., Novitskiy, S.V., et al. (2014). Depletion of carcinoma-associated fibroblasts and fibrosis induces immunosuppression and accelerates pancreas cancer with reduced survival. Cancer Cell 25, 719-734.
- Rhim, A.D., Oberstein, P.E., Thomas, D.H., Mirek, E.T., Palermo, C.F., Sastra, S.A., Dekleva, E.N., Saunders, T., Becerra, C.P., Tattersall, I.W., et al. (2014). Stromal elements act to restrain, rather than support, pancreatic ductal adenocarcinoma. Cancer Cell 25, 735-747.
- Bhattacharjee, S., Hamberger, F., Ravichandra, A., Miller, M., Nair, A., Affo, S., Filliol, A., Chin, L., Savage, T.M., Yin, D., et al. (2021). Tumor restriction by type I collagen opposes tumor-promoting effects of cancerassociated fibroblasts. J. Clin. Invest. 131, e146987.
- Chen, Y., Kim, J., Yang, S., Wang, H., Wu, C.J., Sugimoto, H., LeBleu, V.S., and Kalluri, R. (2021). Type I collagen deletion in
SMA+ myofibroblasts augments immune suppression and accelerates progression of pancreatic cancer. Cancer Cell 39, 548-565.e6. - Chen, Y., McAndrews, K.M., and Kalluri, R. (2021). Clinical and therapeutic relevance of cancer-associated fibroblasts. Nat. Rev. Clin. Oncol. 18, 792-804.
- Valkenburg, K.C., De Groot, A.E., and Pienta, K.J. (2018). Targeting the tumour stroma to improve cancer therapy. Nat. Rev. Clin. Oncol. 15, 366-381.
- Bughda, R., Dimou, P., D’Souza, R.R., and Klampatsa, A. (2021). Fibroblast Activation Protein (FAP)-Targeted CAR-T Cells: Launching an Attack on Tumor Stroma. ImmunoTargets Ther. 10, 313-323.
- Brünker, P., Wartha, K., Friess, T., Grau-Richards, S., Waldhauer, I., Koller, C.F., Weiser, B., Majety, M., Runza, V., Niu, H., et al. (2016). RG7386, a novel tetravalent FAP-DR5 antibody, effectively triggers FAP-dependent, avidity-driven DR5 hyperclustering and tumor cell apoptosis. Mol. Cancer Ther. 15, 946-957.
- Chen, Z., Zhou, L., Liu, L., Hou, Y., Xiong, M., Yang, Y., Hu, J., and Chen, K. (2020). Single-cell RNA sequencing highlights the role of inflammatory cancer-associated fibroblasts in bladder urothelial carcinoma. Nat. Commun. 11, 5077.
- Puram, S.V., Tirosh, I., Parikh, A.S., Patel, A.P., Yizhak, K., Gillespie, S., Rodman, C., Luo, C.L., Mroz, E.A., Emerick, K.S., et al. (2017). Single-cell transcriptomic analysis of primary and metastatic tumor ecosystems in head and neck cancer. Cell 171, 1611-1624.e24.
- Lee, H.-O., Hong, Y., Etlioglu, H.E., Cho, Y.B., Pomella, V., Van den Bosch, B., Vanhecke, J., Verbandt, S., Hong, H., Min, J.W., et al. (2020). Lineagedependent gene expression programs influence the immune landscape of colorectal cancer. Nat. Genet. 52, 594-603.
- Lambrechts, D., Wauters, E., Boeckx, B., Aibar, S., Nittner, D., Burton, O., Bassez, A., Decaluwé, H., Pircher, A., Van den Eynde, K., et al. (2018). Phenotype molding of stromal cells in the lung tumor microenvironment. Nat. Med. 24, 1277-1289.
- Tietscher, S., Wagner, J., Anzeneder, T., Langwieder, C., Rees, M., Sobottka, B., de Souza, N., and Bodenmiller, B. (2023). A comprehensive single-cell map of T cell exhaustion-associated immune environments in human breast cancer. Nat. Commun. 14, 98.
- Friedman, G., Levi-Galibov, O., David, E., Bornstein, C., Giladi, A., Dadiani, M., Mayo, A., Halperin, C., Pevsner-Fischer, M., Lavon, H., et al. (2020). Cancer-associated fibroblast compositions change with breast cancer
progression linking the ratio of S100A4+ and PDPN+ CAFs to clinical outcome. Nat. Cancer 1, 692-708. - Costa, A., Kieffer, Y., Scholer-Dahirel, A., Pelon, F., Bourachot, B., Cardon, M., Sirven, P., Magagna, I., Fuhrmann, L., Bernard, C., et al. (2018). Fibroblast Heterogeneity and Immunosuppressive Environment in Human Breast Cancer. Cancer Cell 33, 463-479.e10.
- Kieffer, Y., Hocine, H.R., Gentric, G., Pelon, F., Bernard, C., Bourachot, B., Lameiras, S., Albergante, L., Bonneau, C., Guyard, A., et al. (2020). Singlecell analysis reveals fibroblast clusters linked to immunotherapy resistance in cancer. Cancer Discov. 10, 1330-1351.
- Buechler, M.B., Pradhan, R.N., Krishnamurty, A.T., Cox, C., Calviello, A.K., Wang, A.W., Yang, Y.A., Tam, L., Caothien, R., Roose-Girma, M., et al. (2021). Cross-tissue organization of the fibroblast lineage. Nature 593, 575-579.
- Bartoschek, M., Oskolkov, N., Bocci, M., Lövrot, J., Larsson, C., Sommarin, M., Madsen, C.D., Lindgren, D., Pekar, G., Karlsson, G., et al. (2018). Spatially and functionally distinct subclasses of breast can-cer-associated fibroblasts revealed by single cell RNA sequencing. Nat. Commun. 9, 5150.
- Cords, L., Tietscher, S., Anzeneder, T., Langwieder, C., Rees, M., de Souza, N., and Bodenmiller, B. (2023). Cancer-associated fibroblast classification in single-cell and spatial proteomics data. Nat. Commun. 14, 4294.
- Elyada, E., Bolisetty, M., Laise, P., Flynn, W.F., Courtois, E.T., Burkhart, R.A., Teinor, J.A., Belleau, P., Biffi, G., Lucito, M.S., et al. (2019). Crossspecies single-cell analysis of pancreatic ductal adenocarcinoma reveals antigen-presenting cancer-associated fibroblasts. Cancer Discov. 9, 1102-1123.
- Öhlund, D., Handly-Santana, A., Biffi, G., Elyada, E., Almeida, A.S., PonzSarvise, M., Corbo, V., Oni, T.E., Hearn, S.A., Lee, E.J., et al. (2017). Distinct populations of inflammatory fibroblasts and myofibroblasts in pancreatic cancer. J. Exp. Med. 214, 579-596.
- Wu, S.Z., Roden, D.L., Wang, C., Holliday, H., Harvey, K., Cazet, A.S., Murphy, K.J., Pereira, B., Al-Eryani, G., Bartonicek, N., et al. (2020). Stromal cell diversity associated with immune evasion in human triplenegative breast cancer. EMBO J. 39, e104063.
- Giesen, C., Wang, H.A.O., Schapiro, D., Zivanovic, N., Jacobs, A., Hattendorf, B., Schüffler, P.J., Grolimund, D., Buhmann, J.M., Brandt, S., et al. (2014). Highly multiplexed imaging of tumor tissues with subcelIular resolution by mass cytometry. Nat. Methods 11, 417-422.
- Gachechiladze, M., Škarda, J., Kolek, V., Grygárková, I., Langová, K., Bouchal, J., Kolář, Z., Baty, F., Stahel, R., Weder, W., et al. (2017). Prognostic and predictive value of loss of nuclear RAD51 immunoreactivity in resected non-small cell lung cancer patients. Lung Cancer 105, 31-38.
- Tischler, V., Pfeifer, M., Hausladen, S., Schirmer, U., Bonde, A.K., Kristiansen, G., Sos, M.L., Weder, W., Moch, H., Altevogt, P., and Soltermann, A. (2011). L1CAM protein expression is associated with poor prognosis in non-small cell lung cancer. Mol. Cancer 10, 127.
- Soltermann, A., Tischler, V., Arbogast, S., Braun, J., Probst-Hensch, N., Weder, W., Moch, H., and Kristiansen, G. (2008). Prognostic significance of epithelial-mesenchymal and mesenchymal- epithelial transition protein expression in non-small cell lung cancer. Clin. Cancer Res. 14, 7430-7437.
- Berg, S., Kutra, D., Kroeger, T., Straehle, C.N., Kausler, B.X., Haubold, C., Schiegg, M., Ales, J., Beier, T., Rudy, M., et al. (2019). Ilastik: interactive machine learning for (bio) image analysis. Nat. Methods 16, 1226-1232.
- McQuin, C., Goodman, A., Chernyshev, V., Kamentsky, L., Cimini, B.A., Karhohs, K.W., Doan, M., Ding, L., Rafelski, S.M., Thirstrup, D., et al. (2018). CellProfiler 3.0: Next-generation image processing for biology. PLoS Biol. 16, e2005970.
- Van Gassen, S., Callebaut, B., Van Helden, M.J., Lambrecht, B.N., Demeester, P., Dhaene, T., and Saeys, Y. (2015). FlowSOM: Using selforganizing maps for visualization and interpretation of cytometry data. Cytometry A. 87, 636-645.
- Schwörer, S., Cimino, F.V., Ros, M., Tsanov, K.M., Ng, C., Lowe, S.W., Carmona-Fontaine, C., and Thompson, C.B. (2023). Hypoxia Potentiates the Inflammatory Fibroblast Phenotype Promoted by Pancreatic Cancer Cell-Derived Cytokines. Cancer Res. 83, 1596-1610.
- Garcia Garcia, C.J., Huang, Y., Fuentes, N.R., Turner, M.C., Monberg, M.E., Lin, D., Nguyen, N.D., Fujimoto, T.N., Zhao, J., Lee, J.J., et al. (2022). Stromal HIF2 Regulates Immune Suppression in the Pancreatic Cancer Microenvironment. Gastroenterology 162, 2018-2031.
- Lodyga, M., and Hinz, B. (2020). TGF-
– A truly transforming growth factor in fibrosis and immunity. Semin. Cell Dev. Biol. 101, 123-139. - Cremasco, V., Astarita, J.L., Grauel, A.L., Keerthivasan, S., Maclsaac, K., Woodruff, M.C., Wu, M., Spel, L., Santoro, S., Amoozgar, Z., et al. (2018). FAP delineates heterogeneous and functionally divergent stromal cells in immune-excluded breast tumors. Cancer Immunol. Res. 6, 1472-1485.
- Canete, N.P., Iyengar, S.S., Ormerod, J.T., Baharlou, H., Harman, A.N., and Patrick, E. (2022). spicyR: spatial analysis of in situ cytometry data in R. Bioinformatics 38, 3099-3105.
- Bejarano, L., Jordāo, M.J.C., and Joyce, J.A. (2021). Therapeutic Targeting of the Tumor Microenvironment. Cancer Discov. 11, 933-959.
- Zhang, M., Yang, H., Wan, L., Wang, Z., Wang, H., Ge, C., Liu, Y., Hao, Y., Zhang, D., Shi, G., et al. (2020). Single-cell transcriptomic architecture and intercellular crosstalk of human intrahepatic cholangiocarcinoma. J. Hepatol. 73, 1118-1130.
- Yu, Z., Zhang, J., Zhang, Q., Wei, S., Shi, R., Zhao, R., An, L., Grose, R., Feng, D., and Wang, H. (2022). Single-cell sequencing reveals the heterogeneity and intratumoral crosstalk in human endometrial cancer. Cell Prolif. 55, e13249.
- Foster, D.S., Januszyk, M., Delitto, D., Yost, K.E., Griffin, M., Guo, J., Guardino, N., Delitto, A.E., Chinta, M., Burcham, A.R., et al. (2022). Multiomic analysis reveals conservation of cancer-associated fibroblast phenotypes across species and tissue of origin. Cancer Cell 40, 13921406.e7.
- Werba, G., Weissinger, D., Kawaler, E.A., Zhao, E., Kalfakakou, D., Dhara, S., Wang, L., Lim, H.B., Oh, G., Jing, X., et al. (2023). Single-cell RNA sequencing reveals the effects of chemotherapy on human pancreatic adenocarcinoma and its tumor microenvironment. Nat. Commun. 14, 3912.
- Keller, P.J., Arendt, L.M., Skibinski, A., Logvinenko, T., Klebba, I., Dong, S., Smith, A.E., Prat, A., Perou, C.M., Gilmore, H., et al. (2012). Defining the cellular precursors to human breast cancer. Proc. Natl. Acad. Sci. USA 109, 2772-2777.
- Cheon, H., Wang, Y., Wightman, S.M., Jackson, M.W., and Stark, G.R. (2023). How cancer cells make and respond to interferon-I. Trends Cancer 9, 83-92.
- Benveniste, E.N., and Qin, H. (2007). Type I interferons as anti-inflammatory mediators. Sci. STKE 2007, pe70.
- Al Bakir, M., Huebner, A., Martínez-Ruiz, C., Grigoriadis, K., Watkins, T.B.K., Pich, O., Moore, D.A., Veeriah, S., Ward, S., Laycock, J., et al. (2023). The evolution of non-small cell lung cancer metastases in TRACERx. Nature 616, 534-542.
- Stagg, J., Divisekera, U., McLaughlin, N., Sharkey, J., Pommey, S., Denoyer, D., Dwyer, K.M., and Smyth, M.J. (2010). Anti-CD73 antibody therapy inhibits breast tumor growth and metastasis. Proc. Natl. Acad. Sci. USA 107, 1547-1552.
- Turcotte, M., Spring, K., Pommey, S., Chouinard, G., Cousineau, I., George, J., Chen, G.M., Gendoo, D.M.A., Haibe-Kains, B., Karn, T., et al. (2015). CD73 is associated with poor prognosis in high-grade serous ovarian cancer. Cancer Res. 75, 4494-4503.
- Wang, L., Zhou, X., Zhou, T., Ma, D., Chen, S., Zhi, X., Yin, L., Shao, Z., Ou, Z., and Zhou, P. (2008). Ecto-5′-nucleotidase promotes invasion, migration and adhesion of human breast cancer cells. J. Cancer Res. Clin. Oncol. 134, 365-372.
- Vijayan, D., Young, A., Teng, M.W.L., and Smyth, M.J. (2017). Targeting immunosuppressive adenosine in cancer. Nat. Rev. Cancer 17, 709-724.
مقالة خلية السرطان
سيل برس
الوصول المفتوح
- فو، ي.، ليو، س.، ين، س.، نيو، و.، شيونغ، و.، تان، م.، لي، ج.، وزو، م. (2017). من المحتمل أن يكون تأثير واربورغ العكسي نقطة ضعف لأمراض السرطان يمكن استغلالها في علاج السرطان. أونكوتارجت 8، 57813-57825.
- بابليدس، س.، ويتاكر-مينيز، د.، كاستيلو-كروس، ر.، فلومينبرغ، ن.، ويتكيفيتش، أ.ك.، فرانك، ب.ج.، كاسيميرو، م.س.، وانغ، س.، فورتينا، ب.، أدي، س.، وآخرون. (2009). تأثير واربورغ العكسي: التحلل الجليكولي الهوائي في الخلايا الليفية المرتبطة بالسرطان وسترومة الورم. دورة الخلية 8، 3984-4001.
- دراغيكا، ج.، سونغ، م.، ووانغ، ل. (2020). أدوار IFN-
في تقدم الورم وتراجعه مراجعة أبحاث العلامات الحيوية، 8 (BioMed Central Ltd ما لم يُذكر خلاف ذلك. جزء من Springer Nature). - Uyttenhove، C.، Pilotte، L.، Théate، I.، Stroobant، V.، Colau، D.، Parmentier، N.، Boon، T.، و Van den Eynde، B.J. (2003). دليل على آلية مقاومة المناعة الورمية تعتمد على تحلل التريبتوفان بواسطة إندولامين 2،3-ديوكسيجيناز. نات. ميد. 9، 1269-1274.
- زيمرمان، ج.أ.، هتياراتشي، م.هـ.، وماكديفيت، ت.س. (2017). تعزيز تثبيط المناعة لخلايا T من خلال العرض المستمر للإنترفيرون النشط بيولوجيًا
داخل هياكل الخلايا الجذعية الميزانشيمية ثلاثية الأبعاد. خلايا الجذع ترانسلات. ميد. 6، 223-237. - هو، هـ.، بيوترونسكا، ز.، هير، ب.ج.، تشين، هـ.، مولفي، هـ.إي.، ميفيلد، أ.، نون، س.، كاترمين، ك.، غرينبرغ، م.، ويليامز، أ.، وآخرون. (2021). ثلاثة أنواع فرعية من ليفات سرطان الرئة تحدد نماذج علاجية متميزة. خلية السرطان 39، 1531-1547.e10.
- يانغ، د.، ليو، ج.، تشيان، هـ.، وزوانغ، ق. (2023). ك. الخلايا الليفية المرتبطة بالسرطان: من العلوم الأساسية إلى العلاج المضاد للسرطان. إكسب. مول. ميد. 55، 1322-1332.
- دومينغيز، سي. إكس.، مولر، س.، كيرثيفاسان، س.، كويبن، هـ.، هونغ، ج.، جيركي، س.، بريارت، ب.، فورمان، أو.، بينبريدج، تي. و.، كاستيجلوني، أ.، وآخرون. (2020). تسلسل RNA على مستوى الخلية الواحدة يكشف عن تطور النسيج الداعم إلى خلايا العضلات الليفية LRRC15+ كعامل محدد لاستجابة المرضى للعلاج المناعي للسرطان. اكتشاف السرطان. 10، 232-253.
- بوندجرز، سي.، كالين، م.، هيلستروم، م.، شيدل، س.ج.، أبرامسون، أ.، رينر، أ.، لينداهل، ب.، تشو، هـ.، كيرل، ج.، وبتشولز، سي. (2003). تحديد ملف النسخ الجيني للأجنة الفأرية التي تفتقر إلى عامل نمو الصفائح الدموية-B، مما يحدد RGS5 كعلامة جديدة للخلايا المحيطية وخلايا العضلات الملساء الوعائية. المجلة الأمريكية للطب الشرعي 162، 721-729.
- شابيرو، د.، جاكسون، هـ.و.، راغورامان، س.، فيشر، ج.ر.، زانوتيلي، ف.ر.ت.، شولتز، د.، جيسن، س.، كاتينا، ر.، فارغا، ز.، وبودنميلر، ب. (2017). هيستو كات: تحليل أنماط الخلايا والتفاعلات في بيانات قياس الصور المتعددة. نات. ميثودز 14، 873-876.
- ويندهاجر، ج.، زانوتيلي، ف.ر.ت.، شولتز، د.، ماير، ل.، دانيال، م.، بودنميلر، ب.، وإلينغ، ن. (2023). سير عمل شامل لمعالجة وتحليل الصور المتعددة. نات. بروتوك. 18، 3565-3613.
- ليفين، ج.هـ.، سيموندز، إ.ف.، بندال، س.س.، ديفيس، ك.ل.، أمير، إ.ا.د.، تادمور، م.د.، ليتفين، أ.، فينبرغ، هـ.ج.، ياجر، أ.، زوندر، إ.ر.، وآخرون. (2015). تحليل ظاهري مدفوع بالبيانات لسرطان الدم الحاد يكشف عن خلايا شبيهة بالسلائف ترتبط بالتشخيص. خلية 162، 184-197.
- جرينوالد، ن.ف.، ميلر، ج.، موين، إ.، كونغ، أ.، كاجيل، أ.، دوغرتي، ت.، فولاوي، س.س.، ماكنتوش، ب.ج.، ليو، ك.إكس.، شوارتز، م.س.، وآخرون. (2022). تقسيم الخلايا الكاملة لصور الأنسجة بأداء على مستوى الإنسان باستخدام توضيح البيانات على نطاق واسع والتعلم العميق. نات. بيوتكنولوجي. 40، 555-565.
- جاكسون، إتش. دبليو.، فيشر، ج. ر.، زانوتيلي، ف. ر. ت.، علي، ح. ر.، ميشيرا، ر.، سويسال، س. د.، موش، هـ.، موانست، س.، فارغا، ز.، ويبر، و. ب.، وبودنميلر، ب. (2020). مشهد علم الأمراض على مستوى الخلية الواحدة لسرطان الثدي. ناتشر 578، 615-620.
- روبنسون، م.د.، مكارثي، د.ج.، وسمايث، ج.ك. (2010). حزمة بايكونداكتور لتحليل التعبير التفاضلي لبيانات التعبير الجيني الرقمي. المعلوماتية الحيوية 26، 139-140.
- Weber، ل.م.، نوويكا، م.، سونيسون، س.، وروبنسون، م.د. (2019). الاكتشاف التفاضلي في علم الخلايا عالي الأبعاد من خلال التجميع عالي الدقة. اتصالات. بيولوجيا. 2، 183.
طرق النجوم
جدول الموارد الرئيسية
| المُعَايِن أو المورد | المصدر | معرف |
| الأجسام المضادة | ||
| أجسام مضادة وحيدة النسيلة من الأرانب ضد الفيمنتين؛ النسخة (EPR3776)؛ الدفعة (GR286525-2) | أبكام | قط#ab193555; RRID: AB_2814713 |
| أجسام مضادة متعددة النسائل من الأرانب ضد p75 (CD271)؛ النسخة (polyclonal_ANT-007)؛ الدفعة (ANT007AN0702) | مختبرات ألومون | القط#ANT-007; RRID: AB_2039968 |
| أرنب مضاد كولاجين I متعدد النسائل؛ النسخة (متعدد النسائل_كولاجين I)؛ الدفعة (GR3271183-3) | أبكام | قط#ab34710; RRID: AB_731684 |
| أجسام مضادة وحيدة النسيلة من نوع الفيبروكتين؛ النسخة (10/فيبروكتين)؛ الدفعة (6251888) | بي دي بيوساينس | القط#610078؛ RRID: AB_397486 |
| أجسام مضادة وحيدة النسيلة من نوع رات ضد بودوبلانين؛ النسخة (NC-08)؛ الدفعة (B260834) | بيوليجند | القط#337002؛ RRID: AB_1595511 |
| أجسام مضادة أحادية النسيلة من نوع الماوس ضد السيتوكيراتين الشامل؛ النسخة (AE1)؛ الدفعة (3252910) | ميلبورو | القط#MAB1612; RRID: AB_2132794 |
| أجسام مضادة متعددة النسائل من الماعز ضد الكربونيك أنهيدراز IX؛ النسخة (polyclonal_CA9_AF2188)؛ الدفعة (VNQ0319011) | أنظمة البحث والتطوير | القط#AF2188; RRID: AB_416562 |
| أجسام مضادة وحيدة النسيلة من الأرانب ضد الهيستون H3؛ النسخة (D1H2)؛ الدفعة (15) | تكنولوجيا إشارات الخلايا | قط#4499BF; RRID: AB_10544537 |
| أرنب متعدد النسائل مضاد لـ CD248؛ النسخة (متعدد النسائل_بروتين تك)؛ الدفعة (00019535) | بروتينتيك | القط#18160؛ RRID: AB_10858230 |
| أجسام مضادة أحادية النسيلة من نوع الماوس ضد CXCL-12؛ النسخة (79018)؛ الدفعة (JOJ0519031) | أنظمة البحث والتطوير | قط#MAB350-100; RRID: AB_2088149 |
| أجسام مضادة متعددة النسائل من الماعز ضد LYVE-1؛ النسخة (متعددة النسائل_LYVE-1)؛ الدفعة (KPY0119052) | أنظمة البحث والتطوير | قط#AF2089; RRID: AB_355144 |
| أجسام مضادة متعددة النسائل من الماعز ضد CCL21؛ النسخة (Polyclonal_CCL21 / 6Ckine)؛ الدفعة (AYJ218071) | أنظمة البحث والتطوير | قط#AF366; RRID: AB_355327 |
| أجسام مضادة وحيدة النسيلة من نوع الماوس ضد كاديرين-11؛ النسخة (283416)؛ الدفعة (VLT0219091) | أنظمة البحث والتطوير | القط#MAB1790; RRID: AB_2076970 |
| أحادي النسيلة من نوع الفأر مضاد CD45RA؛ النسخة (HI100)؛ الدفعة (B291512) | بيوليجند | القط#304102؛ RRID: AB_314406 |
| أجسام مضادة أحادية النسيلة من الأرانب ضد كافولين-1؛ النسخة (D46G3)؛ الدفعة (7) | تكنولوجيا إشارات الخلايا | قط#3267BF; RRID: AB_2275453 |
| أجسام مضادة وحيدة النسيلة من نوع الفأر ضد FOXP3؛ النسخة (236A/E7)؛ الدفعة (2129676) | إي بايوساينس | قط#14-4777-82؛ RRID: AB_467556 |
| أجسام مضادة وحيدة النسيلة من الأرانب ضد MMP9؛ النسخة (D6O3H)؛ الدفعة (4) | تكنولوجيا إشارات الخلايا | القط#A0398; RRID: AB_2798289 |
| أجسام مضادة أحادية النسيلة من الأرانب ضد CD140b/PDGFRb؛ النسخة (Y92)؛ الدفعة (GR296584-4) | أبكام | قط#ab215978; RRID: AB_2894841 |
| أحادي النسيلة من نوع الفأر مضاد لـ CD8a؛ النسخة (C8/144B)؛ الدفعة (2132595) | إي بايوساينس | قط#14-0085-82؛ RRID: AB_11150240 |
| أجسام مضادة متعددة النسائل ضد الميالوبيروكسيداز من الأرانب؛ النسخة (أجسام مضادة متعددة النسائل MPO)؛ الدفعة (20071134) | داكو | القط#AB_2335676; RRID: AB_2335676 |
| أحادي النسيلة من نوع الفأر مضاد لـ CD15؛ النسخة (HI98)؛ الدفعة (B291510) | بيوليجند | القط#301902؛ RRID: AB_314194 |
| أرنب وحيد النسيلة مضاد-CD4؛ النسخة (EPR6855)؛ الدفعة (GR3215375-27) | أبكام | قط#ab181724; RRID: AB_2864377 |
| أجسام مضادة أحادية النسيلة من الأرانب ضد إندولامين 2-3-دي أوكسيجيناز؛ النسخة (SP260)؛ الدفعة (GR3259345-2) | أبكام | قط#ab245737; RRID: AB_2894840 |
| أجسام مضادة أحادية النسيلة من الأرانب ضد CD10؛ النسخة (E5P7S)؛ الدفعة (2) | تكنولوجيا إشارات الخلايا | قط#65534; RRID: AB_2894842 |
| أجسام مضادة متعددة النسائل من الماعز ضد CD146؛ النسخة (متعددة النسائل_ MCAM/CD146_RnD_AF932)؛ الدفعة (ECL0319041) | أنظمة البحث والتطوير | القط#AF932; RRID: AB_355721 |
مقالة خلية السرطان
سيل برس
الوصول المفتوح
| مستمر | ||
| المُعَايِن أو المورد | المصدر | معرف |
| أحادي النسيلة من نوع الفأر ضد SMA؛ النسخة (1A4)؛ الدفعة (2183900) | إي بايوساينس | قط#14-9760-82؛ RRID: AB_2572996 |
| أجسام مضادة أحادية النسيلة من الأرانب ضد CD73؛ النسخة (D7F9A)؛ الدفعة (2) | تكنولوجيا إشارات الخلايا | قط#13160BF; RRID: AB_2716625 |
| أحادي النسيلة من الأجسام المضادة للفأر ضد PNAd؛ النسخة (MECA-79)؛ الدفعة (B257139) | بيوليجند | قط#120802؛ RRID: AB_493555 |
| أجسام مضادة وحيدة النسيلة من الأرانب ضد VCAM-1؛ النسخة (EPR5047)؛ الدفعة (GR3255420-3) | أبكام | قط#ab215380; RRID: AB_2894839 |
| أجسام مضادة أحادية النسيلة من الأرانب ضد CD34؛ النسخة (EP373Y)؛ الدفعة (GR3271518-1) | أبكام | قط#ab198395; RRID: AB_2889381 |
| أجسام مضادة متعددة النسائل ضد vWF من الأرانب؛ النسخة (poly vwf)؛ الدفعة (3322998) | ميلليبور | القطعة#AB7356; RRID: AB_92216 |
| أحادي النسيلة من نوع الفأر مضاد لـ HLA-DR؛ النسخة (TAL 1B5)؛ الدفعة (GR3306331-1) | أبكام | قط#ab20181; RRID: AB_445401 |
| أجسام مضادة متعددة النسائل من الأرانب ضد CD3؛ النسخة (متعددة النسائل_A0452)؛ الدفعة (20073981) | داكو | قط#A0452; RRID: AB_2335677 |
| أجسام مضادة متعددة النسائل ضد MMP11 من الأرانب؛ النسخة (متعددة النسائل)؛ الدفعة (GR111256-10) | أبكام | قط#ab53143; RRID: AB_2042361 |
| أحادي النسيلة من نوع الفأر مضاد للكيراتين الظهاري؛ النسخة (AE3)؛ الدفعة (3255457) | ميلبورو | القط#MAB1611; RRID: AB_2134409 |
| أجسام مضادة وحيدة النسيلة من نوع الماوس ضد CD20؛ النسخة (L26)؛ الدفعة (2172592) | إي بايوساينس | قط#14-0202-82; RRID: AB_10734340 |
| أجسام مضادة أحادية النسيلة من الأرانب ضد CD31؛ النسخة (EPR3094)؛ الدفعة (GR3229164-1) | أبكام | قط#ab207090; RRID: AB_2889382 |
| أجسام مضادة وحيدة النسيلة من الأرانب ضد FSP1 / S100A4؛ النسخة (EPR2761(2))؛ الدفعة (GR317174-3) | أبكام | قط#136784؛ RRID: AB_10807552 |
| أجسام مضادة متعددة النسائل من الأغنام ضد FAP؛ النسخة (polyclonal_FAP)؛ الدفعة (ZKW0619101) | أنظمة البحث والتطوير | القط#AF3715; RRID: AB_2102369 |
| أحادي النسيلة من نوع الفأر مضاد لـ CD68؛ النسخة (KP1)؛ الدفعة (2162103) | إي بايوساينس | قط#14-0688-82; RRID: AB_11151139 |
| أجسام مضادة أحادية النسيلة من الأرانب ضد CD279 (PD-1)؛ النسخة (D4W2J)؛ الدفعة (6) | تكنولوجيا إشارات الخلايا | القط# I44:I47; RRID: AB_2864408 |
| أرنب وحيد النسيلة مضاد-TCF1/TCF7؛ النسخة (C63D9)؛ الدفعة (10) | تكنولوجيا إشارات الخلايا | القط#2203؛ RRID: AB_2534154 |
| أحادي النسيلة من نوع الفأر مضاد CD45RO؛ النسخة (UCHL1)؛ الدفعة (B291511) | بيوليجند | القط#304202؛ RRID: AB_314418 |
| أجسام مضادة وحيدة النسيلة من الأرانب ضد K-Cadherin؛ النسخة (D3T3I)؛ الدفعة (2) | تكنولوجيا إشارات الخلايا | قط#48111; RRID: AB_2799334 |
| أحادي النسيلة من نوع الفأر مضاد Ki-67؛ النسخة (B56)؛ الدفعة (8239549) | بي دي بيوساينس | القط#556003؛ RRID: AB_396287 |
| عينات بيولوجية | ||
| شرائح TMA من سرطان الرئة غير صغير الخلايا البشري | مستشفى جامعة زيورخ، زيورخ، سويسرا | https://www.usz.ch/ |
| المواد الكيميائية، الببتيدات، والبروتينات المؤتلفة | ||
| مُدخل الإريديوم | فلويديجيم | قط#201192B |
| اختبارات تجارية حاسمة | ||
| مجموعة ملصقات ماكس بار X8 متعددة المعادن | فلويديجيم | قط#201300 |
| البيانات المودعة | ||
| صور IMC | هذه الورقة | زينودو: https://doi.org/10.5281/zenodo.7961844 |
| البيانات السريرية | هذه الورقة | زينودو: |
| البرمجيات والخوارزميات | ||
| CellProfiler v3.1.9 | مككوين وآخرون
|
http://cellprofiler.org |
| هيستو كات | شابيرو وآخرون
|
https://github.com/BodenmillerGroup/histoCAT/releases |
| مستمر | ||
| المُعَايِن أو المورد | المصدر | معرف |
| إيلاستيك v.1.3.3 | بيرغ وآخرون
|
https://www.ilastik.org/news/ 2019/09/30/ilastik-1.3.3 |
| بايثون الإصدار 3.4.3 | مؤسسة برمجيات بايثون | https://www.python.org |
| R v4.1 | فريق تطوير R الأساسي | https://www.r-project.org |
| أدوات imctools لمختبر بودنميلر | مختبر بودنميلر | https://github.com/BodenmillerGroup/imctools |
| خط أنابيب تقسيم مختبر بودنميلر | مختبر بودنميلر | https://github.com/BodenmillerGroup/ImcSegmentationPipeline |
| ملحقات بودنميلر سيل بروفايلر | مختبر بودنميلر | https://github.com/BodenmillerGroup/ImcPluginsCP |
| تعويض تسرب مختبر بودنميلر | مختبر بودنميلر | https://github.com/BodenmillerGroup/cyTOFcompensation |
| imcRtools v1.3.7 | ويندهاجر وآخرون
|
https://doi.org/10.1038/s41596-023-00881-0. |
| Rphenoannoy v0.1.0 | ليفين جيه. إتش وآخرون.
|
https://doi.org/10.1016/j.cell.2015.05.047 |
| FLOWSOM v2.2.0 | فان وآخرون
|
https://onlinelibrary.wiley.com/doi/ full/10.1002/cyto.a.22625. |
| رمز التحليل | هذه الورقة | https://doi.org/10.5281/zenodo. 7981268 |
توافر الموارد
جهة الاتصال الرئيسية
توفر المواد
توفر البيانات والشيفرة
تفاصيل النموذج التجريبي وشارك الدراسة
تفاصيل الطريقة
لوحة الأجسام المضادة، التلوين، والتصوير
تم استبعاد كاديرين-6 من جميع التحليلات لأنه لم يظهر أي نمط تلوين محدد. تم اختيار جميع الأجسام المضادة بناءً على دراستنا السابقة حول تغاير CAF بما في ذلك مجموعة بيانات scRNA-seq لسرطان الرئة غير صغير الخلايا.
خط أنابيب التقسيم
تجميع نمط الخلايا
التكميم والتحليل الإحصائي
استبعاد البيانات وتحديد المجموعات
تحليل المجموعات المتطابقة
التحليلات الإحصائية
التحليل المكاني
تحليل كشف بقع نقص الأكسجين
- (C) نموذج CoxPH لمجموعات المرضى، مصحح لنوع الورم ودرجته. مجموعة المرضى 1 تعمل كمرجع. قيم p
مُعَروضة باللون الأزرق، قيم p باللون الرمادي.
(D و E) مخططات كابلان-ماير (للبقاء العام مقارنةً بالمرضى المصنفين كعالي ومنخفض بناءً على النسبة المتوسطة لأنواع CAF المحددة. CAFs ذات التوقعات الجيدة في D، وCAFs ذات التوقعات السيئة في E. جميع المقارنات التي تظهر اختلافات ذات دلالة إحصائية (اختبار لوغ رانك p < 0.05) موضحة.
(F) نموذج كوكس بورتنر المعتمد على لاسو والذي يتضمن متوسط نسب أنواع CAF لكل مريض، وتصنيف المرضى إلى مجموعتين عالية ومنخفضة لكل نوع من أنواع CAF (حسب النسبة المتوسطة)، وجميع البيانات السريرية.
DOI: https://doi.org/10.1016/j.ccell.2023.12.021
PMID: https://pubmed.ncbi.nlm.nih.gov/38242124
Publication Date: 2024-01-18
Cancer-associated fibroblast phenotypes are associated with patient outcome in non-small cell lung cancer
Article
Cancer-associated fibroblast phenotypes are associated with patient outcome in non-small cell lung cancer
Abstract
SUMMARY Despite advances in treatment, lung cancer survival rates remain low. A better understanding of the cellular heterogeneity and interplay of cancer-associated fibroblasts (CAFs) within the tumor microenvironment will support the development of personalized therapies. We report a spatially resolved single-cell imaging mass cytometry (IMC) analysis of CAFs in a non-small cell lung cancer cohort of 1,070 patients. We identify four prognostic patient groups based on 11 CAF phenotypes with distinct spatial distributions and show that CAFs are independent prognostic factors for patient survival. The presence of tumor-like CAFs is strongly correlated with poor prognosis. In contrast, inflammatory CAFs and interferon-response CAFs are associated with inflamed tumor microenvironments and higher patient survival. High density of matrix CAFs is correlated with low immune infiltration and is negatively correlated with patient survival. In summary, our data identify phenotypic and spatial features of CAFs that are associated with patient outcome in NSCLC.
INTRODUCTION
stromal compartment is also important for cancer progression and therapeutic success but is not well understood.
RESULTS
IMC analysis of non-small cell lung cancer samples
CAF phenotypes in NSCLC
Cancer Cell
Article
CellPress
| Table 1. Clinical cohort description | |
| Sex | |
| Male | 710 (66.4%) |
| Female | 348 (32.5%) |
| Missing | 12 (1.1%) |
| Age | |
| Mean (SD) | 64.0 (9.96) |
| Median [Min, Max] | 64.5 [18.0, 87.0] |
| Missing | 12 (1.1%) |
| Tumor type | |
| Adenocarcinoma | 618 (57.8%) |
| Squamous cell carcinoma | 401 (37.5%) |
| Adeno squamous cell carcinoma | 32 (3.0%) |
| Large cell carcinoma | 9 (0.8%) |
| NSCLC | 2 (0.2%) |
| Grade | |
| 1 | 62 (5.8%) |
| 2 | 481 (45.0%) |
| 3 | 515 (48.1%) |
| Missing | 12 (1.1%) |
| Size [cm] | |
| Mean (SD) | 4.04 (2.26) |
| Median [Min, Max] | 3.50 [0.300, 16.0] |
| Missing | 12 (1.1%) |
| T | |
| 1 | 131 (12.2%) |
| 2 | 129 (12.1%) |
| 3 | 381 (35.6%) |
| 4 | 118 (11.0%) |
| 5 | 215 (20.1%) |
| 6 | 84 (7.9%) |
| Missing | 12 (1.1%) |
| N | |
| 0 | 555 (51.9%) |
| 1 | 268 (25.0%) |
| 2 | 220 (20.6%) |
| 3 | 14 (1.3%) |
| Missing | 13 (1.2%) |
| M | |
| 0 | 967 (90.4%) |
| 1 | 71 (6.6%) |
| 2 | 19 (1.8%) |
| Missing | 13 (1.2%) |
| Stage | |
| 1 (IA) | 182 (17.0%) |
| 2 (IB) | 191 (17.9%) |
| 3 (IIA) | 191 (17.9%) |
| 4 (IIB) | 113 (10.6%) |
| 5 (IIIA) | 289 (27.0%) |
| 6 (IIIB) | 32 (3.0%) |
| 7 (IV) | 60 (5.6%) |
| Table 1. Continued | |
| Missing | 12 (1.1%) |
| Chemotherapy (neoadjuvant) | |
| No neoadjuvant chemotherapy | 961 (89.8%) |
| Neoadjuvant chemotherapy | 78 (7.3%) |
| Missing | 31 (2.9%) |
| Radiotherapy (neoadjuvant) | |
| No neoadjuvant radiotherapy | 1035 (96.7%) |
| Neoadjuvant radiotherapy | 4 (0.4%) |
| Missing | 31 (2.9%) |
| Relapse | |
| No relapse | 555 (51.9%) |
| Relapse | 480 (44.9%) |
| Missing | 35 (3.3%) |
| Overall survival | |
| Mean (SD) | 1490 (1190) |
| Median [Min, Max] | 1230 [0, 5730] |
| Missing | 2 (0.2%) |
| Disease free survival | |
| Mean (SD) | 1430 (1220) |
| Median [Min, Max] | 1060 [0, 5100] |
| Missing | 138 (12.9%) |
| Smoking status | |
| No | 86 (8.0%) |
| Currently | 730 (68.2%) |
| Former | 26 (2.4%) |
| Unknown | 197 (18.4%) |
| Missing | 31 (2.9%) |
| Chemotherapy (adjuvant) | |
| Yes | 138 |
| No | 901 |
| Missing | 31 |
more abundant in LUAD, while hypoxic tCAFs, PDPN CAFs, and mCAFs were enriched in LUSC (Figure S3A). We found in addition minor differences between different growth patterns of LUAD, with mCAFs and collagen CAFs enriched in solid and micropapillary tumors, consistent with the relatively high stroma content of these tumor types (Figure S3B). Patients clustered into four patient groups based on proportions of CAF types (Figures 3E, 3F, and S3C), with group 1 enriched in SMA CAFs, group 2 in matrix and collagen CAFs, group 3 in several CAF types including tCAFs, hypoxic CAFs, hypoxic tCAFs and vCAFs, and group 4 in iCAFs and ifnCAFs.
CAF phenotypes are an independent prognostic factor in NSCLC
Cancer Cell
Article


| CAF Marker | CAF Marker | Immune Marker | Endothelia |
| PDPN | p75 | CD45RA/R0 (Immune) | VWF/ CD31 (Blood v.) |
| IDO (ifnCAF) | CXCL13 | CD3 (T cells) | PNAd (HEV) |
| VCAM | CD146 (vCAF) | CD4 (T cells) | LYVE-1 (Lymhatic) |
| CAIX (Hypoxic CAF) | PDGFR-b | CD8 (T cells) | VCAM-1 (Vessel) |
| CXCL12 | CCL21 (rCAF) | FOXP3 (T regs) | CCL21 (Lymphatic) |
| CD10 (tCAF) | CD73 (tCAF) | PD-1 (T cell activation) | Tumour Marker |
| Cadherin-11 | MMP11 (mCAF) | TCF1/7 (T cells) | Pan Cytokeratin |
| FAP | CD248 (iCAF) | IDO (IFN response) | CAIX (Hypoxia) |
| SMA | CAIX (Hypoxia) | CD68 (Macrophages) | |
| FSP-1 | IDO (IFN response) | MPO + MMP9 (N.) | |
| Vimentin | CD20 (B cells) | General marker | |
| Ki-67 (dCAF) | CD11b (Myeloid) | Histone H3 (DNA) | |
| Fibronectin + Col. 1 | CD15 (Neutrophil) | Ki-67 (Division) | |
| CD34 (iCAF) | HLA-DR (Myeloid) | Caveolin-1 |
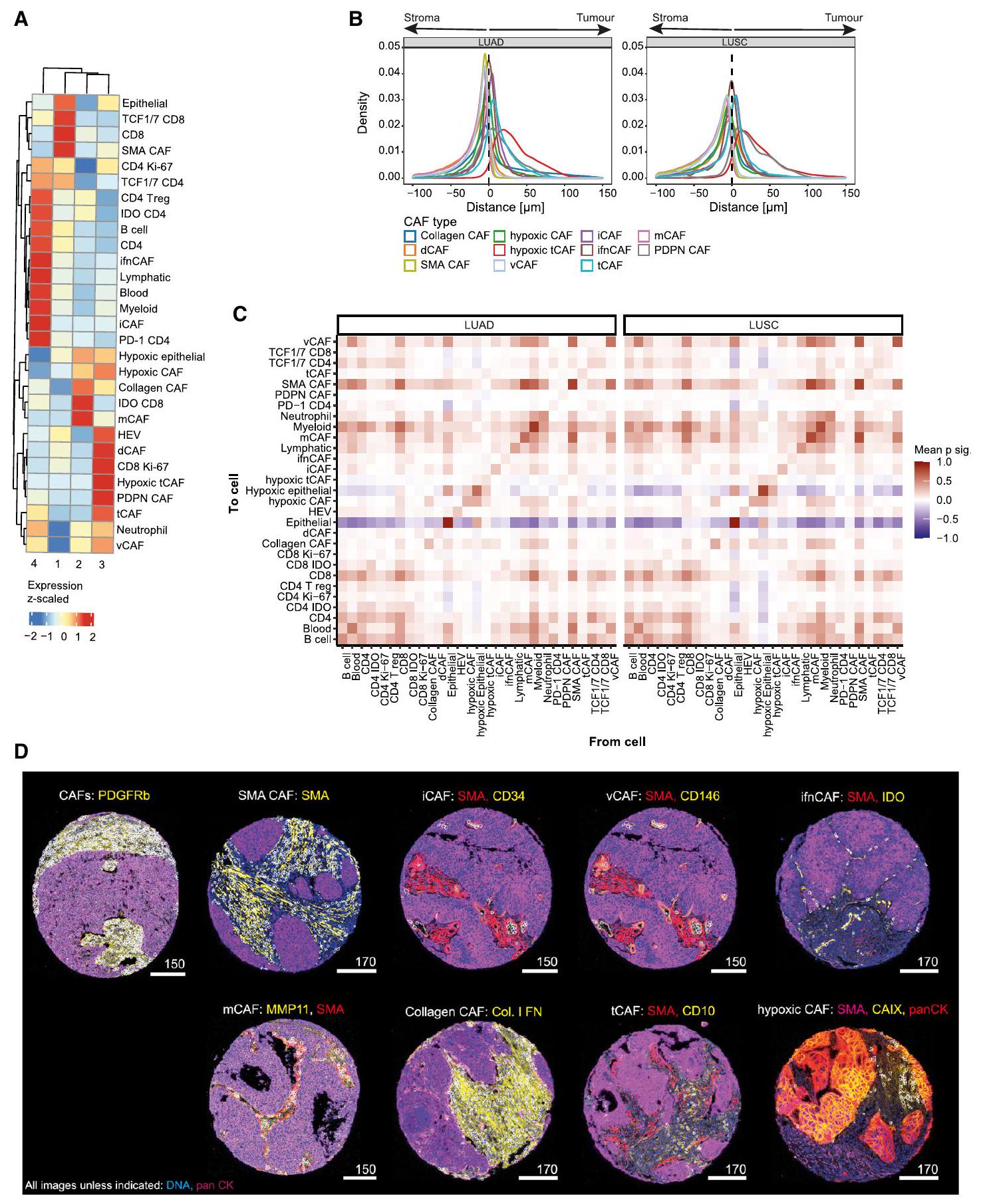
(A) Experimental set-up. NSCLC samples (2070 cores, 1,070 patients) and controls were stained with metal-tagged antibodies and imaged using IMC. Cell and tumor-stroma masks were generated.
(B) Analytical setup. Single-cell data were used to identify cell phenotypes, and to evaluate correlations between cell types and clinical data. CAF based patient stratifications were used for survival and TMA composition analyses. Finally, spatial properties of CAFs and immune cell types were analyzed and integrated with clinical data.
(C) Markers in the antibody panel.

(A) Exemplary LUAD (left) and LUSC cores (right) after cell segmentation based on panCK as a tumor marker and SMA as a stromal marker. Top to bottom, cells colored by average expression of SMA and panCK; cell assignment based on the generated tumor-stroma masks; cell category assignment; and the binned distance of all non-tumor cells to the nearest tumor-stroma border (
nosis (tCAFs, hypoxic tCAFs, collagen CAF, and mCAFs) or better prognosis (ifnCAFs, iCAFs, and SMA CAFs) in NSCLC.
CAF types are associated with chemoresistance and metastasis

(A) Heatmap of mean marker expression (z-scaled from -2 to 3) for all CAF phenotypes identified after FLOWSOM clustering. The bar chart above the heatmap shows the number (square root for visualization) of CAFs per tumor type for each cluster.
(B) UMAP of a random subset (
(C) UMAP of a random subset (
(D) Images showing all CAFs (left) and SMA CAFs (right); CAFs are shown with white masks overlaid on the indicated markers.
(E) Mean proportions (left plot) and absolute numbers (right plot) of each CAF type per patient are shown. Color bar (right) indicates CAF types. The hierarchical clustering tree is colored (different color scale from CAF types) by four meta-clustered patient groups (1, 2, 3, 4).
(F) Heatmap (z-scaled expression) showing enrichment of each CAF type per patient group as defined in E.

(A) Differential abundance testing of all cell types between patient groups defined by long (left) and short(right) overall survival, split by median. p Values
(B) Kaplan-Meier overall survival curves for patient groups (PG) defined by CAF composition (PG 1-4). The table shows patient numbers according to tumor type and p values for comparison between groups (
CAF types are associated with distinct TME compositions
iCAFs were significantly, if weakly, anti-correlated with all hypoxic cells (LUAD
CAFs have defined spatial distributions within the TME
Cancer Cell
Article

(A and B) Boxplots showing the average patient proportions for the indicated CAF types comparing patients that received neoadjuvant therapy versus a matched treatment-naïve cohort for LUAD (A) and LUSC (B). Exact p values are indicated.
(C and D) Differential abundance testing of all CAF types comparing patients receiving adjuvant chemotherapy who did or did not relapse, in LUAD (C) and LUSC (D). p Values < 0.05 are shown in blue, p values > 0.05 in grey.
(E and F) Differential abundance testing of all CAF types comparing patients with (N1/2) and without (N0) lymph node at diagnosis, in LUAD (E) and in LUSC (F). p Values < 0.05 are shown in blue, p values > 0.05 in grey.
(G) Boxplot showing the average proportions per patient of the indicated CAF types in matched patient groups with and without distant metastases at diagnosis. Exact p values are indicated.
as mCAF density low (Figure 7A). Further, all immune cells, including

(A) Heatmap showing enrichment or depletion of cell types between patient groups defined by CAF composition (
(B) Density curves showing the distance per cell of the indicated CAF types to the closest tumor-stroma border for LUAD (left) and LUSC (right).
(C) Neighborhood analysis heatmap showing nearest 15 neighbors of each cell within a radius of
(D) Images with mask overlays (white) of SMA CAFs (SMA
Cancer Cell
Article

(A) SpicyR analysis of cell pairs in patients stratified by median mCAF density. Positive scores (red) indicate higher cellular interactions between cell pairs in patients with low mCAF density, negative scores (blue) indicate higher cellular interactions in patient with high mCAF density; white indicates no difference.
(B) Proportions of B cells (upper) and CD4
(C and D) Images showing the indicated markers for a mCAF density high image (C) and a mCAF density low image (D), together with the corresponding masks colored by tumor cells (magenta), immune cells (yellow) and mCAFs (cyan). Scale bar,
by hypoxic and immune-exhausted microenvironments. In contrast, iCAFs that express CD34 and CD248 as well as ifnCAFs were associated with inflamed tumor microenvironments and higher overall patient survival.
DISCUSSION
whole cohort and in the subset of patients who received chemotherapy post-surgery, suggesting a tumor-suppressing role of ifnCAFs through interferon signaling cues.
potentially immunosuppressed IDO
Limitations of the study
tions that occur at the edges of the cells. Third, only one or two cores were imaged per patient, and all cores were taken from the invasive front. Thus, CAF features and heterogeneity that we describe may be unique to this region of NSCLC tumors. Finally, our study is descriptive and investigated correlation rather than causation, thus, our derived hypotheses are speculative. Nevertheless, this study is the largest multiplex imaging analysis of NSCLC and fibroblasts to date and should serve as a reference and resource for future work.
STAR*METHODS
- KeY RESOURCES TABLE
- RESOURCE AVAILABILITY
- Lead contact
- Material availability
- Data and code availability
- EXPERIMENTAL MODEL AND STUDY PARTICIPANT DETAILS
- METHOD DETAILS
- Antibody panel, staining, and imaging
- Segmentation pipeline
- Cell phenotype clustering
- QUANTIFICATION AND STATISTICAL ANALYSIS
- Data exclusion and subsetting
- Analysis of matched cohorts
- Statistical analyses
- Spatial analysis
- Hypoxia patch detection analysis
SUPPLEMENTAL INFORMATION
ACKNOWLEDGMENTS
AUTHOR CONTRIBUTIONS
Cancer Cell
Article
DECLARATION OF INTERESTS
Revised: November 2, 2023
Accepted: December 21, 2023
Published: January 18, 2024
REFERENCES
- Sung, H., Ferlay, J., Siegel, R.L., Laversanne, M., Soerjomataram, I., Jemal, A., and Bray, F. (2021). Global cancer statistics 2020: GLOBOCAN estimates of incidence and mortality worldwide for 36 cancers in 185 countries. CA. Cancer J. Clin. 71, 209-249.
- Schabath, M.B., and Cote, M.L. (2019). Cancer progress and priorities: Lung cancer. Cancer Epidemiol. Biomarkers Prev. 28, 1563-1579.
- Pikor, L.A., Ramnarine, V.R., Lam, S., and Lam, W.L. (2013). Genetic alterations defining NSCLC subtypes and their therapeutic implications. Lung Cancer 82, 179-189.
- Garon, E.B., Hellmann, M.D., Rizvi, N.A., Carcereny, E., Leighl, N.B., Ahn, M.J., Eder, J.P., Balmanoukian, A.S., Aggarwal, C., Horn, L., et al. (2019). Five-year overall survival for patients with advanced non-small-cell lung cancer treated with pembrolizumab: results from the phase I KEYNOTE001 study. J. Clin. Oncol. 37, 2518-2527.
- Robert, C. (2020). A decade of immune-checkpoint inhibitors in cancer therapy. Nat. Commun. 11, 3801.
- Sahai, E., Astsaturov, I., Cukierman, E., DeNardo, D.G., Egeblad, M., Evans, R.M., Fearon, D., Greten, F.R., Hingorani, S.R., Hunter, T., et al. (2020). A framework for advancing our understanding of cancer-associated fibroblasts. Nat. Rev. Cancer 20, 174-186.
- Hanahan, D., and Weinberg, R.A. (2011). Hallmarks of cancer: the next generation. Cell 144, 646-674.
- Turley, S.J., Cremasco, V., and Astarita, J.L. (2015). Immunological hallmarks of stromal cells in the tumour microenvironment. Nat. Rev. Immunol. 15, 669-682.
- Smyth, M.J., Ngiow, S.F., Ribas, A., and Teng, M.W.L. (2016). Combination cancer immunotherapies tailored to the tumour microenvironment. Nat. Rev. Clin. Oncol. 13, 143-158.
- Altorki, N.K., Markowitz, G.J., Gao, D., Port, J.L., Saxena, A., Stiles, B., McGraw, T., and Mittal, V. (2019). The lung microenvironment: an important regulator of tumour growth and metastasis. Nat. Rev. Cancer 19, 9-31.
- Finak, G., Bertos, N., Pepin, F., Sadekova, S., Souleimanova, M., Zhao, H., Chen, H., Omeroglu, G., Meterissian, S., Omeroglu, A., et al. (2008). Stromal gene expression predicts clinical outcome in breast cancer. Nat. Med. 14, 518-527.
- Bruni, D., Angell, H.K., and Galon, J. (2020). The immune contexture and Immunoscore in cancer prognosis and therapeutic efficacy. Nat. Rev. Cancer 20, 662-680.
- Su, S., Chen, J., Yao, H., Liu, J., Yu, S., Lao, L., Wang, M., Luo, M., Xing, Y., Chen, F., et al. (2018). CD10+GPR77+ Cancer-Associated Fibroblasts Promote Cancer Formation and Chemoresistance by Sustaining Cancer Stemness. Cell 172, 841-856.e16.
- Krishnamurty, A.T., Shyer, J.A., Thai, M., Gandham, V., Buechler, M.B., Yang, Y.A., Pradhan, R.N., Wang, A.W., Sanchez, P.L., Qu, Y., et al. (2022). LRRC15+ myofibroblasts dictate the stromal setpoint to suppress tumour immunity. Nature 611, 148-154.
- Genova, C., Dellepiane, C., Carrega, P., Sommariva, S., Ferlazzo, G., Pronzato, P., Gangemi, R., Filaci, G., Coco, S., and Croce, M. (2021). Therapeutic Implications of Tumor Microenvironment in Lung Cancer: Focus on Immune Checkpoint Blockade. Front. Immunol. 12, 799455.
- Caligiuri, G., and Tuveson, D.A. (2023). Activated fibroblasts in cancer: Perspectives and challenges. Cancer Cell 41, 434-449.
- Grout, J.A., Sirven, P., Leader, A.M., Maskey, S., Hector, E., Puisieux, I., Steffan, F., Cheng, E., Tung, N., Maurin, M., et al. (2022). Spatial Positioning and Matrix Programs of Cancer-Associated Fibroblasts Promote T-cell Exclusion in Human Lung Tumors. Cancer Discov. 12, 2606-2625.
- Ogawa, Y., Masugi, Y., Abe, T., Yamazaki, K., Ueno, A., Fujii-Nishimura, Y., Hori, S., Yagi, H., Abe, Y., Kitago, M., and Sakamoto, M. (2021). Three distinct stroma types in human pancreatic cancer identified by image analysis of fibroblast subpopulations and collagen. Clin. Cancer Res. 27, 107-119.
- Özdemir, B.C., Pentcheva-Hoang, T., Carstens, J.L., Zheng, X., Wu, C.C., Simpson, T.R., Laklai, H., Sugimoto, H., Kahlert, C., Novitskiy, S.V., et al. (2014). Depletion of carcinoma-associated fibroblasts and fibrosis induces immunosuppression and accelerates pancreas cancer with reduced survival. Cancer Cell 25, 719-734.
- Rhim, A.D., Oberstein, P.E., Thomas, D.H., Mirek, E.T., Palermo, C.F., Sastra, S.A., Dekleva, E.N., Saunders, T., Becerra, C.P., Tattersall, I.W., et al. (2014). Stromal elements act to restrain, rather than support, pancreatic ductal adenocarcinoma. Cancer Cell 25, 735-747.
- Bhattacharjee, S., Hamberger, F., Ravichandra, A., Miller, M., Nair, A., Affo, S., Filliol, A., Chin, L., Savage, T.M., Yin, D., et al. (2021). Tumor restriction by type I collagen opposes tumor-promoting effects of cancerassociated fibroblasts. J. Clin. Invest. 131, e146987.
- Chen, Y., Kim, J., Yang, S., Wang, H., Wu, C.J., Sugimoto, H., LeBleu, V.S., and Kalluri, R. (2021). Type I collagen deletion in
SMA+ myofibroblasts augments immune suppression and accelerates progression of pancreatic cancer. Cancer Cell 39, 548-565.e6. - Chen, Y., McAndrews, K.M., and Kalluri, R. (2021). Clinical and therapeutic relevance of cancer-associated fibroblasts. Nat. Rev. Clin. Oncol. 18, 792-804.
- Valkenburg, K.C., De Groot, A.E., and Pienta, K.J. (2018). Targeting the tumour stroma to improve cancer therapy. Nat. Rev. Clin. Oncol. 15, 366-381.
- Bughda, R., Dimou, P., D’Souza, R.R., and Klampatsa, A. (2021). Fibroblast Activation Protein (FAP)-Targeted CAR-T Cells: Launching an Attack on Tumor Stroma. ImmunoTargets Ther. 10, 313-323.
- Brünker, P., Wartha, K., Friess, T., Grau-Richards, S., Waldhauer, I., Koller, C.F., Weiser, B., Majety, M., Runza, V., Niu, H., et al. (2016). RG7386, a novel tetravalent FAP-DR5 antibody, effectively triggers FAP-dependent, avidity-driven DR5 hyperclustering and tumor cell apoptosis. Mol. Cancer Ther. 15, 946-957.
- Chen, Z., Zhou, L., Liu, L., Hou, Y., Xiong, M., Yang, Y., Hu, J., and Chen, K. (2020). Single-cell RNA sequencing highlights the role of inflammatory cancer-associated fibroblasts in bladder urothelial carcinoma. Nat. Commun. 11, 5077.
- Puram, S.V., Tirosh, I., Parikh, A.S., Patel, A.P., Yizhak, K., Gillespie, S., Rodman, C., Luo, C.L., Mroz, E.A., Emerick, K.S., et al. (2017). Single-cell transcriptomic analysis of primary and metastatic tumor ecosystems in head and neck cancer. Cell 171, 1611-1624.e24.
- Lee, H.-O., Hong, Y., Etlioglu, H.E., Cho, Y.B., Pomella, V., Van den Bosch, B., Vanhecke, J., Verbandt, S., Hong, H., Min, J.W., et al. (2020). Lineagedependent gene expression programs influence the immune landscape of colorectal cancer. Nat. Genet. 52, 594-603.
- Lambrechts, D., Wauters, E., Boeckx, B., Aibar, S., Nittner, D., Burton, O., Bassez, A., Decaluwé, H., Pircher, A., Van den Eynde, K., et al. (2018). Phenotype molding of stromal cells in the lung tumor microenvironment. Nat. Med. 24, 1277-1289.
- Tietscher, S., Wagner, J., Anzeneder, T., Langwieder, C., Rees, M., Sobottka, B., de Souza, N., and Bodenmiller, B. (2023). A comprehensive single-cell map of T cell exhaustion-associated immune environments in human breast cancer. Nat. Commun. 14, 98.
- Friedman, G., Levi-Galibov, O., David, E., Bornstein, C., Giladi, A., Dadiani, M., Mayo, A., Halperin, C., Pevsner-Fischer, M., Lavon, H., et al. (2020). Cancer-associated fibroblast compositions change with breast cancer
progression linking the ratio of S100A4+ and PDPN+ CAFs to clinical outcome. Nat. Cancer 1, 692-708. - Costa, A., Kieffer, Y., Scholer-Dahirel, A., Pelon, F., Bourachot, B., Cardon, M., Sirven, P., Magagna, I., Fuhrmann, L., Bernard, C., et al. (2018). Fibroblast Heterogeneity and Immunosuppressive Environment in Human Breast Cancer. Cancer Cell 33, 463-479.e10.
- Kieffer, Y., Hocine, H.R., Gentric, G., Pelon, F., Bernard, C., Bourachot, B., Lameiras, S., Albergante, L., Bonneau, C., Guyard, A., et al. (2020). Singlecell analysis reveals fibroblast clusters linked to immunotherapy resistance in cancer. Cancer Discov. 10, 1330-1351.
- Buechler, M.B., Pradhan, R.N., Krishnamurty, A.T., Cox, C., Calviello, A.K., Wang, A.W., Yang, Y.A., Tam, L., Caothien, R., Roose-Girma, M., et al. (2021). Cross-tissue organization of the fibroblast lineage. Nature 593, 575-579.
- Bartoschek, M., Oskolkov, N., Bocci, M., Lövrot, J., Larsson, C., Sommarin, M., Madsen, C.D., Lindgren, D., Pekar, G., Karlsson, G., et al. (2018). Spatially and functionally distinct subclasses of breast can-cer-associated fibroblasts revealed by single cell RNA sequencing. Nat. Commun. 9, 5150.
- Cords, L., Tietscher, S., Anzeneder, T., Langwieder, C., Rees, M., de Souza, N., and Bodenmiller, B. (2023). Cancer-associated fibroblast classification in single-cell and spatial proteomics data. Nat. Commun. 14, 4294.
- Elyada, E., Bolisetty, M., Laise, P., Flynn, W.F., Courtois, E.T., Burkhart, R.A., Teinor, J.A., Belleau, P., Biffi, G., Lucito, M.S., et al. (2019). Crossspecies single-cell analysis of pancreatic ductal adenocarcinoma reveals antigen-presenting cancer-associated fibroblasts. Cancer Discov. 9, 1102-1123.
- Öhlund, D., Handly-Santana, A., Biffi, G., Elyada, E., Almeida, A.S., PonzSarvise, M., Corbo, V., Oni, T.E., Hearn, S.A., Lee, E.J., et al. (2017). Distinct populations of inflammatory fibroblasts and myofibroblasts in pancreatic cancer. J. Exp. Med. 214, 579-596.
- Wu, S.Z., Roden, D.L., Wang, C., Holliday, H., Harvey, K., Cazet, A.S., Murphy, K.J., Pereira, B., Al-Eryani, G., Bartonicek, N., et al. (2020). Stromal cell diversity associated with immune evasion in human triplenegative breast cancer. EMBO J. 39, e104063.
- Giesen, C., Wang, H.A.O., Schapiro, D., Zivanovic, N., Jacobs, A., Hattendorf, B., Schüffler, P.J., Grolimund, D., Buhmann, J.M., Brandt, S., et al. (2014). Highly multiplexed imaging of tumor tissues with subcelIular resolution by mass cytometry. Nat. Methods 11, 417-422.
- Gachechiladze, M., Škarda, J., Kolek, V., Grygárková, I., Langová, K., Bouchal, J., Kolář, Z., Baty, F., Stahel, R., Weder, W., et al. (2017). Prognostic and predictive value of loss of nuclear RAD51 immunoreactivity in resected non-small cell lung cancer patients. Lung Cancer 105, 31-38.
- Tischler, V., Pfeifer, M., Hausladen, S., Schirmer, U., Bonde, A.K., Kristiansen, G., Sos, M.L., Weder, W., Moch, H., Altevogt, P., and Soltermann, A. (2011). L1CAM protein expression is associated with poor prognosis in non-small cell lung cancer. Mol. Cancer 10, 127.
- Soltermann, A., Tischler, V., Arbogast, S., Braun, J., Probst-Hensch, N., Weder, W., Moch, H., and Kristiansen, G. (2008). Prognostic significance of epithelial-mesenchymal and mesenchymal- epithelial transition protein expression in non-small cell lung cancer. Clin. Cancer Res. 14, 7430-7437.
- Berg, S., Kutra, D., Kroeger, T., Straehle, C.N., Kausler, B.X., Haubold, C., Schiegg, M., Ales, J., Beier, T., Rudy, M., et al. (2019). Ilastik: interactive machine learning for (bio) image analysis. Nat. Methods 16, 1226-1232.
- McQuin, C., Goodman, A., Chernyshev, V., Kamentsky, L., Cimini, B.A., Karhohs, K.W., Doan, M., Ding, L., Rafelski, S.M., Thirstrup, D., et al. (2018). CellProfiler 3.0: Next-generation image processing for biology. PLoS Biol. 16, e2005970.
- Van Gassen, S., Callebaut, B., Van Helden, M.J., Lambrecht, B.N., Demeester, P., Dhaene, T., and Saeys, Y. (2015). FlowSOM: Using selforganizing maps for visualization and interpretation of cytometry data. Cytometry A. 87, 636-645.
- Schwörer, S., Cimino, F.V., Ros, M., Tsanov, K.M., Ng, C., Lowe, S.W., Carmona-Fontaine, C., and Thompson, C.B. (2023). Hypoxia Potentiates the Inflammatory Fibroblast Phenotype Promoted by Pancreatic Cancer Cell-Derived Cytokines. Cancer Res. 83, 1596-1610.
- Garcia Garcia, C.J., Huang, Y., Fuentes, N.R., Turner, M.C., Monberg, M.E., Lin, D., Nguyen, N.D., Fujimoto, T.N., Zhao, J., Lee, J.J., et al. (2022). Stromal HIF2 Regulates Immune Suppression in the Pancreatic Cancer Microenvironment. Gastroenterology 162, 2018-2031.
- Lodyga, M., and Hinz, B. (2020). TGF-
– A truly transforming growth factor in fibrosis and immunity. Semin. Cell Dev. Biol. 101, 123-139. - Cremasco, V., Astarita, J.L., Grauel, A.L., Keerthivasan, S., Maclsaac, K., Woodruff, M.C., Wu, M., Spel, L., Santoro, S., Amoozgar, Z., et al. (2018). FAP delineates heterogeneous and functionally divergent stromal cells in immune-excluded breast tumors. Cancer Immunol. Res. 6, 1472-1485.
- Canete, N.P., Iyengar, S.S., Ormerod, J.T., Baharlou, H., Harman, A.N., and Patrick, E. (2022). spicyR: spatial analysis of in situ cytometry data in R. Bioinformatics 38, 3099-3105.
- Bejarano, L., Jordāo, M.J.C., and Joyce, J.A. (2021). Therapeutic Targeting of the Tumor Microenvironment. Cancer Discov. 11, 933-959.
- Zhang, M., Yang, H., Wan, L., Wang, Z., Wang, H., Ge, C., Liu, Y., Hao, Y., Zhang, D., Shi, G., et al. (2020). Single-cell transcriptomic architecture and intercellular crosstalk of human intrahepatic cholangiocarcinoma. J. Hepatol. 73, 1118-1130.
- Yu, Z., Zhang, J., Zhang, Q., Wei, S., Shi, R., Zhao, R., An, L., Grose, R., Feng, D., and Wang, H. (2022). Single-cell sequencing reveals the heterogeneity and intratumoral crosstalk in human endometrial cancer. Cell Prolif. 55, e13249.
- Foster, D.S., Januszyk, M., Delitto, D., Yost, K.E., Griffin, M., Guo, J., Guardino, N., Delitto, A.E., Chinta, M., Burcham, A.R., et al. (2022). Multiomic analysis reveals conservation of cancer-associated fibroblast phenotypes across species and tissue of origin. Cancer Cell 40, 13921406.e7.
- Werba, G., Weissinger, D., Kawaler, E.A., Zhao, E., Kalfakakou, D., Dhara, S., Wang, L., Lim, H.B., Oh, G., Jing, X., et al. (2023). Single-cell RNA sequencing reveals the effects of chemotherapy on human pancreatic adenocarcinoma and its tumor microenvironment. Nat. Commun. 14, 3912.
- Keller, P.J., Arendt, L.M., Skibinski, A., Logvinenko, T., Klebba, I., Dong, S., Smith, A.E., Prat, A., Perou, C.M., Gilmore, H., et al. (2012). Defining the cellular precursors to human breast cancer. Proc. Natl. Acad. Sci. USA 109, 2772-2777.
- Cheon, H., Wang, Y., Wightman, S.M., Jackson, M.W., and Stark, G.R. (2023). How cancer cells make and respond to interferon-I. Trends Cancer 9, 83-92.
- Benveniste, E.N., and Qin, H. (2007). Type I interferons as anti-inflammatory mediators. Sci. STKE 2007, pe70.
- Al Bakir, M., Huebner, A., Martínez-Ruiz, C., Grigoriadis, K., Watkins, T.B.K., Pich, O., Moore, D.A., Veeriah, S., Ward, S., Laycock, J., et al. (2023). The evolution of non-small cell lung cancer metastases in TRACERx. Nature 616, 534-542.
- Stagg, J., Divisekera, U., McLaughlin, N., Sharkey, J., Pommey, S., Denoyer, D., Dwyer, K.M., and Smyth, M.J. (2010). Anti-CD73 antibody therapy inhibits breast tumor growth and metastasis. Proc. Natl. Acad. Sci. USA 107, 1547-1552.
- Turcotte, M., Spring, K., Pommey, S., Chouinard, G., Cousineau, I., George, J., Chen, G.M., Gendoo, D.M.A., Haibe-Kains, B., Karn, T., et al. (2015). CD73 is associated with poor prognosis in high-grade serous ovarian cancer. Cancer Res. 75, 4494-4503.
- Wang, L., Zhou, X., Zhou, T., Ma, D., Chen, S., Zhi, X., Yin, L., Shao, Z., Ou, Z., and Zhou, P. (2008). Ecto-5′-nucleotidase promotes invasion, migration and adhesion of human breast cancer cells. J. Cancer Res. Clin. Oncol. 134, 365-372.
- Vijayan, D., Young, A., Teng, M.W.L., and Smyth, M.J. (2017). Targeting immunosuppressive adenosine in cancer. Nat. Rev. Cancer 17, 709-724.
Cancer Cell Article
CellPress
OPEN ACCESS
- Fu, Y., Liu, S., Yin, S., Niu, W., Xiong, W., Tan, M., Li, G., and Zhou, M. (2017). The reverse Warburg effect is likely to be an Achilles’ heel of cancer that can be exploited for cancer therapy. Oncotarget 8, 57813-57825.
- Pavlides, S., Whitaker-Menezes, D., Castello-Cros, R., Flomenberg, N., Witkiewicz, A.K., Frank, P.G., Casimiro, M.C., Wang, C., Fortina, P., Addya, S., et al. (2009). The reverse Warburg effect: Aerobic glycolysis in cancer associated fibroblasts and the tumor stroma. Cell Cycle 8, 3984-4001.
- Dragica, J., Song, M., and Wang, L. (2020). Roles of IFN-
in Tumor Progression and Regression a Review Biomarker Research, 8 (BioMed Central Ltd unless otherwise stated. Part of Springer Nature). - Uyttenhove, C., Pilotte, L., Théate, I., Stroobant, V., Colau, D., Parmentier, N., Boon, T., and Van den Eynde, B.J. (2003). Evidence for a tumoral immune resistance mechanism based on tryptophan degradation by indoleamine 2,3-dioxygenase. Nat. Med. 9, 1269-1274.
- Zimmermann, J.A., Hettiaratchi, M.H., and McDevitt, T.C. (2017). Enhanced Immunosuppression of T Cells by Sustained Presentation of Bioactive Interferon-
Within Three-Dimensional Mesenchymal Stem Cell Constructs. Stem Cells Transl. Med. 6, 223-237. - Hu, H., Piotrowska, Z., Hare, P.J., Chen, H., Mulvey, H.E., Mayfield, A., Noeen, S., Kattermann, K., Greenberg, M., Williams, A., et al. (2021). Three subtypes of lung cancer fibroblasts define distinct therapeutic paradigms. Cancer Cell 39, 1531-1547.e10.
- Yang, D., Liu, J., Qian, H., and Zhuang, Q. (2023). Q. Cancer-associated fibroblasts: from basic science to anticancer therapy. Exp. Mol. Med. 55, 1322-1332.
- Dominguez, C.X., Müller, S., Keerthivasan, S., Koeppen, H., Hung, J., Gierke, S., Breart, B., Foreman, O., Bainbridge, T.W., Castiglioni, A., et al. (2020). Single-cell RNA sequencing reveals stromal evolution into LRRC15+ myofibroblasts as a determinant of patient response to cancer immunotherapy. Cancer Discov. 10, 232-253.
- Bondjers, C., Kalén, M., Hellström, M., Scheidl, S.J., Abramsson, A., Renner, O., Lindahl, P., Cho, H., Kehrl, J., and Betsholtz, C. (2003). Transcription profiling of platelet-derived growth factor-B-deficient mouse embryos identifies RGS5 as a novel marker for pericytes and vascular smooth muscle cells. Am. J. Pathol. 162, 721-729.
- Schapiro, D., Jackson, H.W., Raghuraman, S., Fischer, J.R., Zanotelli, V.R.T., Schulz, D., Giesen, C., Catena, R., Varga, Z., and Bodenmiller, B. (2017). histoCAT: analysis of cell phenotypes and interactions in multiplex image cytometry data. Nat. Methods 14, 873-876.
- Windhager, J., Zanotelli, V.R.T., Schulz, D., Meyer, L., Daniel, M., Bodenmiller, B., and Eling, N. (2023). An end-to-end workflow for multiplexed image processing and analysis. Nat. Protoc. 18, 3565-3613.
- Levine, J.H., Simonds, E.F., Bendall, S.C., Davis, K.L., Amir, E.a.D., Tadmor, M.D., Litvin, O., Fienberg, H.G., Jager, A., Zunder, E.R., et al. (2015). Data-Driven Phenotypic Dissection of AML Reveals Progenitorlike Cells that Correlate with Prognosis. Cell 162, 184-197.
- Greenwald, N.F., Miller, G., Moen, E., Kong, A., Kagel, A., Dougherty, T., Fullaway, C.C., McIntosh, B.J., Leow, K.X., Schwartz, M.S., et al. (2022). Whole-cell segmentation of tissue images with human-level performance using large-scale data annotation and deep learning. Nat. Biotechnol. 40, 555-565.
- Jackson, H.W., Fischer, J.R., Zanotelli, V.R.T., Ali, H.R., Mechera, R., Soysal, S.D., Moch, H., Muenst, S., Varga, Z., Weber, W.P., and Bodenmiller, B. (2020). The single-cell pathology landscape of breast cancer. Nature 578, 615-620.
- Robinson, M.D., McCarthy, D.J., and Smyth, G.K. (2010). A Bioconductor package for differential expression analysis of digital gene expression data. Bioinformatics 26, 139-140.
- Weber, L.M., Nowicka, M., Soneson, C., and Robinson, M.D. (2019). Differential discovery in high-dimensional cytometry via high-resolution clustering. Commun. Biol. 2, 183.
STAR*METHODS
KEY RESOURCES TABLE
| REAGENT or RESOURCE | SOURCE | IDENTIFIER |
| Antibodies | ||
| Rabbit monoclonal anti-Vimentin; Clone (EPR3776); Lot(GR286525-2) | Abcam | Cat#ab193555; RRID: AB_2814713 |
| Rabbit polyclonal anti-p75 (CD271); Clone (polyclonal_ANT-007); Lot(ANT007AN0702) | alomone labs | Cat#ANT-007; RRID: AB_2039968 |
| Rabbit polyclonal anti-Collagen I; Clone (Polyclonal_Collagen I); Lot(GR3271183-3) | Abcam | Cat#ab34710; RRID: AB_731684 |
| Mouse monoclonal anti-Fibronectin; Clone (10/Fibronectin); Lot(6251888) | BD Biosciences | Cat#610078; RRID: AB_397486 |
| Rat monoclonal anti-Podoplanin; Clone (NC-08); Lot(B260834) | Biolegend | Cat#337002; RRID: AB_1595511 |
| Mouse monoclonal anti-pan Cytokeratin; Clone (AE1); Lot(3252910) | Millipore | Cat#MAB1612; RRID: AB_2132794 |
| Goat polyclonal anti-Carbonic Anhydrase IX; Clone (polyclonal_CA9_AF2188); Lot(VNQ0319011) | R&D Systems | Cat#AF2188; RRID: AB_416562 |
| Rabbit monoclonal anti-Histone H3; Clone (D1H2); Lot(15) | Cell Signal Technology | Cat#4499BF; RRID: AB_10544537 |
| Rabbit polyclonal anti-CD248; Clone (Polyclonal_Proteintech); Lot(00019535) | Proteintech | Cat#18160; RRID: AB_10858230 |
| Mouse monoclonal anti-CXCL-12; Clone (79018); Lot(JOJ0519031) | R&D Systems | Cat#MAB350-100; RRID: AB_2088149 |
| Goat polyclonal anti-LYVE-1; Clone (Polyclonal_LYVE-1); Lot(KPY0119052) | R&D Systems | Cat#AF2089; RRID: AB_355144 |
| Goat polyclonal anti-CCL21 ; Clone (Polyclonal_CCL21 / 6Ckine); Lot(AYJ218071) | R&D Systems | Cat#AF366; RRID: AB_355327 |
| Mouse monoclonal anti-Cadherin-11; Clone (283416); Lot(VLT0219091) | R&D Systems | Cat#MAB1790; RRID: AB_2076970 |
| Mouse monoclonal anti-CD45RA; Clone (HI100); Lot(B291512) | Biolegend | Cat#304102; RRID: AB_314406 |
| Rabbit monoclonal anti-Caveolin-1; Clone (D46G3); Lot(7) | Cell Signal Technology | Cat#3267BF; RRID: AB_2275453 |
| Mouse monoclonal anti-FOXP3; Clone (236A/E7); Lot(2129676) | eBioscience | Cat#14-4777-82; RRID: AB_467556 |
| Rabbit monoclonal anti-MMP9; Clone (D6O3H); Lot(4) | Cell Signal Technology | Cat#A0398; RRID: AB_2798289 |
| Rabbit monoclonal anti-CD140b/PDGFRb; Clone (Y92); Lot(GR296584-4) | Abcam | Cat#ab215978; RRID: AB_2894841 |
| Mouse monoclonal anti-CD8a; Clone (C8/144B); Lot(2132595) | eBioscience | Cat#14-0085-82; RRID: AB_11150240 |
| Rabbit polyclonal anti-Myeloperoxidase; Clone (Polyclonal MPO); Lot(20071134) | Dako | Cat#AB_2335676; RRID: AB_2335676 |
| Mouse monoclonal anti-CD15; Clone (HI98); Lot(B291510) | Biolegend | Cat#301902; RRID: AB_314194 |
| Rabbit monoclonal anti-CD4; Clone (EPR6855); Lot(GR3215375-27) | Abcam | Cat#ab181724; RRID: AB_2864377 |
| Rabbit monoclonal anti-Indoleamine 2-3-dioxygenase; Clone (SP260); Lot(GR3259345-2) | Abcam | Cat#ab245737; RRID: AB_2894840 |
| Rabbit monoclonal anti-CD10; Clone (E5P7S); Lot(2) | Cell Signal Technology | Cat#65534; RRID: AB_2894842 |
| Goat polyclonal anti-CD146; Clone (Polyclonal_ MCAM/CD146_RnD_AF932); Lot(ECL0319041) | R&D Systems | Cat#AF932; RRID: AB_355721 |
Cancer Cell Article
CellPress
OPEN ACCESS
| Continued | ||
| REAGENT or RESOURCE | SOURCE | IDENTIFIER |
| Mouse monoclonal anti-SMA; Clone (1A4); Lot(2183900) | eBioscience | Cat#14-9760-82; RRID: AB_2572996 |
| Rabbit monoclonal anti-CD73; Clone (D7F9A); Lot(2) | Cell Signal Technology | Cat#13160BF; RRID: AB_2716625 |
| Rat monoclonal anti-PNAd; Clone (MECA-79); Lot(B257139) | Biolegend | Cat#120802; RRID: AB_493555 |
| Rabbit monoclonal anti-VCAM-1; Clone (EPR5047); Lot(GR3255420-3) | Abcam | Cat#ab215380; RRID: AB_2894839 |
| Rabbit monoclonal anti-CD34; Clone (EP373Y); Lot(GR3271518-1) | Abcam | Cat#ab198395; RRID: AB_2889381 |
| Rabbit polyclonal anti-vWF; Clone (poly vwf); Lot(3322998) | Millipore | Cat#AB7356; RRID: AB_92216 |
| Mouse monoclonal anti-HLA-DR; Clone (TAL 1B5); Lot(GR3306331-1) | Abcam | Cat#ab20181; RRID: AB_445401 |
| Rabbit polyclonal anti-CD3; Clone (polyclonal_A0452); Lot(20073981) | Dako | Cat#A0452; RRID: AB_2335677 |
| Rabbit polyclonal anti-MMP11; Clone (polyclonal); Lot(GR111256-10) | Abcam | Cat#ab53143; RRID: AB_2042361 |
| Mouse monoclonal anti-Keratin Epithelial; Clone (AE3); Lot(3255457) | Millipore | Cat#MAB1611; RRID: AB_2134409 |
| Mouse monoclonal anti-CD20; Clone (L26); Lot(2172592) | eBioscience | Cat#14-0202-82; RRID: AB_10734340 |
| Rabbit monoclonal anti-CD31; Clone (EPR3094); Lot(GR3229164-1) | Abcam | Cat#ab207090; RRID: AB_2889382 |
| Rabbit monoclonal anti-FSP1 / S100A4; Clone (EPR2761(2)); Lot(GR317174-3) | Abcam | Cat#136784; RRID: AB_10807552 |
| Sheep polyclonal anti-FAP; Clone (polyclonal_FAP); Lot(ZKW0619101) | R&D Systems | Cat#AF3715; RRID: AB_2102369 |
| Mouse monoclonal anti-CD68; Clone (KP1); Lot(2162103) | eBioscience | Cat#14-0688-82; RRID: AB_11151139 |
| Rabbit monoclonal anti-CD279 (PD-1); Clone (D4W2J); Lot(6) | Cell Signal Technology | Cat# I44:I47; RRID: AB_2864408 |
| Rabbit monoclonal anti-TCF1/TCF7; Clone (C63D9); Lot(10) | Cell Signal Technology | Cat#2203; RRID: AB_2534154 |
| Mouse monoclonal anti-CD45RO; Clone (UCHL1); Lot(B291511) | Biolegend | Cat#304202; RRID: AB_314418 |
| Rabbit monoclonal anti-K-Cadherin ; Clone (D3T3I); Lot(2) | Cell Signal Technology | Cat#48111; RRID: AB_2799334 |
| Mouse monoclonal anti-Ki-67; Clone (B56); Lot(8239549) | BD Biosciences | Cat#556003; RRID: AB_396287 |
| Biological samples | ||
| Human NSCLC TMA sections | University Hospital Zurich, Zurich, Switzerland | https://www.usz.ch/ |
| Chemicals, peptides, and recombinant proteins | ||
| Iridium intercalator | Fluidigm | Cat#201192B |
| Critical commercial assays | ||
| MaxPar X8 Multimetal Labeling Kit | Fluidigm | Cat#201300 |
| Deposited data | ||
| IMC images | This paper | Zenodo: https://doi.org/10.5281/zenodo. 7961844 |
| Clinical data | This paper | Zenodo: |
| Software and algorithms | ||
| CellProfiler v3.1.9 | McQuin et al.
|
http://cellprofiler.org |
| HistoCAT | Schapiro et al.
|
https://github.com/BodenmillerGroup/ histoCAT/releases |
| Continued | ||
| REAGENT or RESOURCE | SOURCE | IDENTIFIER |
| Ilastik v.1.3.3 | Berg et al.
|
https://www.ilastik.org/news/ 2019/09/30/ilastik-1.3.3 |
| Python v 3.4.3 | Python Software Foundation | https://www.python.org |
| R v4.1 | R Development Core Team | https://www.r-project.org |
| Bodenmiller lab imctools | Bodenmiller lab | https://github.com/BodenmillerGroup/ imctools |
| Bodenmiller lab segmentation pipeline | Bodenmiller lab | https://github.com/BodenmillerGroup/ ImcSegmentationPipeline |
| Bodenmiller CellProfiler plugins | Bodenmiller lab | https://github.com/BodenmillerGroup/ ImcPluginsCP |
| Bodenmiller lab spillover compensation | Bodenmiller lab | https://github.com/BodenmillerGroup/ cyTOFcompensation |
| imcRtools v1.3.7 | Windhager et al.
|
https://doi.org/10.1038/s41596-023-00881-0. |
| Rphenoannoy v0.1.0 | Levine JH et al.
|
https://doi.org/10.1016/j.cell.2015.05.047 |
| FLOWSOM v2.2.0 | Van et al.
|
https://onlinelibrary.wiley.com/doi/ full/10.1002/cyto.a.22625. |
| Analysis code | This paper | https://doi.org/10.5281/zenodo. 7981268 |
RESOURCE AVAILABILITY
Lead contact
Material availability
Data and code availability
EXPERIMENTAL MODEL AND STUDY PARTICIPANT DETAILS
METHOD DETAILS
Antibody panel, staining, and imaging
segmentation but excluded from subsequent clustering analyses. Cadherin-6 was excluded from all analyses as it did not show any specific staining pattern. All antibodies were selected based on our previous study of CAF heterogeneity including a scRNA-seq dataset of NSCLC.
Segmentation pipeline
Cell phenotype clustering
QUANTIFICATION AND STATISTICAL ANALYSIS
Data exclusion and subsetting
Analysis of matched cohorts
Statistical analyses
Spatial analysis
Hypoxia patch detection analysis
- (C) CoxPH-model for patient groups, corrected for tumor type and grade. Patient group 1 serves as a reference. p Values
are shown in blue, p values in grey.
(D and E) Kaplan-Meier (plots for overall survival comparing patients stratified as high and low based on the median proportion of indicated CAF types. Good prognosis CAFs are in D, poor prognosis CAFs in E. All comparisons with significant differences (log rank testing p < 0.05) are shown.
(F) Lasso-regressed CoxPH model including mean CAF type proportions per patient, patient stratification into high and low for each CAF type (by median proportion), and all clinical data.
