DOI: https://doi.org/10.1007/s40747-024-01764-x
تاريخ النشر: 2025-01-15
إطار هجين لاكتشاف وتصنيف أمراض أوراق النباتات باستخدام الشبكات العصبية التلافيفية ومحولات الرؤية
© المؤلف(ون) 2024
الملخص
مؤخراً، استخدم العلماء بشكل واسع أساليب الذكاء الاصطناعي في الزراعة الذكية لزيادة إنتاجية قطاع الزراعة والتغلب على مجموعة واسعة من المشكلات. يعد اكتشاف وتصنيف أمراض النباتات مشكلة صعبة بسبب الأعداد الكبيرة من النباتات في جميع أنحاء العالم والعديد من الأمراض التي تؤثر سلباً على إنتاج المحاصيل المختلفة. الهدف من أي نظام يعتمد على الذكاء الاصطناعي هو الكشف المبكر والتصنيف الدقيق لأمراض النباتات. تقترح هذه الورقة إطار عمل هجين لتحسين دقة التصنيف لأمراض أوراق النباتات بشكل كبير. يستفيد هذا النموذج المقترح من قوة الشبكات العصبية التلافيفية (CNNs) ومحولات الرؤية (ViT)، حيث يتم استخدام نموذج تجميعي يتكون من الهياكل المعروفة للشبكات العصبية التلافيفية VGG16 وInception-V3 وDenseNet20 لاستخراج ميزات عالمية قوية. ثم يتم استخدام نموذج ViT لاستخراج ميزات محلية للكشف بدقة عن أمراض النباتات. يتم تقييم أداء النموذج المقترح باستخدام مجموعتين من البيانات متاحة للجمهور (التفاح والذرة). تتكون كل مجموعة بيانات من أربع فئات. يكتشف النموذج الهجين المقترح بنجاح ويصنف أمراض أوراق النباتات متعددة الفئات ويتفوق على طرق مشابهة تم نشرها مؤخراً، حيث حقق النموذج الهجين المقترح معدل دقة قدره
الاختصارات
شبكات CNN العصبية التلافيفية
محول الرؤية فيت
تعلم الآلة
تعلم عميق
| SVM | آلة الدعم الناقل |
| الشبكات العصبية الاصطناعية | الشبكات العصبية الاصطناعية |
| ملاحظة | بايزي الساذج |
| إم إل بي | الإدراك متعدد الطبقات |
| MHA | الانتباه الذاتي متعدد الرؤوس |
| طبيب عام | التجميع العالمي |
| FFN | شبكة التغذية الأمامية |
| NL | طبقة التطبيع |
| LR | معدل التعلم |
| الجامعة الأمريكية في القاهرة | المساحة تحت المنحنى |
| ROC | الخصائص التشغيلية للمستقبل |
| رئيس الوزراء | مصفوفة الالتباس |
مقدمة
بشكل فعال مع صانعي السياسات. بالإضافة إلى ذلك، تؤثر التغيرات العالمية مثل تغير المناخ على أنواع التهديدات المرضية التي تواجهها، ونتائجها المحتملة، والطرق المتبعة لمعالجتها. قد يواجه المزارعون صعوبة في تشخيص أمراض النباتات بدقة بسبب ميزاتها الصغيرة. علاوة على ذلك، يفتقر العديد من المزارعين إلى المعرفة لتشخيص الأمراض، لذا يمكن أن تساعدهم الذكاء الاصطناعي في تشخيص الأمراض بدقة أكبر.
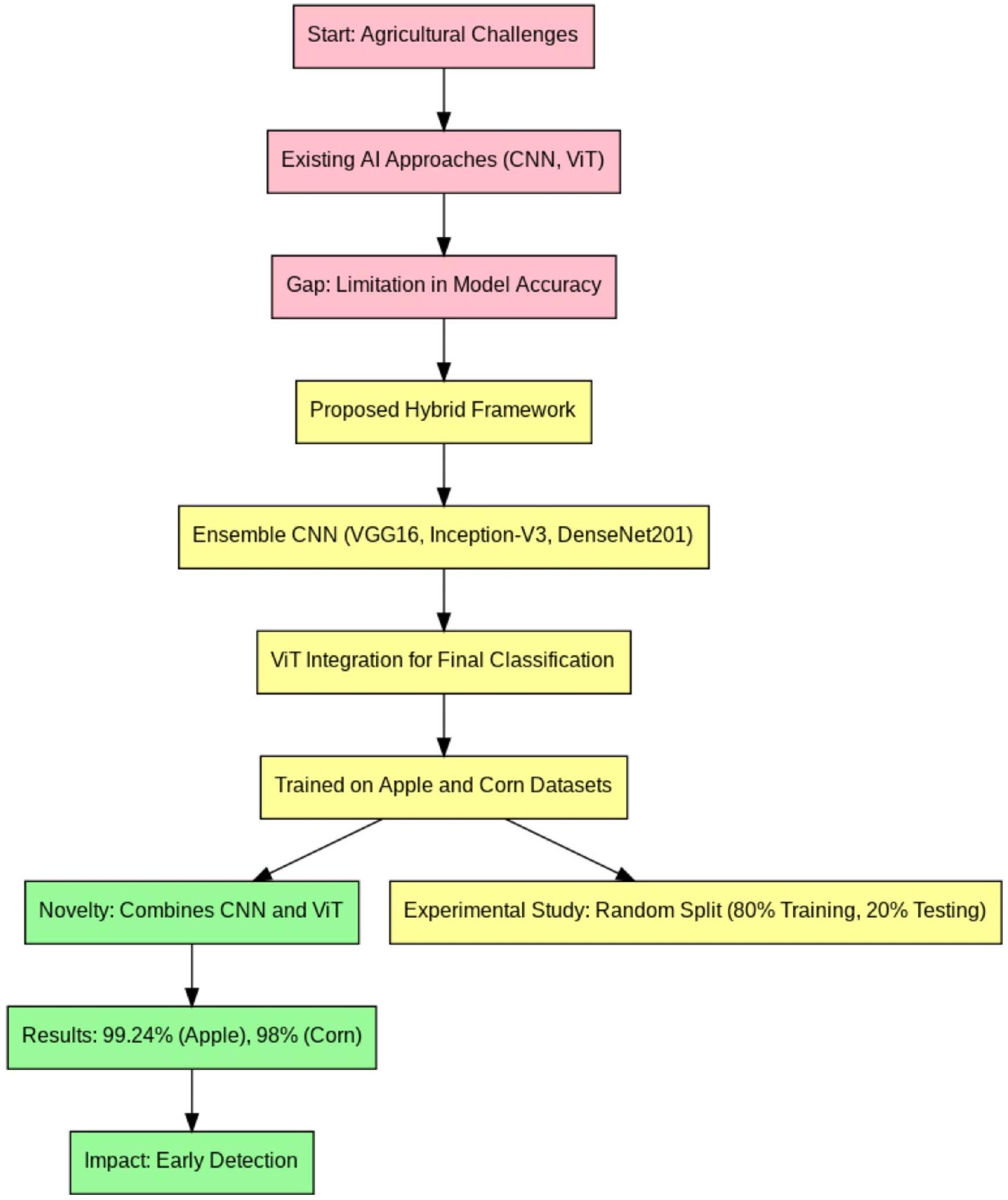
الأنواع العديدة من الأمراض والمحاصيل المتنوعة المعنية. تشابه الأعراض بين الأمراض المختلفة وأنماط هذه الأمراض المتغيرة مع مرور الوقت يزيد من تعقيد الوضع. التحدي هو أن الأساليب التقليدية قد واجهت صعوبة في استخراج الميزات ذات الصلة من الصور المدخلة. وبالتالي، يفضل الباحثون نماذج التعلم العميق، وخاصة الشبكات العصبية التلافيفية (CNNs)، التي يمكنها استخراج الميزات ذات الصلة والمعلوماتية تلقائيًا من البيانات المدخلة، مما يؤدي إلى أداء متفوق. ومع ذلك، فإن نماذج CNN محدودة في قدرتها على تحليل العلاقات بين البكسلات البعيدة، حيث إنها تأخذ في الاعتبار فقط الارتباط بين البكسلات المجاورة مكانيًا ضمن المجال الاستقبالي المحدد بحجم الفلتر. علاوة على ذلك، على الرغم من فعاليتها، غالبًا ما تتطلب نماذج CNN الحالية مجموعات بيانات كبيرة وموارد حسابية كبيرة، مما يمثل تحديات للتطبيقات في الوقت الحقيقي في البيئات ذات الموارد المحدودة. استكشف الباحثون مؤخرًا استخدام آليات الانتباه لمعالجة التحديات المرتبطة بالقدرة المحدودة لنموذج CNN في التقاط العلاقات بين البكسلات البعيدة. بالإضافة إلى ذلك، ركزت معظم الدراسات الحالية على نماذج CNN الفردية، مع تحقيق محدود في الفوائد المحتملة لأساليب النمذجة الجماعية لتحسين تعميم النتائج. وبالتالي، أنشأنا إطار عمل هجين يجمع بين عدة هياكل DL ومحولات الرؤية لاستخراج ميزات عميقة من خلال نهج هجين، كما هو موضح في الشكل 1. يمكن تلخيص النقاط الرئيسية في هذه الورقة كما يلي:
- يتم اقتراح إطار هجين، يتضمن الشبكات العصبية التلافيفية العميقة (CNNs) ومحولات الرؤية، للكشف عن أمراض أوراق النباتات وتصنيفها.
- الإطار المقترح من البداية إلى النهاية يلتقط أبرز الميزات، مما يسمح بالكشف الدقيق والتصنيف متعدد الفئات لأمراض أوراق النباتات.
- تمت تجربة النموذج المقترح بشكل مكثف مع نوعين مختلفين من مجموعات بيانات أمراض أوراق النباتات (الذرة والتفاح) وتمت مقارنته بالنماذج المتطورة باستخدام نفس مجموعات البيانات.
الأعمال ذات الصلة
من خلال الاستفادة من المحولات وآليات الانتباه الذاتي. يقوم النموذج بتقسيم البيانات البصرية إلى أجزاء صغيرة ومحلية، ويحسب الارتباطات بين هذه المناطق باستخدام آلية الانتباه، ثم يجمع المعلومات الأكبر والأكثر شمولاً لإبلاغ مهمة التصنيف [35]. أظهر ريدا وآخرون [36] فعالية نموذج المحول البصري الخالي من الالتفاف، الذي يستفيد من آلية الانتباه الذاتي لمعالجة الصورة كسلسلة من الأجزاء من خلال مشفر المحول القياسي. على الرغم من استخدام مجموعة بيانات صغيرة نسبيًا، حققوا أداءً عاليًا، والذي نسبوه إلى تقنيات مثل زيادة البيانات، والتعلم الانتقالي، وعدد محدود من الفئات [36]. استخدم وو وآخرون [37] نهجًا مزدوج النطاق من خلال تمرير مجموعة البيانات عبر نموذجين من ViT في وقت واحد، أحدهما بحجم جزء صغير والآخر بحجم جزء كبير. ثم تم دمج مخرجات هذين النموذجين من ViT باستخدام نموذج دمج وتم تغذيتها إلى رأس متعدد الطبقات. استنتج الباحثون أنه من خلال دمج مقاييس مختلفة من تسلسلات الانتباه الذاتي، يمكن للنموذج استخراج معلومات أغنى من الصور على مستويات مختلفة من الدقة [37]. تم تلخيص تحليل الطرق الحالية المستخدمة لتحديد أمراض النباتات في الجدول 1. كما هو موضح في الجدول 1، تشير أداء التصنيف إلى الحاجة إلى مزيد من التحسينات.
المواد والطرق
العمارة المقترحة
| مرجع | هدف | طريقة | النتائج الرئيسية | القيود | ||
| جيانغ وآخرون (2019) [20] | الكشف الفوري عن أمراض أوراق التفاح | شبكة عصبية تلافيفية محسّنة | – توفير حل عالي الأداء مع كشف في الوقت الحقيقي |
|
||
| بكوميرا وآخرون (2022) [21] | تصنيف الجزر استنادًا إلى رؤية الكمبيوتر وشبكة الأعصاب التلافيفية الرسومية (GCNN) | شبكة CNN ذات الرسم البياني المتسلسل مع تحسين بايزي | – فعال في التقييم الآلي للجزر |
|
||
| فو وآخرون (2022) [22] | نموذج خفيف الوزن لاكتشاف أمراض أوراق التفاح | شبكة عصبية خفيفة الوزن | – دقة عالية مع تقليل تعقيد النموذج | – مركّز على أوراق التفاح؛ قد يفتقر النموذج إلى المتانة بالنسبة لمحاصيل أخرى. | ||
| بانسال وآخرون (2021) [23] | كشف مرض أوراق التفاح | مجموعة من نماذج الشبكات العصبية التلافيفية العميقة المدربة مسبقًا | -تحسين دقة تصنيف أمراض التفاح | – دقة التعرف تتدهور لعدة أمراض | ||
| خان وآخرون (2022) [24] | نظام الكشف عن أمراض أوراق التفاح في الوقت الحقيقي | شبكة CNN خفيفة الوزن وYolov4 | – نموذج خفيف الوزن مع أداء في الوقت الحقيقي | – أداء التصنيف يحتاج إلى تحسين | ||
| تاجي وآخرون (2024) [27] | تصنيف أمراض النباتات باستخدام ميزات الشبكات العصبية التلافيفية الهجينة | إطار عمل هجين يعتمد على الخوارزميات الميتاهيرستية والشبكات العصبية التلافيفية | – تحسين الأداء من خلال تحسين الميتاهيرستيك | تعديل الميتاهيرستيك معقد، مما قد يؤدي إلى تكاليف حسابية عالية ووقت طويل لت优化 المعلمات. | ||
| حسني وآخرون (2023) [29] | تصنيف أمراض أوراق النباتات متعددة الفئات | دمج الميزات بين الشبكات العصبية التلافيفية ونمط الثنائي المحلي | -تحسين دقة التصنيف متعدد الفئات | – قد تعيق التعقيدات في دمج الميزات قابلية تفسير النموذج ومرونته. | ||
| بريادارshini وآخرون (2019) [30] | تصنيف أمراض أوراق الذرة | لي نت المعدل | -نموذج خفيف الوزن مع أداء في الوقت الحقيقي | – تم تقييمه فقط لتصنيف أمراض أوراق الذرة | ||
| وحيد وآخرون (2020) [31] | التعرف على مرض أوراق الذرة وتصنيفه | – نموذج DenseNet المحسن | – تحسين كفاءة ودقة التصنيف | – قد تؤدي البنية المعمارية الكثيفة إلى زيادة استهلاك الذاكرة، مما قد يؤثر على النشر على الأجهزة ذات الموارد المحدودة. | ||
| وو وآخرون (2019) [32] | الكشف الذاتي عن أمراض النباتات | نموذج CNN على الصور الجوية | – فعال في الكشف عن الأمراض على نطاق واسع | – يقتصر على الصور الجوية، التي قد تفوت الأعراض الدقيقة للمرض. | ||
| زينغ ولي (2020) [33] | التعرف على أمراض أوراق المحاصيل | نموذج CNN مع الانتباه الذاتي | – تحسين الدقة باستخدام آلية الانتباه الذاتي | – تقييمات محدودة على مجموعات بيانات المحاصيل. | ||
| تشن وآخرون (2021) [34] | كشف أمراض نبات الأرز | MobileNet-V2 المدرب مسبقًا مع آلية الانتباه | – فعال ودقيق لأمراض الأرز | – الهيكل الخفيف قد يضحي بالتفاصيل في مجموعات البيانات المتنوعة أو عالية الدقة. | ||
| كيان وآخرون (2022) [35] | تحديد أمراض أوراق الذرة | شبكة عصبية تلافيفية قائمة على آلية الانتباه | – دقة محسّنة مع الانتباه الذاتي | – التكلفة الحسابية العالية بسبب طبقات الانتباه قد تحد من التطبيق في الوقت الحقيقي. | ||
| ريدها وآخرون (2022) [36] | تصنيف الأعشاب والمحاصيل | شبكة عميقة قائمة على الانتباه | – أداء عالي، خاصة مع مجموعات بيانات تدريب صغيرة | معالجة الصور عالية الدقة تتطلب موارد حسابية كبيرة، مما قد يعيق الاستخدام في الوقت الحقيقي. | ||
| وو وآخرون (2021) [37] | التعرف على أمراض أوراق الطماطم | فيت | – نموذج استخراج الميزات متعدد الدقة | – قد تحدد التعقيد الحسابي العالي لمحوّلات الرؤية من الاستخدام في الوقت الحقيقي |
والميزات العالمية، مما يعزز من قابلية تفسير النموذج.
1.1. هياكل التعلم العميق المدربة مسبقًا
1.1.1.VGG16
نماذج مدربة مسبقًا
(استخراج الميزات العميقة)
1.1.2. جوجل نت
منطقة محلية. بعد ذلك، يتم جمع هذه الميزات المتنوعة ودمجها على مستوى محدد قبل أن يتم نقلها إلى الطبقة التالية.
1.1.3. DenseNet201
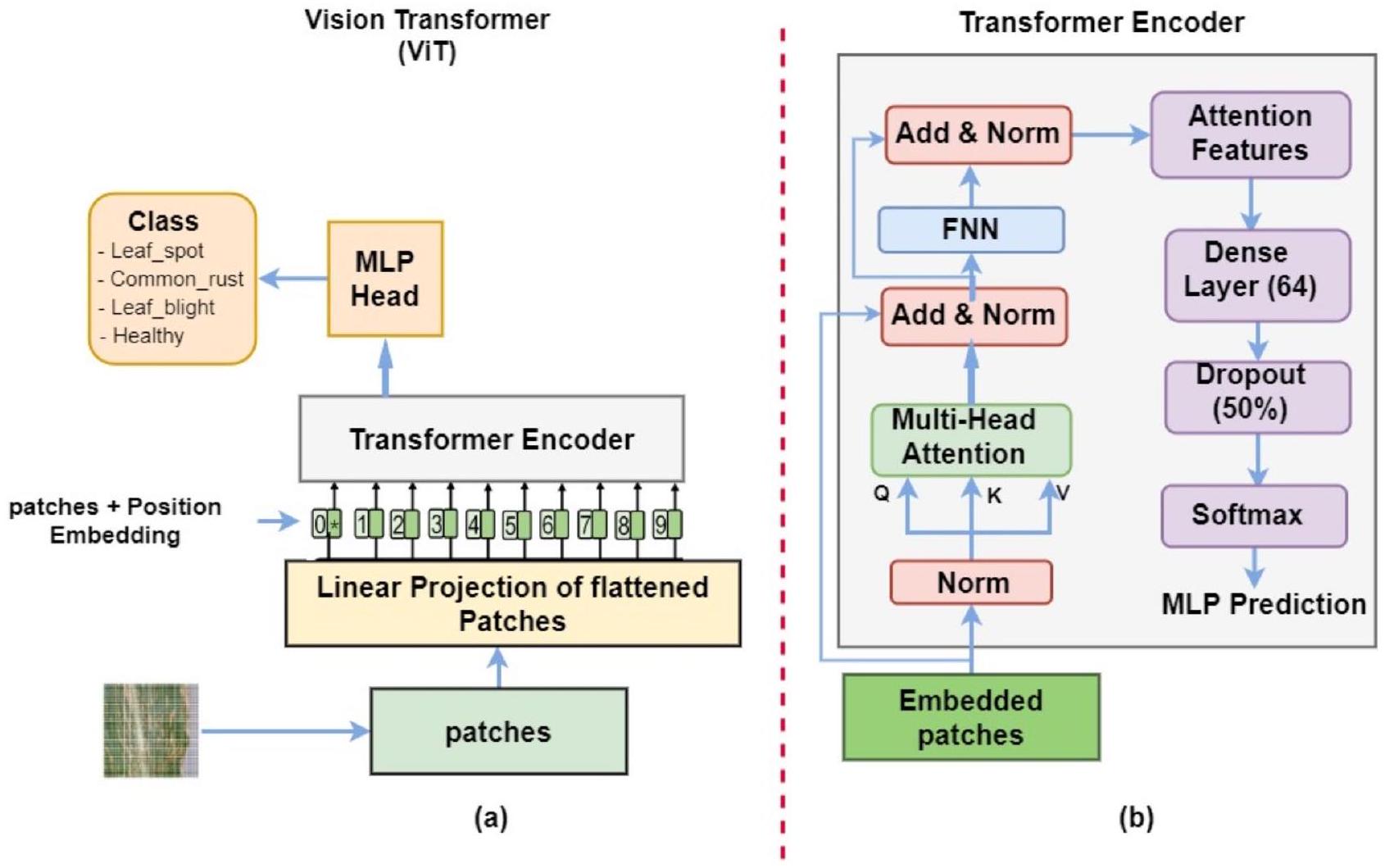
1.2. محول الرؤية (ViT)
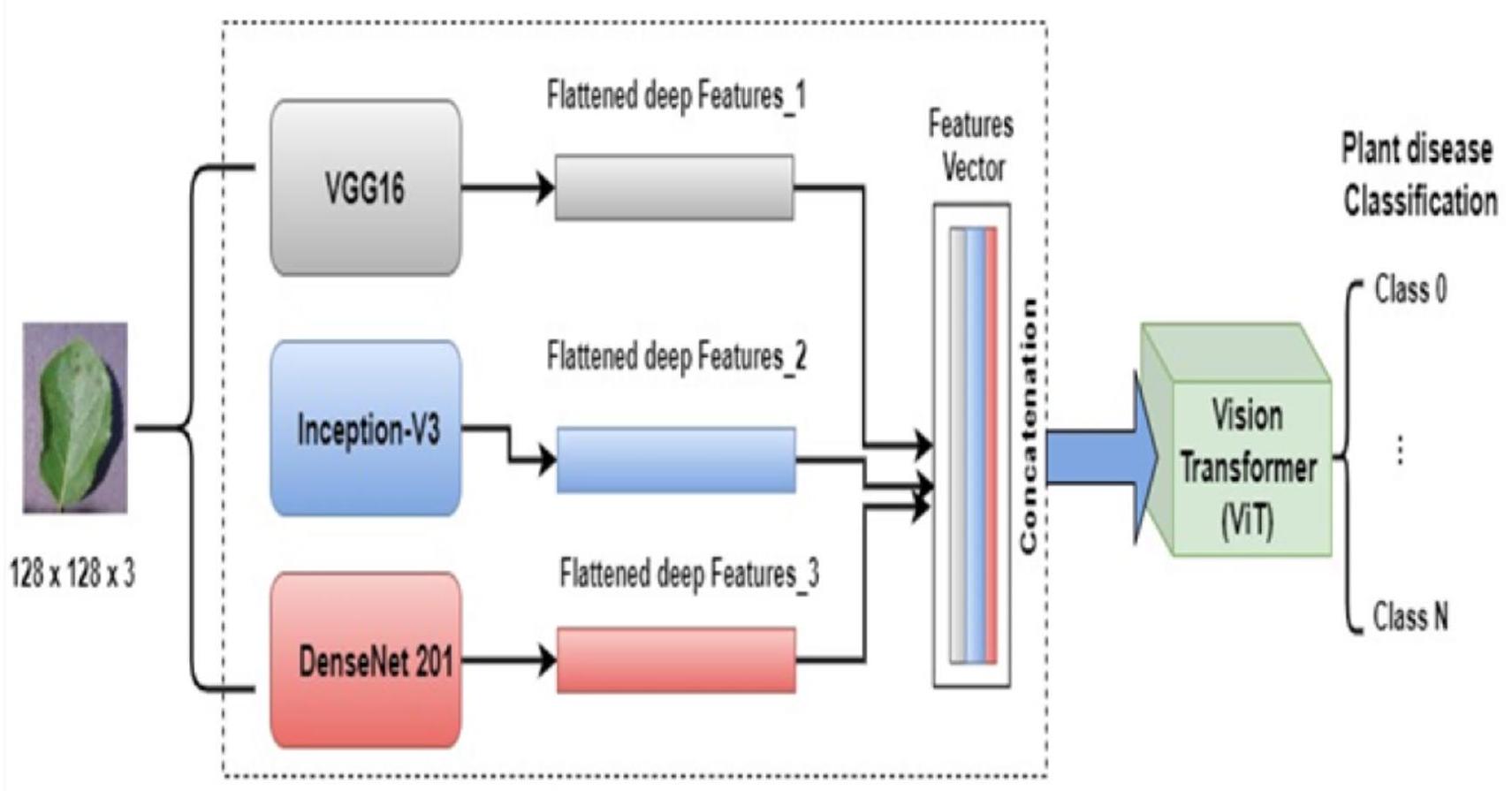
1.3. استخراج ودمج الميزات العميقة
| فهرس الصف | اسم الصف | حجم مجموعة التدريب (80%) | حجم مجموعة الاختبار (20%) | إجمالي |
| 0 | صدأ التفاح | ٨٠٠ | ٢٠٠ | ١٠٠٠ |
| 1 | تعفن أسود | ٨٠٠ | ٢٠٠ | ١٠٠٠ |
| 2 | صدأ تفاح الأرز | ٨٠٠ | ٢٠٠ | 1,000 |
| ٣ | صحي | ١٣١٦ | ٣٢٩ | 1,645 |
| إجمالي | ٣٧١٦ | 929 | ٤٦٤٥ |
| فهرس الصف | اسم الصف | حجم مجموعة التدريب (80%) | حجم مجموعة الاختبار (20%) | إجمالي |
| 0 | بقعة الأوراق الرمادية | ١٣٦٣ | ٣٤١ | ١٧٠٤ |
| 1 | صدأ شائع | 1,358 | ٣٤٠ | ١٦٩٨ |
| 2 | تعفن الأوراق الشمالية | ١٣٦٣ | ٣٤١ | ١٧٠٤ |
| ٣ | صحي | ١٣٣٤ | ٣٣٤ | 1,668 |
| إجمالي | ٥٤١٨ | 1,356 | ٦٧٧٤ |
تضمن ذلك إضافة حشو صفري إلى بنية GoogleNet قبل دمج الميزات.
1.4. مجموعات البيانات
1.5. إعداد التجارب

| وظيفة | معامل | قيمة |
| معلمات التدريب | محسّن | آدم |
| معدل التعلم | 0.0001 | |
| عصور | 50 | |
| حجم الدفعة | ٣٢ | |
| معلمات ViT | حجم الدفعة | ٢ |
| البعد المدمج | 64 | |
| عدد رؤوس الانتباه | ٨ | |
| عدد طبقات الشبكة العصبية متعددة الطبقات | 256 |
1.6. تقييم النموذج
| نموذج التعلم العميق | مقاييس التقييم | |||
| الدقة (%) | الدقة (%) | استرجاع (%) | درجة F1 (%) | |
| تفاح | 96 | 96 | 96 | 96 |
| VGG16 | ||||
| إنسيبشن-3 | 94 | 94 | 94 | 94 |
| دينس نت 201 | 97 | 97 | 97 | 97 |
| VGG1VGG16 + InceptionV3 + DenseNet201 | 97.6 | ٩٨ | 98 | 98 |
| النموذج الهجين المقترح | 99.24 | 99 | 99 | 99 |
| ذرة | 96 | 96 | 96 | 96 |
| في جي جي 16 | ||||
| إنسيبشن-3 | 93.87 | 94 | 94 | 94 |
| دينس نت 201 | 97 | 97 | 97 | 97 |
| VGG16 + InceptionV3 + DenseNet201 | 97 | 97 | 97 | 97 |
| النموذج الهجين المقترح | 98 | 98 | 98 | 98 |
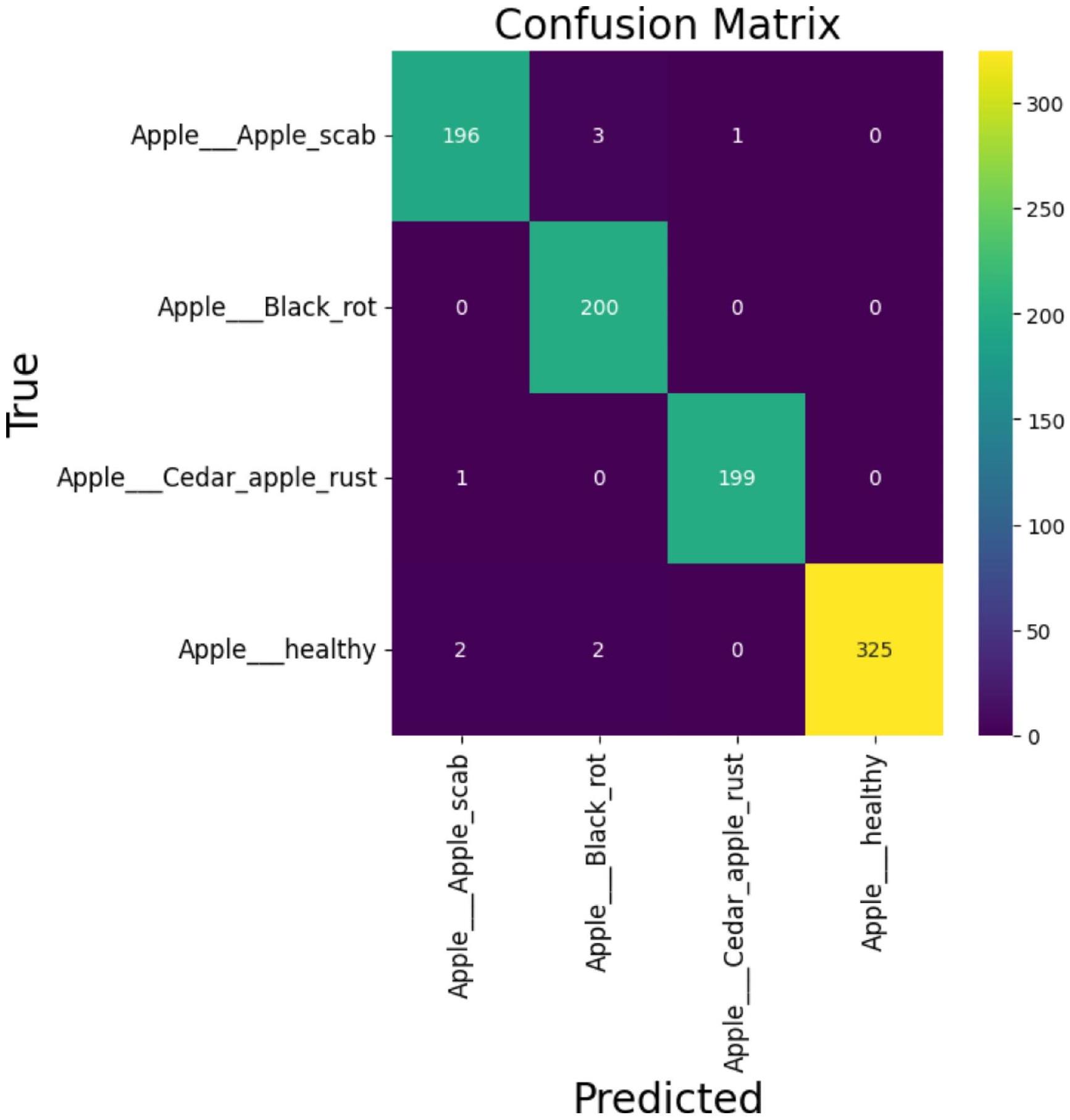
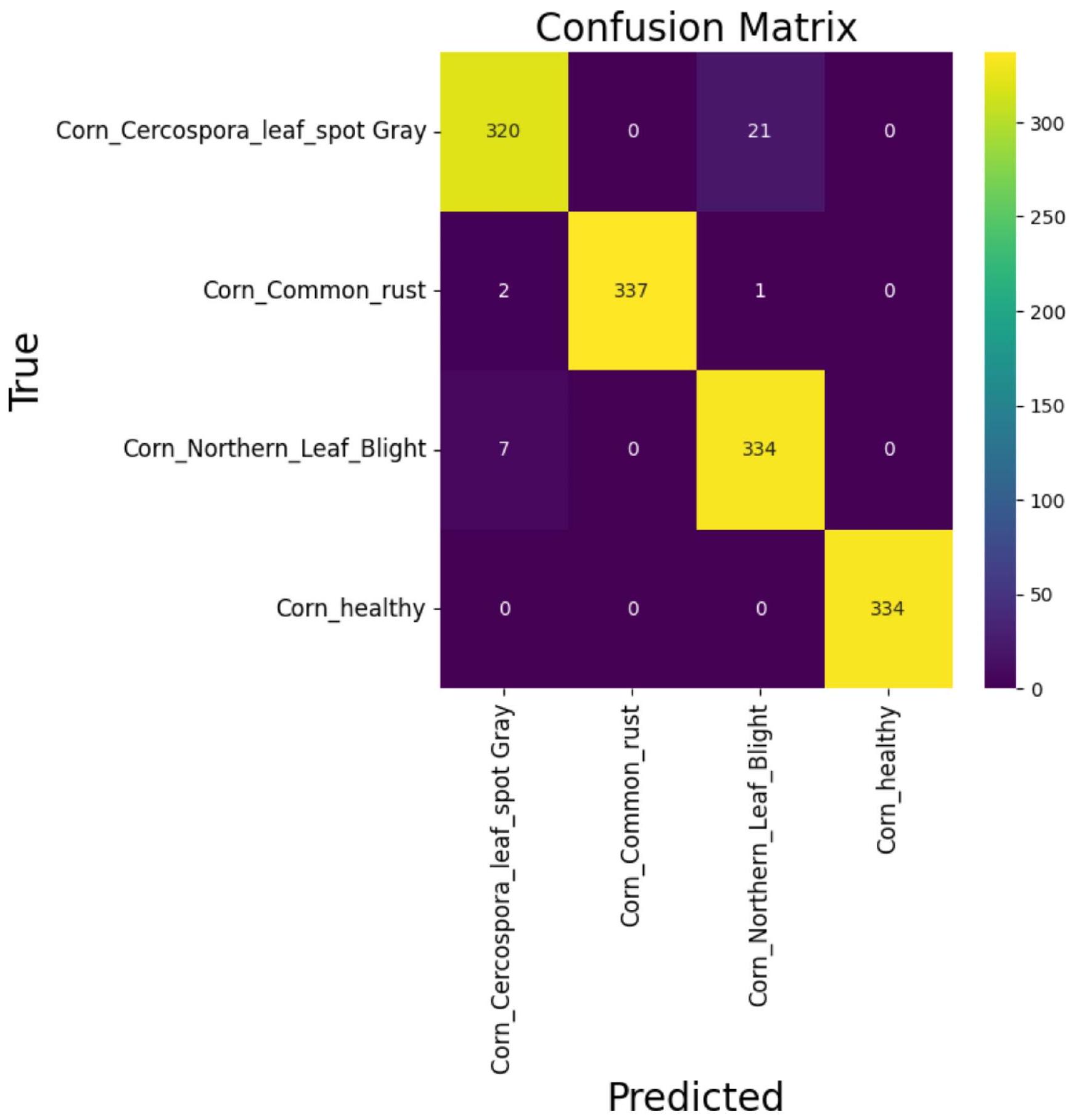
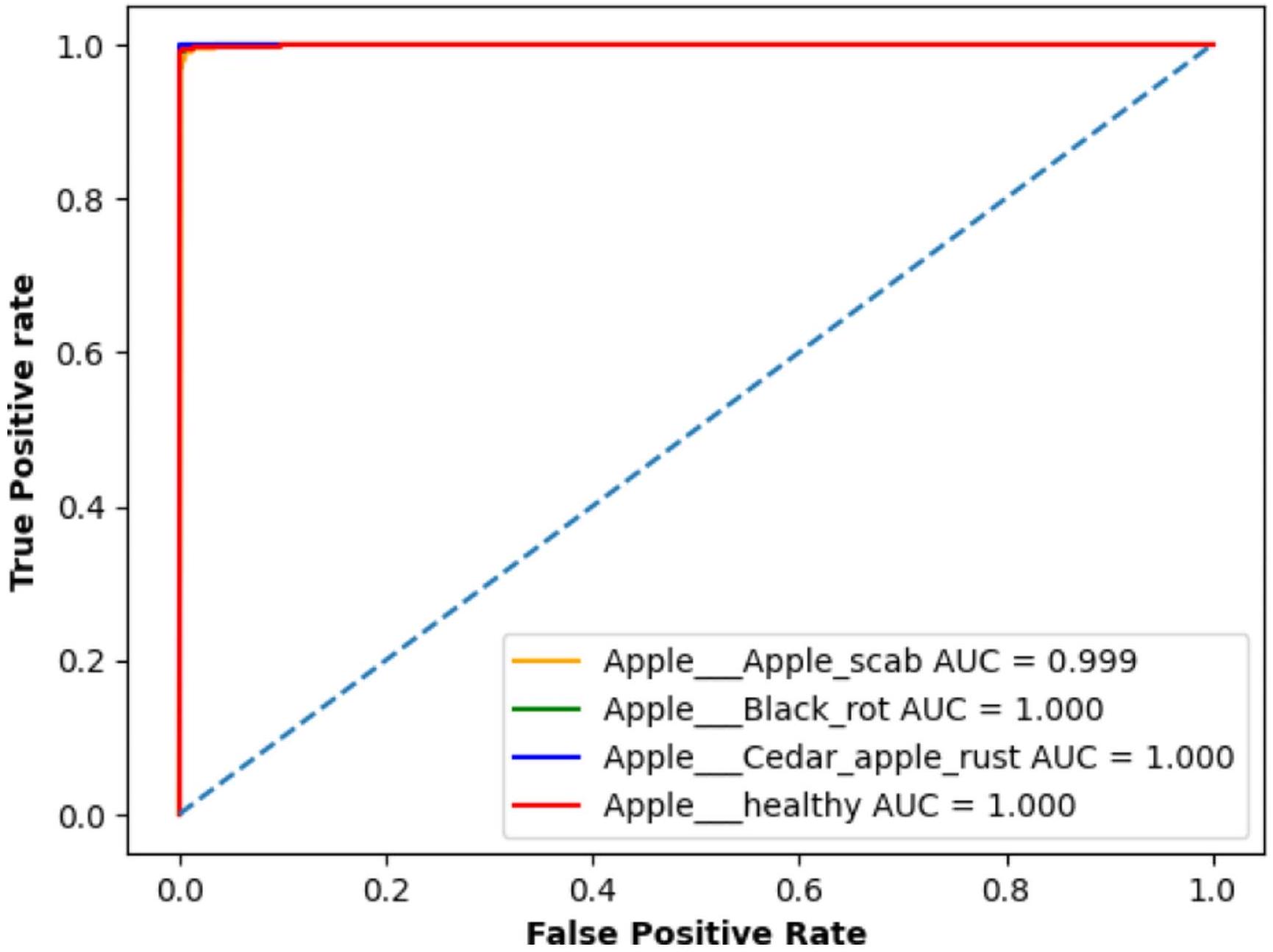
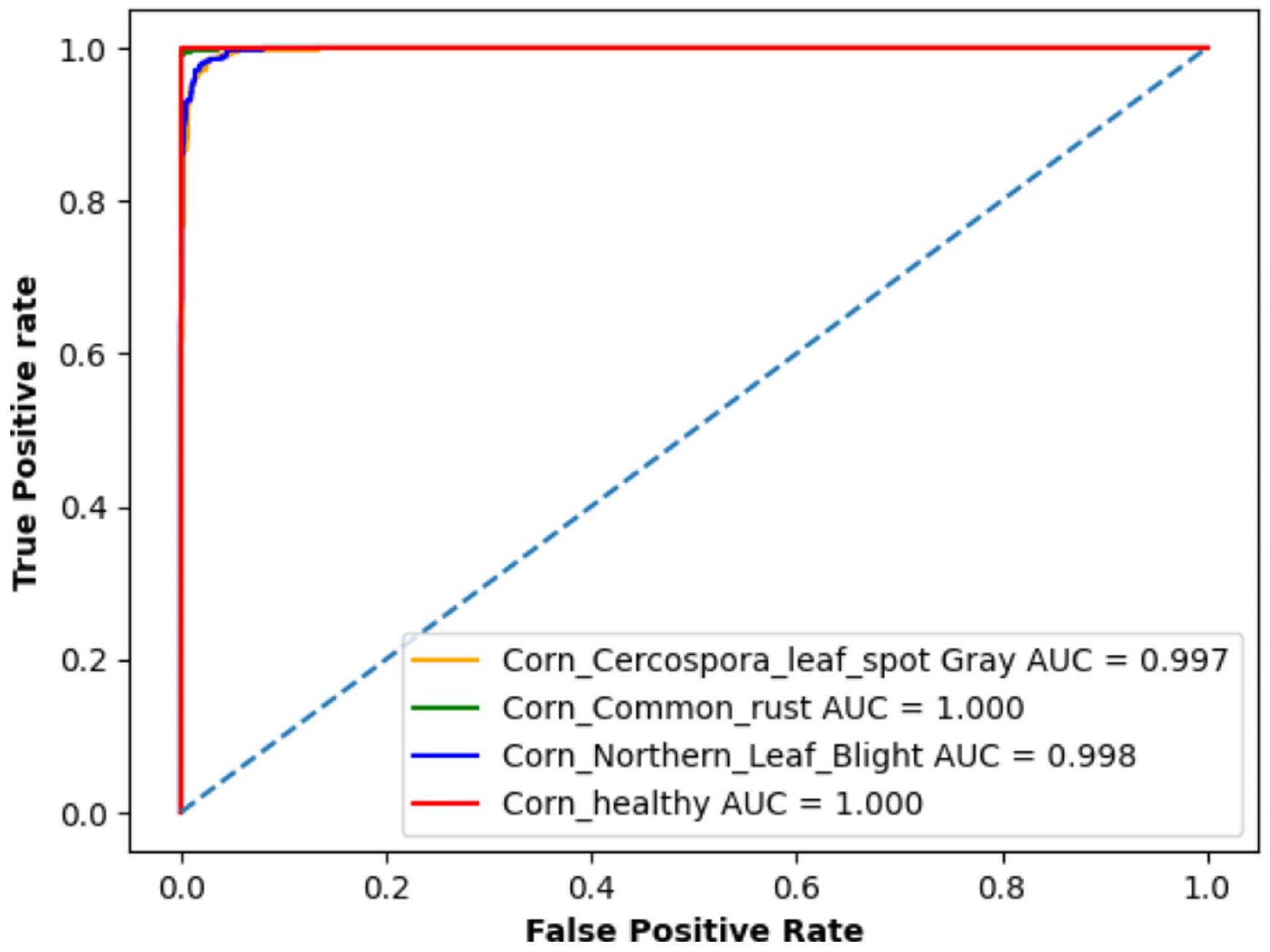
النتائج والمناقشة
درجة F1 من
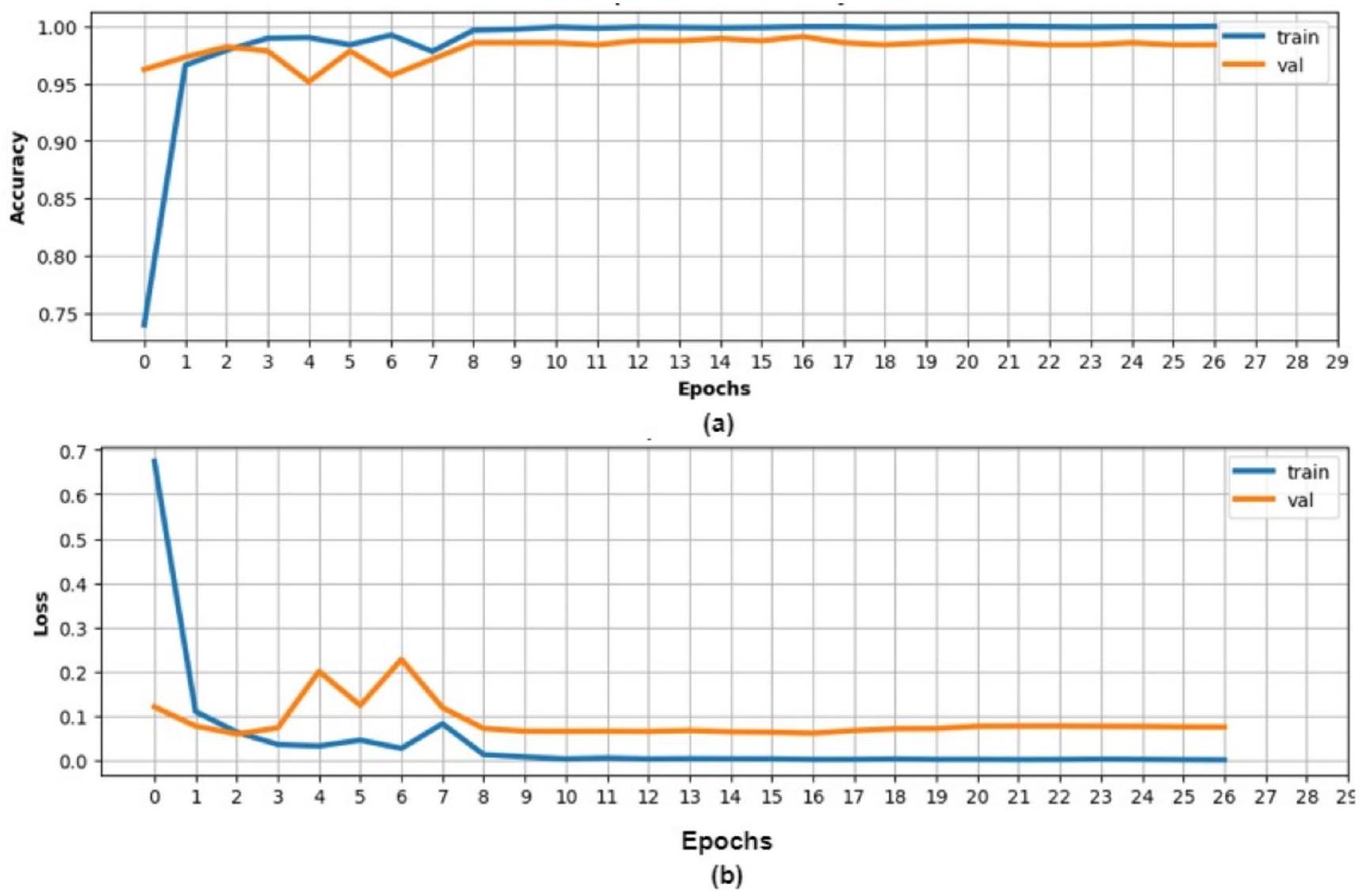
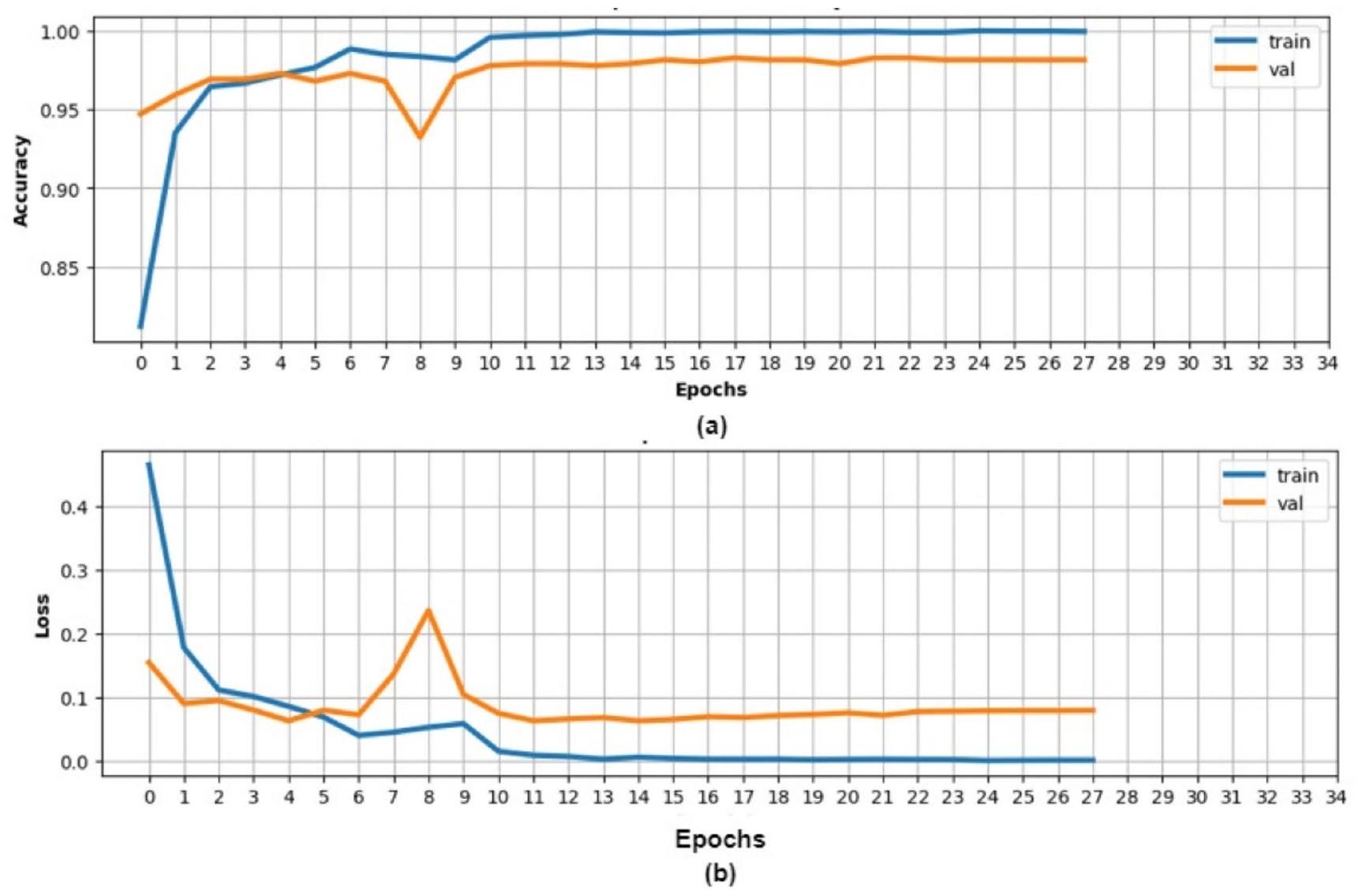
أن دقة التدريب والتحقق لمجموعة بيانات الذرة وصلت إلى
(VGG16 و Inception-V3 و DenseNet201) ودمج تقنية ViT.
الآثار الإدارية
تحدي ندرة البيانات. يمكن اعتماد هذا الإطار الهجين لإنشاء أنظمة تحديد أمراض النباتات بشكل آلي، مما يؤدي إلى تحسين إدارة المحاصيل وزيادة الإنتاجية مع التخفيف من تأثير أمراض النباتات على إنتاج الغذاء. علاوة على ذلك، فإن نجاح هذا الإطار يبرز الفرص للبحث والتطوير الإضافي، بما في ذلك إمكانيته للتوسع.
تنفيذ في الوقت الحقيقي، وقابلية التكيف مع محاصيل وأمراض أخرى.
الخاتمة
عدم الدقة في
| مجموعة بيانات | المؤلفون | نهج | الدقة (%) |
| تفاح | ثاكور وآخرون (2021) [48] | سي إن إن + في تي | 98.6 |
| لي وآخرون (2022) [49] | سي إن إن + في تي | 96.7 | |
| أرشاد وآخرون (2023) [44] | سي إن إن + في تي | ٩٦.٤٢ | |
| النموذج الهجين المقترح | مجموعة من نماذج التعلم العميق المدربة مسبقًا مع ViT | 99.24 | |
| ذرة | وحيد وآخرون (2020) [31] | نموذج DenseNet المحسن | ٩٨ |
| Qian وآخرون (2022) [35] | مبني على الانتباه والمحولات | 97.7 | |
| ميشرا وآخرون (2020) [50] | DCCN | ٨٨.٤٦ | |
| النموذج الهجين المقترح | مجموعة من نماذج التعلم العميق المدربة مسبقًا مع ViT | 98 |
الإعلانات
إذا كانت اللوائح أو الاستخدامات تتجاوز الاستخدام المسموح به، ستحتاج إلى الحصول على إذن مباشرة من صاحب حقوق الطبع والنشر. لعرض نسخة من هذه الرخصة، قم بزيارة http://creativecommons.org/licenses/by-nc-nd/4.0/.
References
- Savary S, Bregaglio S, Willocquet L, Gustafson D, Mason D’Croz D, Sparks A, Garrett K (2017) Crop health and its global impacts on the components of food security. Food Secur 9:311-327
- Borhani Y, Khoramdel J, Najafi E (2022) A deep learning-based approach for automated plant disease classification using vision transformer. Sci Rep 12(1):11554
- Shoaib M, Shah B, Ei-Sappagh S, Ali A, Ullah A, Alenezi F, Ali F (2023) An advanced deep learning models-based plant disease detection: a review of recent research. Front Plant Sci 14:1158933
- Hou C, Zhuang J, Tang Y, He Y, Miao A, Huang H, Luo S (2021) Recognition of early blight and late blight diseases on potato leaves based on graph cut segmentation. J Agric Food Res 5:100154
- Sun Y, Jiang Z, Zhang L, Dong W, Rao Y (2019) SLIC_SVM based leaf diseases saliency map extraction of tea plant. Comput Electron Agric 157:102-109
- Zhang S, Wang Z (2016) Cucumber disease recognition based on global-local singular value decomposition. Neurocomputing 205:341-348
- Hamdani H, Septiarini A, Sunyoto A, Suyanto S, Utaminingrum F (2021) Detection of oil palm leaf disease based on color histogram and supervised classifier. Optik 245:167753
- Ramesh S, Vydeki D (2020) Recognition and classification of paddy leaf diseases using optimized deep neural network with Jaya algorithm. Inform Process Agric 7(2):249-260
- Johannes A, Picon A, Alvarez-Gila A, Echazarra J, RodriguezVaamonde S, Navajas AD, Ortiz-Barredo A (2017) Automatic plant disease diagnosis using mobile capture devices, applied on a wheat use case. Comput Electron Agric 138:200-209
- Mohanty SP, Hughes DP, Salathé M (2016) Using deep learning for image-based plant disease detection. Front Plant Sci 7:1419
- Barbedo JGA (2018) Impact of dataset size and variety on the effectiveness of deep learning and transfer learning for plant disease classification. Comput Electron Agric 153:46-53
- Chen J, Chen J, Zhang D, Sun Y, Nanehkaran YA (2020) Using deep transfer learning for image-based plant disease identification. Comput Electron Agric 173:105393
- Thakur PS, Sheorey T, Ojha A (2023) VGG-ICNN: a lightweight CNN model for crop disease identification. Multimedia Tools Appl 82(1):497-520
- Shah SR, Qadri S, Bibi H, Shah SMW, Sharif MI, Marinello F (2023) Comparing inception V3, VGG 16, VGG 19, CNN, and ResNet 50: a case study on early detection of a Rice Disease. Agronomy 13(6): 1633
- Thakur PS, Chaturvedi S, Khanna P, Sheorey T, Ojha A (2023) Vision transformer meets convolutional neural network for plant disease classification. Ecol Inf 77:102245
- Li G, Wang Y, Zhao Q, Yuan P, Chang B (2023) PMVT: a lightweight vision transformer for plant disease identification on mobile devices. Front Plant Sci 14:1256773
- Yu S, Xie L, Huang Q (2023) Inception convolutional vision transformers for plant disease identification. Internet Things 21:100650
- De Silva M, Brown D (2023) Multispectral plant Disease Detection with Vision transformer-convolutional neural network hybrid approaches. Sensors 23(20):8531
- Boukabouya RA, Moussaoui A, Berrimi M (2022), November Vision Transformer Based Models for Plant Disease Detection
and Diagnosis. In 2022 5th International Symposium on Informatics and its Applications (ISIA) (pp. 1-6). IEEE - Jiang P, Chen Y, Liu B, He D, Liang C (2019) Real-time detection of apple leaf diseases using deep learning approach based on improved convolutional neural networks. IEEE Access 7:59069-59080
- Bukumira M, Antonijevic M, Jovanovic D, Zivkovic M, Mladenovic D, Kunjadic G (2022) Carrot grading system using computer vision feature parameters and a cascaded graph convolutional neural network. J Electron Imaging 31(6):061815-061815
- Fu L, Li S, Sun Y, Mu Y, Hu T, Gong H (2022) Lightweightconvolutional neural network for apple leaf disease identification. Front Plant Sci 13:831219
- Bansal P, Kumar R, Kumar S (2021) Disease detection in apple leaves using deep convolutional neural network. Agriculture 11(7):617
- Khan AI, Quadri SMK, Banday S, Shah JL (2022) Deep diagnosis: a real-time apple leaf disease detection system based on deep learning. Comput Electron Agric 198:107093
- Zhang C, Wang J, Yan T et al (2023) An instance-based deep transfer learning method for quality identification of Longjing tea from multiple geographical origins. Complex Intell Syst 9:3409-3428
- Albattah W, Nawaz M, Javed A et al (2022) A novel deep learning method for detection and classification of plant diseases. Complex Intell Syst 8:507-524
- Taji K, Sohail A, Shahzad T, Khan BS, Khan MA, Ouahada K (2024) An Ensemble Hybrid Framework: a comparative analysis of Metaheuristic algorithms for Ensemble Hybrid CNN features for plants Disease classification. IEEE Access 12:61886-61906
- Mohamed M (2023) Agricultural sustainability in the age of Deep Learning: current trends, challenges, and future trajectories. Sustainable Mach Intell J 4:2-1
- Hosny KM, El-Hady WM, Samy FM, Vrochidou E, Papakostas GA (2023) Multi-class classification of Plant Leaf diseases using Feature Fusion of deep convolutional neural network and local binary pattern. IEEE Access 11:62307-62317
- Ahila Priyadharshini R, Arivazhagan S, Arun M, Mirnalini A (2019) Maize leaf disease classification using deep convolutional neural networks. Neural Comput Appl 31:8887-8895
- Waheed A, Goyal M, Gupta D, Khanna A, Hassanien AE, Pandey HM (2020) An optimized dense convolutional neural network model for disease recognition and classification in corn leaf. Comput Electron Agric 175:105456
- Wu H, Wiesner-Hanks T, Stewart EL, DeChant C, Kaczmar N, Gore MA, Lipson H (2019) Autonomous detection of plant disease symptoms directly from aerial imagery. Plant Phenome J 2(1):1-9
- Zeng W, Li M (2020) Crop leaf disease recognition based on selfattention convolutional neural network. Comput Electron Agric 172:105341
- Chen J, Zhang D, Zeb A, Nanehkaran YA (2021) Identification of rice plant diseases using lightweight attention networks. Expert Syst Appl 169:114514
- Qian X, Zhang C, Chen L, Li K (2022) Deep learning-based identification of maize leaf diseases is improved by an attention mechanism: self-attention. Front Plant Sci 13:864486
- Reedha R, Dericquebourg E, Canals R, Hafiane A (2022) Transformer neural network for weed and crop classification of highresolution UAV images. Remote Sens 14(3):592
- Wu S, Sun Y, Huang H (2021), December Multi-granularity feature extraction based on vision transformer for tomato leaf disease recognition. In 2021 3rd International Academic Exchange Conference on Science and Technology Innovation (IAECST) (pp. 387-390). IEEE
- Dosovitskiy, A., Beyer, L., Kolesnikov, A., Weissenborn, D., Zhai, X., Unterthiner,T., … Houlsby, N. (2020). An image is worth 16×16 words: Transformers for image recognition at scale. arXiv preprint arXiv:2010.11929
- Ukwuoma CC, Qin Z, Heyat MBB, Akhtar F, Bamisile O, Muaad AY, Al-Antari MA (2023) A hybrid explainable ensemble transformer encoder for pneumonia identification from chest X-ray images. J Adv Res 48:191-211
- Simonyan K, Zisserman A (2014) Very deep convolutional networks for large-scale image recognition. arXiv preprint arXiv:1409.1556
- Szegedy, C., Liu, W., Jia, Y., Sermanet, P., Reed, S., Anguelov, D., … Rabinovich,A. (2015). Going deeper with convolutions. In Proceedings of the IEEE conference on computer vision and pattern recognition (pp. 1-9)
- He K, Zhang X, Ren S, Sun J (2016) Deep residual learning for image recognition. In Proceedings of the IEEE conference on computer vision and pattern recognition (pp. 770-778)
- Al-Hejri AM, Al-Tam RM, Fazea M, Sable AH, Lee S, Al-Antari MA (2022) ETECADx: Ensemble self-attention transformer encoder for breast cancer diagnosis using full-field digital X-ray breast images. Diagnostics 13(1):89
- Arshad F, Mateen M, Hayat S, Wardah M, Al-Huda Z, Gu YH, Al-antari MA (2023) PLDPNet: end-to-end hybrid deep learning framework for potato leaf disease prediction. Alexandria Eng J 78:406-418
- Al-Tam RM, Al-Hejri AM, Narangale SM, Samee NA, Mahmoud NF, Al-Masni MA, Al-Antari MA (2022) A hybrid workflow of residual convolutional transformer encoder for breast cancer classification using digital X-ray mammograms. Biomedicines 10(11):2971
- Oluwafemi ET (2019) PlantVillage Dataset. Accessed: Dec. 30, 2023. [Online]. Available: https://www.kaggle.com/datasets/em marex/plantdisease
- Stojanović M, Apostolović M, Stojanović D, Milošević Z, Toplaović A, Mitić-Lakušić V, Golubović M (2014) Understanding sensitivity, specificity, and predictive values. Vojnosanit Pregl 71(11):1062-1065
- Thakur PS, Khanna P, Sheorey T, Ojha A (2021), December Vision Transformer for Plant Disease Detection: PlantViT. In International Conference on Computer Vision and Image Processing (pp. 501-511). Cham: Springer International Publishing
- Li X, Li S (2022) Transformer help CNN see better: a lightweight hybrid apple disease identification model based on transformers. Agriculture 12(6):884
- Mishra S, Sachan R, Rajpal D (2020) Deep convolutional neural network-based detection system for real-time corn plant disease recognition. Procedia Comput Sci 167:2003-2010
DOI: https://doi.org/10.1007/s40747-024-01764-x
Publication Date: 2025-01-15
A hybrid Framework for plant leaf disease detection and classification using convolutional neural networks and vision transformer
© The Author(s) 2024
Abstract
Recently, scientists have widely utilized Artificial Intelligence (AI) approaches in intelligent agriculture to increase the productivity of the agriculture sector and overcome a wide range of problems. Detection and classification of plant diseases is a challenging problem due to the vast numbers of plants worldwide and the numerous diseases that negatively affect the production of different crops. Early detection and accurate classification of plant diseases is the goal of any AI-based system. This paper proposes a hybrid framework to improve classification accuracy for plant leaf diseases significantly. This proposed model leverages the strength of Convolutional Neural Networks (CNNs) and Vision Transformers (ViT), where an ensemble model, which consists of the well-known CNN architectures VGG16, Inception-V3, and DenseNet20, is used to extract robust global features. Then, a ViT model is used to extract local features to detect plant diseases precisely. The performance proposed model is evaluated using two publicly available datasets (Apple and Corn). Each dataset consists of four classes. The proposed hybrid model successfully detects and classifies multi-class plant leaf diseases and outperforms similar recently published methods, where the proposed hybrid model achieved an accuracy rate of
Abbreviations
CNNs Convolutional Neural Networks
ViT Vision Transformer
ML Machine Learning
DL Deep Learning
| SVM | Support Vector Machine |
| ANNs | Artificial Neural Networks |
| NB | Naive Bayes |
| MLP | Multilayer Perception |
| MHA | Multi-Head self-Attention |
| GP | Global Pooling |
| FFN | Feed Forward Network |
| NL | Normalization Layer |
| LR | Learning Rate |
| AUC | Area Under Curve |
| ROC | Receiver Operating Characteristic |
| CM | Confusion Matrix |
Introduction
effectively with policymakers. Additionally, global changes such as climate change impact the types of disease threats faced, their potential outcomes, and the approaches for addressing them [1]. Farmers may struggle to diagnose plant diseases accurately due to their tiny features. Furthermore, many farmers lack the knowledge to diagnose diseases, so artificial intelligence can help them diagnose diseases more accurately [2].
the numerous disease species and diverse crops involved. The similarity in symptoms between different diseases and the changing patterns of these diseases over time further compounds the complexity. The limitation is that traditional approaches have struggled to extract relevant features from input images. Consequently, researchers prefer deep learning models, especially CNNs, which can automatically extract relevant and informative features from the input data, leading to superior performance. However, CNN models are limited in their ability to analyze the relationships between distant pixels, as they only consider the correlation between spatially neighboring pixels within the receptive field defined by the filter size. Furthermore, while effective, existing CNN models often require large datasets and substantial computational resources, presenting challenges for real-time applications in resource-constrained environments. Researchers have recently explored the use of attention mechanisms to address the challenges associated with the CNN model’s limited ability to capture relationships between distant pixels. Additionally, most existing studies have focused on individual CNN models, with a limited investigation into the potential benefits of ensemble modeling approaches for improved generalization of results. Consequently, we have created a hybrid framework that combines multiple DL architectures and vision transformers to extract profound features through a hybrid approach, as shown in Fig. 1. The key points of this paper can be stated as follows:
- A hybrid framework, including deep-learning CNNs and vision transformers, is suggested for detecting and classifying plant leaf diseases.
- The suggested end-to-end framework captures the most distinct features, allowing for precise detection and multi-class classification of plant leaf diseases.
- The suggested model has been extensively experimented with two different types of plant leaf disease datasets (Corn and Apple) and compared to the cutting-edge models using the same datasets.

Related works
by leveraging transformers and self-attention mechanisms. The model partitions visual data into small, localized segments, computes correlations between these regions using an attention mechanism, and then aggregates larger, global information to inform the classification task [35]. Reedha et al. [36] demonstrated the effectiveness of the convolu-tional-free Vision Transformer model, which leverages the self-attention mechanism to process an image as a sequence of patches through a standard transformer encoder. Despite using a relatively small dataset, they achieved high performance, which they attributed to techniques such as data augmentation, transfer learning, and a limited number of classes [36]. Wu et al. [37] employed a dual-scale approach by passing the dataset through two ViT models simultaneously, one with a small patch size and the other with a large patch size. The outputs of these two ViT models were then combined using a fusion model and fed into a multilayer perceptron header. The researchers concluded that by integrating different scales of self-attention sequences, the model could extract richer information from the images at various levels of granularity [37]. The analysis of current methods utilized to identify plant diseases is summarized in Table 1. As shown in Table 1, the classification performance indicates a need for further improvements.
Materials and methods
The proposed architecture
| Reference | Objective | Method | Key Findings | Limitations | ||
| Jiang et al. (2019) [20] | Real-time detection of apple leaf diseases | Improved CNN | – provide a high-performance solution with realtime detection |
|
||
| Bukumira et al. (2022) [21] | Carrot grading based on computer vision and Graph Convolutional Neural Network (GCNN) | Cascaded Graph CNN with Bayesian optimization | – Effective in automated grading of carrots |
|
||
| Fu et al. (2022) [22] | Lightweight model for apple leaf disease detection | Lightweight-CNN | – High accuracy with reduced model complexity | – Focused on apple leaves; the model might lack robustness for other crops. | ||
| Bansal et al. (2021) [23] | Apple leaf disease detection | ensemble of pretrained deep CNN models | -Improved classification accuracy for apple diseases | – identification accuracy degrades for multiple diseases | ||
| Khan et al. (2022) [24] | Real-time apple leaf disease detection system | Lightweight-CNN and Yolov4 | – Lightweight model with Real-time performance | – Classification performance needs improvement | ||
| Taji et al. (2024) [27] | Plant disease classification with hybrid CNN features | Metaheuristicbased hybrid CNN framework | – Enhanced performance with metaheuristic optimization | – Metaheuristic tuning is complex, which may lead to high computational costs and time-consuming parameter optimization. | ||
| Hosny et al. (2023) [29] | Multi-class plant leaf disease classification | Feature fusion of CNN and Local Binary Pattern | -Enhanced multi-class classification accuracy | – Complexity in feature fusion may impede model interpretability and adaptability. | ||
| Priyadharshini et al. (2019) [30] | Classification of maize leaf diseases | modified LeNet | -Lightweight model with Real-time performance | – Only evaluated for maize leaf disease classification | ||
| Waheed et al. (2020) [31] | corn leaf disease recognition and classification | – Optimized DenseNet model | – Improved classification efficiency and accuracy | – The dense architectural structure may elevate memory consumption, potentially impacting the deployment on devices with limited resources. | ||
| Wu et al. (2019) [32] | Autonomous detection of plant disease | CNN model on aerial imagery | – Effective in large-scale disease detection | – Limited to aerial imagery, which may miss finer disease symptoms. | ||
| Zeng & Li (2020) [33] | Crop leaf disease recognition | CNN model with Self-Attention | – Improved accuracy with self-attention mechanism | – Limited evaluations on crop datasets. | ||
| Chen et al. (2021) [34] | Rice plant disease detection | Pre-trained MobileNet-V2 with Attention Mechanism | – Efficient and accurate for rice diseases | – Lightweight structure may sacrifice detail on diverse or high-resolution datasets. | ||
| Qian et al. (2022) [35] | Maize leaf disease identification | Attention mecha-nism-based CNN | – Enhanced accuracy with self-attention | – High computational cost due to attention layers may limit real-time application. | ||
| Reedha et al. (2022) [36] | Weed and crop classification | attention-based deep network | – high performance, especially with small training datasets | – High-resolution image processing is computationally demanding, which may hinder real-time use. | ||
| Wu et al. (2021) [37] | Tomato leaf disease recognition | ViT | – multi-granularity feature extraction model | – High computational complexity of Vision Transformers may limit real-time use |
and global features, thereby enhancing the interpretability of the model.
1.1. Pre-trained deep learning architectures
1.1.1.VGG16
Pre-trained Models
(Deep Features Extraction)

1.1.2. GoogleNet
a local area. Subsequently, these diverse features are gathered and combined at a specific level before being transmitted to the next layer.
1.1.3. DenseNet201
1.2. Vision Transformer (ViT)

1.3. Extraction and fusion of deep features
| Class Index | Class Name | Training set size (80%) | Testing set size (20%) | Total |
| 0 | Apple_scab | 800 | 200 | 1,000 |
| 1 | Black_rot | 800 | 200 | 1,000 |
| 2 | Cedar_apple_rust | 800 | 200 | 1,000 |
| 3 | Healthy | 1,316 | 329 | 1,645 |
| Total | 3,716 | 929 | 4,645 |
| Class Index | Class Name | Training set size (80%) | Testing set size (20%) | Total |
| 0 | Gray_leaf_spot | 1,363 | 341 | 1,704 |
| 1 | Common_rust | 1,358 | 340 | 1,698 |
| 2 | Northern_leaf_blight | 1,363 | 341 | 1,704 |
| 3 | Healthy | 1,334 | 334 | 1,668 |
| Total | 5,418 | 1,356 | 6,774 |
involved zero-padding the GoogleNet architecture before concatenating the features.
1.4. Datasets
1.5. Experiments setup

| Function | Parameter | Value |
| Training Parameters | Optimizer | Adam |
| Learning Rate | 0.0001 | |
| Epochs | 50 | |
| Batch Size | 32 | |
| ViT Parameters | Batch Size | 2 |
| Embedded Dimension | 64 | |
| Number of Attention Heads | 8 | |
| Number of MLP Layers | 256 |
1.6. Model evaluation
| DL Model | Evaluation metrics | |||
| Accuracy (%) | Precision (%) | Recall (%) | F1-score (%) | |
| Apple | 96 | 96 | 96 | 96 |
| VGG16 | ||||
| Inception-v3 | 94 | 94 | 94 | 94 |
| DenseNet201 | 97 | 97 | 97 | 97 |
| VGG1VGG16 + InceptionV3 + DenseNet201 | 97.6 | 98 | 98 | 98 |
| Proposed Hybrid Model | 99.24 | 99 | 99 | 99 |
| Corn | 96 | 96 | 96 | 96 |
| VGG16 | ||||
| Inception-v3 | 93.87 | 94 | 94 | 94 |
| DenseNet201 | 97 | 97 | 97 | 97 |
| VGG16 + InceptionV3 + DenseNet201 | 97 | 97 | 97 | 97 |
| Proposed Hybrid Model | 98 | 98 | 98 | 98 |
Results and discussion
an F1-score of
that the training and validation accuracy for the corn dataset reached


(VGG16, Inception-V3, and DenseNet201) and the incorporation of ViT technology.

Managerial implications

the challenge of data scarcity. This hybrid framework can be adopted to create automated plant disease identification systems, leading to improved crop management and yield optimization while mitigating the impact of plant diseases on food production. Furthermore, the success of this framework underscores opportunities for additional research and development, including its potential for scalability,

real-time implementation, and adaptability to other crops and diseases.
Conclusion
accuracies of

| Dataset | Authors | Approach | Accuracy (%) |
| Apple | Thakur et al. (2021) [48] | CNN+ViT | 98.6 |
| Li et al. (2022) [49] | CNN+ViT | 96.7 | |
| Arshad et al. (2023) [44] | CNN+ViT | 96.42 | |
| The proposed hybrid model | An ensemble of pre-trained DL models with ViT | 99.24 | |
| Corn | Waheed et al. (2020) [31] | optimized DenseNet model | 98 |
| Qian et al. (2022) [35] | Attention-based and transformer | 97.7 | |
| Mishra et al. (2020) [50] | DCCN | 88.46 | |
| The proposed hybrid model | An ensemble of pre-trained DL models with ViT | 98 |
Declarations
regulation or exceeds the permitted use, you will need to obtain permission directly from the copyright holder. To view a copy of this licence, visit http://creativecommons.org/licenses/by-nc-nd/4.0/.
References
- Savary S, Bregaglio S, Willocquet L, Gustafson D, Mason D’Croz D, Sparks A, Garrett K (2017) Crop health and its global impacts on the components of food security. Food Secur 9:311-327
- Borhani Y, Khoramdel J, Najafi E (2022) A deep learning-based approach for automated plant disease classification using vision transformer. Sci Rep 12(1):11554
- Shoaib M, Shah B, Ei-Sappagh S, Ali A, Ullah A, Alenezi F, Ali F (2023) An advanced deep learning models-based plant disease detection: a review of recent research. Front Plant Sci 14:1158933
- Hou C, Zhuang J, Tang Y, He Y, Miao A, Huang H, Luo S (2021) Recognition of early blight and late blight diseases on potato leaves based on graph cut segmentation. J Agric Food Res 5:100154
- Sun Y, Jiang Z, Zhang L, Dong W, Rao Y (2019) SLIC_SVM based leaf diseases saliency map extraction of tea plant. Comput Electron Agric 157:102-109
- Zhang S, Wang Z (2016) Cucumber disease recognition based on global-local singular value decomposition. Neurocomputing 205:341-348
- Hamdani H, Septiarini A, Sunyoto A, Suyanto S, Utaminingrum F (2021) Detection of oil palm leaf disease based on color histogram and supervised classifier. Optik 245:167753
- Ramesh S, Vydeki D (2020) Recognition and classification of paddy leaf diseases using optimized deep neural network with Jaya algorithm. Inform Process Agric 7(2):249-260
- Johannes A, Picon A, Alvarez-Gila A, Echazarra J, RodriguezVaamonde S, Navajas AD, Ortiz-Barredo A (2017) Automatic plant disease diagnosis using mobile capture devices, applied on a wheat use case. Comput Electron Agric 138:200-209
- Mohanty SP, Hughes DP, Salathé M (2016) Using deep learning for image-based plant disease detection. Front Plant Sci 7:1419
- Barbedo JGA (2018) Impact of dataset size and variety on the effectiveness of deep learning and transfer learning for plant disease classification. Comput Electron Agric 153:46-53
- Chen J, Chen J, Zhang D, Sun Y, Nanehkaran YA (2020) Using deep transfer learning for image-based plant disease identification. Comput Electron Agric 173:105393
- Thakur PS, Sheorey T, Ojha A (2023) VGG-ICNN: a lightweight CNN model for crop disease identification. Multimedia Tools Appl 82(1):497-520
- Shah SR, Qadri S, Bibi H, Shah SMW, Sharif MI, Marinello F (2023) Comparing inception V3, VGG 16, VGG 19, CNN, and ResNet 50: a case study on early detection of a Rice Disease. Agronomy 13(6): 1633
- Thakur PS, Chaturvedi S, Khanna P, Sheorey T, Ojha A (2023) Vision transformer meets convolutional neural network for plant disease classification. Ecol Inf 77:102245
- Li G, Wang Y, Zhao Q, Yuan P, Chang B (2023) PMVT: a lightweight vision transformer for plant disease identification on mobile devices. Front Plant Sci 14:1256773
- Yu S, Xie L, Huang Q (2023) Inception convolutional vision transformers for plant disease identification. Internet Things 21:100650
- De Silva M, Brown D (2023) Multispectral plant Disease Detection with Vision transformer-convolutional neural network hybrid approaches. Sensors 23(20):8531
- Boukabouya RA, Moussaoui A, Berrimi M (2022), November Vision Transformer Based Models for Plant Disease Detection
and Diagnosis. In 2022 5th International Symposium on Informatics and its Applications (ISIA) (pp. 1-6). IEEE - Jiang P, Chen Y, Liu B, He D, Liang C (2019) Real-time detection of apple leaf diseases using deep learning approach based on improved convolutional neural networks. IEEE Access 7:59069-59080
- Bukumira M, Antonijevic M, Jovanovic D, Zivkovic M, Mladenovic D, Kunjadic G (2022) Carrot grading system using computer vision feature parameters and a cascaded graph convolutional neural network. J Electron Imaging 31(6):061815-061815
- Fu L, Li S, Sun Y, Mu Y, Hu T, Gong H (2022) Lightweightconvolutional neural network for apple leaf disease identification. Front Plant Sci 13:831219
- Bansal P, Kumar R, Kumar S (2021) Disease detection in apple leaves using deep convolutional neural network. Agriculture 11(7):617
- Khan AI, Quadri SMK, Banday S, Shah JL (2022) Deep diagnosis: a real-time apple leaf disease detection system based on deep learning. Comput Electron Agric 198:107093
- Zhang C, Wang J, Yan T et al (2023) An instance-based deep transfer learning method for quality identification of Longjing tea from multiple geographical origins. Complex Intell Syst 9:3409-3428
- Albattah W, Nawaz M, Javed A et al (2022) A novel deep learning method for detection and classification of plant diseases. Complex Intell Syst 8:507-524
- Taji K, Sohail A, Shahzad T, Khan BS, Khan MA, Ouahada K (2024) An Ensemble Hybrid Framework: a comparative analysis of Metaheuristic algorithms for Ensemble Hybrid CNN features for plants Disease classification. IEEE Access 12:61886-61906
- Mohamed M (2023) Agricultural sustainability in the age of Deep Learning: current trends, challenges, and future trajectories. Sustainable Mach Intell J 4:2-1
- Hosny KM, El-Hady WM, Samy FM, Vrochidou E, Papakostas GA (2023) Multi-class classification of Plant Leaf diseases using Feature Fusion of deep convolutional neural network and local binary pattern. IEEE Access 11:62307-62317
- Ahila Priyadharshini R, Arivazhagan S, Arun M, Mirnalini A (2019) Maize leaf disease classification using deep convolutional neural networks. Neural Comput Appl 31:8887-8895
- Waheed A, Goyal M, Gupta D, Khanna A, Hassanien AE, Pandey HM (2020) An optimized dense convolutional neural network model for disease recognition and classification in corn leaf. Comput Electron Agric 175:105456
- Wu H, Wiesner-Hanks T, Stewart EL, DeChant C, Kaczmar N, Gore MA, Lipson H (2019) Autonomous detection of plant disease symptoms directly from aerial imagery. Plant Phenome J 2(1):1-9
- Zeng W, Li M (2020) Crop leaf disease recognition based on selfattention convolutional neural network. Comput Electron Agric 172:105341
- Chen J, Zhang D, Zeb A, Nanehkaran YA (2021) Identification of rice plant diseases using lightweight attention networks. Expert Syst Appl 169:114514
- Qian X, Zhang C, Chen L, Li K (2022) Deep learning-based identification of maize leaf diseases is improved by an attention mechanism: self-attention. Front Plant Sci 13:864486
- Reedha R, Dericquebourg E, Canals R, Hafiane A (2022) Transformer neural network for weed and crop classification of highresolution UAV images. Remote Sens 14(3):592
- Wu S, Sun Y, Huang H (2021), December Multi-granularity feature extraction based on vision transformer for tomato leaf disease recognition. In 2021 3rd International Academic Exchange Conference on Science and Technology Innovation (IAECST) (pp. 387-390). IEEE
- Dosovitskiy, A., Beyer, L., Kolesnikov, A., Weissenborn, D., Zhai, X., Unterthiner,T., … Houlsby, N. (2020). An image is worth 16×16 words: Transformers for image recognition at scale. arXiv preprint arXiv:2010.11929
- Ukwuoma CC, Qin Z, Heyat MBB, Akhtar F, Bamisile O, Muaad AY, Al-Antari MA (2023) A hybrid explainable ensemble transformer encoder for pneumonia identification from chest X-ray images. J Adv Res 48:191-211
- Simonyan K, Zisserman A (2014) Very deep convolutional networks for large-scale image recognition. arXiv preprint arXiv:1409.1556
- Szegedy, C., Liu, W., Jia, Y., Sermanet, P., Reed, S., Anguelov, D., … Rabinovich,A. (2015). Going deeper with convolutions. In Proceedings of the IEEE conference on computer vision and pattern recognition (pp. 1-9)
- He K, Zhang X, Ren S, Sun J (2016) Deep residual learning for image recognition. In Proceedings of the IEEE conference on computer vision and pattern recognition (pp. 770-778)
- Al-Hejri AM, Al-Tam RM, Fazea M, Sable AH, Lee S, Al-Antari MA (2022) ETECADx: Ensemble self-attention transformer encoder for breast cancer diagnosis using full-field digital X-ray breast images. Diagnostics 13(1):89
- Arshad F, Mateen M, Hayat S, Wardah M, Al-Huda Z, Gu YH, Al-antari MA (2023) PLDPNet: end-to-end hybrid deep learning framework for potato leaf disease prediction. Alexandria Eng J 78:406-418
- Al-Tam RM, Al-Hejri AM, Narangale SM, Samee NA, Mahmoud NF, Al-Masni MA, Al-Antari MA (2022) A hybrid workflow of residual convolutional transformer encoder for breast cancer classification using digital X-ray mammograms. Biomedicines 10(11):2971
- Oluwafemi ET (2019) PlantVillage Dataset. Accessed: Dec. 30, 2023. [Online]. Available: https://www.kaggle.com/datasets/em marex/plantdisease
- Stojanović M, Apostolović M, Stojanović D, Milošević Z, Toplaović A, Mitić-Lakušić V, Golubović M (2014) Understanding sensitivity, specificity, and predictive values. Vojnosanit Pregl 71(11):1062-1065
- Thakur PS, Khanna P, Sheorey T, Ojha A (2021), December Vision Transformer for Plant Disease Detection: PlantViT. In International Conference on Computer Vision and Image Processing (pp. 501-511). Cham: Springer International Publishing
- Li X, Li S (2022) Transformer help CNN see better: a lightweight hybrid apple disease identification model based on transformers. Agriculture 12(6):884
- Mishra S, Sachan R, Rajpal D (2020) Deep convolutional neural network-based detection system for real-time corn plant disease recognition. Procedia Comput Sci 167:2003-2010
