DOI: https://doi.org/10.1038/s41586-025-09263-w
PMID: https://pubmed.ncbi.nlm.nih.gov/40670786
تاريخ النشر: 2025-07-16
الإيميدازول بروبيونات هو محرك وهدف علاجي في تصلب الشرايين
تم الاستلام: 1 يونيو 2023
تم القبول: 9 يونيو 2025
تم النشر على الإنترنت: 16 يوليو 2025
الوصول المفتوح
Annalaura Mastrangelo ${ }^{mathbf{1 , 3 0}}$, Iñaki Robles-Vera ${ }^{mathbf{1 , 3 0}}$, Diego Mañanes ${ }^{mathbf{1 , 2}}$, Miguel Galán ${ }^{mathbf{1 , 2}}$, Marcos Femenía-Muiña ${ }^{1,3}$, Ana Redondo-Urzainqui ${ }^{1,2}$, Rafael Barrero-Rodríguez ${ }^{1,2}$, Eleftheria Papaioannou ${ }^{4,5,6}$, Joaquín Amores-Iniesta ${ }^{1}$, Ana Devesa ${ }^{1,7,8}$, Manuel Lobo-González ${ }^{1}$, Alba Carreras ${ }^{9}$, Katharina R. Beck ${ }^{9}$, Sophie Ivarsson ${ }^{9}$, Anders Gummesson ${ }^{9,10}$, Georgios Georgiopoulos ${ }^{11,12,13}$, Manuel Rodrigo-Tapias ${ }^{1,14}$, Sarai Martínez-Cano ${ }^{mathbf{1 , 1 4}}$, Ivan Fernández-López ${ }^{mathbf{1}}$, Vanessa Nuñez ${ }^{mathbf{1}}$, Alessia Ferrarini ${ }^{mathbf{1}}$, Naohiro Inohara ${ }^{15}$, Kimon Stamatelopoulos ${ }^{11}$, Alberto Benguría ${ }^{1}$, Danay Cibrian ${ }^{1,8,16}$, Francisco Sánchez-Madrid ${ }^{1,8,16,17}$, Vanesa Alonso-Herranz ${ }^{18}$, Ana Dopazo ${ }^{1,8}$, Coral Barbas ${ }^{19}$, Jesús Vázquez ${ }^{1,8}$, Juan Antonio López ${ }^{1,8}$, Alicia González-Martín ${ }^{4,5,6}$, Gabriel Nuñez ${ }^{15,19,20}$, Konstantinos Stellos ${ }^{21,22,23,24}$, Göran Bergström ${ }^{9,25}$, Fredrik Bäckhed ${ }^{9,25,26,27}$, Valentín Fuster ${ }^{1,28}$, Borja Ibañez ${ }^{1,8,29}$ & David Sancho ${ }^{1 boxtimes text { ® }}$
الملخص
تصلب الشرايين هو السبب الرئيسي الكامن وراء الأمراض القلبية الوعائية. تعتمد الوقاية منه على الكشف عن عوامل الخطر القلبية الوعائية التقليدية وعلاجها
بالمراحل المبكرة من مرض الأوعية الدموية التصلبية التي يمكن أن تساهم في تقدم تصلب الشرايين.
التحليل الأيضي غير المستهدف يربط ImP بتصلب الشرايين في الفئران
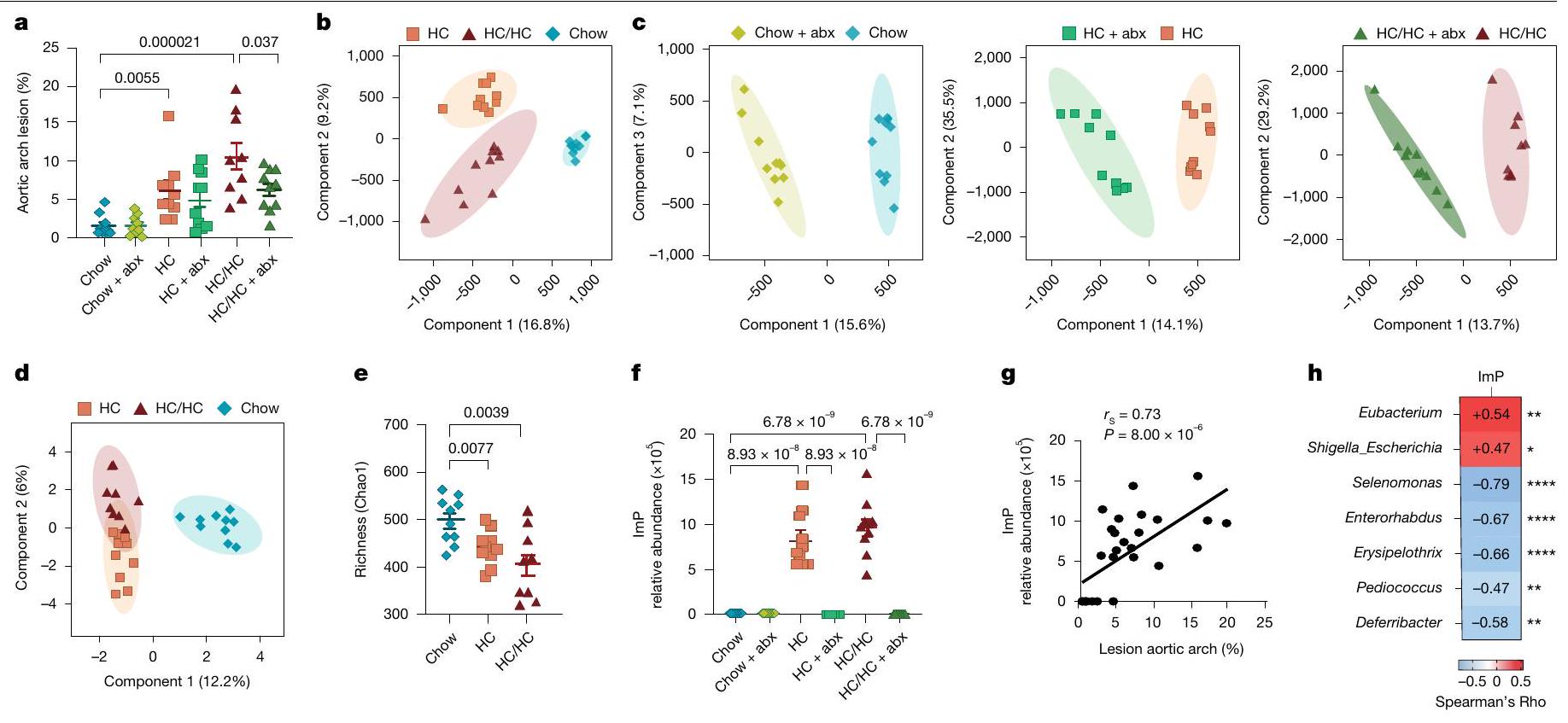
مع تصلب الشرايين بطريقة تعتمد على الميكروبيوتا
ImP مرتبط بتصلب الشرايين تحت السريري في البشر
(الشكل 2b). تم قياس ImP بعد ذلك في مجموعة مستقلة، مجموعة تحمل تحمل الجلوكوز (IGT) (الجدول التمديدي 2a,b)، والتي تضم متطوعين غير مصابين، بما في ذلك 529 ضابطًا و1,315 فردًا يعانون من تصلب الشرايين تحت السريري
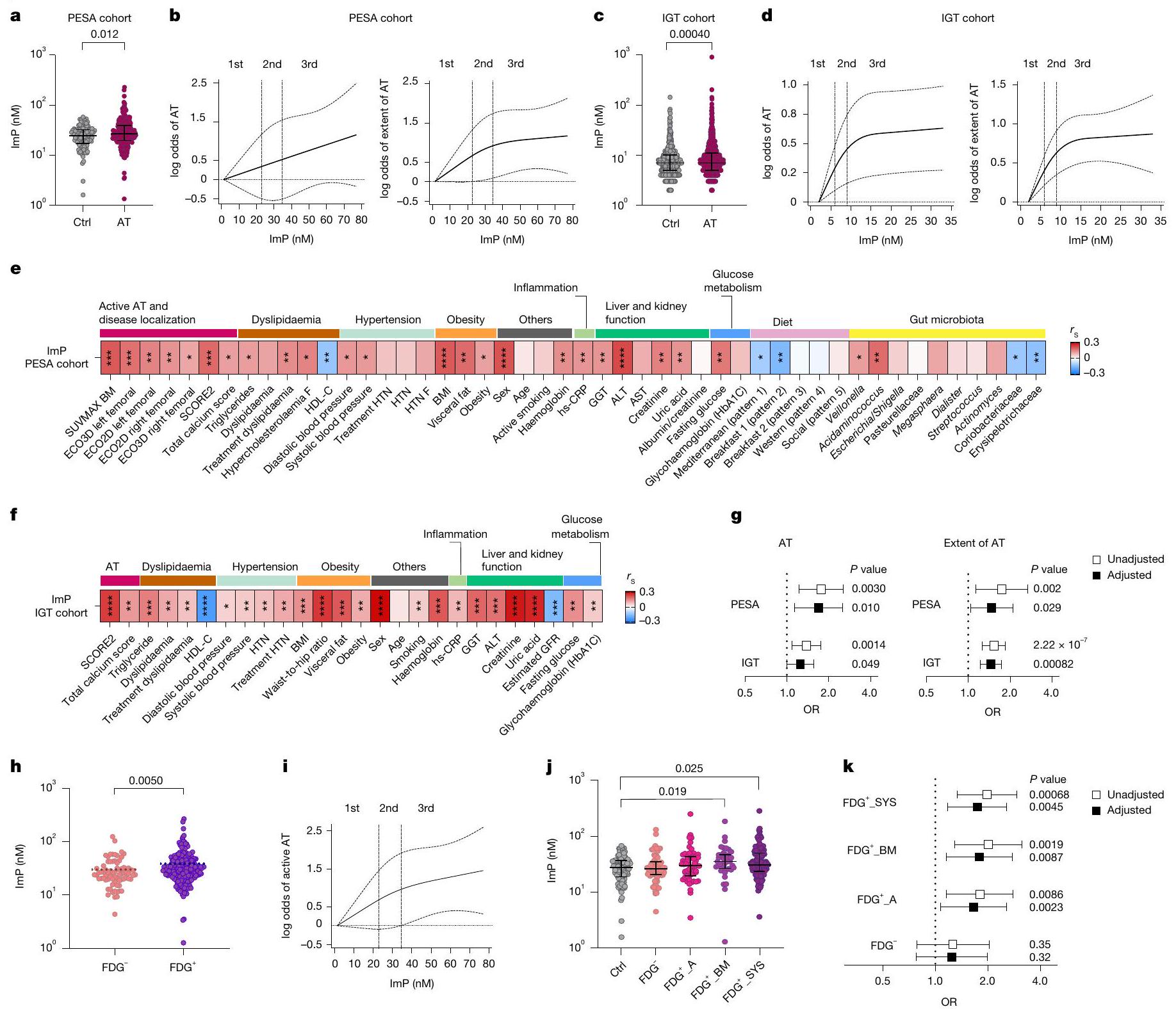
عرض فترات الثقة 95%. أو، نسبة الأرجحية. ج، مستوى ImP في البلازما لدى الأفراد الذين يعانون من تصلب الشرايين غير النشط (FDG
الكوليسترول منخفض الكثافة (LDL) و hs-CRP في تمييز انتشار تصلب الشرايين في كلا المجموعتين (الشكل البياني الموسع 2c، d).
تصلب الشرايين النشط (
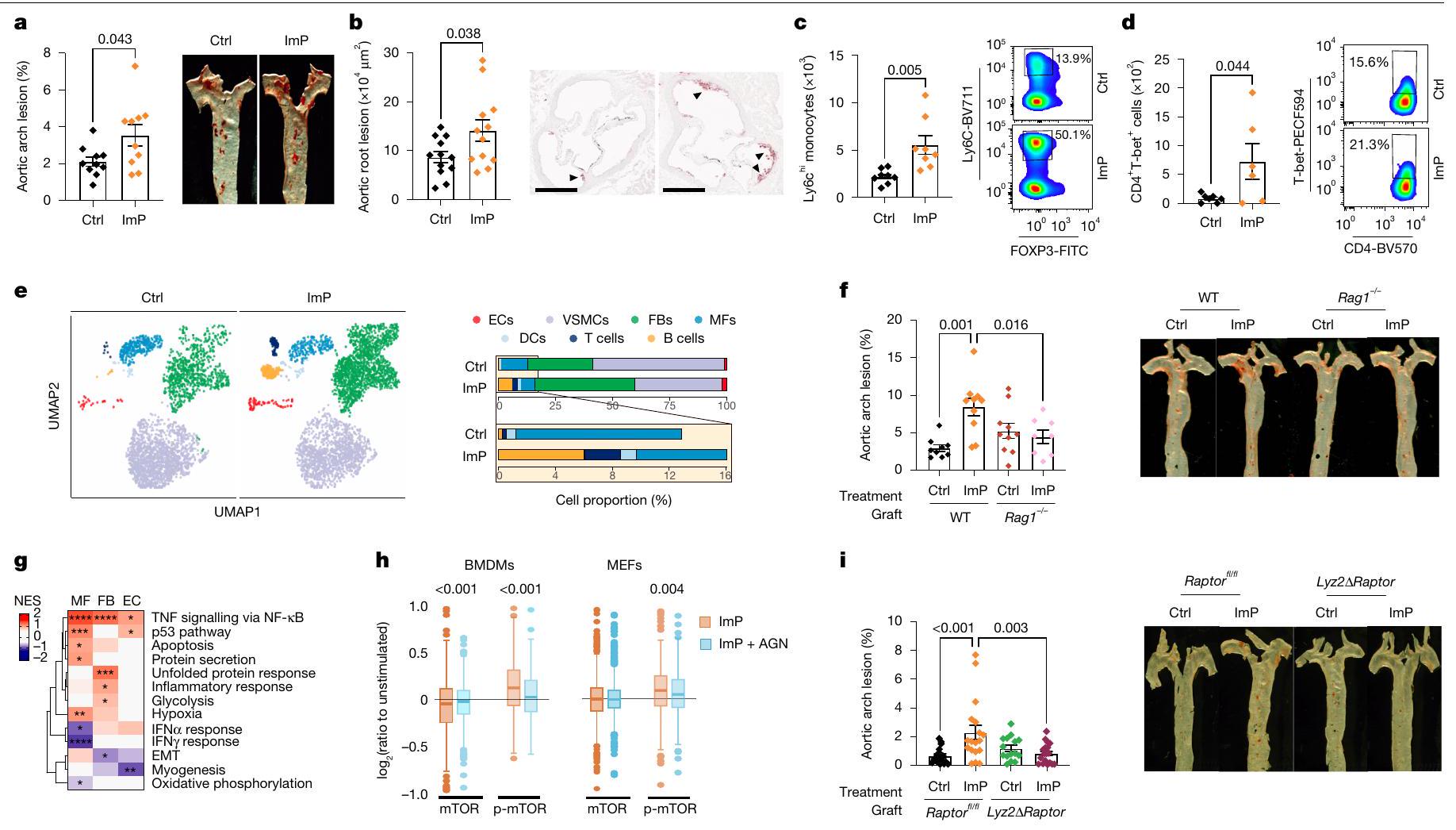
صور الشريان الأورطي (يمين). ج، خريطة حرارية لتحليل المسار الجيني المقارن بين ImP والمجموعة الضابطة لخلايا البطانية، الألياف، والبلعميات من بيانات تسلسل RNA أحادي الخلية لـ Apoe
تنشيط النخاع
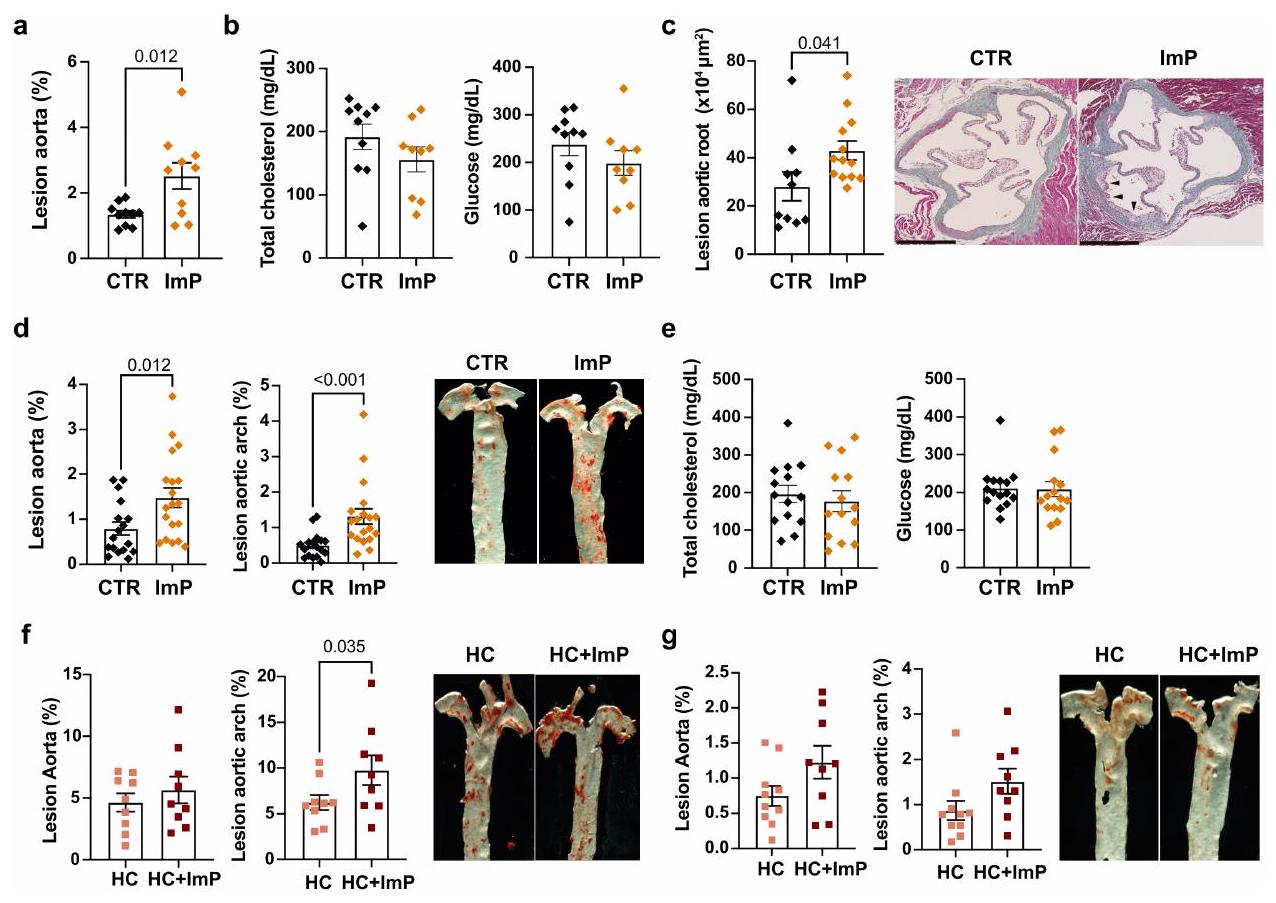
ImP يسبب تصلب الشرايين دون تغيير في مستوى الكوليسترول
و الشكل الإضافي للبيانات 3c-e). في كلا النموذجين، زادت مكملات ImP من تطور تصلب الشرايين في الشريان الأورطي وجذر الشريان الأورطي دون التأثير على تركيز الكوليسترول أو الجلوكوز في الدم (الشكل 3a,b والشكل الإضافي للبيانات 3a-e). أدى التغذية بنظام HC الغذائي وحده إلى إنتاج ImP (الشكل 1f). لم تؤدِ مكملات ImP الإضافية إلى تفاقم تصلب الشرايين بشكل كبير في المجموعة التي تغذت على HC.
تكرار تجمع الخلايا في الشريان الأورطي (الشكل التوضيحي 4e)، دون تغييرات في الخلايا المناعية الدائرة (الشكل التوضيحي 4g، h). بالمقابل، أظهر تسلسل RNA أحادي الخلية للشرايين الأورطية من الفئران المعالجة بـ ImP لمدة ثمانية أسابيع زيادة في العدد النسبي للأرومات الليفية، والخلايا البطانية، والخلايا المناعية (الشكل 3e). تم تأكيد العدد المتزايد من الخلايا المناعية في الشريان الأورطي من خلال تحليل تدفق الخلايا (الشكل التوضيحي 4i) والتحليل النسيجي لجذر الشريان الأورطي، الذي أظهر زيادة في تسلل خلايا T في طبقة الإنتيما-ميديا وزيادة في صبغة MAC2 للماكروفاجات الالتهابية في لويحة التصلب العصيدي للفئران المعالجة بـ ImP مقارنة بالمجموعة الضابطة (الشكل التوضيحي 4j، k). من المRemarkably، كانت قدرة ImP على تحفيز تصلب الشرايين منخفضة بشكل ملحوظ في
في الجسم الحي، يغير ImP التركيب الجيني للشريان الأورطي
يُفعّل استجابة التهابية تعتمد على mTOR في الخلايا النخاعية، مما يساهم في تكوين اللويحات.
ImP يقود تصلب الشرايين عبر IIR في الخلايا النخاعية
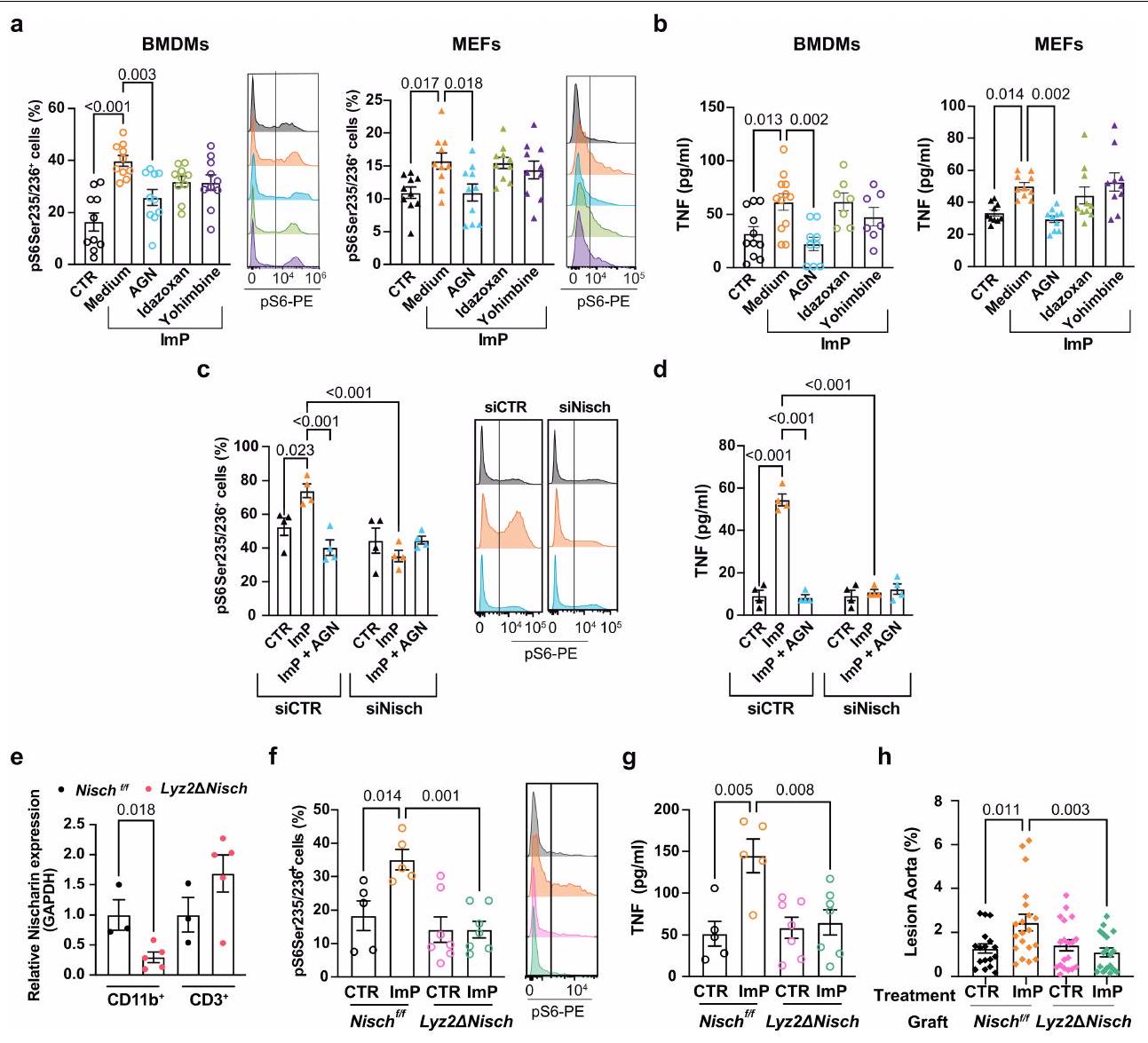
لتحليل الاستخدام المحتمل لـ AGN192403 لمنع تصلب الشرايين الناتج عن ImP، قمنا بإعطاء ImP و AGN192403 معًا في مياه الشرب لجرذان Apoe التي تتغذى على العلف.
حجب ImP-I1R يمنع تقدم تصلب الشرايين
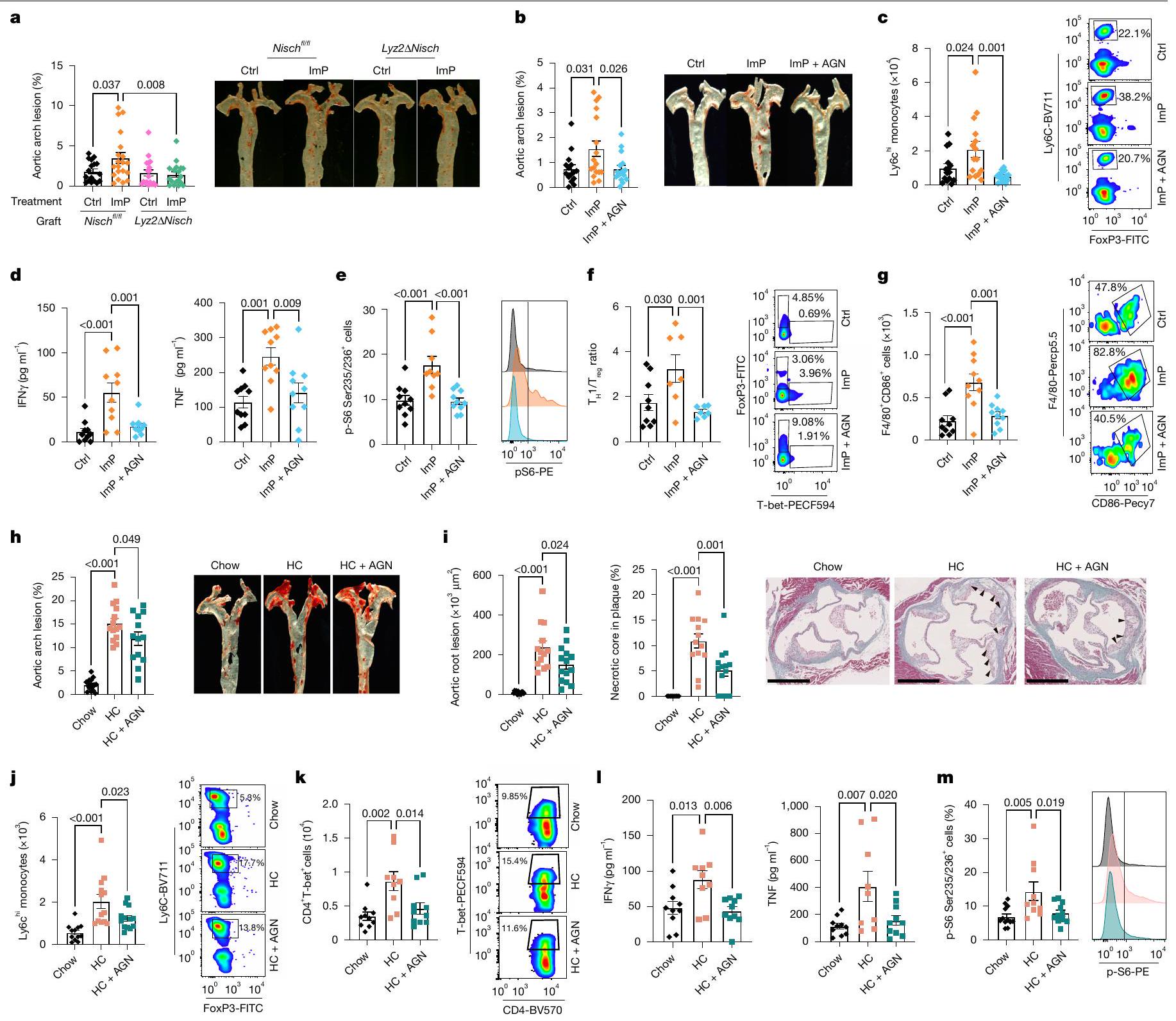
في الفئران التي تم تغذيتها بنظام HC الغذائي (الشكل التوضيحي الممتد 8e). علاوة على ذلك، فإن علاج AGN192403 منع التحفيز المعتمد على نظام HC الغذائي لمستويات Ly6C في الدم.
تركيز الكوليسترول في البلازما، مما يشير إلى نهج جديد لعلاج تصلب الشرايين.
نقاش
المحتوى عبر الإنترنت
5. ريدكر، ب. م. وآخرون. العلاج المضاد للالتهابات باستخدام كاناكنوماب لمرض تصلب الشرايين. نيو إنجلاند جورنال أوف ميديسين 377، 1119-1131 (2017).
6. نيلسون، ك.، فوستر، ف. & ريدكر، ب. م. الكولشيسين بجرعة منخفضة للوقاية الثانوية من مرض الشريان التاجي. مجلة الكلية الأمريكية لأمراض القلب 82، 648-660 (2023).
7. ساباتين، م. س. وآخرون. إيفولوكوماب والنتائج السريرية لدى المرضى الذين يعانون من أمراض القلب والأوعية الدموية. نيو إنجلند جورنال أوف ميديسين 376، 1713-1722 (2017).
8. كاسينبروود، ل. وآخرون. توزيع تقدير خطر الأحداث الوعائية المتكررة على مدى 10 سنوات والخطر المتبقي في مجموعة الوقاية الثانوية. الدورة الدموية 134، 1419-1429 (2016).
9. تشاكرون، ر. م.، أولسون، ل. م. وباكهيد، ف. إمكانيات تخصيص الميكروبيوم المعوي للوقاية من الأمراض القلبية الأيضية وعلاجها. نات. ريف. كارديو. 20، 217-235 (2023).
10. نيمت، إ. وآخرون. أطلس لمنتجات الميكروبات المعوية المستمدة من الأحماض الأمينية العطرية ومخاطر الأمراض القلبية الوعائية والوفيات. مجلة القلب الأوروبية 44، 3085-3096 (2023).
11. ويتكوفسكي، م.، ويكس، ت. ل. & هازن، س. ل. ميكروبيوتا الأمعاء وأمراض القلب والأوعية الدموية. دائرة. بحث. 127، 553-570 (2020).
12. نيمت، إ. وآخرون. مستقلب ميكروبي معوي مرتبط بأمراض القلب والأوعية الدموية يعمل عبر مستقبلات الأدرينالين. خلية 180، 862-877.e22 (2020).
13. مارتينيز-لوبيز، م. وآخرون. استشعار الميكروبيوتا بواسطة محور مينكل-سايك في الخلايا الشجرية ينظم إنتاج الإنترلوكين-17 و-22 ويعزز سلامة الحاجز المعوي. المناعة 50، 446-461.e9 (2019).
14. وانغ، ز. وآخرون. استقلاب فلورا الأمعاء للفوسفاتيديل كولين يعزز أمراض القلب والأوعية الدموية. نيتشر 472، 57-63 (2011).
15. كو، أ. وآخرون. الإيميدازول بروبيونات المنتج ميكروبيًا يعيق إشارات الأنسولين من خلال mTORC1. خلية 175، 947-961.e17 (2018).
16. رويير، ب. وآخرون. بروتيوميات البلازما لتوقع التكلسات الشريانية التاجية تحت السريرية في الوقاية الأولية. مجلة القلب الأمريكية 271، 55-67 (2024).
17. مولينارو، أ. وآخرون. تم زيادة بروبيونات الإيميدازول في مرض السكري وهو مرتبط بأنماط النظام الغذائي وبيئة الميكروبات المتغيرة. نات. كوميونيك. 11، 5881 (2020).
18. كوري، أ. وآخرون. الميكروبات الفموية والمعوية واللويحات في المرضى الذين يعانون من تصلب الشرايين. وقائع الأكاديمية الوطنية للعلوم 108، 4592-4598 (2011).
19. ترسيد، م.، أندرسن، ج. Ø.، بروخ، ك. & هوف، ج. ر. الميكروبيوم المعوي في مرض الشريان التاجي وفشل القلب: المعرفة الحالية والاتجاهات المستقبلية. إي بايو ميديسين 52، 102649 (2020).
20. فرنانديز-فرييرا، ل. وآخرون. الالتهاب الوعائي في تصلب الشرايين تحت السريري المكتشف بواسطة PET/MRI الهجين. مجلة الكلية الأمريكية لأمراض القلب 73، 1371-1382 (2019).
21. ديفيسا، أ. وآخرون. تنشيط نخاع العظام استجابةً لمتلازمة الأيض وتصلب الشرايين المبكر. مجلة القلب الأوروبية 43، 1809-1828 (2022).
22. ما، ج.، ليو، ج.، صن، ي. وزهاو، ز. السيتوكينات المرتبطة بالاستجابة المناعية في تصلب الشرايين. المجلة الأمريكية للبحوث الانتقالية 14، 6424-6444 (2022).
23. غولد، إي. إس. وآخرون. ATF3 يحمي من تصلب الشرايين عن طريق قمع تكوين أجسام الدهون المستحث بواسطة 25-هيدروكسي كوليسترول. ج. إكسب. ميد. 209، 807-817 (2012).
24. وكيليك، س. ك.، دنفي، ج.، هيراس-مورييلو، إ.، ماسترنغو، أ. وسانشو، د. استقلاب البلعميات النسيجية في التوازن والمرض. الخلية. المناعة الجزيئية. 19، 384-408 (2022).
25. تشين، ج. ي.، كورتيز، س. و فيريرا، ف. ب. بروبردين: جزيء متعدد الأوجه مرتبط بالالتهابات والأمراض. مناعة جزيئية 102، 58-72 (2018).
26. كيس، م. ج. وبايندر، س. ج. التأثير المتعدد الأوجه للمكمل على تصلب الشرايين. تصلب الشرايين 351، 29-40 (2022).
27. كان، هـ. وآخرون. تحليل النسخ الجيني على مستوى الخلية الواحدة يكشف عن تباين خلوي في الشرايين الأورطية الصاعدة للفئران العادية والفئران التي تتبع نظام غذائي عالي الدهون. التجارب. مول. ميد. 53، 1379-1389 (2021).
28. زهاو، ج. وآخرون. تقنيات النسخ الجيني على مستوى الخلية الواحدة تكشف عن مرونة البطانة خلال تكوين تصلب الشرايين السكري. Front. Cell Dev. Biol. 9، 689469 (2021).
29. غوتشيفا، ف.، تشين، إكس.، بيترز، سي.، رينهكل، تي. وجويس، ج. أ. حذف الكاتيبسين H يعيق التحول الوعائي، التوعية ونمو الأورام في نموذج فأر لسرطان خلايا جزر البنكرياس. الكيمياء الحيوية 391، 937-945 (2010).
30. شيه، س.-سي. وآخرون. بروتين L6 TM4SF1 حاسم لوظيفة الخلايا البطانية وتكوين الأوعية الدموية في الأورام. أبحاث السرطان 69، 3272-3277 (2009).
31. شنييلر، د. وآخرون. السيتوكين الشبيه 1 هو عامل جديد محفز لتكوين الأوعية يتم إفرازه بواسطة خلايا السلف البطانية ويقوم بوساطة وظائفها. دائرة. بحث. 124، 243-255 (2019).
32. داير، ل. أ.، بي، إكس. و باترسون، س. دور BMPs في وظيفة الخلايا البطانية وعدم وظيفتها. اتجاهات الغدد الصماء والتمثيل الغذائي 25، 472-480 (2014).
33. آييلو، ر. ج. وآخرون. بروتين كيميائي جذب وحيدات النوى-1 يسرع تصلب الشرايين في الفئران التي تفتقر إلى بروتين الأبوليبوبروتين E. علم الأوعية الدموية. 19، 1518-1525 (1999).
34. وانغ، ل.، لورانس، ج. س.، ستورغيل، ت. و. & هاريس، ت. إ. نشاط مركب الهدف الثديي من الراباميسين 1 (mTORC1) مرتبط بفوسفاتة الرابتور بواسطة mTOR. ج. كيمياء حيوية. 284، 14693-14697 (2009).
35. بوسكيه، ب.، هودسون، أ.، غارسيا-سيفيلا، ج. أ. ولي، ج.-إكس. نظام مستقبلات الإيميدازولين: الماضي، الحاضر، والمستقبل. مراجعة علم الأدوية 72، 50-79 (2020).
36. هيد، ج. أ. ومايوروف، د. ن. مستقبلات الإيميدازولين، عوامل جديدة والإمكانات العلاجية. عوامل الأدوية القلبية الوعائية والدموية. 4، 17-32 (2006).
37. زانغ، إكس. وآخرون. الأنظمة الغذائية الغنية بالبروتين تزيد من خطر الإصابة بأمراض القلب والأوعية الدموية من خلال تنشيط الماكرومير mTOR لقمع الميتوفاجي. نات. ميتاب. 2، 110-125 (2020).
38. مولينارو، أ. وآخرون. الإيميدازول بروبيونات المنتج ميكروبيًا مرتبط بفشل القلب والوفيات. مجلة جACC لفشل القلب. 11، 810-821 (2023).
39. زو، و. وآخرون. المستقلبات الميكروبية المعوية TMAO تعزز فرط تفاعل الصفائح الدموية وزيادة خطر thrombosis. خلية 165، 111-124 (2016).
40. وانغ، ز. وآخرون. ميكروبيوتا الأمعاء، وعوامل الالتهاب الدائرة، والمواد الأيضية، وتصلب الشرايين السباتية في عدوى فيروس نقص المناعة البشرية. ميكروبيوم 11، 119 (2023).
41. فان سون، ج. وآخرون. بروبيونات الإيميدازول في البلازما مرتبط إيجابيًا بضغط الدم لدى البشر الذين يعانون من زيادة الوزن والسمنة. المغذيات 13، 2706 (2021).
42. ترواسيد، م. وآخرون. التغيرات في ميكروبيوم الأمعاء ومستويات الإيميدازول بروبيونات المتداولة مرتبطة بمرض الشريان التاجي الانسدادي لدى الأشخاص المصابين بفيروس نقص المناعة البشرية. مجلة الأمراض المعدية 229، 898-907 (2024).
43. ناجيسواران، ف. وآخرون. المستقلبات الميكروبية المعوية إيميدازول بروبيونات تؤثر سلبًا على وظيفة الخلايا البطانية وتعزز تطور تصلب الشرايين. علم الأوعية الدموية. 45، 823-839 (2025).
44. ويست، ر. س. أ. وآخرون. تقنيات التصوير بالرنين المغناطيسي الناشئة لتصوير تصلب الشرايين. علم الأوعية الدموية. 39، 841-849 (2019).
45. جيمبرون، م. أ. وغارسيا-كاردينا، ج. خلل وظيفة الخلايا البطانية وعلم الأمراض المرتبط بتصلب الشرايين. دائرة. بحث. 118، 620-636 (2016).
46. روي، ب.، أوريكشوني، م. ولي، ك. كيف يشكل الجهاز المناعي تصلب الشرايين: أدوار المناعة الفطرية والتكيفية. نات. ريف. إيمونول. 22، 251-265 (2022).
47. فين، أ. ف.، ناكانو، م.، نارولا، ج.، كولودجي، ف. د. وفيرماني، ر. مفهوم اللويحة القابلة للخطر/ غير المستقرة. علم الأوعية الدموية والتخثر. 30، 1282-1292 (2010).
48. وولف، د. ولي، ك. المناعة والالتهاب في تصلب الشرايين. دائرة. بحث. 124، 315-327 (2019).
49. زانغ، إكس. وآخرون. تحديد تأثير العتبة الذي يتوسطه الليوسين والذي يحكم إشارة الماكرومول الماكرومول والمخاطر القلبية الوعائية. نات. ميتاب. 6، 359-377 (2024).
50. لويد-جونز، د. م. وآخرون. 2017 تحديث مركّز لمسار قرار الإجماع من خبراء ACC لعام 2016 حول دور العلاجات غير الستاتينية في خفض كوليسترول LDL في إدارة مخاطر مرض القلب والأوعية الدموية التصلبي: تقرير من مجموعة العمل التابعة للكلية الأمريكية لأمراض القلب حول مسارات قرار الإجماع من الخبراء. ج. أم. كول. كارد. 70، 1785-1822 (2017).
51. غوبتا، أ. وآخرون. الأحداث السلبية المرتبطة بالعلاج بالستاتين غير المموه، ولكن ليس مع العلاج المموه، في تجربة النتائج القلبية الأنجلو-سكندنافية – ذراع خفض الدهون (ASCOT-LLA): تجربة عشوائية مزدوجة التعمية خاضعة للرقابة الوهمية ومرحلتها غير العشوائية وغير المموهة. لانسيت 389، 2473-2481 (2017).
© المؤلفون 2025، نشر معدل 2025
طرق
فئران
إعطاء المضادات الحيوية
تحديد كمية الآفات في الشريان الأورطي
علم الأنسجة لجذور الشريان الأورطي
جمع محتوى القولون
تحليل المصل
التحليل الأيوني الكتلي السائل غير المستهدف
وتم فصل المستقلبات عند
تحليل تدفق الخلايا
إيلاستاز
مجموعات الدراسة
الشريان الأورطي (نقاط: 0، لا؛ 1، نعم)؛ (4) درجة CAC (نقاط: 0، 0؛ 1، 0.1-99؛ 2، 100-399؛ 3، 400-الحد الأقصى). بالنسبة للتحليل الأيضي، تم جمع عينات الدم الطرفية بعد صيام ليلة كاملة وتمت عملية الطرد المركزي في
الميتabolوميات المستهدفة
استخراج الحمض النووي، تسلسل الأمبليكون 16S rDNA وتحليل بيانات الميكروبيوتا
شريحة bioanalyzer DNA1000 (Agilent Technologies). تم تجميع العينات بكميات متساوية وتم تسلسلها باستخدام مجموعة MiSeq Reagent v.3، على منصة MiSeq Illumina (خدمة تسلسل FISABIO).
تقييم نمط النظام الغذائي
تسلسل scRNA-seq
إجمالي عد UMI (
خطوط الخلايا
تسلسل RNA بالجملة
البروتيوميات والفوسفورتيوميات بواسطة الوسم المتساوي
تم إجراء البيانات الكمية باستخدام
تلوين p-S6 بواسطة تحليل تدفق الخلايا
جمع البلعميات البريتونية
قياس TNF
قياس MCP1
ترانسويل
قياسات السيتوكينات في المصل
علاج ImP في الفئران المزروعة
تخفيض التعبير الجيني بواسطة siRNA I1R
تفاعل البوليميراز المتسلسل الكمي مع النسخ العكسي
الإحصائيات
ملخص التقرير
توفر البيانات
52. راكوف-ناهوم، س.، باجلينو، ج.، إسلامي-فارزانه، ف.، إدبرغ، س. وميدزهيتوف، ر. التعرف على الميكروفلورا المتعايشة بواسطة مستقبلات شبيهة التول ضروري للحفاظ على توازن الأمعاء. خلية 118، 229-241 (2004).
53. سنتا، م.، كيتلهوت، د. ف. ج.، مالين، س. وجيسترا، أ. قياس تصلب الشرايين في الفئران. ج. فيس. إكسب.https://doi.org/10.3791/59828 (2019).
54. ماسترنجيلو، أ. وآخرون. مقاومة الأنسولين في الأطفال البدينين قبل البلوغ ترتبط بتغيرات الميتابولوم المبكرة المعتمدة على الجنس. المجلة الدولية للسمنة 40، 1494-1502 (2016).
55. لورينزو، سي. وآخرون. ALDH4A1 هو مستضد ذاتي للتصلب العصيدي يستهدفه الأجسام المضادة الواقية. ناتشر 589، 287-292 (2021).
56. بانغ، ز. وآخرون. استخدام MetaboAnalyst 5.0 لمعالجة طيف LC-HRMS، ودمج البيانات متعددة الأوميات، وضبط المتغيرات في بيانات الميتابولوميات العالمية. بروتوكولات الطبيعة 17، 1735-1761 (2022).
57. جيل دي لا فوانتي، أ. وآخرون. CEU Mass Mediator 3.0: أداة لتوصيف المستقلبات. مجلة أبحاث البروتينات 18، 797-802 (2019).
58. باريرو-رودريغيز، ر. وآخرون. TurboPutative: خادم ويب لمعالجة البيانات وتصنيف المستقلبات في علم المستقلبات غير المستهدفة. Front. Mol. Biosci. 9، 952149 (2022).
59. فرنانديز-أورتيز، أ. وآخرون. دراسة تقدم واكتشاف مبكر للتصلب العصيدي تحت السريري (PESA): الأساس والتصميم. مجلة القلب الأمريكية 166، 990-998 (2013).
60. سانغوينتي، إ. وآخرون. بصمات الميكروبيوم-الميتوبولوم في الفئران المعرضة وراثياً لتطوير الخرف، التي تتغذى على نظام غذائي عادي أو دهني. ساي. ريب. 8، 4907 (2018).
61. كاروسو، ر.، أونو، م.، بانكر، م. إ.، نونيز، ج. & إينوهارا، ن. تغييرات ديناميكية وغير متكافئة في المجتمعات الميكروبية بعد العيش المشترك في الفئران المخبرية. تقارير الخلايا 27، 3401-3412.e3 (2019).
62. كوزيتش، ج. ج.، ويستكوت، س. ل.، باكستر، ن. ت.، هايلاندر، س. ك. وشلوس، ب. د. تطوير استراتيجية تسلسل ذات فهرسين وخط أنابيب تنسيق لتحليل بيانات تسلسل الأمبليكون على منصة تسلسل ميسيك إلومينا. الميكروبيولوجيا البيئية التطبيقية 79، 5112-5120 (2013).
63. كالاها، ب. ج. وآخرون. DADA2: استنتاج عينة عالي الدقة من بيانات أمبليكون إيلومينا. نات. ميثودز 13، 581-583 (2016).
64. شلوس، ب. د. وآخرون. تقديم موثور: برنامج مفتوح المصدر، مستقل عن المنصات، مدعوم من المجتمع لوصف ومقارنة المجتمعات الميكروبية. تطبيقات. ميكروبيولوجيا البيئة. 75، 7537-7541 (2009).
65. دي لا فوانتي-أريلاج، سي.، رويز، ز. ف.، بيس-راسترويلو، م.، سامبسون، ل. ومارتينيز-غونزاليس، م. أ. قابلية إعادة إنتاج استبيان تكرار تناول الطعام تم التحقق منه في إسبانيا. التغذية والصحة العامة. 13، 1364-1372 (2010).
66. أميزكيتا، ر. أ. وآخرون. تنسيق تحليل الخلايا الفردية باستخدام بايكونداكتور. نات. ميثودز 17، 137-145 (2020).
67. هاو، ي. وآخرون. تحليل متكامل لبيانات الخلايا الفردية متعددة الأنماط. خلية 184، 3573-3587.e29 (2021).
68. بوغي، ج. ج. وآخرون. BFF وcellhashR: أدوات تحليل لتفكيك بيانات تجزئة الخلايا بدقة. المعلوماتية الحيوية 38، 2791-2801 (2022).
69. تورال، م. وآخرون. تنظيم نقل الكارنيتين بالميتويل بواسطة PPAR-
70. غارود، ج. وآخرون. التكيفات في سلسلة التنفس الميتوكوندرية في البلعميات تساهم في الدفاع المضاد للبكتيريا. نات. إيمونول. 17، 1037-1045 (2016).
71. دوبين، أ. وآخرون. ستار: محاذي تسلسل RNA عالمي فائق السرعة. المعلوماتية الحيوية 29، 15-21 (2013).
72. لي، ب. ودووي، س. ن. RSEM: تقدير دقيق لنسخ الجينات من بيانات RNA-seq مع أو بدون جينوم مرجعي. BMC Bioinformatics 12، 323 (2011).
73. لوف، م. آي.، هوبر، و. & أندرس، س. تقدير معتدل لتغير الطي والتشتت لبيانات RNA-seq باستخدام DESeq2. جينوم بيو. 15، 550 (2014).
74. Gu، ز.، Eils، ر. و Schlesner، م. الخرائط الحرارية المعقدة تكشف عن الأنماط والارتباطات في البيانات الجينومية متعددة الأبعاد. المعلوماتية الحيوية 32، 2847-2849 (2016).
75. روميرو-بيسيرا، ر. وآخرون.
76. رودريغيز، ج. م. وآخرون. iSanXoT: تطبيق مستقل لتحليل البيانات الكمية للبروتينات المستندة إلى مطيافية الكتلة. مجلة الحوسبة والهندسة الحيوية 23، 452-459 (2024).
77. سوبرا مانيان، أ. وآخرون. تحليل إثراء مجموعة الجينات: نهج قائم على المعرفة لتفسير ملفات التعبير الجينومي الشامل. وقائع الأكاديمية الوطنية للعلوم في الولايات المتحدة الأمريكية 102، 15545-15550 (2005).
78. ستاماتيلوبولوس، ك. وآخرون. أميلويد-
79. ستاماتيلوبولوس، ك. وآخرون. مستويات الكاتيبسين S والبقاء على قيد الحياة بين المرضى الذين يعانون من متلازمات الشريان التاجي الحادة غير المرتبطة بارتفاع مقطع ST. مجلة الكلية الأمريكية لأمراض القلب 80، 998-1010 (2022).
80. والد، أ. اختبارات متسلسلة للفرضيات الإحصائية. آن. ماث. ستات. 16، 117-186 (1945).
81. هاسي، ت.، تيبشيراني، ر. وواينرايت، م. التعلم الإحصائي مع الندرة: اللاسو والتعميمات (تشابمان وهال، 2015).
82. دي لونغ، إ. ر.، دي لونغ، د. م. وكلارك-بيرسون، د. ل. مقارنة المساحات تحت منحنيات خصائص التشغيل المستقبلي المرتبطة: نهج غير معلمي. البيومترية 44، 837-845 (1988).
معلومات إضافية
يجب توجيه المراسلات والطلبات للحصول على المواد إلى ديفيد سانشو. تشكر مجلة Nature سُرَيّا طالب والمراجعين الآخرين المجهولين على مساهمتهم في مراجعة الأقران لهذا العمل. تقارير مراجعي الأقران متاحة.
معلومات إعادة الطبع والتصاريح متاحة علىhttp://www.nature.com/reprints.
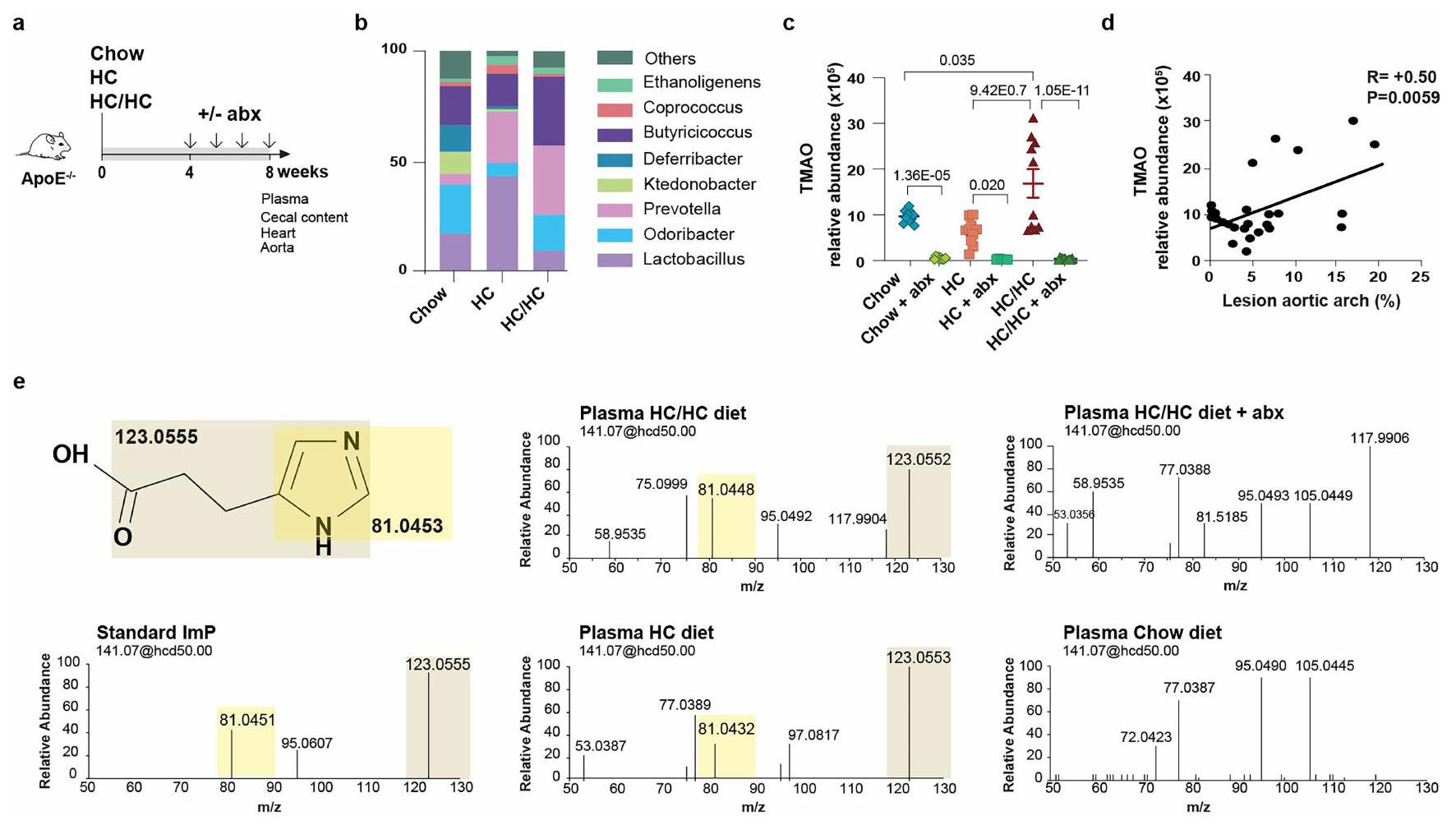
ج، الوفرة النسبية لـ TMAO في البلازما تم قياسها بواسطة LC-MS غير المستهدف.
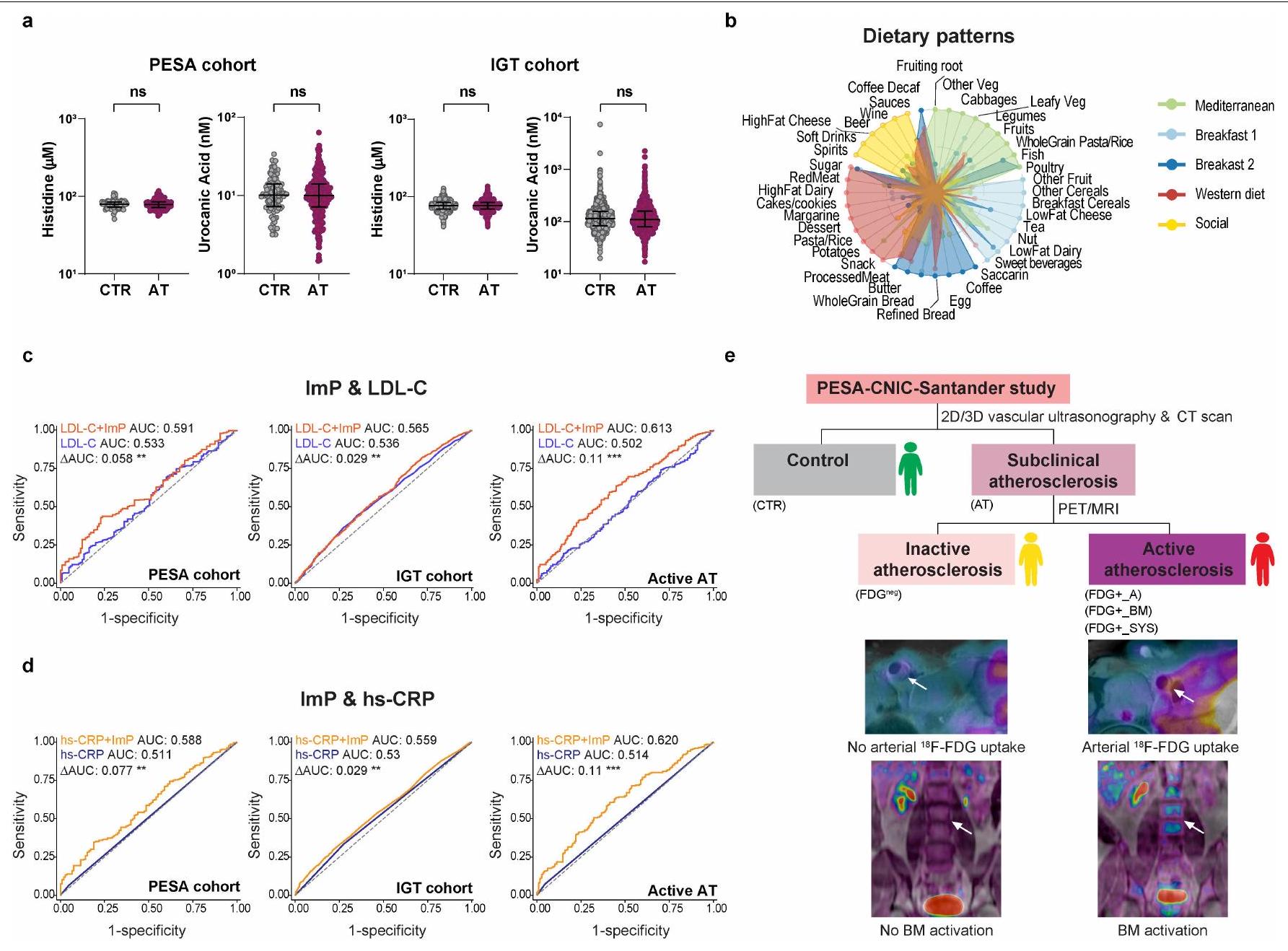
مجموعة: AUC
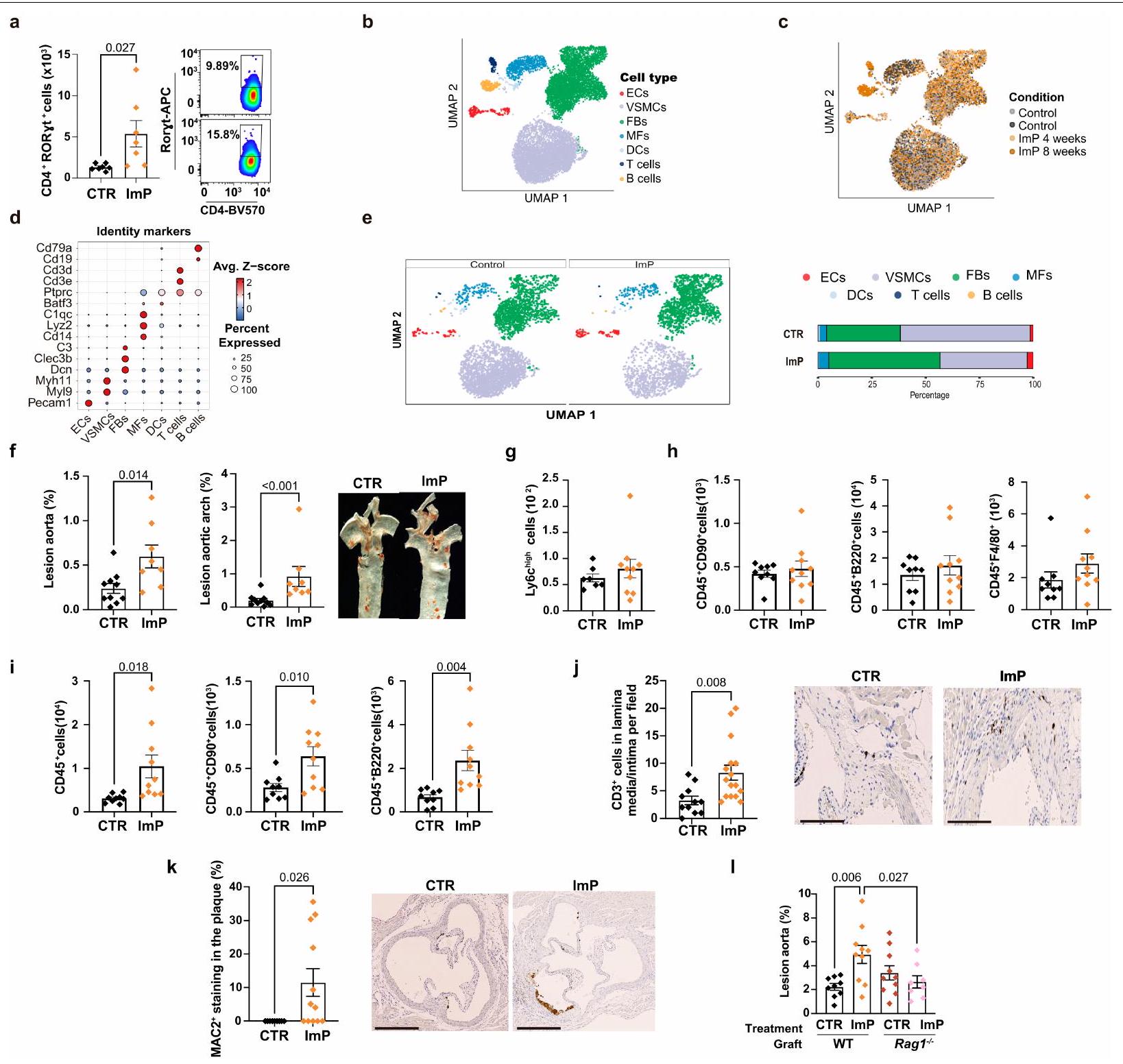
محدد بواسطة تحليل تدفق الخلايا. إجمالي أعداد CD45
مقالة
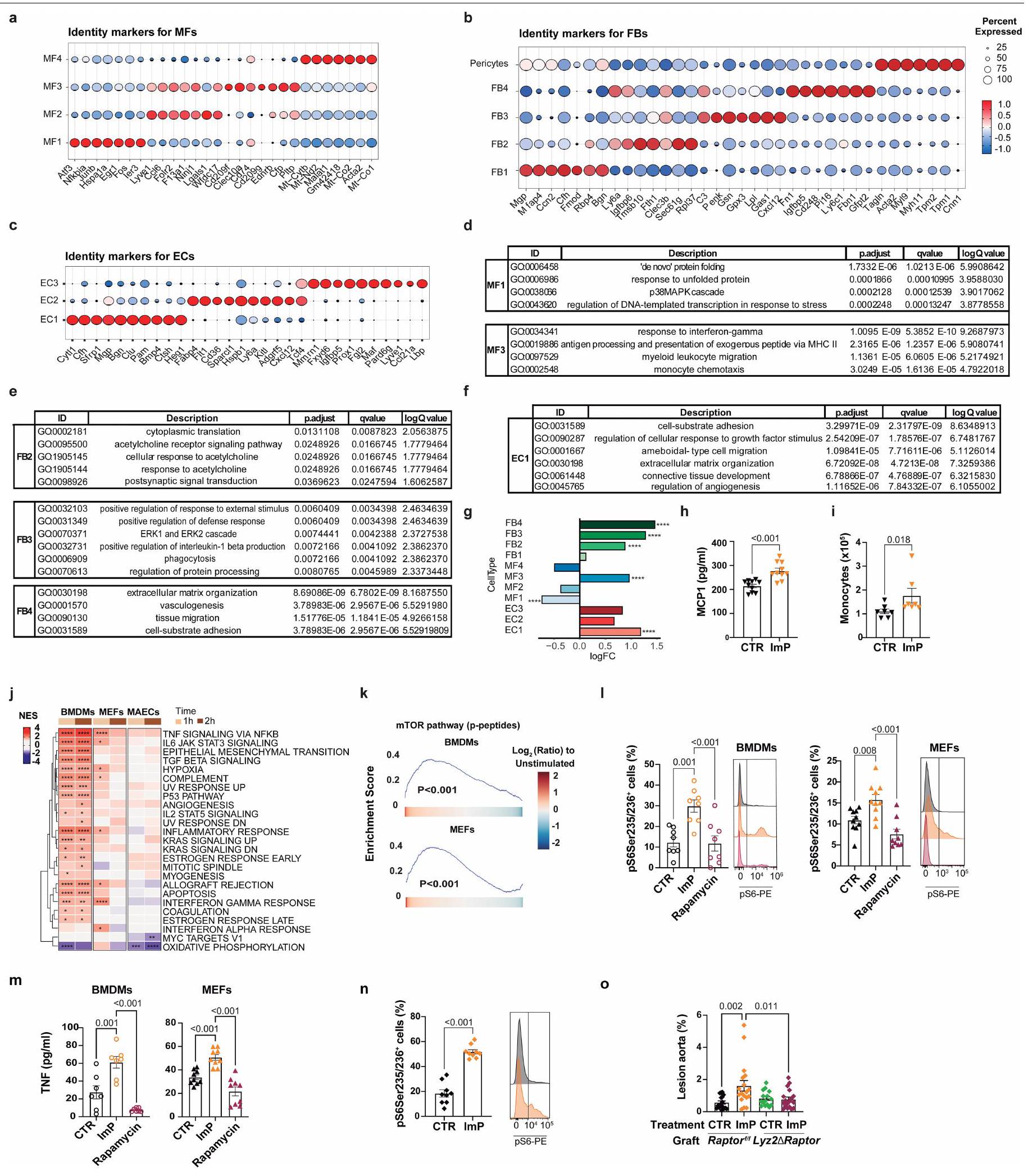
بعد تحفيز ImP لمدة 24 ساعة مع أو بدون تزامن مع الراباميسين. 1، صبغة تدفق الخلايا لبروتين ribosomal pS6. اليسار: BMDMn = 8؛ MEF CTR = 10؛ ImP = 10؛ ImP + AGN = 9؛ اليمين: هيستوجرامات تمثيلية لـ CTR (الأعلى)، ImP (الوسط) و ImP + راباميسين (الأسفل). m، إنتاج TNF تم قياسه بواسطة ELISA. BMDM
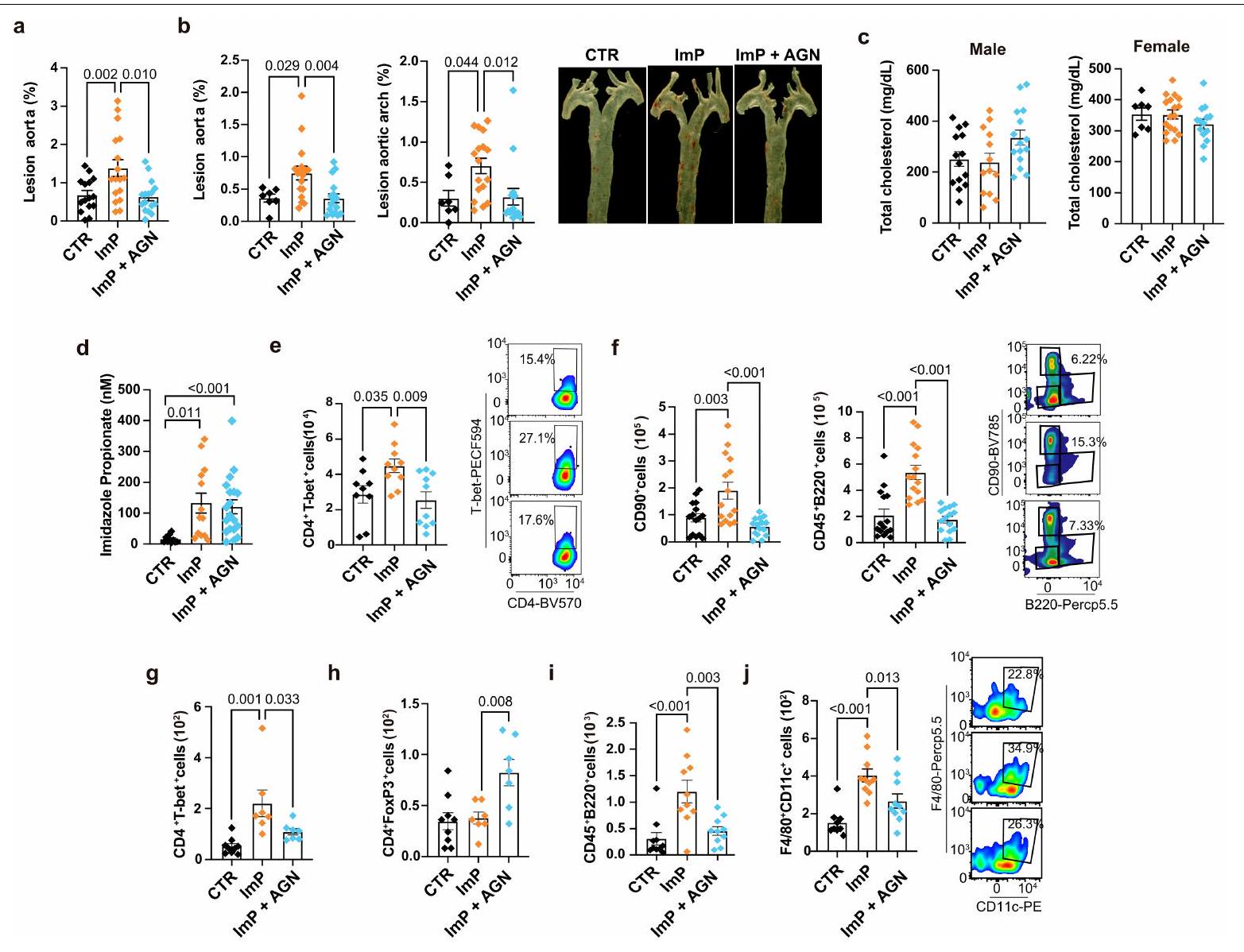
صبغة الشريان الأورطي في إناث الفئران تظهر تقدير الآفة التصلبية في الشريان الأورطي بالكامل (يسار)، قوس الشريان الأورطي (وسط) وصور تمثيلية (يمين). تشاو
محفظة الطبيعة
ديفيد سانشو
آخر تحديث من المؤلفين: 3 يونيو 2025
ملخص التقرير
الإحصائيات
غير متوفر
تم التأكيد
□

□

□

يجب أن تُوصف الاختبارات الشائعة فقط بالاسم؛ واصفًا التقنيات الأكثر تعقيدًا في قسم الطرق.
□

□

□

□

□ لتحليل بايزي، معلومات حول اختيار القيم الأولية وإعدادات سلسلة ماركوف مونت كارلو

□ لتصميمات هرمية ومعقدة، تحديد المستوى المناسب للاختبارات والتقارير الكاملة عن النتائج

□ تقديرات أحجام التأثير (مثل حجم تأثير كوهين)
البرمجيات والشيفرة
جمع البيانات
تم جمع بيانات الميتابولوميات غير المستهدفة بواسطة نظام HPLC Ultimate 3000 المتصل بجهاز LTQ Orbitrap XL.
تم إنشاء خريطة الحرارة وجميع الرسوم البيانية باستخدام برنامج GraphPad Prism الإصدار 9.0 (GraphPad Software، سان دييغو، كاليفورنيا). تم استخدام الإصدار 23.0 من SPSS والإصدار 18 من Stata (StataCorp LP، كوليدج ستيشن، تكساس) للتحليل الإحصائي. تم إجراء تحليل التمييز الجزئي (PLS-DA) وتحليل خصائص التشغيل المستقبلية (ROC) في MetaboAnalyst V5 (https://www.metaboanalyst.ca/MetaboAnalyst/home.xhtml) وStata. تم استخدام اختبار كولموغوروف-سميرنوف واختبار شابيرو-ويلك لتحديد التوزيع الطبيعي للعينات. تم تقديم المتغيرات الاسمية كنسب وتكرارات. تم إجراء المقارنات بين مجموعتين باستخدام اختبارات t لطلاب غير المقترنة ذات الذيلين (التوزيع الطبيعي) أو اختبارات مان-ويتني U (التوزيع غير الطبيعي) للمتغيرات المستمرة أو اختبار فيشر الدقيق للمتغيرات الفئوية. ما لم يُذكر خلاف ذلك، تم تقييم المقارنات عبر مجموعات متعددة باستخدام ANOVA أحادي الاتجاه (التوزيع الطبيعي) تليها اختبار توكي بعد ذلك، أو اختبار كروسكال-واليس (التوزيع غير الطبيعي) تليه اختبار دن كما هو موضح في كل أسطورة الشكل. تم مقارنة المتغيرات المستمرة عبر الثلثيات من ImP بواسطة ANOVA أو اختبارات كروسكال واليس غير المعلمية حسب الاقتضاء، بينما تم تطبيق المتغيرات الاسمية باستخدام اختبار كوهرا-أرمتيج (المتغيرات الثنائية) واختبار جونكهير-تيربسترا (المتغير الترتيبي) للاتجاه. تم تطبيق المنحنيات المكعبة المقيدة (المنحنيات الطبيعية، RCS) مع 3 عقد عبر توزيع ImP لإنتاج منحنيات الاستجابة للجرعة ولتشكيل نموذج مرن للارتباط غير الخطي المحتمل لـ ImP المستمر مع النقاط النهائية الرئيسية، كما تم وصفه سابقًا (PMID: 29799975؛ PMID: 36049808). في كلا العينتين، تم تثبيت 3 عقد لـ RCS عند النسبة المئوية العشرية، الخمسين والتسعين من ImP
محفظة الطبيعة | ملخص التقرير أبريل 2023
التوزيع. تم اختيار عدد العقد بناءً على التحسين في احتمال اللوغاريتم بين نماذج الانحدار مع 3 – أو 4 – عقد RCS. لتعزيز الوضوح البصري، تم قطع الرسوم البيانية مع RCS عند القيم التي تتجاوز النسبة المئوية 95 من
بالنسبة لتحليل تسلسل جين 16S rRNA من كل من عينات الفئران والبشر، تم تنسيق التسلسلات ذات الطرفين، وتجميعها في وحدات تصنيف تشغيلية عند مستوى هوية >97%، وتم التعليق باستخدام إصدار SILVA v. 132 باستخدام DADA2 لبيانات الفئران وإصدار v. 138 وقواعد بيانات RDP الإصدار 18 باستخدام Mothur (v.1.40.5) لبيانات البشر.
لتقييم نمط النظام الغذائي، تم إجراء تحليل عامل في SPSS V.23.0 (SPSS Inc.، أرمونك، نيويورك، الولايات المتحدة الأمريكية) لتحديد الأبعاد الأساسية المشتركة (العوامل أو الأنماط) لاستهلاك الطعام من خلال اشتقاق تحميلات العوامل لكل مجموعة غذائية محددة مسبقًا. تم بعد ذلك تدوير العوامل باستخدام إجراء Varimax للحفاظ على العوامل غير المرتبطة. تم استخدام تحليل القيم الذاتية، ورسم بياني للانحدار، وقابلية تفسير حل العامل لدعم القرار النهائي بشأن الاحتفاظ بحل 5 عوامل، حيث كان لكل عامل قيمة ذاتية
بالنسبة لبيانات تسلسل RNA أحادية الخلية، تم إجراء معالجة بيانات التسلسل الخام من ملف FASTQ من كل منفذ باستخدام Cell Ranger (v6.1.2) مع المعلمات الافتراضية ومرجع جينوم الفئران mm 10 (GRCm38.p6) المقدم من 10x Genomics. تم تحميل مصفوفات العد الفريدة للمؤشرات الجزيئية (UMI) للخلايا المعتمدة الصالحة لكل منفذ إلى
بالنسبة لبيانات RNA-seq، تم رسم القراءات الناتجة على الجينوم المرجعي GRCm38.102 باستخدام STAR25 وتم تقدير مستويات التعبير الجيني باستخدام RSEM26. تم إجراء تحليلات إضافية في
البيانات
معلومات السياسة حول توفر البيانات
- رموز الوصول، معرفات فريدة، أو روابط ويب لمجموعات البيانات المتاحة للجمهور
- وصف لأي قيود على توفر البيانات
- بالنسبة لمجموعات البيانات السريرية أو بيانات الطرف الثالث، يرجى التأكد من أن البيان يتماشى مع سياستنا
nature portfolio | ملخص التقرير أبريل 2023
البحث الذي يشمل المشاركين البشريين، بياناتهم، أو المواد البيولوجية
التقرير عن الجنس والنوع
خصائص السكان
تتكون مجموعة IGT التي تم استخدامها للتحقق من النتائج في مجموعة مستقلة من الرجال والنساء الذين تتراوح أعمارهم بين 50-64 عامًا من منطقة غوتنبرغ (السويد). في هذه الحالة، شملت المجموعة 1844 مشاركًا من بينهم
تم إجراء هذه الدراسة في مجموعة فرعية من المشاركين من دراسة PESA (تقدم تصلب الشرايين تحت السريرية المبكرة)، وهي دراسة جماعية مستقبلية رصدية مستمرة لموظفين غير مصابين من أصل قوقازي في بنك سانتاندير في مدريد (PMID: 34238438). تم جمع المعلومات المتعلقة بالعرق ومجموعات اجتماعية ذات صلة أخرى لكل مشارك من خلال الإبلاغ الذاتي عبر استبيان ولم يتم استخدامها كعامل استبعاد. في دراسة IGT، تشمل المجموعة رجالًا ونساءً تتراوح أعمارهم بين 50-64 عامًا من منطقة غوتنبرغ (السويد)، تم تجنيدهم على أساس عشوائي من سجل السكان السويدي. من بين معايير الاستبعاد، تم تضمين الاضطراب المعرفي وعدم القدرة على فهم اللغة السويدية المكتوبة والم spoken وكذلك الأشخاص المولودين خارج السويد.
تتكون مجموعة دراسة IGT من رجال ونساء تتراوح أعمارهم بين 50-64 عامًا من منطقة غوتنبرغ (السويد)، تم تجنيدهم على أساس عشوائي من سجل السكان السويدي وشملوا بناءً على حالة الجلوكوز لديهم. تضمنت معايير الاستبعاد المطبقة: داء السكري المعروف، الأمراض الالتهابية (مثل مرض كرون، التهاب القولون التقرحي، الأمراض الروماتيزمية)، العلاج بالستيرويدات أو الأدوية المناعية، السرطان (ما لم يكن خاليًا من الانتكاس لمدة 5 سنوات سابقة)، الاضطراب المعرفي، العلاج للأمراض المعدية ومع المضادات الحيوية في الأشهر الثلاثة الماضية، عدم القدرة على فهم اللغة السويدية المكتوبة والم spoken وكذلك الأشخاص المولودين خارج السويد.
تم تقييم تصلب الشرايين في الشرايين السباتية باستخدام التصوير بالموجات فوق الصوتية وفقًا لبروتوكول موحد يتضمن جهاز تصوير بالموجات فوق الصوتية Siemens Acuson S2000 ومن خلال وجود الكالسيوم في الشريان التاجي الذي تم تقييمه بواسطة التصوير المقطعي المحوسب. تم قياس ضغط الدم الانقباضي (SBP) مرتين باستخدام جهاز تلقائي (Omron M10-IT، Omron Health care Co، كيوتو، اليابان) وتم استخدام متوسط القياسات. تم استخدام استبيان لجمع معلومات مفصلة حول الأدوية والتاريخ العائلي. تم جمع عينة دم وريدية
محفظة الطبيعة | ملخص التقرير أبريل 2023
التنوع المهني. علاوة على ذلك، قد يؤدي الإدراج الحصري للأفراد الإسبان في مجموعة PESA والأفراد السويديين في مجموعة IGT إلى إدخال تحيز جغرافي وسكاني. نتيجة لذلك، قد لا تمثل النتائج بشكل كامل طيف المخاطر القلبية الوعائية أو تقديم المرض الذي يُرى في السكان الأكثر تنوعًا عرقيًا واجتماعيًا أو غير الأوروبيين. في مجموعة IGT، على الرغم من أن التجنيد كان قائمًا على السكان، قد يكون استبعاد الأفراد غير المولودين في السويد وأولئك غير القادرين على فهم اللغة السويدية قد أدخل تحيزًا ثقافيًا أو اجتماعيًا إضافيًا.
وافقت لجنة الأخلاقيات البحثية في معهد كارلوس الثالث للصحة على بروتوكول الدراسة (CEI PI 32_2018) وتم تزويد جميع المشاركين بموافقة مستنيرة مكتوبة.
قدم جميع المشاركين موافقة مستنيرة، وتمت الموافقة على دراسة IGT من قبل مجلس مراجعة الأخلاقيات في غوتنبرغ (560-13)
يرجى ملاحظة أنه يجب أيضًا تقديم معلومات كاملة حول الموافقة على بروتوكول الدراسة في المخطوطة.
التقارير الخاصة بالمجال
علوم الحياة
العلوم السلوكية والاجتماعية
العلوم البيئية والتطورية والإيكولوجية
لنسخة مرجعية من الوثيقة مع جميع الأقسام، انظر nature.com/documents/nr-reporting-summary-flat.pdf
تصميم دراسة علوم الحياة
n : حجم العينة لكل مجموعة
SD: الانحراف المعياري (يفترض أنه متساوي بين المجموعات)
d: حجم التأثير (الفرق الأدنى القابل للاكتشاف بين متوسطات المجموعات)
بالنسبة لمعاملاتنا، سنحتاج إلى حوالي 15.7 فأرًا لكل مجموعة لتحقيق مستوى دلالة 0.05، وقوة 0.8، واكتشاف فرق لا يقل عن 1 انحراف معياري. نزيد حجم العينة بفأر واحد لنكون قادرين على استبعاد فأر حيث قد تفشل التقنية التجريبية. عادةً ما يكون الحجم الإجمالي للعينة حوالي 17 فأرًا لاختبارها في (على الأقل) تجربتين مستقلتين.
كانت الفئران متجانسة من حيث العمر (8 أسابيع) وتم تعيينها عشوائيًا إلى أقفاص مختلفة للعلاجات التجريبية (المضادات الحيوية، إيميدازول بروبيونات وإدارة AGN192403). تم تحليل عينات البشر والحيوانات بطريقة عشوائية على مطياف الكتلة.
بالنسبة للدراسات الحيوانية والدراسات الخلوية المعتمدة على المختبر، تم تعيين أرقام لكل عينة تجريبية وتم تعمية التحليل عن تخصيص المجموعة أثناء جمع البيانات.
بالنسبة للدراسة البشرية، تم جمع العينات بواسطة موظفين سريريين معميين وتم توليد جميع البيانات بواسطة باحثين كانوا معميين عن
البيانات الوصفية. بمجرد توليد البيانات، تم إجراء التحليل الحسابي مع جميع المعلومات السريرية اللازمة لاختبار الفروق بين المجموعات.
تصميم دراسة العلوم السلوكية والاجتماعية
| وصف الدراسة | وصف بإيجاز نوع الدراسة بما في ذلك ما إذا كانت البيانات كمية أو نوعية أو مختلطة (مثل: دراسة نوعية مقطعية، دراسة تجريبية كمية، دراسة حالة مختلطة). |
| عينة البحث | حدد عينة البحث (مثل: طلاب جامعة هارفارد، القرويين في الهند الريفية) وقدم معلومات ديموغرافية ذات صلة (مثل: العمر، الجنس) وأشر إلى ما إذا كانت العينة تمثل المجتمع. قدم مبررات لعينة الدراسة المختارة. بالنسبة للدراسات التي تتضمن مجموعات بيانات موجودة، يرجى وصف مجموعة البيانات والمصدر. |
| استراتيجية أخذ العينات | وصف إجراء أخذ العينات (مثل: عشوائي، كرة الثلج، طبقي، ملائم). وصف الأساليب الإحصائية التي تم استخدامها لتحديد حجم العينة مسبقًا.
|
| جمع البيانات | قدم تفاصيل حول إجراء جمع البيانات، بما في ذلك الأدوات أو الأجهزة المستخدمة لتسجيل البيانات (مثل القلم والورق، الكمبيوتر، جهاز تتبع العين، معدات الفيديو أو الصوت) وما إذا كان هناك أي شخص حاضر بخلاف المشاركين والباحث، وما إذا كان الباحث غير مدرك لحالة التجربة و/أو فرضية الدراسة أثناء جمع البيانات. |
| توقيت | حدد تواريخ بدء وانتهاء جمع البيانات. إذا كان هناك فجوة بين فترات الجمع، اذكر التواريخ لكل مجموعة عينة. |
| استثناءات البيانات | إذا لم يتم استبعاد أي بيانات من التحليلات، يرجى ذكر ذلك أو إذا تم استبعاد بيانات، يرجى تقديم العدد الدقيق للاستبعادات والأسباب وراءها، مع الإشارة إلى ما إذا كانت معايير الاستبعاد قد تم تحديدها مسبقًا. |
| عدم المشاركة | حدد عدد المشاركين الذين انسحبوا/رفضوا المشاركة والأسباب المقدمة لذلك
|
| التوزيع العشوائي | إذا لم يتم تخصيص المشاركين في مجموعات تجريبية، يرجى ذكر ذلك أو وصف كيفية تخصيص المشاركين في المجموعات، وإذا لم يكن التخصيص عشوائيًا، يرجى وصف كيفية التحكم في المتغيرات المشتركة. |
تصميم دراسة العلوم البيئية والتطورية والبيئية
| وصف الدراسة | وصف الدراسة بإيجاز. بالنسبة للبيانات الكمية، تشمل عوامل العلاج والتفاعلات، هيكل التصميم (مثل: عامل، متداخل، هرمي)، طبيعة وعدد الوحدات التجريبية والتكرارات. |
| عينة البحث | وصف عينة البحث (مثل مجموعة من طيور الدوري المنزلي المعلّمة، أو جميع نباتات ستينوسيريوس ثوربيري داخل نصب أنبوب الصبار الوطني)، وقدم مبررًا لاختيار العينة. عند الاقتضاء، وصف تصنيفات الكائنات، المصدر، الجنس، نطاق العمر وأي تعديلات. اذكر أي مجموعة سكانية من المفترض أن تمثلها العينة عند الاقتضاء. بالنسبة للدراسات التي تتضمن مجموعات بيانات موجودة، وصف البيانات ومصدرها. |
| استراتيجية أخذ العينات | يرجى ملاحظة إجراء أخذ العينات. وصف الطرق الإحصائية التي تم استخدامها لتحديد حجم العينة مسبقًا أو إذا لم يتم إجراء حساب لحجم العينة، وصف كيف تم اختيار أحجام العينات وتقديم مبرر لسبب كفاية هذه الأحجام. |
| جمع البيانات | وصف إجراء جمع البيانات، بما في ذلك من قام بتسجيل البيانات وكيف. |
| توقيت ومقياس مكاني | حدد تواريخ بدء وانتهاء جمع البيانات، مع الإشارة إلى تكرار ومدة أخذ العينات وتقديم مبرر لهذه الخيارات. إذا كان هناك فجوة بين فترات الجمع، اذكر التواريخ لكل مجموعة عينة. حدد النطاق المكاني الذي تم أخذ البيانات منه. |
| استثناءات البيانات | إذا لم يتم استبعاد أي بيانات من التحليلات، يرجى ذكر ذلك أو إذا تم استبعاد بيانات، يرجى وصف الاستبعادات والمبررات وراءها، مع الإشارة إلى ما إذا كانت معايير الاستبعاد قد تم تحديدها مسبقًا. |
| إعادة الإنتاج | وصف التدابير المتخذة للتحقق من قابلية تكرار النتائج التجريبية. لكل تجربة، اذكر ما إذا كانت هناك أي محاولات لتكرار التجربة قد فشلت أو اذكر أن جميع المحاولات لتكرار التجربة كانت ناجحة. |
| العشوائية | وصف كيفية تخصيص العينات/الكائنات/المشاركين إلى مجموعات. إذا لم يكن التخصيص عشوائيًا، فاشرح كيف تم التحكم في المتغيرات المشتركة. إذا لم يكن هذا ذا صلة بدراستك، فاشرح لماذا. |
| عمى | صف مدى استخدام التعمية أثناء جمع البيانات وتحليلها. إذا لم يكن من الممكن استخدام التعمية، فاشرح السبب أو اشرح لماذا لم تكن التعمية ذات صلة بدراستك. |
| هل شمل البحث العمل الميداني؟ □ نعم
|
| ظروف الميدان | وصف ظروف الدراسة للعمل الميداني، مع تقديم المعايير ذات الصلة (مثل: درجة الحرارة، هطول الأمطار). |
| الموقع | حدد موقع العينة أو التجربة، مع تقديم المعلمات ذات الصلة (مثل: خط العرض وخط الطول، الارتفاع، عمق الماء). |
| الوصول والاستيراد/التصدير | صف الجهود التي بذلتها للوصول إلى المواطن وجمع عيناتك واستيرادها/تصديرها بطريقة مسؤولة وامتثالاً للقوانين المحلية والوطنية والدولية، مع الإشارة إلى أي تصاريح تم الحصول عليها (اذكر اسم الجهة المصدرة، تاريخ الإصدار، وأي معلومات تعريفية). |
| اضطراب | صف أي إزعاج ناتج عن الدراسة وكيف تم تقليله. |
التقارير عن مواد وأنظمة وطرق محددة
| المواد والأنظمة التجريبية | طرق | |
| غير متوفر | مشارك في الدراسة | غير متوفر |
| □ | – الأجسام المضادة |  |
| □ | خطوط خلايا حقيقية النواة X | □ |
 |
□ علم الحفريات وعلم الآثار | إكس |
| □ | X الحيوانات وغيرها من الكائنات الحية | |
| □ | ||
| □ | ||
الأجسام المضادة
أجسام مضادة مضادة للفأر CDllc Brilliant Violet 650m; Biolegend; 117339; Clone N418 PE مضاد CDllc; BD bioscience; 553802; Clone HL3 PE مضاد للفأر NK-1.I; Biolegend; I56504; Clone S17016D
FITC مضاد للفأر CD3e; تونبو; 35-0031-U500; النسخة 145-2C11
مضاد الفأر B220-FITC؛ eBioscience؛ ll-0452-82؛ النسخة RA3-6B2
أجسام مضادة PerCP المضادة للفأر/البشر CD45R/B220؛ بايو ليجند؛ I03234؛ النسخة RA3-6B2
مضاد للبشر/الفئران CD45R (B220) PerCP-Cy5.5؛ eBioscience؛ 45-0452-82؛ النسخة RA3-6B2
أجسام مضادة مضادة للفأر/البشر CDllb Brilliant Violet 605m؛ Biolegend؛ I01237؛ النسخة M1/70
مضاد CDllb الفلوري 660؛ eBioscience؛ 50-0112-80؛ النسخة M1/70
مضاد CDIIb الفأري PerCP-Cyanine5.5؛ eBioscience؛ 45-0112-82؛ النسخة M1/70
مضاد CD206-eF450 للفأر؛ eBioscience؛ 48-2069-42؛ النسخة 19.2
أجسام مضادة PE مضادة للفأر CD31; بيو ليجند; | 02407; النسخة 390
مضاد CD4-BV570 للفئران؛ بايو ليجند؛ I00542؛ النسخة RM4-5
أجسام مضادة مضادة للفأر CD4 PE/Cy7؛ بيو ليجند؛ I00528؛ النسخة RM4-5
مضاد CD45 الفأري APC-eFluor 780; eBioscience; 47-0451-82; النسخة 30-F11
أجسام مضادة CD49b المضادة للفأر (خلايا NK الشاملة)؛ بايو ليجند؛ l08908؛ النسخة DX5
مضاد CD8 الفأري-PE; eBioscience; 12-0081-83; النسخة 53-6.7
أجسام مضادة أحادية النسيلة CD86 (B7-2) (GII)، PE-Cyanine7؛ eBioscience؛ 25-0862-80؛ النسخة GL1
CD90.2 فأر مضاد للفأر، BUV805، النسخة: 53-2.I؛ BD بيوساينس؛ BDB741908؛ النسخة 53-2.1
BV786 فأر مضاد للفأر CD90.2 النسخة 30-H12 (للاستخدام البحثي فقط)؛ BD Biosciences؛ 740841؛ النسخة 53-21
مضاد F4/80 مضاد للفأر 5 PerCP/CyS؛ بيو ليجند؛ 123128؛ النسخة BM8
مضاد فوكسب 3 لفأر/جرذ FITC FJK-16s; eBioscience; ll-5773-82; النسخة FJK-16s
مضاد الفأر Ly6C-BV711; بايوليجند; 128037; النسخة HK1.4
مضاد الماوس LY6G PE; BD فارماجين; 551461; النسخة 1A8
مضاد الفأر Ly6G من APC; BD PHARMINGEN; 560599; النسخة 1A8
مضاد-Ly6G v450; BD PHARMINGEN; 560603; النسخة 1A8
أليكسا فلور 700 مضاد للفأر I-A/1-E [M5/114.15.2]؛ بايوليجند؛ 107622؛ النسخة M5/114.15.2
مضاد MHCII للفأر-UV737; BD Biosciences; 367-5321-82; النسخة M5/114.15.2
بروتين الريبوسوم فوسفو-S6 (سير235/236) (D57.2.2E) XP
غاما (ت) APC; eBioscience; l 7-6981-80; Clone B2D
فأر مضاد لجرذان PE Rat Anti-Mouse Siglec-F Clone ES0-2440 (RUO)؛ BD PHARMINGEN؛ 552126؛ النسخة E50-2440
مضاد الماوس SiglecH FITC؛ eBioscience؛ II-0333-81؛ eBIO440c
مضاد الفأر T-bet-PECF594؛ BD Biosciences؛ 562467؛ النسخة O4-46
أجسام مضادة موصولة بألكسا فلور® 594 لمستقبل VCAM-1/CD106 للفئران؛ بيولوجيا التكنولوجيا؛ FAB6432T؛ النسخة 112734
أجسام مضادة XCRI المضادة للفأر/الجرذ Brilliant Violet 78S’M؛ Biolegend؛ 148225؛ النسخة ZET
https://www.biolegend.com/ar/products/brilliant-violet-650-anti-mouse-cd11c-antibody-8840
https://www.fishersci.com/shop/products/anti-cd11c-r-pe-clone-hl3bd/BDB553802
https://www.biolegend.com/ar/products/pe-anti-mouse-nk-1-1-antibody-16926?GroupID=GROUP20
https://www.citeab.com/antibodies/2204479-35-0031-u500-anti-mouse-cd3e
https://www.thermofisher.com/antibody/product/CD45R-B220-Antibody-clone-RA3-6B2-Monoclonal/11-0452-82
https://www.biolegend.com/fr-ch/search-results/percp-anti-mouse-human-cd45r-b220-antibody-4266?Clone=RA3-6B2
https://www.thermofisher.com/antibody/product/CD45R-B220-Antibody-clone-RA3-6B2-Monoclonal/45-0452-82
https://www.biolegend.com/ar/products/brilliant-violet-605-anti-mouse-human-cd11b-antibody-7637?GroupID=BLG10530
https://www.thermofisher.com/antibody/product/CD11b-Antibody-clone-M1-70-Monoclonal/50-0112-82
https://www.thermofisher.com/antibody/product/CD11b-Antibody-clone-M1-70-Monoclonal/45-0112-82
https://www.thermofisher.com/antibody/product/CD206-MMR-Antibody-clone-19-2-Monoclonal/48-2069-42
https://www.biolegend.com/nl-nl/products/pe-anti-mouse-cd31-antibody-122
https://www.biolegend.com/nl-be/products/brilliant-violet-570-anti-mouse-cd4-antibody-7379
https://www.biolegend.com/ar/فصل-الخلايا/أجسام-مضادة-سي-دي-4-مضاد-للفأر-بي-سيانين-7-1932
https://www.thermofisher.com/antibody/product/CD45-Antibody-clone-30-F11-Monoclonal/47-0451-82
https://www.biolegend.com/ar/products/pe-anti-mouse-cd49b-pan-nk-cells-antibody-234?GroupID=BLG4768
https://www.citeab.com/antibodies/2037994-12-0081-82-cd8a-monoclonal-antibody-53-6-7-pe-eb
https://www.thermofisher.com/antibody/product/CD86-B7-2-Antibody-clone-GL1-Monoclonal/25-0862-82
https://www.fishersci.com/shop/products/cd90-2-rat-anti-mouse-buv805-clone-53-2-1-bd-biosciences/BDB741908
https://www.biolegend.com/ar/products/percp-cyanine5-5-anti-mouse-f480-antibody-4303?GroupID=BLG5319
https://www.citeab.com/antibodies/2041744-11-5773-82-foxp3-monoclonal-antibody-fjk-16s-fitc
https://www.biolegend.com/ar/products/brilliant-violet-711-anti-mouse-ly-6c-antibody-8935?GroupID=BLG7242
https://www.citeab.com/antibodies/2410368-551461-bd-pharmingen-pe-rat-anti-mouse-ly-6g
https://www.citeab.com/antibodies/2410938-560599-bd-pharmingen-apc-rat-anti-mouse-ly-6g
https://www.citeab.com/antibodies/2408945-560603-bd-horizon-v450-rat-anti-mouse-ly-6g
https://www.biolegend.com/fr-ch/products/alexa-fluor-700-anti-mouse-i-a-i-e-antibody-3413
https://www.bdbiosciences.com/ar/products/reagents/flow-cytometry-reagents/research-reagents/single-color-antibodies-ruo/bv786-rat-anti-mouse-i-a-i-e. 742894
https://www.cellsignal.com/products/primary-antibodies/phospho-s6-ribosomal-protein-ser235-236-d57-2-2e-xp-rabbit-mab/4858 https://www.labome.com/product/Invitrogen/17-6981-80.html
https://www.citeab.com/antibodies/2411876-552126-bd-pharmingen-pe-rat-anti-mouse-siglec-f
https://www.labome.com/product/Invitrogen/11-0333-82.html
https://www.bdbiosciences.com/en-de/products/reagents/flow-cytometry-reagents/research-reagents/single-color-antibodies-ruo/بي-سي-إف594-ماوس-أنتي-تي-بيت. 562467
https://www.bio-techne.com/p/antibodies/mouse-vcam-1-cd106-alexa-fluor-594-conjugated-antibody-112734_fab6432t
https://www.biolegend.com/fr-ch/products/brilliant-violet-785-anti-mouse-rat-xcr1-antibody-16711
خطوط خلايا حقيقية النواة
معلومات السياسة حول خطوط الخلايا والجنس والنوع في البحث
مصدر(s) خط الخلية
تمت مراقبة جميع خطوط الخلايا بشكل مستمر واختبارها سلبياً لوجود تلوث بالميوكوبلازما بواسطة تفاعل البوليميراز المتسلسل (PCR).
□
المصادقة
تلوث الميكوبلازما
محفظة الطبيعة | ملخص التقرير أبريل 2023
أصل العينة
إيداع العينة
طرق التأريخ
□ ضع علامة في هذا المربع لتأكيد أن التواريخ الخام والمعايرة متاحة في الورقة أو في المعلومات التكميلية.
الإشراف الأخلاقي
الحيوانات وغيرها من الكائنات البحثية
الحيوانات المخبرية
فئران Rag1tm1Mom/J (مختبر جاكسون، سلالة 002216، Rag1-/-)؛ فئران B6.Cg-Rptortm1.1Dmsa/J (مختبر جاكسون، سلالة 013188، RaploxP) تم تزاوجها مع فئران B6.129P2-Lyz2tm1(cre)lfo/J (مختبر جاكسون، سلالة 004781، Lyz2-Cre) لإنتاج Lyz2
الحيوانات البرية
الإبلاغ عن الجنس
عينات تم جمعها من الميدان
الإشراف الأخلاقي
البيانات السريرية
معلومات السياسة حول الدراسات السريرية
تسجيل التجارب السريرية
تمت الموافقة على مجموعة IGT المستخدمة للتحقق من صحة من قبل مجلس مراجعة الأخلاقيات في غوتنبرغ (IGTM [560-13])
جمع البيانات
كانت معايير الاستبعاد لدراسة IGT هي: داء السكري المعروف، الأمراض الالتهابية (مثل مرض كرون، التهاب القولون التقرحي، الأمراض الروماتيزمية)، العلاج بالستيرويدات أو الأدوية المناعية، السرطان (ما لم يكن خاليًا من الانتكاس لمدة 5 سنوات سابقة)، الاضطراب المعرفي، العلاج للأمراض المعدية واستخدام المضادات الحيوية في الأشهر الثلاثة الماضية، عدم القدرة على فهم اللغة السويدية المكتوبة والم spoken وكذلك الأشخاص المولودين خارج السويد.
بالنسبة لمجموعة IGT، تم جمع عينات الدم من المشاركين تلاها قياس الطيف الكتلي المستهدف. تم تقييم تصلب الشرايين في الشرايين السباتية بواسطة التصوير بالموجات فوق الصوتية باستخدام بروتوكول موحد بما في ذلك جهاز الموجات فوق الصوتية Siemens Acuson S2000 ومن خلال وجود الكالسيوم في الشرايين التاجية الذي تم تقييمه بواسطة الأشعة المقطعية. تم قياس ضغط الدم الانقباضي (SBP) مرتين باستخدام جهاز تلقائي (Omron M10-IT، Omron Health care Co، كيوتو، اليابان) وتم استخدام متوسط القياسات. تم استخدام استبيان لجمع معلومات مفصلة عن الأدوية والتاريخ العائلي. تم حساب SCORE2 (تقييم المخاطر التاجية المنهجي) كما هو موضح سابقًا. تم قياس وزن الجسم والطول ومحيط الخصر ومحيط الورك باستخدام معدات معايرة مع ارتداء الأشخاص ملابس خفيفة بدون أحذية ووفقًا لتوصيات منظمة الصحة العالمية الحالية.
البحث المزدوج الاستخدام من القلق
معلومات السياسة حول البحث المزدوج الاستخدام من القلق
المخاطر
نعم
□
□ الصحة العامة
□
□ الأمن القومي
□
□ المحاصيل و/أو الماشية
□
□ النظم البيئية
□ □ أي مجال آخر مهم
التجارب المثيرة للقلق
لا
نعم
□ □ إظهار كيفية جعل لقاح غير فعال
□ □ منح مقاومة للمضادات الحيوية أو العوامل المضادة للفيروسات المفيدة علاجياً
□
□ تعزيز شدة مسببات الأمراض أو جعل مسببات غير ضارة ضارة
□
□ زيادة قابلية انتقال مسببات الأمراض
□
□ تغيير نطاق المضيف لمسبب المرض
□
□ تمكين التهرب من طرق التشخيص/الكشف
□
□ تمكين تسليح عامل بيولوجي أو سم
□
□ أي مجموعة أخرى من التجارب والمواد المحتملة الضارة
تسلسل شريحة الكروماتين
إيداع البيانات
□ تأكد من أنك قد قمت بإيداع أو توفير الوصول إلى ملفات الرسم البياني (مثل ملفات BED) للقيم المحددة.
روابط الوصول إلى البيانات
جلسة متصفح الجينوم (مثل UCSC)
المنهجية
نسخ
الأجسام المضادة
معلمات استدعاء القمة
برمجيات
وصف البرنامج المستخدم لجمع وتحليل بيانات ChIP-seq. بالنسبة للكود المخصص الذي تم إيداعه في مستودع مجتمعي، يرجى تقديم تفاصيل الوصول.
تدفق الخلايا
المؤامرات
أكد أن:
المقاييس على المحاور مرئية بوضوح. قم بتضمين الأرقام على المحاور فقط للرسم البياني في أسفل اليسار من المجموعة (المجموعة هي تحليل للعلامات المتطابقة).
جميع الرسوم البيانية هي رسوم بيانية متساوية الارتفاع مع نقاط شاذة أو رسوم بيانية بالألوان الزائفة.
تم توفير قيمة عددية لعدد الخلايا أو النسبة المئوية (مع الإحصائيات).
المنهجية
تحضير العينة
تم الاحتفاظ بالشرايين الأورطية في وسط دولبيكو المعدل Eagle (DMEM) البارد ليتم هضمها. تمت إزالة الدهون المحيطة بالأوعية الدموية وتم فتح الشريان الأورطي الصدري مع القوس طوليًا وقطعه إلى قطع أصغر تم حضنها لمدة 30 دقيقة عند 37 درجة مئوية في حمام مائي في محلول الهضم (كولاجيناز A).
فاصل خلايا FACSAria (بي دي بيوساينس)
برنامج FlowJo الإصدار 10 (TreeStar)
استراتيجية التصفية على أنسجة الدم والأبهر:
- FSC و SSC
- FSC-H و FSC-A للبوابة على مجموعة خلايا مفردة
- قابلية البوابة على الخلايا الحية، يتم تعريف ذلك على أنه خلايا سلبية للتلوين
- خلايا CD45+
- خلايا CD90 لخلايا T، وخلايا B220 لخلايا B، والخلايا غير T أو B كخلايا سلبية مزدوجة.
- تم تصنيف CD90-;B220-; بشكل إضافي على المونوسيتات عالية Ly6C
- تم تصنيف CD90-;B220-؛ بشكل إضافي على F4/80+CD86+ و F4/80+CD11c+ (البلاعم)
- تم تصنيف خلايا CD90 بشكل إضافي إلى CD4+Rorg-t+ (Th17)، CD4+t-Bet+ (Th1)، CD4+Foxp3+ (Treg)
- FSC و SSC
- FSC-H و FSC-A للدخول على مجموعة خلايا مفردة
- قابلية البوابة على الخلايا الحية، يتم تعريف ذلك على أنه خلايا سلبية للتلوين
- خلايا CD45+
- خلايا CD3-، B220-، NK1.1-، LY6G-، CD49-، SiglecF-
- تم تحديد المونوسيتات كـ CD11B+ و Ly6C+/-
التصوير بالرنين المغناطيسي
تصميم تجريبي
استحواذ
□ مستخدم
□ غير مستخدم
التحضير المسبق
قدم تفاصيل عن إصدار البرنامج ورقم المراجعة والمعلمات المحددة (النموذج/الوظائف، استخراج الدماغ، التقسيم، حجم نواة التنعيم، إلخ).
التطبيع
| قالب التطبيع | وصف القالب المستخدم للتطبيع/التحويل، مع تحديد مساحة الموضوع أو مساحة المجموعة الموحدة (مثل تاليراش الأصلية، MNI305، ICBM152) أو الإشارة إلى أن البيانات لم يتم تطبيعها. | |||
| إزالة الضوضاء والعيوب | وصف إجراءاتك لإزالة العيوب والضوضاء الهيكلية، مع تحديد معلمات الحركة، وإشارات الأنسجة، والإشارات الفسيولوجية (معدل ضربات القلب، التنفس). | |||
| تقييد الحجم | حدد برنامجك و/أو طريقتك ومعاييرك لتصفية الحجم، وبيّن مدى هذه التصفية. | |||
| النمذجة الإحصائية والاستدلال | ||||
| نوع النموذج والإعدادات | حدد النوع (وحيد المتغير الكتلي، متعدد المتغيرات، تحليل الأنماط المتكررة، التنبؤ، إلخ) واذكر التفاصيل الأساسية للنموذج في المستويين الأول والثاني (مثل: تأثيرات ثابتة، عشوائية أو مختلطة؛ انحراف أو ارتباط ذاتي). | |||
| التأثيرات المختبرة | حدد التأثير الدقيق من حيث شروط المهمة أو المحفز بدلاً من المفاهيم النفسية وأشر إلى ما إذا كانت ANOVA أو التصاميم العاملية قد استخدمت. | |||
| حدد نوع التحليل: | ||||
| نوع الإحصائيات للاستدلال (انظر إكلوند وآخرون 2016) | حدد على مستوى الفوكسل أو على مستوى الكتلة وقدم جميع المعلمات ذات الصلة لطرق الكتلة. | |||
| تصحيح | وصف نوع التصحيح وكيفية الحصول عليه للمقارنات المتعددة (مثل FWE، FDR، التبديل أو مونت كارلو). | |||
|
||||
| الاتصال الوظيفي و/أو الفعال | أبلغ عن مقاييس الاعتماد المستخدمة وتفاصيل النموذج (مثل ارتباط بيرسون، الارتباط الجزئي، المعلومات المتبادلة). | |||
| تحليل الرسوم البيانية | أبلغ عن المتغير التابع ومقياس الاتصال، مع تحديد الرسم البياني الموزون أو الرسم البياني الثنائي، على مستوى الموضوع أو المجموعة، والملخصات العالمية و/أو العقد المستخدمة (مثل معامل التجميع، الكفاءة، إلخ). | |||
| النمذجة متعددة المتغيرات والتحليل التنبؤي | حدد المتغيرات المستقلة، استخراج الميزات وتقليل الأبعاد، النموذج، مقاييس التدريب والتقييم. | |||
- مجموعة العمل SCORE2 وتعاون مخاطر القلب والأوعية الدموية ESC. خوارزميات توقع مخاطر SCORE2: نماذج جديدة لتقدير مخاطر الإصابة بأمراض القلب والأوعية الدموية على مدى 10 سنوات في أوروبا. Eur. Heart J. 42, 2439-2454 (2021).
- فرناندز-فرييرا، ل. وآخرون. انتشار، توزيع الأوعية، ومدى الأوعية المتعددة من تصلب الشرايين تحت السريرية في مجموعة متوسطة العمر. الدورة الدموية 131، 2104-2113 (2015).
- ليبي، ب. المشهد المتغير لتصلب الشرايين. الطبيعة 592، 524-533 (2021).
- بالنسبة لدراسة PESA، تم جمع عينات البلازما والبراز من ديسمبر 2018 إلى مايو 2021. تم جمع وتحليل بيانات الميتابولوميات في عام 2021. تم جمع وتحليل بيانات تسلسل جين 16S في عام 2022.
في مجموعة IGT، تم فحص الرجال والنساء الذين تتراوح أعمارهم بين 50 و64 عامًا والذين وُلِدوا في منطقة غوتنبرغ، السويد، بين عامي 2013
DOI: https://doi.org/10.1038/s41586-025-09263-w
PMID: https://pubmed.ncbi.nlm.nih.gov/40670786
Publication Date: 2025-07-16
Imidazole propionate is a driver and therapeutic target in atherosclerosis
Received: 1 June 2023
Accepted: 9 June 2025
Published online: 16 July 2025
Open access
Annalaura Mastrangelo ${ }^{mathbf{1 , 3 0}}$, Iñaki Robles-Vera ${ }^{mathbf{1 , 3 0}}$, Diego Mañanes ${ }^{mathbf{1 , 2}}$, Miguel Galán ${ }^{mathbf{1 , 2}}$, Marcos Femenía-Muiña ${ }^{1,3}$, Ana Redondo-Urzainqui ${ }^{1,2}$, Rafael Barrero-Rodríguez ${ }^{1,2}$, Eleftheria Papaioannou ${ }^{4,5,6}$, Joaquín Amores-Iniesta ${ }^{1}$, Ana Devesa ${ }^{1,7,8}$, Manuel Lobo-González ${ }^{1}$, Alba Carreras ${ }^{9}$, Katharina R. Beck ${ }^{9}$, Sophie Ivarsson ${ }^{9}$, Anders Gummesson ${ }^{9,10}$, Georgios Georgiopoulos ${ }^{11,12,13}$, Manuel Rodrigo-Tapias ${ }^{1,14}$, Sarai Martínez-Cano ${ }^{mathbf{1 , 1 4}}$, Ivan Fernández-López ${ }^{mathbf{1}}$, Vanessa Nuñez ${ }^{mathbf{1}}$, Alessia Ferrarini ${ }^{mathbf{1}}$, Naohiro Inohara ${ }^{15}$, Kimon Stamatelopoulos ${ }^{11}$, Alberto Benguría ${ }^{1}$, Danay Cibrian ${ }^{1,8,16}$, Francisco Sánchez-Madrid ${ }^{1,8,16,17}$, Vanesa Alonso-Herranz ${ }^{18}$, Ana Dopazo ${ }^{1,8}$, Coral Barbas ${ }^{19}$, Jesús Vázquez ${ }^{1,8}$, Juan Antonio López ${ }^{1,8}$, Alicia González-Martín ${ }^{4,5,6}$, Gabriel Nuñez ${ }^{15,19,20}$, Konstantinos Stellos ${ }^{21,22,23,24}$, Göran Bergström ${ }^{9,25}$, Fredrik Bäckhed ${ }^{9,25,26,27}$, Valentín Fuster ${ }^{1,28}$, Borja Ibañez ${ }^{1,8,29}$ & David Sancho ${ }^{1 boxtimes text { ® }}$
Abstract
Atherosclerosis is the main underlying cause of cardiovascular diseases. Its prevention is based on the detection and treatment of traditional cardiovascular risk factors
with early stages of atherosclerotic vascular disease that could contribute to atherosclerosis progression.
Untargeted metabolomics links ImP to atherosclerosis in mice

with atherosclerosis in a microbiota-dependent manner
ImP is linked to subclinical atherosclerosis in humans
(Fig. 2b). ImP was subsequently measured in an independent cohort, the impaired glucose tolerance (IGT) cohort (Supplementary Table 2a,b), comprising asymptomatic volunteers, including 529 controls and 1,315 individuals with subclinical atherosclerosis

show 95% confidence intervals. OR, odds ratio.h,j, Plasma ImP in individuals with inactive atherosclerosis (FDG
low-density lipoprotein (LDL)-cholesterol and hs-CRP in discriminating atherosclerosis prevalence in both cohorts (Extended Data Fig. 2c,d).
active atherosclerosis (

images of aorta (right). g, Heat map of GSEA comparing ImP versus control for endothelial cells, fibroblasts and macrophages from scRNA-seq data of Apoe
marrow activation (
ImP causes atherosclerosis without cholesterol change
and Extended Data Fig. 3c-e). In both models, ImP supplementation increased atherosclerosis development in the aorta and aortic root without affecting circulating cholesterol or glucose concentration (Fig. 3a,b and Extended Data Fig. 3a-e). Feeding with HC diet alone induced ImP production (Fig.1f). Additional ImP supplementation did not significantly exacerbate atherosclerosis in HC -fed
cell population frequency in the aorta (Extended Data Fig. 4e), without changes in circulating immune cells (Extended Data Fig. 4g,h). By contrast, scRNA-seq of aortas from mice treated with ImP for eight weeks revealed an increase in the relative number of fibroblasts, endothelial cells and immune cells (Fig. 3e). The increased number of immune cells in the aorta was confirmed by flow cytometry analysis (Extended Data Fig. 4i) and histological analysis of the aortic root, which showed increased T cell infiltration in the intima-media layer and increased MAC2 staining for inflammatory macrophages in the atheroma plaque of ImP-treated mice compared with controls (Extended Data Fig. 4j,k). Remarkably, the ability of ImP to induce atherosclerosis was markedly reduced in
In vivo ImP alters the aortic transcriptome
activates an mTOR-dependent inflammatory response in myeloid cells, contributing to atherogenesis.
ImP drives atherosclerosis via IIR in myeloid cells
To analyse the potential use of AGN192403 to prevent ImP-induced atherosclerosis we co-administered ImP and AGN192403 in drinking water to chow-fed Apoe
ImP-I1R blockade prevents atherosclerosis progression

in mice fed the HC diet (Extended Data Fig. 8e). Moreover, AGN192403 treatment prevented HC diet-dependent induction of blood Ly6C
cholesterol concentration in plasma, suggesting a novel approach for atherosclerosis therapy.
Discussion
Online content
5. Ridker, P. M. et al. Antiinflammatory therapy with canakinumab for atherosclerotic disease. N. Engl. J. Med. 377, 1119-1131 (2017).
6. Nelson, K., Fuster, V. & Ridker, P. M. Low-dose colchicine for secondary prevention of coronary artery disease. J. Am. Coll. Cardiol. 82, 648-660 (2023).
7. Sabatine, M. S. et al. Evolocumab and clinical outcomes in patients with cardiovascular disease. N. Engl. J. Med. 376, 1713-1722 (2017).
8. Kaasenbrood, L. et al. Distribution of estimated 10 -year risk of recurrent vascular events and residual risk in a secondary prevention population. Circulation 134, 1419-1429 (2016).
9. Chakaroun, R. M., Olsson, L. M. & Bäckhed, F. The potential of tailoring the gut microbiome to prevent and treat cardiometabolic disease. Nat. Rev. Cardiol. 20, 217-235 (2023).
10. Nemet, I. et al. Atlas of gut microbe-derived products from aromatic amino acids and risk of cardiovascular morbidity and mortality. Eur. Heart J. 44, 3085-3096 (2023).
11. Witkowski, M., Weeks, T. L. & Hazen, S. L. Gut microbiota and cardiovascular disease. Circ. Res. 127, 553-570 (2020).
12. Nemet, I. et al. A cardiovascular disease-linked gut microbial metabolite acts via adrenergic receptors. Cell 180, 862-877.e22 (2020).
13. Martínez-López, M. et al. Microbiota sensing by Mincle-Syk axis in dendritic cells regulates interleukin-17 and -22 production and promotes intestinal barrier integrity. Immunity 50, 446-461.e9 (2019).
14. Wang, Z. et al. Gut flora metabolism of phosphatidylcholine promotes cardiovascular disease. Nature 472, 57-63 (2011).
15. Koh, A. et al. Microbially produced imidazole propionate impairs insulin signaling through mTORC1. Cell 175, 947-961.e17 (2018).
16. Royer, P. et al. Plasma proteomics for prediction of subclinical coronary artery calcifications in primary prevention. Am. Heart J. 271, 55-67 (2024).
17. Molinaro, A. et al. Imidazole propionate is increased in diabetes and associated with dietary patterns and altered microbial ecology. Nat. Commun. 11, 5881 (2020).
18. Koren, O. et al. Human oral, gut, and plaque microbiota in patients with atherosclerosis. Proc. Natl Acad. Sci. 108, 4592-4598 (2011).
19. Trøseid, M., Andersen, G. Ø., Broch, K. & Hov, J. R. The gut microbiome in coronary artery disease and heart failure: current knowledge and future directions. eBioMedicine 52, 102649 (2020).
20. Fernández-Friera, L. et al. Vascular inflammation in subclinical atherosclerosis detected by hybrid PET/MRI. J. Am. Coll. Cardiol. 73, 1371-1382 (2019).
21. Devesa, A. et al. Bone marrow activation in response to metabolic syndrome and early atherosclerosis. Eur. Heart J. 43, 1809-1828 (2022).
22. Ma, J., Luo, J., Sun, Y. & Zhao, Z. Cytokines associated with immune response in atherosclerosis. Am. J. Transl. Res. 14, 6424-6444 (2022).
23. Gold, E. S. et al. ATF3 protects against atherosclerosis by suppressing 25-hydroxycholesterol-induced lipid body formation. J. Exp. Med. 209, 807-817 (2012).
24. Wculek, S. K., Dunphy, G., Heras-Murillo, I., Mastrangelo, A. & Sancho, D. Metabolism of tissue macrophages in homeostasis and pathology. Cell. Mol. Immunol. 19, 384-408 (2022).
25. Chen, J. Y., Cortes, C. & Ferreira, V. P. Properdin: a multifaceted molecule involved in inflammation and diseases. Mol. Immunol. 102, 58-72 (2018).
26. Kiss, M. G. & Binder, C. J. The multifaceted impact of complement on atherosclerosis. Atherosclerosis 351, 29-40 (2022).
27. Kan, H. et al. Single-cell transcriptome analysis reveals cellular heterogeneity in the ascending aortas of normal and high-fat diet-fed mice. Exp. Mol. Med. 53, 1379-1389 (2021).
28. Zhao, G. et al. Single-cell transcriptomics reveals endothelial plasticity during diabetic atherogenesis. Front. Cell Dev. Biol. 9, 689469 (2021).
29. Gocheva, V., Chen, X., Peters, C., Reinheckel, T. & Joyce, J. A. Deletion of cathepsin H perturbs angiogenic switching, vascularization and growth of tumors in a mouse model of pancreatic islet cell cancer. Biol. Chem. 391, 937-945 (2010).
30. Shih, S.-C. et al. The L6 protein TM4SF1 is critical for endothelial cell function and tumor angiogenesis. Cancer Res. 69, 3272-3277 (2009).
31. Schneller, D. et al. Cytokine-like 1 is a novel proangiogenic factor secreted by and mediating functions of endothelial progenitor cells. Circ. Res. 124, 243-255 (2019).
32. Dyer, L. A., Pi, X. & Patterson, C. The role of BMPs in endothelial cell function and dysfunction. Trends Endocrinol. Metab. 25, 472-480 (2014).
33. Aiello, R. J. et al. Monocyte chemoattractant protein-1 accelerates atherosclerosis in apolipoprotein E-deficient mice. Arterioscler. Thromb. Vasc. Biol. 19, 1518-1525 (1999).
34. Wang, L., Lawrence, J. C., Sturgill, T. W. & Harris, T. E. Mammalian target of rapamycin complex 1 (mTORC1) activity is associated with phosphorylation of raptor by mTOR. J. Biol. Chem. 284, 14693-14697 (2009).
35. Bousquet, P., Hudson, A., García-Sevilla, J. A. & Li, J.-X. Imidazoline receptor system: the past, the present, and the future. Pharmacol. Rev. 72, 50-79 (2020).
36. Head, G. A. & Mayorov, D. N. Imidazoline receptors, novel agents and therapeutic potential. Cardiovasc. Hematol. Agents Med. Chem. 4, 17-32 (2006).
37. Zhang, X. et al. High-protein diets increase cardiovascular risk by activating macrophage mTOR to suppress mitophagy. Nat. Metab. 2, 110-125 (2020).
38. Molinaro, A. et al. Microbially produced imidazole propionate is associated with heart failure and mortality. JACC Heart Fail. 11, 810-821 (2023).
39. Zhu, W. et al. Gut microbial metabolite TMAO enhances platelet hyperreactivity and thrombosis risk. Cell 165, 111-124 (2016).
40. Wang, Z. et al. Gut microbiota, circulating inflammatory markers and metabolites, and carotid artery atherosclerosis in HIV infection. Microbiome 11, 119 (2023).
41. van Son, J. et al. Plasma imidazole propionate is positively correlated with blood pressure in overweight and obese humans. Nutrients 13, 2706 (2021).
42. Trøseid, M. et al. Gut microbiota alterations and circulating imidazole propionate levels are associated with obstructive coronary artery disease in people with HIV. J. Infect. Dis. 229, 898-907 (2024).
43. Nageswaran, V. et al. Gut microbial metabolite imidazole propionate impairs endothelial cell function and promotes the development of atherosclerosis. Arterioscler. Thromb. Vasc. Biol. 45, 823-839 (2025).
44. Wüst, R. C. I. et al. Emerging magnetic resonance imaging techniques for atherosclerosis imaging. Arterioscler. Thromb. Vasc. Biol. 39, 841-849 (2019).
45. Gimbrone, M. A. & García-Cardeña, G. Endothelial cell dysfunction and the pathobiology of atherosclerosis. Circ. Res. 118, 620-636 (2016).
46. Roy, P., Orecchioni, M. & Ley, K. How the immune system shapes atherosclerosis: roles of innate and adaptive immunity. Nat. Rev. Immunol. 22, 251-265 (2022).
47. Finn, A. V., Nakano, M., Narula, J., Kolodgie, F. D. & Virmani, R. Concept of vulnerable/ unstable plaque. Arterioscler. Thromb. Vasc. Biol. 30, 1282-1292 (2010).
48. Wolf, D. & Ley, K. Immunity and inflammation in atherosclerosis. Circ. Res. 124, 315-327 (2019).
49. Zhang, X. et al. Identification of a leucine-mediated threshold effect governing macrophage mTOR signalling and cardiovascular risk. Nat. Metab. 6, 359-377 (2024).
50. Lloyd-Jones, D. M. et al. 2017 focused update of the 2016 ACC expert consensus decision pathway on the role of non-statin therapies for LDL-cholesterol lowering in the management of atherosclerotic cardiovascular disease risk: a report of the American College of Cardiology Task Force on Expert Consensus Decision Pathways. J. Am. Coll. Cardiol. 70, 1785-1822 (2017).
51. Gupta, A. et al. Adverse events associated with unblinded, but not with blinded, statin therapy in the Anglo-Scandinavian Cardiac Outcomes Trial-Lipid-Lowering Arm (ASCOT-LLA): a randomised double-blind placebo-controlled trial and its nonrandomised non-blind extension phase. Lancet 389, 2473-2481 (2017).
© The Author(s) 2025, modified publication 2025
Methods
Mice
Antibiotic administration
Quantification of lesions in the aorta
Aortic roots histology
Caecal content collection
Serum analysis
LC-MS untargeted metabolomics
and metabolites were eluted at
Flow cytometry analysis
elastase (
Study populations
aorta (points: 0, no;1, yes); (4) CAC score (points: 0, 0;1, 0.1-99;2, 100-399;3, 400-maximum). For the metabolomic analysis, peripheral blood samples collected after overnight fasting were centrifuged at
Targeted metabolomics
DNA extraction, 16S rDNA amplicon sequencing and microbiota data analysis
bioanalyzer DNA1000 chip (Agilent Technologies). The samples were pooled in equimolar amounts and sequenced with the MiSeq Reagent kit v.3, on a MiSeq Illumina platform (FISABIO sequencing service).
Dietary pattern assessment
scRNA-seq
number of UMI counts (
Cell lines
Bulk RNA-seq
Proteomics and phosphoproteomics by isobaric labelling
quantitative data was performed using
p-S6 staining by flow cytometry
Peritoneal macrophages collection
TNF measurement
MCP1 measurement
Transwell
Serum cytokines measure
ImP treatment in grafted mice
I1R siRNA silencing
Quantitative PCR with reverse transcription
Statistics
Reporting summary
Data availability
52. Rakoff-Nahoum, S., Paglino, J., Eslami-Varzaneh, F., Edberg, S. & Medzhitov, R. Recognition of commensal microflora by toll-like receptors is required for intestinal homeostasis. Cell 118, 229-241 (2004).
53. Centa, M., Ketelhuth, D. F. J., Malin, S. & Gisterå, A. Quantification of atherosclerosis in mice. J. Vis. Exp. https://doi.org/10.3791/59828 (2019).
54. Mastrangelo, A. et al. Insulin resistance in prepubertal obese children correlates with sex-dependent early onset metabolomic alterations. Int. J. Obes. 40, 1494-1502 (2016).
55. Lorenzo, C. et al. ALDH4A1 is an atherosclerosis auto-antigen targeted by protective antibodies. Nature 589, 287-292 (2021).
56. Pang, Z. et al. Using MetaboAnalyst 5.0 for LC-HRMS spectra processing, multi-omics integration and covariate adjustment of global metabolomics data. Nat. Protoc. 17, 1735-1761 (2022).
57. Gil-de-la-Fuente, A. et al. CEU Mass Mediator 3.0: a metabolite annotation tool. J. Proteome Res. 18, 797-802 (2019).
58. Barrero-Rodríguez, R. et al. TurboPutative: a web server for data handling and metabolite classification in untargeted metabolomics. Front. Mol. Biosci. 9, 952149 (2022).
59. Fernández-Ortiz, A. et al. The Progression and Early detection of Subclinical Atherosclerosis (PESA) study: rationale and design. Am. Heart J. 166, 990-998 (2013).
60. Sanguinetti, E. et al. Microbiome-metabolome signatures in mice genetically prone to develop dementia, fed a normal or fatty diet. Sci. Rep. 8, 4907 (2018).
61. Caruso, R., Ono, M., Bunker, M. E., Núñez, G. & Inohara, N. Dynamic and asymmetric changes of the microbial communities after cohousing in laboratory mice. Cell Rep. 27, 3401-3412.e3 (2019).
62. Kozich, J. J., Westcott, S. L., Baxter, N. T., Highlander, S. K. & Schloss, P. D. Development of a dual-index sequencing strategy and curation pipeline for analyzing amplicon sequence data on the MiSeq Illumina sequencing platform. Appl. Environ. Microbiol. 79, 5112-5120 (2013).
63. Callahan, B. J. et al. DADA2: high-resolution sample inference from Illumina amplicon data. Nat. Methods 13, 581-583 (2016).
64. Schloss, P. D. et al. Introducing mothur: open-source, platform-independent, communitysupported software for describing and comparing microbial communities. Appl. Environ. Microbiol. 75, 7537-7541 (2009).
65. de la Fuente-Arrillaga, C., Ruiz, Z. V., Bes-Rastrollo, M., Sampson, L. & Martinez-González, M. A. Reproducibility of an FFQ validated in Spain. Public Health Nutr. 13, 1364-1372 (2010).
66. Amezquita, R. A. et al. Orchestrating single-cell analysis with Bioconductor. Nat. Methods 17, 137-145 (2020).
67. Hao, Y. et al. Integrated analysis of multimodal single-cell data. Cell 184, 3573-3587.e29 (2021).
68. Boggy, G. J. et al. BFF and cellhashR: analysis tools for accurate demultiplexing of cell hashing data. Bioinformatics 38, 2791-2801 (2022).
69. Toral, M. et al. Carnitine palmitoyltransferase-1 up-regulation by PPAR-
70. Garaude, J. et al. Mitochondrial respiratory-chain adaptations in macrophages contribute to antibacterial host defense. Nat. Immunol. 17, 1037-1045 (2016).
71. Dobin, A. et al. STAR: ultrafast universal RNA-seq aligner. Bioinformatics 29, 15-21 (2013).
72. Li, B. & Dewey, C. N. RSEM: accurate transcript quantification from RNA-seq data with or without a reference genome. BMC Bioinformatics 12, 323 (2011).
73. Love, M. I., Huber, W. & Anders, S. Moderated estimation of fold change and dispersion for RNA-seq data with DESeq2. Genome Biol. 15, 550 (2014).
74. Gu, Z., Eils, R. & Schlesner, M. Complex heatmaps reveal patterns and correlations in multidimensional genomic data. Bioinformatics 32, 2847-2849 (2016).
75. Romero-Becerra, R. et al.
76. Rodríguez, J. M. et al. iSanXoT: a standalone application for the integrative analysis of mass spectrometry-based quantitative proteomics data. Comput. Struct. Biotechnol. J. 23, 452-459 (2024).
77. Subramanian, A. et al. Gene set enrichment analysis: a knowledge-based approach for interpreting genome-wide expression profiles. Proc. Natl Acad. Sci. USA 102, 15545-15550 (2005).
78. Stamatelopoulos, K. et al. Amyloid-
79. Stamatelopoulos, K. et al. Cathepsin S levels and survival among patients with non-ST-segment elevation acute coronary syndromes. J. Am. Coll. Cardiol. 80, 998-1010 (2022).
80. Wald, A. Sequential tests of statistical hypotheses. Ann. Math. Stat. 16, 117-186 (1945).
81. Hastie, T., Tibshirani, R. & Wainwright, M. Statistical Learning with Sparsity: The Lasso and Generalizations (Chapman & Hall, 2015).
82. DeLong, E. R., DeLong, D. M. & Clarke-Pearson, D. L. Comparing the areas under two or more correlated receiver operating characteristic curves: a nonparametric approach. Biometrics 44, 837-845 (1988).
Additional information
Correspondence and requests for materials should be addressed to David Sancho. Peer review information Nature thanks Soraya Taleb and the other, anonymous, reviewer(s) for their contribution to the peer review of this work. Peer reviewer reports are available.
Reprints and permissions information is available at http://www.nature.com/reprints.

c, Relative abundance of TMAO in plasma measured by untargeted LC-MS.

cohort: AUC


determined by flow cytometry.i, Total numbers of CD45

Article
(right) after ImP stimulation for 24 h with or without rapamycin co-incubation. 1, Flow cytometry staining for pS6 ribosomal protein. Left:BMDMn = 8; MEF CTR = 10; ImP=10; ImP + AGN = 9; Right: representative histograms for CTR (top), ImP (middle) and ImP + rapamycin (bottom). m,TNF production measured by ELISA. BMDM



staining of the aorta in female mice showing quantification of atherosclerotic lesion in the whole aorta (left), aortic arch (middle) and representative images (right). Chow
natureportfolio
David Sancho
Last updated by author(s): Jun 3, 2025
Reporting Summary
Statistics
n/a
Confirmed
□

□

□

Only common tests should be described solely by name; describe more complex techniques in the Methods section.
□

□

□

□

□ For Bayesian analysis, information on the choice of priors and Markov chain Monte Carlo settings

□ For hierarchical and complex designs, identification of the appropriate level for tests and full reporting of outcomes

□ Estimates of effect sizes (e.g. Cohen’s
Software and code
Data collection
Untargeted metabolomics data was collected by an Ultimate 3000 HPLC system coupled to a LTQ Orbitrap XL
Heatmap and all charts were generated using GraphPad Prism software v.9.0 (GraphPad Software, San Diego, CA). SPSS version 23.0 and Stata version 18 (StataCorp LP, College Station, TX) were used for statistical analysis. Partial least squares discriminant analysis (PLS-DA) and receiver operating characteristic (ROC) analyses were performed in MetaboAnalyst V5 (https://www.metaboanalyst.ca/MetaboAnalyst/ home.xhtml) and Stata. The Kolmogorov-Smirnov test and Shapiro-Wilk normality test were used to determine the normal distribution of samples. Nominal variables were presented as percentages and frequencies. Comparisons between two groups were performed using twotailed unpaired Student’s t-tests (normal distribution) or Mann-Whitney U-tests (non-normal distribution) for continous variables or Fisher exact test for categorical variables. Unless otherwise stated, comparisons across multiple groups were assessed using one-way ANOVA (normal distribution) followed by Tukey’s post hoc test, or Kruskal-Wallis test (non-normal distribution) followed by Dunn’s test as indicated in each figure legend. Continuous variables were compared across tertiles of ImP by ANOVA or non-parametric Kruskall Wallis tests as appropriate, whereas nominal variables by applying the Cohran-Armitage test (dichotomous variables) and the Jonckheere-Terpstra test (ordinal variable) for trend. Restricted cubic splines (natural splines, RCS) with 3 knots across ImP distribution were applied to produce a doseresponse curves and to flexibly model the potential nonlinear association of continuous ImP with the main endpoints, as previously described (PMID: 29799975; PMID: 36049808). In both cohorts, the 3 knots for RCS were fixed at the 10th, 50th and 90th percentile of the ImP
nature portfolio | reporting summary April2023
distribution. Number of knots was selected on the basis of improvement in log likelihood among regression models with 3 – or 4 -knot RCS. To enhance visual clarity, plots with RCS were truncated at values exceeding the 95th perecentile of the
For 16S rRNA gene sequencing analysis from both mice and human samples, the paired-end sequences were curated, binned into operational taxonomic units at >97% identity level, and annotated with SILVA release v. 132 using DADA2 for mice data and v. 138 and RDP version 18 databases using Mothur (v.1.40.5) for human data.
For dietary pattern assessment a factor analysis was performed in SPSS V.23.0 (SPSS Inc., Armonk, NY, USA) to identify common underlying dimensions (factors or patterns) of food consumption by deriving factor loadings for each predefined food group. Factors were subsequently rotated using a Varimax procedure to maintain uncorrelated factors. Analysis of eigenvalues, scree plot, and the interpretability of the factor solution were used to support a final decision on retaining a 5 -factor solution, where each factor had an eigenvalue
For single-cell RNA seq data raw sequencing data processing was performed from FASTQ file from each port using Cell Ranger (v6.1.2) with default parameters and the mm 10 (GRCm38.p6) mouse genome reference provided by 10x Genomics. Obtained raw unique molecular identifier (UMI) count matrices of valid barcoded cells for each port were loaded into
For RNA-seq resulted reads were mapped to the reference transcriptome GRCm38.102 using STAR25 and gene expression levels were estimated using RSEM26. Further analyses were performed in
Data
Policy information about availability of data
- Accession codes, unique identifiers, or web links for publicly available datasets
- A description of any restrictions on data availability
- For clinical datasets or third party data, please ensure that the statement adheres to our policy
nature portfolio | reporting summary April 2023
Research involving human participants, their data, or biological material
Reporting on sex and gender
Population characteristics
The IGT cohort that was used to validate the results in an independent cohort comprises men and women aged 50-64 years from the Gothenburg area (Sweden). In this case the cohort included 1844 participants of which
This study was conducted in a subset of participants from the PESA study (Progression of Early Subclinical Atherosclerosis), an ongoing observational prospective cohort study of caucasian asymptomatic employees of the Santander Bank in Madrid (PMID: 34238438). The information regarding race and other socially relevant groupings of each participant was collected by self-reporting via a questionnaire and was not used as exclusion factor. In the IGT study, the population includes men and women aged 50-64 years from the Gothenburg area (Sweden), recruited on a random basis from the Swedish population register. Among the exclusion criteria, cognitive dysfunction and inability to understand written and spoken Swedish as well as subjects born outside Sweden were included.
The IGT study population is composed of men and women aged 50-64 years from the Gothenburg area (Sweden), recruited on a random basis from the Swedish population register and included based on their glucose status. The applied exclusion criteria included: known diabetes, inflammatory diseases (e.g. Crohn’s disease, ulcerative colitis, rheumatic diseases), treatment with steroids or immunomodulatory drugs, cancer (unless relapse free for the preceding 5 years), cognitive dysfunction, treatment for infectious diseases and with antibiotics in the past three months, inability to understand written and spoken Swedish as well as subjects born outside Sweden.
Atherosclerosis in the carotid arteries was assessed by ultrasound imaging using a standardized protocol including a Siemens Acuson S2000 ultrasound scanner and by presence of coronary artery calcium assessed by CT scan. Systolic blood pressure (SBP) was measured twice with an automatic device (Omron M10-IT, Omron Health care Co, Kyoto, Japan) and the mean of the measurements was used. A questionnaire was used to collect detailed information on medication and family history. A venous blood sample
nature portfolio | reporting summary April2023
occupational diversity. Moreover, the exclusive inclusion of Spanish individuals in the PESA cohort and Swedish individuals in the IGT cohort may introduce geographic and population-related bias. As a result, the findings may not fully represent the spectrum of cardiovascular risk or disease presentation seen in more ethnically and socioeconomically diverse or nonEuropean populations. In the IGT cohort, although recruitment was population-based, the exclusion of non-Swedish-born individuals and those unable to understand Swedish may have introduced additional cultural or socioeconomic bias.
The Instituto de Salud Carlos III research ethics committee approved the study protocol (CEI PI 32_2018) and all participants were provided with a written informed consent.
All participants gave informed consent, and IGT study was approved by the Ethics Review Board in Gothenburg (560-13)
Note that full information on the approval of the study protocol must also be provided in the manuscript.
Field-specific reporting
Life sciences
Behavioural & social sciences
Ecological, evolutionary & environmental sciences
For a reference copy of the document with all sections, see nature.com/documents/nr-reporting-summary-flat.pdf
Life sciences study design
n : Sample size per group
SD: Standard deviation (assumed equal between groups)
d: Effect size (minimum detectable difference between group means)
For our parameters, we would need approximately 15.7 mice per group to achieve a significance level of 0.05 , a power of 0.8 , and detect at least a 1 standard deviation difference. We increase the sample group in 1 mouse to be able to discard a mouse where the experimental technique may fail. Total sample size is usually about 17 mice to be tested in (at least) two independent experiments.
Mice were homogeneous in terms of age ( 8 weeks) and randomly assigned to different cages for experimental treatments (antibiotics, imidazole propionate and AGN192403 administration). Human and animal samples were analysed in a randomized fashion on the mass spectrometer.
For animal studies and cell-based in vitro studies, numbers were assigned to each experimental sample and analysis was blinded to group allocation during data collection.
For human study, samples were collected by blinded clinical staff and all data were generated by investigators that were blinded to the
metadata. Once data were generated, computational analysis was performed with all of the necessary clinical information to test between groups.
Behavioural & social sciences study design
| Study description | Briefly describe the study type including whether data are quantitative, qualitative, or mixed-methods (e.g. qualitative cross-sectional, quantitative experimental, mixed-methods case study). |
| Research sample | State the research sample (e.g. Harvard university undergraduates, villagers in rural India) and provide relevant demographic information (e.g. age, sex) and indicate whether the sample is representative. Provide a rationale for the study sample chosen. For studies involving existing datasets, please describe the dataset and source. |
| Sampling strategy | Describe the sampling procedure (e.g. random, snowball, stratified, convenience). Describe the statistical methods that were used to predetermine sample size
|
| Data collection | Provide details about the data collection procedure, including the instruments or devices used to record the data (e.g. pen and paper, computer, eye tracker, video or audio equipment) whether anyone was present besides the participant(s) and the researcher, and whether the researcher was blind to experimental condition and/or the study hypothesis during data collection. |
| Timing | Indicate the start and stop dates of data collection. If there is a gap between collection periods, state the dates for each sample cohort. |
| Data exclusions | If no data were excluded from the analyses, state so OR if data were excluded, provide the exact number of exclusions and the rationale behind them, indicating whether exclusion criteria were pre-established. |
| Non-participation | State how many participants dropped out/declined participation and the reason(s) given
|
| Randomization | If participants were not allocated into experimental groups, state so OR describe how participants were allocated to groups, and if allocation was not random, describe how covariates were controlled. |
Ecological, evolutionary & environmental sciences study design
| Study description | Briefly describe the study. For quantitative data include treatment factors and interactions, design structure (e.g. factorial, nested, hierarchical), nature and number of experimental units and replicates. |
| Research sample | Describe the research sample (e.g. a group of tagged Passer domesticus, all Stenocereus thurberi within Organ Pipe Cactus National Monument), and provide a rationale for the sample choice. When relevant, describe the organism taxa, source, sex, age range and any manipulations. State what population the sample is meant to represent when applicable. For studies involving existing datasets, describe the data and its source. |
| Sampling strategy | Note the sampling procedure. Describe the statistical methods that were used to predetermine sample size OR if no sample-size calculation was performed, describe how sample sizes were chosen and provide a rationale for why these sample sizes are sufficient. |
| Data collection | Describe the data collection procedure, including who recorded the data and how. |
| Timing and spatial scale | Indicate the start and stop dates of data collection, noting the frequency and periodicity of sampling and providing a rationale for these choices. If there is a gap between collection periods, state the dates for each sample cohort. Specify the spatial scale from which the data are taken |
| Data exclusions | If no data were excluded from the analyses, state so OR if data were excluded, describe the exclusions and the rationale behind them, indicating whether exclusion criteria were pre-established. |
| Reproducibility | Describe the measures taken to verify the reproducibility of experimental findings. For each experiment, note whether any attempts to repeat the experiment failed OR state that all attempts to repeat the experiment were successful. |
| Randomization | Describe how samples/organisms/participants were allocated into groups. If allocation was not random, describe how covariates were controlled. If this is not relevant to your study, explain why. |
| Blinding | Describe the extent of blinding used during data acquisition and analysis. If blinding was not possible, describe why OR explain why blinding was not relevant to your study. |
| Did the study involve field work? □ Yes
|
| Field conditions | Describe the study conditions for field work, providing relevant parameters (e.g. temperature, rainfall). |
| Location | State the location of the sampling or experiment, providing relevant parameters (e.g. latitude and longitude, elevation, water depth). |
| Access & import/export | Describe the efforts you have made to access habitats and to collect and import/export your samples in a responsible manner and in compliance with local, national and international laws, noting any permits that were obtained (give the name of the issuing authority, the date of issue, and any identifying information). |
| Disturbance | Describe any disturbance caused by the study and how it was minimized. |
Reporting for specific materials, systems and methods
| Materials & experimental systems | Methods | |
| n/a | Involved in the study | n/a |
| □ | – Antibodies |  |
| □ | X Eukaryotic cell lines | □ |
 |
□ Palaeontology and archaeology | X |
| □ | X Animals and other organisms | |
| □ | ||
| □ | ||
Antibodies
Brilliant Violet 650m anti-mouse CDllc Antibody ;Biolegend;117339; Clone N418 PE anti CDllc;BD bioscience;553802; Clone HL3 PE anti-mouse NK-1.I Antibody ;Biolegend;I56504; Clone S17016D
FITC Anti-Mouse CD3e;Tonbo;35-0031-U500; clone 145-2C11
Anti-mouse B220-FITC;eBioscience;ll-0452-82; clone RA3-6B2
PerCP anti-mouse/human CD45R/B220 Antibody;Biolegend;I03234; Clone RA3-6B2
Anti-Human/Mouse CD45R (B220) PerCP-Cy5.5;eBioscience;45-0452-82; clone RA3-6B2
Brilliant Violet 605m anti-mouse/human CDllb Antibody;Biolegend;I01237; Clone M1/70
anti-mouse CDllb eFluor 660;eBioscience;50-0112-80; Clone M1/70
Anti-Mouse CDIIb PerCP-Cyanine5.5;eBioscience;45-0112-82; Clone M1/70
Anti-mouse CD206-eF450 ;eBioscience;48-2069-42; Clone 19.2
PE anti-mouse CD31 Antibody;Biolegend;|02407; Clone 390
Anti-mouse CD4-BV570;Biolegend;I00542; Clone RM4-5
PE/Cy7 anti-mouse CD4 Antibody;Biolegend;I00528; Clone RM4-5
Anti-Mouse CD45 APC-eFluor” 780;eBioscience;47-0451-82; Clone 30-F11
PE anti-mouse CD49b (pan-NK cells) Antibody;Biolegend;l08908; Clone DX5
Anti-mouse CD8-PE;eBioscience;12-0081-83; Clone 53-6.7
CD86 (B7-2) Monoclonal Antibody (GII), PE-Cyanine7;eBioscience;25-0862-80; Clone GL1
CD90.2 Rat anti-Mouse, BUV805, Clone: 53-2.I;BD Biosciences;BDB741908; Clone 53-2.1
BV786 Rat Anti-Mouse CD90.2 Clone 30-H12 (RUO);BD Biosciences; 740841; Clone 53-21
PerCP/CyS. 5 anti-mouse F4/80 Antibody;Biolegend;123128; Clone BM8
anti-mouse/rat Foxp3 FITC FJK-16s;eBioscience;ll-5773-82; Clone FJK-16s
Anti-mouse Ly6C-BV711;Biolegend;128037 ; Clone HK1.4
ANTI-MOUSE LY6G PE ;BD PHARMINGEN;551461; Clone 1A8
APC rat anti-Mouse Ly6G ;BD PHARMINGEN;560599; Clone 1A8
anti-Ly6G v450;BD PHARMINGEN;560603; Clone 1A8
Alexa Fluor” 700 anti-mouse I-A/1-E [M5/114.15.2];Biolegend; 107622 ; Clone M5/114.15.2
Anti-mouse MHCII-UV737;BD Biosciences;367-5321-82; Clone M5/114.15.2
Phospho-S6 Ribosomal Protein (Ser235/236) (D57.2.2E) XP
gamma (t) APC;eBioscience;l 7-6981-80 ; Clone B2D
PE Rat Anti-Mouse Siglec-F Clone ES0-2440 (RUO);BD PHARMINGEN;552126; Clone E50-2440
Anti-mouse SiglecH FITC;eBioscience;II-0333-81; eBIO440c
Anti-mouse T-bet-PECF594;BD Biosciences;562467; Clone O4-46
Mouse VCAM-1/CD106 Alexa Fluor® 594-conjugated Antibody;biotechne;FAB6432T; Clone 112734
Brilliant Violet 78S’M anti-mouse/rat XCRI Antibody;Biolegend;148225; Clone ZET
https://www.biolegend.com/en-gb/products/brilliant-violet-650-anti-mouse-cd11c-antibody-8840
https://www.fishersci.com/shop/products/anti-cd11c-r-pe-clone-hl3bd/BDB553802
https://www.biolegend.com/en-us/products/pe-anti-mouse-nk-1-1-antibody-16926?GroupID=GROUP20
https://www.citeab.com/antibodies/2204479-35-0031-u500-anti-mouse-cd3e
https://www.thermofisher.com/antibody/product/CD45R-B220-Antibody-clone-RA3-6B2-Monoclonal/11-0452-82
https://www.biolegend.com/fr-ch/search-results/percp-anti-mouse-human-cd45r-b220-antibody-4266?Clone=RA3-6B2
https://www.thermofisher.com/antibody/product/CD45R-B220-Antibody-clone-RA3-6B2-Monoclonal/45-0452-82
https://www.biolegend.com/en-us/products/brilliant-violet-605-anti-mouse-human-cd11b-antibody-7637?GroupID=BLG10530
https://www.thermofisher.com/antibody/product/CD11b-Antibody-clone-M1-70-Monoclonal/50-0112-82
https://www.thermofisher.com/antibody/product/CD11b-Antibody-clone-M1-70-Monoclonal/45-0112-82
https://www.thermofisher.com/antibody/product/CD206-MMR-Antibody-clone-19-2-Monoclonal/48-2069-42
https://www.biolegend.com/nl-nl/products/pe-anti-mouse-cd31-antibody-122
https://www.biolegend.com/nl-be/products/brilliant-violet-570-anti-mouse-cd4-antibody-7379
https://www.biolegend.com/en-us/cell-separation/pe-cyanine7-anti-mouse-cd4-antibody-1932
https://www.thermofisher.com/antibody/product/CD45-Antibody-clone-30-F11-Monoclonal/47-0451-82
https://www.biolegend.com/en-gb/products/pe-anti-mouse-cd49b-pan-nk-cells-antibody-234?GroupID=BLG4768
https://www.citeab.com/antibodies/2037994-12-0081-82-cd8a-monoclonal-antibody-53-6-7-pe-eb
https://www.thermofisher.com/antibody/product/CD86-B7-2-Antibody-clone-GL1-Monoclonal/25-0862-82
https://www.fishersci.com/shop/products/cd90-2-rat-anti-mouse-buv805-clone-53-2-1-bd-biosciences/BDB741908
https://www.biolegend.com/en-us/products/percp-cyanine5-5-anti-mouse-f480-antibody-4303?GroupID=BLG5319
https://www.citeab.com/antibodies/2041744-11-5773-82-foxp3-monoclonal-antibody-fjk-16s-fitc
https://www.biolegend.com/en-us/products/brilliant-violet-711-anti-mouse-ly-6c-antibody-8935?GroupID=BLG7242
https://www.citeab.com/antibodies/2410368-551461-bd-pharmingen-pe-rat-anti-mouse-ly-6g
https://www.citeab.com/antibodies/2410938-560599-bd-pharmingen-apc-rat-anti-mouse-ly-6g
https://www.citeab.com/antibodies/2408945-560603-bd-horizon-v450-rat-anti-mouse-ly-6g
https://www.biolegend.com/fr-ch/products/alexa-fluor-700-anti-mouse-i-a-i-e-antibody-3413
https://www.bdbiosciences.com/en-ca/products/reagents/flow-cytometry-reagents/research-reagents/single-color-antibodies-ruo/ bv786-rat-anti-mouse-i-a-i-e. 742894
https://www.cellsignal.com/products/primary-antibodies/phospho-s6-ribosomal-protein-ser235-236-d57-2-2e-xp-rabbit-mab/4858 https://www.labome.com/product/Invitrogen/17-6981-80.html
https://www.citeab.com/antibodies/2411876-552126-bd-pharmingen-pe-rat-anti-mouse-siglec-f
https://www.labome.com/product/Invitrogen/11-0333-82.html
https://www.bdbiosciences.com/en-de/products/reagents/flow-cytometry-reagents/research-reagents/single-color-antibodies-ruo/ pe-cf594-mouse-anti-t-bet. 562467
https://www.bio-techne.com/p/antibodies/mouse-vcam-1-cd106-alexa-fluor-594-conjugated-antibody-112734_fab6432t
https://www.biolegend.com/fr-ch/products/brilliant-violet-785-anti-mouse-rat-xcr1-antibody-16711
Eukaryotic cell lines
Policy information about cell lines and Sex and Gender in Research
Cell line source(s)
All cells lines were continuously surveyed and tested negative for mycoplasma contamination by PCR.
□
Authentication
Mycoplasma contamination
nature portfolio | reporting summary April 2023
Specimen provenance
Specimen deposition
Dating methods
□ Tick this box to confirm that the raw and calibrated dates are available in the paper or in Supplementary Information.
Ethics oversight
Animals and other research organisms
Laboratory animals
Rag1tm1Mom/J mice (The Jackson Laboratory, strain 002216, Rag1-/-); B6.Cg-Rptortm1.1Dmsa/J mice (The Jackson Laboratory, strain 013188, RaploxP) were mated with B6.129P2-Lyz2tm1(cre)lfo/J mice (The Jackson Laboratory, strain 004781, Lyz2-Cre) to generate Lyz2
Wild animals
Reporting on sex
Field-collected samples
Ethics oversight
Clinical data
Policy information about clinical studies
Clinical trial registration
The IGT cohort used for the validation was approved by Ethics Review Board in Gothenburg (IGTM [560-13])
Data collection
Exclusion criteria for the IGT study were: known diabetes, inflammatory diseases (e.g. Crohn’s disease, ulcerative colitis, rheumatic diseases), treatment with steroids or immunomodulatory drugs, cancer (unless relapse free for the preceding 5 years), cognitive dysfunction, treatment for infectious diseases and with antibiotics in the past three months, inability to understand written and spoken Swedish as well as subjects born outside Sweden.
For the IGT cohort blood samples were collected from the participants followed by targeted mass spectrometry. Atherosclerosis in the carotid arteries was assessed by ultrasound imaging using a standardized protocol including a Siemens Acuson S2000 ultrasound scanner and by presence of coronary artery calcium assessed by CT scan. Systolic blood pressure (SBP) was measured twice with an automatic device (Omron M10-IT, Omron Health care Co, Kyoto, Japan) and the mean of the measurements was used. A questionnaire was used to collect detailed information on medication and family history. SCORE2 (Systematic Coronary Risk Evaluation) was calculated as previously. Body weight, height waist and hip circumferences were measured on calibrated equipment with subjects dressed in light clothing without shoes and according to current WHO recommendations.
Dual use research of concern
Policy information about dual use research of concern
Hazards
Yes
□
□ Public health
□
□ National security
□
□ Crops and/or livestock
□
□ Ecosystems
□ □ Any other significant area
Experiments of concern
No
Yes
□ □ Demonstrate how to render a vaccine ineffective
□ □ Confer resistance to therapeutically useful antibiotics or antiviral agents
□
□ Enhance the virulence of a pathogen or render a nonpathogen virulent
□
□ Increase transmissibility of a pathogen
□
□ Alter the host range of a pathogen
□
□ Enable evasion of diagnostic/detection modalities
□
□ Enable the weaponization of a biological agent or toxin
□
□ Any other potentially harmful combination of experiments and agents
ChIP-seq
Data deposition
□ Confirm that you have deposited or provided access to graph files (e.g. BED files) for the called peaks.
Data access links
Genome browser session (e.g. UCSC)
Methodology
Replicates
Antibodies
Peak calling parameters
Software
Describe the software used to collect and analyze the ChIP-seq data. For custom code that has been deposited into a community repository, provide accession details.
Flow Cytometry
Plots
Confirm that:
The axis scales are clearly visible. Include numbers along axes only for bottom left plot of group (a ‘group’ is an analysis of identical markers).
All plots are contour plots with outliers or pseudocolor plots.
A numerical value for number of cells or percentage (with statistics) is provided.
Methodology
Sample preparation
The aortas were kept in cold Dulbecco’s Modified Eagle Medium (DMEM) to be digested. Perivascular fat was removed and the thoracic aorta with arch was opened longitudinally and cut into smaller pieces that were incubated for 30 min at 37 oC in water bath in digestion buffer (Collagenase A
FACSAria Cell Sorter (BD Biosciences)
FlowJo software version 10 (TreeStar)
Gating strategy on blood and aorta tissues:
- FSC and SSC
- FSC-H and FSC-A to gate on single cell population
- Viability to gate on live cells, this is defined as negative cells to staining
- CD45+ cells
- CD90 cells for T cells, B220 cell for B cells and non t or b cells as double negative.
- In CD90-;B220-; was further gated on Ly6C high Monocytes
- In CD90-;B220-; was further gated on F4/80+CD86+ and F4/80+CD11c+ (Macrophages)
- CD90 cells was further gated CD4+Rorg-t+ (Th17), CD4+t-Bet+ (Th1), CD4+Foxp3+ (Treg)
- FSC and SSC
- FSC-H and FSC-A to gate on single cell population
- Viability to gate on live cells, this is defined as negative cells to staining
- CD45+ cells
- CD3-, B220-, NK1.1-, LY6G-, CD49-, SiglecF- cells
- Monocytes was gated as CD11B+ and Ly6C+/-
Magnetic resonance imaging
Experimental design
Acquisition
□ Used
□ Not used
Preprocessing
Provide detail on software version and revision number and on specific parameters (model/functions, brain extraction, segmentation, smoothing kernel size, etc.).
Normalization
| Normalization template | Describe the template used for normalization/transformation, specifying subject space or group standardized space (e.g. original Talairach, MNI305, ICBM152) OR indicate that the data were not normalized. | |||
| Noise and artifact removal | Describe your procedure(s) for artifact and structured noise removal, specifying motion parameters, tissue signals and physiological signals (heart rate, respiration). | |||
| Volume censoring | Define your software and/or method and criteria for volume censoring, and state the extent of such censoring. | |||
| Statistical modeling & inference | ||||
| Model type and settings | Specify type (mass univariate, multivariate, RSA, predictive, etc.) and describe essential details of the model at the first and second levels (e.g. fixed, random or mixed effects; drift or auto-correlation). | |||
| Effect(s) tested | Define precise effect in terms of the task or stimulus conditions instead of psychological concepts and indicate whether ANOVA or factorial designs were used. | |||
| Specify type of analysis: | ||||
| Statistic type for inference (See Eklund et al. 2016) | Specify voxel-wise or cluster-wise and report all relevant parameters for cluster-wise methods. | |||
| Correction | Describe the type of correction and how it is obtained for multiple comparisons (e.g. FWE, FDR, permutation or Monte Carlo). | |||
|
||||
| Functional and/or effective connectivity | Report the measures of dependence used and the model details (e.g. Pearson correlation, partial correlation, mutual information). | |||
| Graph analysis | Report the dependent variable and connectivity measure, specifying weighted graph or binarized graph, subject- or group-level, and the global and/or node summaries used (e.g. clustering coefficient, efficiency, etc.). | |||
| Multivariate modeling and predictive analysis | Specify independent variables, features extraction and dimension reduction, model, training and evaluation metrics. | |||
- SCORE2 working group and ESC Cardiovascular risk collaboration. SCORE2 risk prediction algorithms: new models to estimate 10-year risk of cardiovascular disease in Europe. Eur. Heart J. 42, 2439-2454 (2021).
- Fernández-Friera, L. et al. Prevalence, vascular distribution, and multiterritorial extent of subclinical atherosclerosis in a middle-aged cohort. Circulation 131, 2104-2113 (2015).
- Libby, P. The changing landscape of atherosclerosis. Nature 592, 524-533 (2021).
- For the PESA study, plasma and faecal samples were collected from December 2018 to May 2021. Metabolomics data were collected and analysed in 2021. 16S gene sequencing data was collected and analysed in 2022.
In the IGT cohort, men and women aged 50 to 64 years and born in the region of Gothenburg, Sweden, were screened between 2013
