المجلة: New Phytologist، المجلد: 242، العدد: 4
DOI: https://doi.org/10.1111/nph.19541
PMID: https://pubmed.ncbi.nlm.nih.gov/38297461
تاريخ النشر: 2024-01-31
DOI: https://doi.org/10.1111/nph.19541
PMID: https://pubmed.ncbi.nlm.nih.gov/38297461
تاريخ النشر: 2024-01-31
السنة: 2024
التعايش الفطري الجذري: آفاق البحث في الجينوميات، والبيئة، وتطبيقات الزراعة
معرف الوثيقة الرقمي: https://doi.org/10.1111/nph. 19541
مراجعة تانزلي
التعايش الفطري الجذري: آفاق البحث في الجينوميات، والبيئة، وتطبيقات الزراعة
المؤلفون للتواصل:
فرانسيس مارتن
البريد الإلكتروني: francis.martin@inrae.fr
مارسيل ج. أ. فان دير هايدن
البريد الإلكتروني: marcel.vanderheijden@agroscope.admin.ch
فرانسيس مارتن
البريد الإلكتروني: francis.martin@inrae.fr
مارسيل ج. أ. فان دير هايدن
البريد الإلكتروني: marcel.vanderheijden@agroscope.admin.ch
المحتويات
| ملخص | 1 | السادس: التعايش الفطري الجذري وديناميات مجتمع النباتات | 11 | ||
| المقدمة | 2 | السابع. تطبيقات الفطريات الجذرية | 12 | ||
| ثانياً. استغلال الجينوميات لتعزيز فهمنا لتطور ووظيفة التعايش الفطري | ٣ |
|
12 | ||
| III. الشبكات الفطرية | ٨ | الشكر والتقدير | 15 | ||
| الرابع. رسم خرائط تنوع الفطريات الجذرية | 9 | المراجع | 15 | ||
| V. التعايش الفطري في النظم البيئية | 10 |
نيو فيتولوجيست (2024)
doi: 10.1111/nph. 19541
doi: 10.1111/nph. 19541
الكلمات الرئيسية: الفطريات الميكوريزية الجذرية، التنوع البيولوجي، الفطريات الميكوريزية الخارجية، الفطريات الميكوريزية الإريكية، التعايش، الفطريات الميكوريزية الأوركيدية، تفاعلات النبات والميكروبات، علم الوراثة السكانية.
ملخص
تعتبر التعايشات الفطرية الجذرية بين النباتات والفطريات ضرورية لهيكل التربة، ودورة المغذيات، وتنوع النباتات، واستدامة النظام البيئي. أكثر من 250000 نوع من النباتات مرتبط بالفطريات الجذرية. لقد أحدثت التقدمات الأخيرة في علم الجينوم والأساليب ذات الصلة ثورة في فهمنا لبيولوجيا وبيئة التعايشات الجذرية. تم إصدار جينومات أكثر من 250 فطرًا جذرًا وتم تحديد مئات الجينات التي تلعب أدوارًا محورية في تنظيم تطوير التعايش والتمثيل الغذائي. توفر تقنيات تحليل الحمض النووي الريبوزي (rDNA) وتحليل التعبير الجيني (metatranscriptomics) رؤى جديدة حول الإشارات البيئية التي تحرك المجتمعات الفطرية والوظائف التي تعبر عنها هذه التعايشات، مما يربط الجينات بالخصائص البيئية مثل اكتساب المغذيات وتحلل المواد العضوية في التربة. هنا، نستعرض الدراسات الجينومية التي كشفت عن الجينات المعنية في امتصاص المغذيات وتطوير التعايش، ونناقش التكيفات التي تعتبر أساسية لتطور أنماط الحياة الفطرية الجذرية. كما قمنا بتقييم الخدمات البيئية التي تقدمها الشبكات الفطرية الجذرية ونناقش كيف أن التعايشات الفطرية الجذرية تحمل وعدًا للزراعة المستدامة والغابات من خلال تعزيز اكتساب المغذيات وتحمل الضغوط. بشكل عام، فإن فك رموز الديناميات المعقدة للتعايشات الفطرية الجذرية أمر بالغ الأهمية.
لتعزيز الاستدامة البيئية ومعالجة القضايا البيئية الملحة الحالية. تنتهي هذه المراجعة بأهم المجالات للبحث المستقبلي.
لتعزيز الاستدامة البيئية ومعالجة القضايا البيئية الملحة الحالية. تنتهي هذه المراجعة بأهم المجالات للبحث المستقبلي.
المقدمة
من المعروف على نطاق واسع أن الارتباطات الفطرية موجودة في معظم النظم البيئية الأرضية، بدءًا من الصحاري والغابات الاستوائية إلى السافانا والأراضي الزراعية (Tedersoo et al.، 2014؛ van der Heijden et al.، 2015). أكثر من
الفوسفور (P)، بالإضافة إلى مجموعة واسعة من العناصر الدقيقة (مثل Fe،
الفوسفور (P)، بالإضافة إلى مجموعة واسعة من العناصر الدقيقة (مثل Fe،
تعتبر التعايشات الفطرية الجذرية ذات أهمية كبيرة لعلماء الأحياء والبيئة بسبب تأثيرها العميق على نمو النباتات والفطريات، واللياقة، والتنوع (فان دير هايدن وآخرون، 2015؛ بروندريت وتيدرسو، 2018). علاوة على ذلك، تلعب هذه العلاقات التبادلية دورًا محوريًا في دورات النيتروجين والفوسفور داخل النظم البيئية (سايتو وإزاوا، 2016؛ نيهلس وبلاسارد، 2018؛ بيلتييه وزاك، 2018). أظهرت العديد من الدراسات أن الفطريات الجذرية الم mycorrhizal AMF و ECM يمكن أن تستنزف نسبة كبيرة من مركبات الكربون في العائل، حيث تمثل 5-30% من تدفق الكربون. على مستوى العالم، يتدفق

الشكل 1 الهياكل النموذجية لأنواع مختلفة من التعايش الفطري. (أ) أبواغ الفطريات الميكورية الجذرية (AMF) من Rhizophagus irregularis تحتوي على مئات من النوى الملونة؛ الصورة بإذن من فاسيليس كوكوريس (الجامعة الحرة، أمستردام). النوى مرئية بسبب التلوين بصبغة الحمض النووي الفلورية الخضراء SYTO 13. كل صورة هي نتيجة لـ
دخول الكربون الثابت من النبات إلى الفطريات الحية يعادل
على مدى العقدين الماضيين، ظهرت الجينوميات، كأداة لتحليل المعلومات الجينية للكائنات، كنهج أساسي لفهم البيولوجيا والتطور والبيئة للارتباطات الميكوريزية. بالإضافة إلى توفير رؤى آلية حاسمة، تحدد المعلومات الجينية أيضًا الأهداف التي يمكن إعطاؤها الأولوية لتطبيق الارتباطات الميكوريزية في الزراعة والغابات. في هذه المراجعة، نناقش الدراسات الحديثة التي توضح تأثير الجينوميات والنهج ذات الصلة (مثل تحليل الحمض النووي ونسخ الجينات) على فهمنا للارتباطات الميكوريزية. أولاً، نلخص المعرفة الحالية حول جينومات الفطريات الميكوريزية وآثارها على فهم الانتقال من السابروتروفية إلى التعايش عبر شجرة الحياة الفطرية. ثانيًا، نفحص كيف تم استخدام جينومات الفطريات الميكوريزية وأشجارها المضيفة لفهم الآليات الجزيئية التي تكمن وراء إنشاء وظيفة التعايش. لقد سهلت هذه الموارد تحديد الجينات والمسارات المعنية في تطوير التعايش، وتبادل المغذيات، وتحمل الضغوط. ثالثًا، نناقش كيف كشفت أدوات تحليل الحمض النووي عالية الإنتاجية عن التنوع البيولوجي الرائع للفطريات التربة، كاشفة عن توزيعها من نوى التربة الصغيرة إلى القارة بأكملها. في مناقشتنا، نؤكد على أهمية الشبكات الميكوريزية في النظم البيئية الطبيعية ونبرز أدوارها في توفير خدمات النظام البيئي المختلفة. كما نناقش بإيجاز التطبيقات المحتملة للتعايش الميكوريزي في الزراعة المستدامة واستعادة النظام البيئي، مؤكدين على أهمية فهم الخصائص الوظيفية والأدوار البيئية للفطريات الميكوريزية. من خلال تجميع أحدث نتائج الأبحاث، تهدف هذه المراجعة إلى تحديد فجوات المعرفة واقتراح اتجاهات مستقبلية لأبحاث الميكوريز لمواجهة التحديات العالمية في الزراعة والغابات واستدامة البيئة.
II. استغلال الجينوميات لتعزيز فهمنا لتطوير وتطور ووظيفة التعايشات الميكوريزية
قاعدة بيانات معهد الجينوم المشترك (JGI) MycoCosm تحتوي حاليًا على 2701 جينوم فطري (اعتبارًا من نوفمبر 2023)، بما في ذلك أكثر من 220 فطر ميكوريزي، ومركز المعلومات الوطنية للتكنولوجيا الحيوية GenBank لديه تعليقات توضيحية لـ 4770 جينوم (اعتبارًا من نوفمبر 2023)، مما يدل على التقدم الهائل الذي تم إحرازه في هذا المجال (مارتن وآخرون، 2011؛ غريغوريف وآخرون، 2014). الأنواع الفطرية التي تم تسلسلها متنوعة للغاية وتضم فصائل رئيسية من Mycota. من بين الجينومات المتاحة في قاعدة بيانات MycoCosm، كان 1693 ينتمي إلى Ascomycota، و696 إلى
Basidiomycota، و181 إلى Mucoromycota (بما في ذلك 13 إلى Glomeromycotina).
فتح إصدار جينومات الفطريات ECM Laccaria bicolor وTuber melanosporum (مارتن وآخرون، 2008، 2010) وفطريات AM Rhizophagus irregularis (تيزرانت وآخرون، 2013) آفاقًا جديدة للوراثيين لمقارنة المخطط الجينومي الميكوريزي مع تلك الأنواع السابروتروفية والمرضية، مما يعزز فهمنا لتطور المتعايشات الميكوريزية. يعتمد الاختيار الحالي للفطريات الميكوريزية التي تم تسلسلها على قدرتها على إنشاء ارتباطات ميكوريزية مختلفة، وأهميتها البيئية، والعلاقات النشوء والتطور، والاعتبارات العملية. توفر هذه المجموعة من تسلسلات الجينوم المتاحة فرصًا للتحقيق في الأنماط التطورية والخصائص الوظيفية للتعايشات الميكوريزية عبر العشرات من العائلات الفطرية (وولف وآخرون، 2012؛ كوهلر وآخرون، 2015؛ بيتر وآخرون، 2016؛ مراد وآخرون، 2018؛ مياوتشي وآخرون، 2020؛ ماركيز غالفز وآخرون، 2021؛ ليبرتون وآخرون، 2022؛ لوني وآخرون، 2022؛ كوباياشي وآخرون، 2023؛ بليت وآخرون، 2023). بالنسبة لعدة عائلات، مثل Boletales، أبرزت البيانات ليس فقط أي المناطق من الجينومات كانت متشابهة بين الأنواع المختلفة ولكن أيضًا متى تباينت تسلسلاتها على نطاق ملايين السنين (مياوتشي وآخرون، 2020؛ لوفغرين وآخرون، 2021؛ وو وآخرون، 2022). كانت الأهداف الرئيسية لهذه الدراسات المعتمدة على الجينوم هي (1) مراقبة الانتقالات المستقلة المختلفة من السابروتروفية إلى التعايش في سلالات الفطريات (مثل Agaricales وBoletales وRussulales)، (2) إقامة روابط بين خصائص الجينوم والخصائص البيئية المهمة، مثل امتصاص المغذيات والمواد العضوية في التربة (SOM)، و(3) تحديد مجموعات الجينات المعنية في تطوير ووظيفة الارتباطات الميكوريزية. في الأقسام التالية، نقدم ملخصًا للنتائج الرئيسية لهذه الدراسات بشأن الأنواع الرئيسية من التعايشات الميكوريزية.
1. الفطريات الميكوريزية الجذرية
لقد أسرت الفطريات الميكوريزية الجذرية (AMF) علماء الأحياء التطورية بسبب أصولها القديمة، التي تعود إلى
تمتلك الفطريات الميكوريزية الجذرية خيوطًا متشابكة تحتوي على آلاف النوى وتتكاثر كجراثيم كبيرة متعددة النوى (الشكل 1). حتى الآن، تم إصدار تجميعات الجينوم النووي من الأنواع الممثلة لمختلف أوامر AMF، من Glomerales إلى Diversisporales وArchaeosporales. لديها جينومات كبيرة بشكل غير عادي: حوالي 150 ميغابايت لأنواع Rhizophagus إلى 784 ميغابايت لأنواع Gigaspora (تيزرانت وآخرون، 2013؛ كوباياشي وآخرون، 2018؛ مورين وآخرون، 2019؛ فينيس وآخرون، 2020)، والتي تحتوي على تسلسلات متكررة بكثرة. الفطريات AMF المبكرة المتباينة Paraglomus
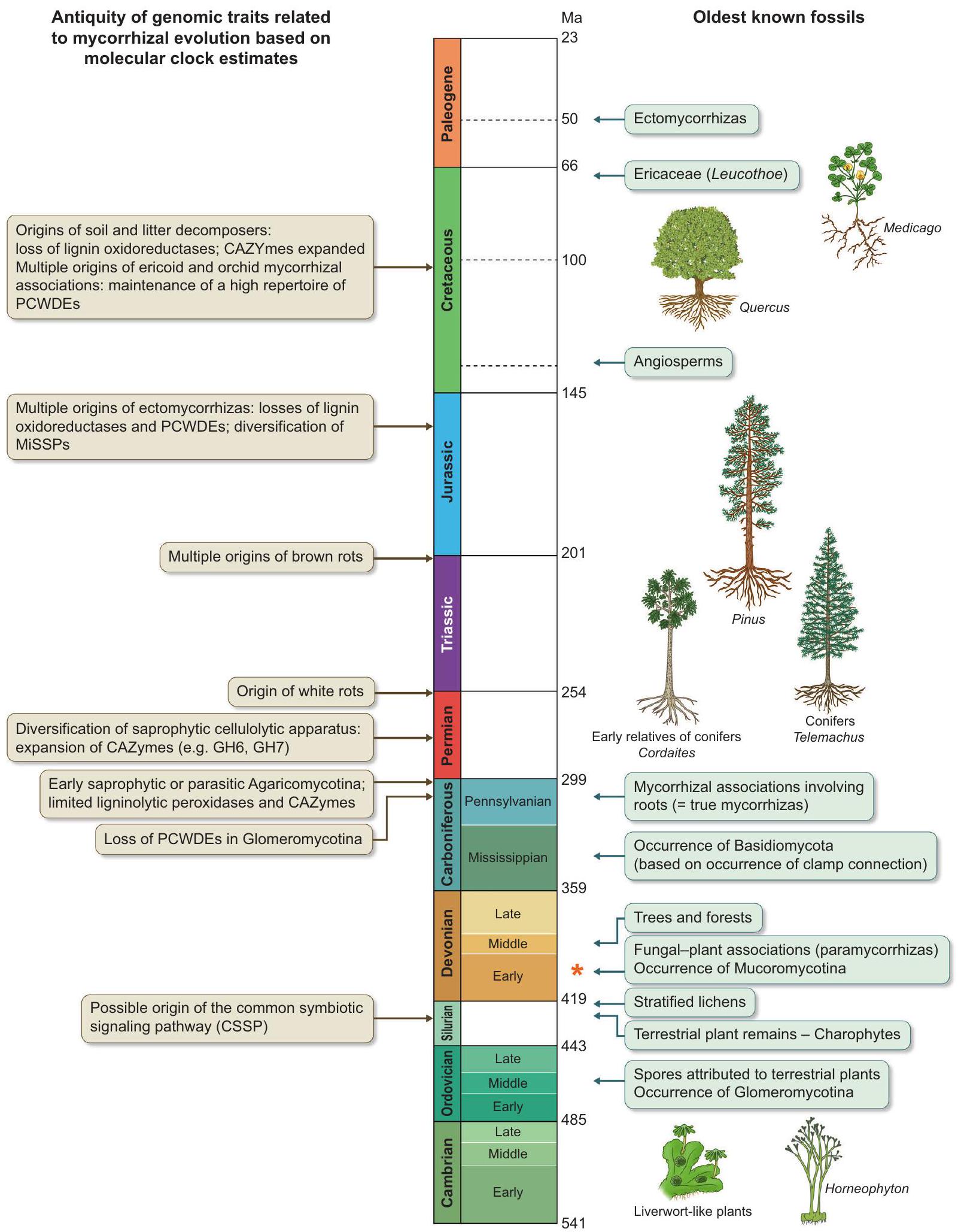
الشكل 2 ظهور السمات الجينومية التعايشية الرئيسية المتعلقة بتطور التعايش الميكوريزي (يسار) بناءً على تقديرات الساعة الجزيئية على طول المقياس الزمني الجيولوجي/العصور الجيولوجية من الكامبري إلى الباليوجيني (الوسط) مع أقدم الحفريات المعروفة (يمين). يمثل النجم البرتقالي تشكيل شرت ريني في 407 مليون سنة. تُظهر الرسوم التوضيحية للنباتات الأرضية المبكرة (نباتات شبيهة بالكبد، هورنيوفيتون)، الأقارب الأوائل للصنوبريات (كورداتيس)، الصنوبريات (تيليماخوس، بينوس)، والنباتات المزهرة (كواركوس، ميدicago). CAZymes، إنزيمات نشطة على الكربوهيدرات؛ GH، جليكوزيل هيدراز؛ MiSSPs، بروتينات صغيرة سرية مستحثة بالميكورزا؛ PCWDEs، إنزيمات تحلل جدران خلايا النباتات. تم التكيف من سترو-ديرين وآخرون (2018).
occultum (Paraglomerales) يمتلك حجم جينوم أصغر بكثير
تم تحدي الفكرة القائلة بأن الفطريات المائية (AMF) هي كائنات قديمة غير جنسية من خلال التحليل الجينومي (Ropars et al., 2016). تم تحديد مناطق جينومية وتنظيمات نووية مرتبطة بالتكاثر الجنسي، بما في ذلك جينات محددة للانقسام الاختزالي، ومواقع محتملة لنوع التزاوج، ومراحل حياة متجانسة-ثنائية النوى (Kokkoris et al., 2021; Reinhardt et al., 2021; Yildirir et al., 2022).
يمكن لفطريات Mucoromycotina المختلفة أن تقيم علاقات تكافلية مشابهة لعلاقات الفطريات الم mycorrhizal مع نباتات الكبد، ونباتات القرن، وغيرها من النباتات الأرضية. ومع ذلك، لم يتم تسلسل جينومات هذه المتكافلات بعد. يمكن أن تعزز هذه الفطريات المعروفة باسم endophytes الجذرية الدقيقة نمو النبات والتغذية (Hoysted et al., 2023) وغالبًا ما تكون أكثر وفرة في الحقول الزراعية مقارنة بالبيئات الأصلية (Albornoz et al., 2022). وقد تم اقتراح تمايز في النطاق بين الفطريات Mucoromycotinian و Glomeromycotinian (Mansfield et al., 2023)، مما يشير إلى أهميتها الزراعية والبيئية، حيث قد يكون لها وظائف تكاملية في النظم البيئية. سيسلط تسلسل جينوم endophytes الجذرية الدقيقة الضوء على تطورها وبيولوجيتها وبيئتها.
تتطلب تطوير الهياكل التبادلية والنشاط الأيضي في الأنسجة التبادلية تنسيقًا متكاملًا وتواصلًا بين المتعايشين، مما يؤدي إلى تغييرات في التعبير الجيني وتنشيط مسارات الإشارة. إن الحفاظ على ‘مسار الإشارة التبادلي المشترك’ (CSSP) بين أنواع مختلفة من النباتات الزهرية هو أمر جدير بالملاحظة (الشكل 2؛ دلاوكس وآخرون، 2015). وهذا يبرز الأصل التطوري المشترك لهذا المسار الإشاري التبادلي القديم. وقد اقترحت الدراسات الجينومية التطورية أن بعض ميزات هذا المسار قد تكون سبقت تطور أولى النباتات الأرضية في الفترة الأوردوفيسية الوسطى إلى المتأخرة وساهمت في إقامة أقدم الارتباطات مع النباتات الأرضية السلفية (دلاوكس وآخرون، 2015). حتى الآن، لم تقم سوى دراسات قليلة بقياس استجابات النسخ الفطرية لظروف بيئية مختلفة (غرونوالد وآخرون، 2009؛ هونيك وآخرون، 2005؛ زينغ وآخرون، 2018). استخدم سيرانو وآخرون (2023) كل من
تقنيات تسلسل RNA النووي والمكاني لاستكشاف النسخ الجينية للتعايش مع الفطريات في مستوى الخلية والدقة المكانية. وقد حددوا استجابات محددة لنوع الخلية تجاه التعايش، بما يتماشى مع الدراسات السابقة التي استخدمت تحليلات النسخ الجينية المعتمدة على التقاط الليزر (Gaude et al., 2012; Hogekamp & Küster, 2013). وقد حددت هذه الدراسات الخاصة بتوصيف RNA مجموعة من الجينات المستجيبة للتعايش، بما في ذلك العديد من الجينات المميزة التي تعتبر حاسمة للتعايش، مثل تلك المعنية بتمثيل الفوسفور (مثل MtPT4) وتلك التي تشفر بروتينات شبيهة بالعوامل المؤثرة (الشكل 2). على الرغم من أن هوية ووظيفة معظم النسخ الجينية المعبر عنها بشكل مختلف من قبل النباتات والفطريات خلال التفاعلات التعايشية لم يتم تحديدها بعد، فإن هذه الدراسات تقدم رؤى قيمة حول الآليات الجزيئية الكامنة وراء التعايش مع الفطريات (انظر Lanfranco & Bonfante، 2023 لمراجعة).
تقنيات تسلسل RNA النووي والمكاني لاستكشاف النسخ الجينية للتعايش مع الفطريات في مستوى الخلية والدقة المكانية. وقد حددوا استجابات محددة لنوع الخلية تجاه التعايش، بما يتماشى مع الدراسات السابقة التي استخدمت تحليلات النسخ الجينية المعتمدة على التقاط الليزر (Gaude et al., 2012; Hogekamp & Küster, 2013). وقد حددت هذه الدراسات الخاصة بتوصيف RNA مجموعة من الجينات المستجيبة للتعايش، بما في ذلك العديد من الجينات المميزة التي تعتبر حاسمة للتعايش، مثل تلك المعنية بتمثيل الفوسفور (مثل MtPT4) وتلك التي تشفر بروتينات شبيهة بالعوامل المؤثرة (الشكل 2). على الرغم من أن هوية ووظيفة معظم النسخ الجينية المعبر عنها بشكل مختلف من قبل النباتات والفطريات خلال التفاعلات التعايشية لم يتم تحديدها بعد، فإن هذه الدراسات تقدم رؤى قيمة حول الآليات الجزيئية الكامنة وراء التعايش مع الفطريات (انظر Lanfranco & Bonfante، 2023 لمراجعة).
نظرًا لأن البروتينات الصغيرة المفرزة الشبيهة بالعوامل والبروتينات الصغيرة RNA تلعب دورًا رئيسيًا في تفاعلات النبات مع مسببات الأمراض، فقد تم التحقيق فيها أيضًا في ارتباطات الفطريات الم mycorrhiza (Ledford et al.، 2023). حدد Aparicio Chacón et al. (2023) مجموعة من البروتينات المفرزة الشبيهة بالعوامل التي تحفزها الفطريات الم mycorrhiza، والتي ينظم بعضها تعبير الجينات المضيفة. وجد Teulet et al. (2023) بروتينات ذات تشابه هيكلي مع العوامل ثنائية المجال المعروفة من Fusarium oxysporum. من بين المئات من البروتينات المتوقعة للعوامل من الفطر الم mycorrhiza
توجد معلومات محدودة حول كيفية تأثير التvariations الجينية للنباتات أو الفطريات على الاستجابات النسخية للنباتات تجاه الفطريات (ماتيوس وآخرون، 2019). لا يزال تأثير البكتيريا الداخلية للفطريات الم mycorrhizal والفيروسات والمجتمعات الميكروبية المرتبطة بالخيوط على تنظيم التعبير الجيني والقدرات الفسيولوجية للرابطة غير واضح ويتطلب مزيدًا من التحقيق (لانفرانكو وبونفانتي، 2023).
2. الفطريات الجذرية الخارجية
الانتقال من السابروتروفية إلى التعايش مع الفطريات المكونة للغلاف الجذري هو حدث تطوري رئيسي حدث عدة مرات خلال تطور الفطريات (تيدرسو وآخرون، 2010؛ سترولو-ديرين وآخرون، 2018؛ الشكل 2). على الرغم من أن الآليات الدقيقة التي تحرك هذه الانتقالات لا تزال غير واضحة، فإن الدراسات الجينومية المقارنة والدراسات النشئية قد قدمت فرضيات وأنماط لمزيد من التحقيقات (كوهلر وآخرون، 2015؛ مياوتشي وآخرون، 2020؛ ليبرتون وآخرون، 2021). من المحتمل أن الانتقال إلى نمط حياة الفطريات المكونة للغلاف الجذري حدث من خلال سلسلة من الخطوات، بدءًا من استعمار الفطريات السابروتروفية للتربة/الفضلات لجذور النباتات وتشكيل علاقة تعاونية تدريجية مع المضيف. قد تكون العوامل البيئية مثل الوصول السهل إلى الكربوهيدرات القابلة للذوبان قد دفعت هذا الانتقال. في البيئات الفقيرة بالمغذيات، يسمح تشكيل الجمعيات التعاونية مع النباتات للفطريات المكونة للغلاف الجذري بالوصول إلى الكربوهيدرات القابلة للذوبان والفيتامينات، مما يوسع نطاقها البيئي. تشير قدرة الفطريات المكونة للغلاف الجذري على تشكيل جمعيات تعايشية مع مجموعة متنوعة من أنواع الأشجار إلى الإشعاع التكيفي، وآليات تعايش محددة، وتخصص في النطاق خلال تطورها.
التاريخ (مارتن وآخرون، 2016، 2017؛ ساتو، 2023؛ الشكل 2). قد تكون سلالات الفطريات المتكافلة مع الجذور قد انحرفت في أواخر العصر الطباشيري ولديها فرص للتطور المشترك مع النباتات الزهرية المتكافلة (على سبيل المثال، من خلال تطوير أنظمة التعرف على المضيف؛ تانغ وآخرون، 2021؛ ليبرتون وآخرون، 2022؛ لوني وآخرون، 2022) وكبح دفاعات النباتات (بلت وآخرون، 2011، 2014؛ كانغ وآخرون، 2020)، مما أدى إلى توسعها في مساحات جديدة وتنوع سريع لاحق (مارتن وآخرون، 2016؛ ساتو، 2023). تبرز التطورات المتكررة لنمط الحياة المتكافل مع الجذور (وغياب العودة إلى الحالة السلفية) والتطور المتقارب في مئات من سلالات الفطريات أهميتها التكيفية وتؤكد الضغوط الانتقائية التي تدفع هذا التطور التطوري (وولف وآخرون، 2012؛ ليبرتون وآخرون، 2021).
التاريخ (مارتن وآخرون، 2016، 2017؛ ساتو، 2023؛ الشكل 2). قد تكون سلالات الفطريات المتكافلة مع الجذور قد انحرفت في أواخر العصر الطباشيري ولديها فرص للتطور المشترك مع النباتات الزهرية المتكافلة (على سبيل المثال، من خلال تطوير أنظمة التعرف على المضيف؛ تانغ وآخرون، 2021؛ ليبرتون وآخرون، 2022؛ لوني وآخرون، 2022) وكبح دفاعات النباتات (بلت وآخرون، 2011، 2014؛ كانغ وآخرون، 2020)، مما أدى إلى توسعها في مساحات جديدة وتنوع سريع لاحق (مارتن وآخرون، 2016؛ ساتو، 2023). تبرز التطورات المتكررة لنمط الحياة المتكافل مع الجذور (وغياب العودة إلى الحالة السلفية) والتطور المتقارب في مئات من سلالات الفطريات أهميتها التكيفية وتؤكد الضغوط الانتقائية التي تدفع هذا التطور التطوري (وولف وآخرون، 2012؛ ليبرتون وآخرون، 2021).
تميل أعداد نسخ الجينات الخاصة بالإنزيمات الميكروبية القابلة للتحلل (PCWDEs) إلى إظهار أنماط مميزة مرتبطة بموائل بيئية محددة في الفطريات (Floudas et al., 2012, 2020; Kohler et al., 2015) بسبب دورها في تحلل مواد جدران خلايا النباتات (الشكل 2). يُعتبر الانكماش الحاد في عائلات الجينات التي تشفر الإنزيمات الميكروبية القابلة للتحلل سمة مميزة للفطريات المتكافلة (ECM) والفطريات الميكوريزية (AM) (الشكل 3). يفتقر معظمها إلى البيروكسيدازات من الفئة الثانية (PODs) القابلة للتحلل اللجنيني، والإنفرتازات، والإنزيمات الداخلية للسليلوز، والإنزيمات المحللة للسليلوز، والعناصر المرتبطة بالسليلوز، مما يقيّد قدرتها على تحلل المواد العضوية السطحية (SOM) وجدران خلايا النباتات التي تعيش عليها. مع تحول السلالات القديمة للفطريات المتكافلة نحو العلاقات التبادلية، انخفضت الحاجة إلى هذه الإنزيمات التحليلية، مما أدى إلى فقدانها التدريجي على مر الزمن التطوري (Martin & Selosse, 2008; Wolfe et al., 2012؛
ليبريتون وآخرون، 2021). هذا يجعل الفطريات الميكوريزية تعتمد على نباتاتها المضيفة للحصول على الكربون. ومع ذلك، فإن العديد من الفطريات التي تم تسلسلها قد حافظت على مجموعة فريدة من إنزيمات تحلل جدران الخلايا النباتية، بما في ذلك الإندوجلوكاناز والأكسيدوريدوكتاز/لاكاز، مما يشير إلى أن العديد من الفطريات الميكوريزية الخارجية تمتلك قدرات متنوعة لجمع الحطام النباتي والميكروبي (النيكرومس) من التربة والفضلات (بوديكر وآخرون، 2014؛ فينول فوراي وآخرون، 2014؛ أوب دي بيك وآخرون، 2018؛ بيليتييه وزاك، 2018؛ نيكولاس وآخرون، 2019؛ فلوداس وآخرون، 2020؛ مايار وآخرون، 2023). من الضروري تنظيم هذه الجينات الخاصة بإنزيمات تحلل جدران الخلايا النباتية بدقة لتجنب تحفيز استجابات الدفاع النباتية (مياوتشي وآخرون، 2020).
في Mucoromycota، تُعتبر Endogonales مجموعة غامضة من الفطريات التي تُقيم تآزرًا إكتوميكوريزيًا وعلاقات حيوية مع السرخس، والليكوفيت، والهورنورت، واللبلاب، بالإضافة إلى كونها تضم فطريات محتملة من النوع السابروتروفي المرتبطة بالخشب المتعفن ورقائق الأوراق (Desirò et al.، 2017). لفهم أفضل لتطور التآزر الميكوريزي والبيئة الغذائية في Endogonales، تم تسلسل جينومات أربع مجموعات من Endogonaceae، بما في ذلك ثلاث مجموعات من ECM ومجموعة واحدة محتملة من السابروتروف (Chang et al.، 2019). تُظهر جينومات العزلات ECM خصائص نموذجية لفطريات ECM، بما في ذلك مجموعة محدودة من جينات PCWDE. علاوة على ذلك، أشارت تحليلات التأريخ إلى أن Endogonaceae نشأت في حدود العصر البرمي-الترياسي، مما يدل على أن العلاقة الميكوريزية بين Endogonales والنباتات قد لعبت دورًا حاسمًا في استعمار اليابسة بواسطة النباتات.
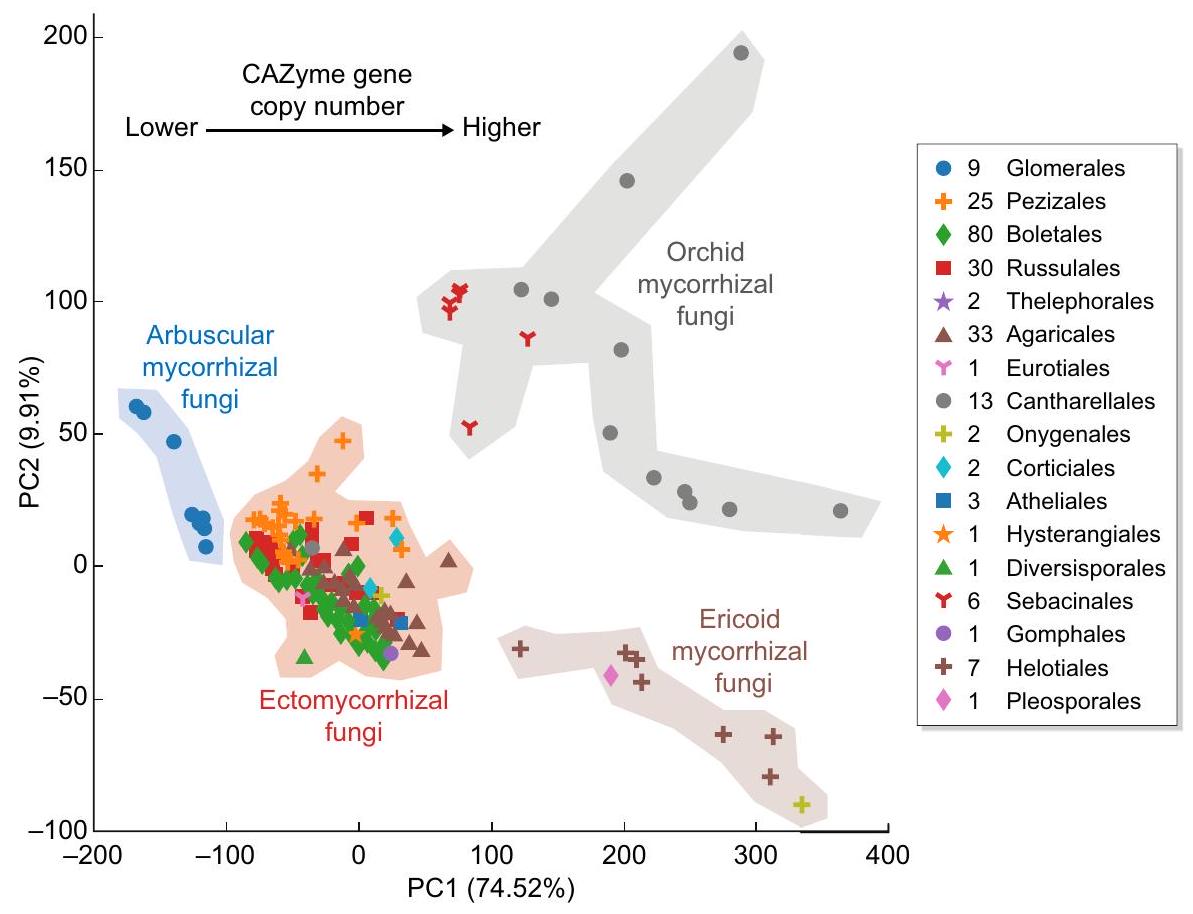
الشكل 3 التوزيع التفاضلي لعدد نسخ الجينات لجينات CAZyme التي تعمل على البوليسكاريدات الميكروبية والفطرية والنباتية ومشتقات اللجنين في جينومات 217 نوعًا من الفطريات الميكوريزية الجذرية، والفطريات الميكوريزية الخارجية، والفطريات الميكوريزية الأوركيدية أو الفطريات الميكوريزية الإريكية ذات البيئات المتباينة. تم تحديد تحليل المكونات الرئيسية لعدد نسخ جينات CAZyme الكلي الموجودة في الفطريات الميكوريزية الجذرية، والفطريات الميكوريزية الخارجية، والفطريات الميكوريزية الإريكية، والفطريات الميكوريزية الأوركيدية. كل رمز يتوافق مع جينوم نوع من الأنواع الميكوريزية المتاحة في قاعدة بيانات JGI MycoCosm (mycocosm.jgi.doe.gov). يتم الإشارة إلى الترتيب التصنيفي للأنواع المتسلسلة بواسطة رموز ملونة مختلفة كما هو موضح في الأسطورة (اللوحة اليمنى). لاحظ التوزيعات المتباينة في مجموعات جينات CAZyme في أنماط الحياة الميكوريزية المختلفة مع زيادة مجموعة جينات CAZyme من الفطريات الميكوريزية الجذرية (السفلى) إلى الفطريات الميكوريزية الإريكية (العليا؛ من اليسار إلى اليمين). تم إجراء تحليل المكونات الرئيسية (PCA) باستخدام أداة MycoCosm PCA (mycocosm.jgi.doe.gov). تم الحصول على مجموعات جينات CAZyme بعد تنسيق شبه يدوي لتسلسلات البروتين بواسطة فريق CAZy (www.cazy.org) (Drula et al., 2022).
لقد انحرف مجال وراثة الفطريات الميكوريزية عن مجرد مقارنة الجينوم واكتشاف الجينات إلى مجال علم الأنظمة، مستكشفًا الأسس الخلوية والجزيئية للتعايش. وقد قاد هذا التطور توفر مجموعات بيانات التعبير الجيني على نطاق واسع (Plett et al., 2014, 2019; Liao et al., 2018; Bouffaud et al., 2020; Ruytinx et al., 2021). لقد سهلت هذه البيانات تحديد شبكات الجينات والمراكز المعنية في العمليات البيولوجية الرئيسية مثل امتصاص المغذيات، والتمثيل الغذائي، والإشارات في التعايش مع الفطريات الميكوريزية. تتميز أنواع مختلفة من التعايش مع الفطريات الميكوريزية بزيادة تنظيم الجينات من فئات وظيفية متداخلة، مثل تخزين المعلومات ومعالجتها، والنقل، والتمثيل الغذائي. ومع ذلك، تعبر كل علاقة عن مجموعة مميزة من الجينات المرتبطة بالتعايش المعنية في تفاعلات الأكسدة والاختزال، ونقل المغذيات، والتمثيل الغذائي. عدد كبير من هذه الجينات له نظائر في الأنواع المتحللة، مما يشير إلى أنها ليست فريدة من نوعها في الفطريات الميكوريزية ولكنها مرتبطة بمسارات التمثيل الغذائي الأساسية الضرورية (Lebreton et al., 2022; Looney et al., 2022; Wu et al., 2022). بسبب هذه التغيرات في التعبير الجيني، تؤدي التفاعلات الميكوريزية الخارجية إلى تغييرات في مستويات المستقلبات الفطرية ومستقلبات نبات المضيف، بما في ذلك التحولات الملحوظة في التمثيل الغذائي ونقل الكربوهيدرات، والأحماض الأمينية، والأحماض العطرية، والأحماض العضوية، والأحماض الدهنية، والماء (Hortal et al., 2017; Plett et al., 2021, 2023; Tang et al., 2021; Bogar, 2023; Stuart et al., 2023).
ظهرت جينات جديدة خاصة بنمط حياة الفطريات الميكوريزية (ECM)، مثل تلك التي تشفر للبروتينات الصغيرة المفرزة نتيجة لتحفيز الفطريات الميكوريزية (Plett et al., 2011, 2014; Plett & Martin, 2011; Pellegrin et al., 2015) والـ RNA الصغيرة (Wong-Bajracharya et al., 2022)، مما يعزز القدرات التبادلية للفطريات الميكوريزية ويضبط مناعة النبات المضيف بالإضافة إلى دخول ميكروبات أخرى تستعمر النبات المضيف. تم تحديد عدة جينات قد تسهم في التوافق بين النباتات والفطريات (Lofgren et al., 2021; Tang et al., 2021; Lebreton et al., 2022).
تحديد جينات النباتات التي تساهم في وتحتاج إلى التعايش مع الفطريات الميكورية الخارجية هو مهمة شاقة، وقد تم تحديد عدد محدود من الجينات وتوصيفها (تاجو وآخرون، 2005؛ لابيه وآخرون، 2019). ومن الجدير بالذكر أن كوبر وآخرين (2019) قد أظهروا أن الفطر الميكوري الخارجي
3. فطريات الميكوريزا للأوركيد
مع ما يقرب من 30000 نوع، تقدم عائلة الأوركيد موارد قيمة لدراسة تطور النباتات والتكيف البيئي، وتعتبر نباتات زينة مهمة اقتصاديًا وثقافيًا في المجتمع البشري. يبدو أن الأوركيد ظهرت منذ حوالي 112 مليون سنة (م.س؛ الشكل 2). تباعدت تحت العائلات Orchidoideae وEpidendroideae عن بعضها البعض في نهاية العصر الطباشيري، وتباعدت قبائل الإيبيدندرويد العليا بسرعة بين 37.9 و30.8 م.س (Givnish et al.، 2015؛ Zhang et al.، 2017). تشتهر الأوركيد اعتمادها على الفطريات الميكوريزية خلال الإنبات، وتأسيس الشتلات، واكتساب العناصر الغذائية، خاصة خلال المراحل المبكرة من دورة حياتها. تعتمد البروتوكورمات للأوركيد المنبتة بشكل كامل.
عدم استعمار الفطريات الميكوريزية من أجل تأسيسها ونموها (Perotto & Balestrini، 2023). وبالتالي، لا يمكن أن تعيش الأوركيد بدون الفطريات الميكوريزية (Rasmussen & Rasmussen، 2014). في السنوات الأخيرة، زاد الاهتمام بعلم الجينوم للأوركيد وفطريات ORM لفك الشفرات الجينية التي تكمن وراء تفاعلات الأوركيد والفطريات وخصوصية هذه العلاقات التبادلية (Chen et al.، 2022؛ Song et al.، 2022). تم تسلسل جينومات عدة أنواع من الأوركيد (Cai et al.، 2015؛ Zhang et al.، 2016، 2017؛ Yang et al.، 2021؛ Jiang et al.، 2022؛ Li et al.، 2022). تم مقارنة تجميعات الجينوم على مستوى الكروموسومات لاثنين من أنواع الأوركيد القريبة،
عدم استعمار الفطريات الميكوريزية من أجل تأسيسها ونموها (Perotto & Balestrini، 2023). وبالتالي، لا يمكن أن تعيش الأوركيد بدون الفطريات الميكوريزية (Rasmussen & Rasmussen، 2014). في السنوات الأخيرة، زاد الاهتمام بعلم الجينوم للأوركيد وفطريات ORM لفك الشفرات الجينية التي تكمن وراء تفاعلات الأوركيد والفطريات وخصوصية هذه العلاقات التبادلية (Chen et al.، 2022؛ Song et al.، 2022). تم تسلسل جينومات عدة أنواع من الأوركيد (Cai et al.، 2015؛ Zhang et al.، 2016، 2017؛ Yang et al.، 2021؛ Jiang et al.، 2022؛ Li et al.، 2022). تم مقارنة تجميعات الجينوم على مستوى الكروموسومات لاثنين من أنواع الأوركيد القريبة،
استهدفت جهود تسلسل الجينوم فطريات ORM محددة للحصول على فهم أعمق لمجموعاتها الجينية وقدراتها التعايشية. تم نشر جينومات Tulasnella calospora وSebacina vermifera وCeratobasidium spp. (Miyauchi et al., 2020)، وتم تسلسل عشرة فطريات ORM إضافية في Tulasnellaceae وSerendipitaceae وCeratobasidiaceae في JGI. على عكس ECM وAMF، تمتلك فطريات ORM مجموعة كبيرة من PCWDEs التي يتم التعبير عنها خلال تفاعلاتها (Balestrini et al., 2014؛ Chen et al., 2017، 2022؛ Ghirardo et al., 2020؛ الشكل 3). مشابهة لفطريات ERM، غالبًا ما تمتلك أنواع ORM مجموعة أكبر من PCWDEs مقارنةً بفطريات تحلل الخشب ومحللات التربة/الفضلات (Miyauchi et al., 2020)، مما يدعم أنماط حياتها المزدوجة السابروتروفية/التعايشية. افترضنا أن حجم مجموعة PCWDE يعكس عمر التعايش الفطري على طول استمرارية السابروتروفية إلى التعايش، مع ظهور التعايش AMF في الديفوني المبكر (
4. الفطريات الإريكويدية
تشكل فطريات الميكورايزا الإريكويدية تعايشات متبادلة مع مجموعة واسعة من نباتات Ericaceae، بما في ذلك الخلنج، والتوت الأزرق، والرودودندرون، وتلعب أدوارًا حاسمة في اكتساب العناصر الغذائية وتحمل الضغوط في التربة الفقيرة بالعناصر الغذائية والحمضية. ركز تسلسل الجينوم على عدة أنواع رئيسية من ERM، بما في ذلك الفطريات الإريكويدية النموذجية Rhizoscyphus ericae وOidiodendron maius (Kohler et al., 2015؛ Grelet et al., 2016؛ Martino et al., 2018). وفرت توفر هذه الجينومات رؤى حول المجموعة الجينية
والسمات الوظيفية لفطريات ERM. وفقًا للتحليل النشوي الجيني، تم تقدير أن أحدث سلف مشترك للفطريات ERM المتسلسلة، بما في ذلك
والسمات الوظيفية لفطريات ERM. وفقًا للتحليل النشوي الجيني، تم تقدير أن أحدث سلف مشترك للفطريات ERM المتسلسلة، بما في ذلك
III. الشبكات الفطرية
تعد الشبكات الفطرية تحت الأرض ضرورية لاكتساب العناصر الغذائية ونقلها بواسطة النباتات. تتكون من خيوط فطرية مترابطة تمتد من جذور الميكورايزا إلى التربة المحيطة، مكونة خيوط فطرية خارجية (الشكل 4). في التربة الطبيعية، تتطور العديد من الشبكات الفطرية المتداخلة بأحجام مختلفة حيث يتم استعمار كل نبات مضيف بواسطة عدة متعايشات فطرية، تصل إلى عدة عشرات لكل شجرة بالغة (Rog et al., 2020). تلعب دورًا حيويًا في استكشاف التربة بكفاءة، وجمع العناصر الغذائية، ونقلها إلى جذور المضيف، والمساهمة في خدمات النظام البيئي الأساسية، بما في ذلك تعزيز هيكل التربة وامتصاص الماء (الجدول 1). علاوة على ذلك، تلعب الشبكات الفطرية دورًا حاسمًا في دورة الكربون وتخزينه (Hawkins et al., 2023؛ الجدول 1). يعتمد تأسيس هذه الشبكات الفطرية ونشاطها على إمداد مستمر من مركبات الكربون النباتية.
داخل الشبكات الفطرية التي أسستها الفطريات الميكورايزية، يتم تشكيل مجموعة فرعية محددة تسمى الشبكات الفطرية المشتركة (CMNs) عندما يقيم الميكوبونت اتصالات فعلية بين جذور نوعين أو أكثر من النباتات. تم اقتراح نقل السكريات والعناصر الغذائية الأخرى بين النباتات المترابطة، غالبًا ما تشمل الشتلات، من خلال CMNs في عدة دراسات (Finlay & Read, 1986؛ Arnebrant et al. 1993؛ Simard
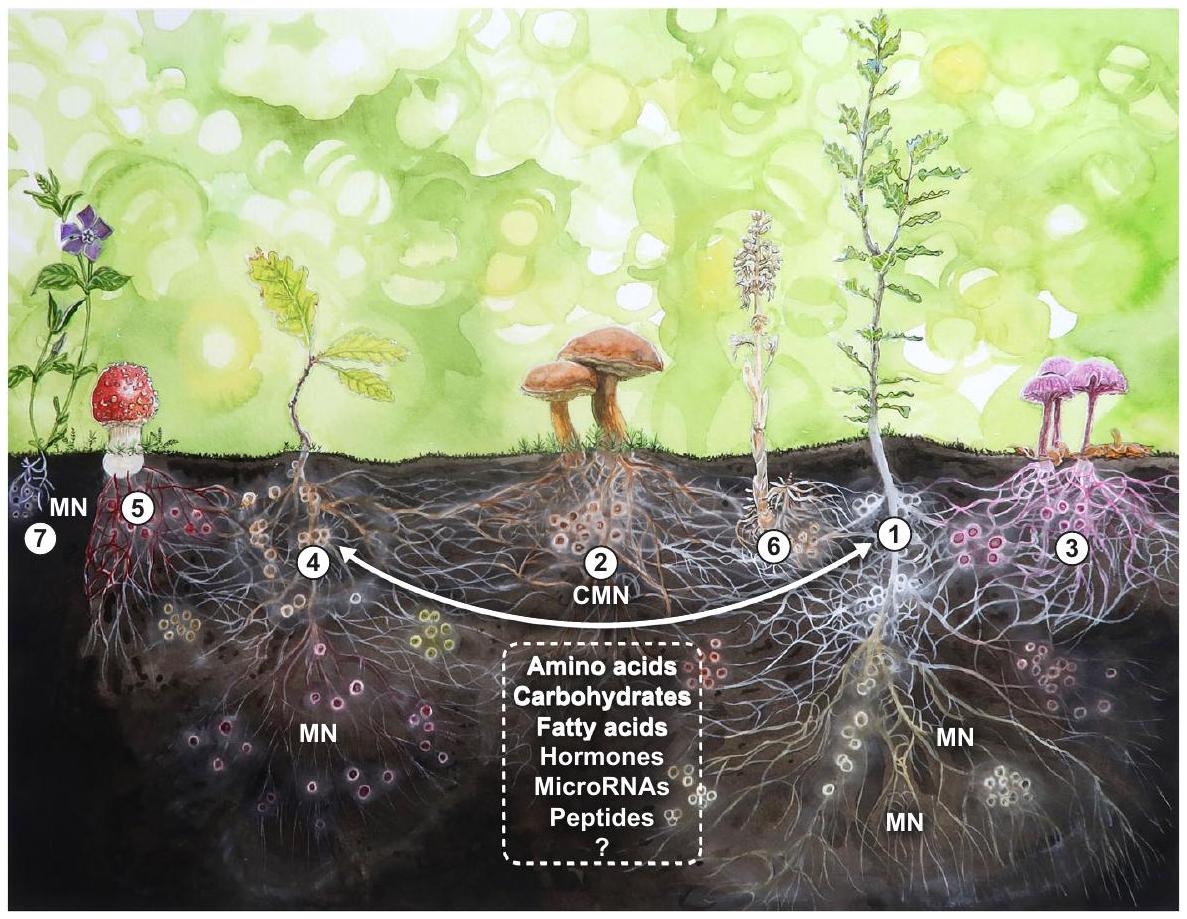
الشكل 4 عرض لمجتمع التربة الفطرية في غابة افتراضية تهيمن عليها أنواع البلوط المرتبطة بأنواع مختلفة من الفطريات الميكورايزية التي تشكل شبكات تحت الأرض منفصلة (MN) قد تستخدم لنقل الأحماض الأمينية، والكربوهيدرات، والأحماض الدهنية، والميكروRNAs الإشارية أو الببتيدات والهرمونات. يشكل بلوط دائم الخضرة (Quercus ilex) (1) جذور ميكورايزية خارجية وشبكات فطرية مع Xerocomellus chrysentheron (البوليت الأحمر المتشقق) (2) وLaccaria amethystina (المخادع الأرجواني) (3)، بينما يؤسس بلوط ثابت (Quercus petrea) (4) ميكورايزا خارجية (ECM) وشبكات فطرية مع Xerocomellus chrysentheron (2) وAmanita muscaria (5). تظهر جذور ECM كنقاط ملونة على أنظمة الجذر. تربط شبكات X. chrysentheron بين Q. ilex وQ. petrea من خلال شبكة فطرية مشتركة (CMN). تتطفل الأوركيد غير الضوئية Neottia nidus-avis وشبكتها الفطرية (ORM) (6) على شبكة الميكورايزا للبلوط الدائم الخضرة. يتم استعمار البيروينكل الأصغر (Vinca minor) بواسطة AMF، مما يشكل شبكة ميكورايزية مستقلة (7). تمثل الألوان المختلفة شبكات فطرية ميكورايزية مختلفة، مع جذور ECM كنقاط من نفس اللون. لاحظ أن تركيبات أخرى ممكنة (مثل الغابات الشمالية التي تحتوي على أشجار ECM غالبًا ما تحتوي على طبقة تحتية من الشجيرات (مثل Vaccinium) التي تشكل ارتباطات ميكورايزا إريكويدية (ERM)؛ توضيح بفضل فلوريان غادين).
الجدول 1 خدمات النظام البيئي المقدمة بواسطة الفطريات الميكورايزية والشبكات.
| خدمة النظام البيئي | الأهمية البيئية | المراجع المختارة |
| امتصاص العناصر الغذائية وتحفيز نمو النبات | يمكن أن تعزز الفطريات الميكورايزية نمو النبات وامتصاص العناصر الغذائية. يمكن أن يتم الحصول على ما يصل إلى 90% من الفوسفور والنيتروجين في النبات بواسطة الفطريات الميكورايزية. تعتمد بعض النباتات بالكامل على العناصر الغذائية التي تحصل عليها من الفطريات الميكورايزية. | van der Heijden et al. (2008) |
| تخزين الكربون تحت الأرض ودورة الكربون | تخصص النباتات 5-30% من منتجاتها الضوئية للمتعايشات الميكورايزية. يتم تخصيص ما يصل إلى 13 جيجا طن من الكربون
|
van der Heijden et al. (2008)؛ Hawkins et al. (2023) |
| تشكيل هيكل التربة والتجميع | تقوم الخيوط الفطرية والمخلفات الفطرية بربط جزيئات التربة معًا في تجمعات أكبر وتساهم بشكل كبير في تعزيز هيكل التربة. | Jastrow et al. (1998) |
| امتصاص الماء وتقليل فقد العناصر الغذائية بالتسرب | تقليل فقد العناصر الغذائية بالتسرب بواسطة الميكورايزات. امتصاص الماء بواسطة الخيوط الفطرية. الأدلة من الأنظمة الطبيعية مفقودة إلى حد كبير. | Ahonen-Jonnarth et al. (2003)؛ Cavagnaro et al. (2015)؛ Kakouridis et al. (2022) |
| تعزيز تأسيس الشتلات | يمكن أن تستعمر الفطريات الميكوريزية الجذرية بسرعة جذور الشتلات الناشئة ويمكن أن يكون لها تأثيرات قوية على نجاح تأسيس الشتلات. كما يتم الإبلاغ بانتظام عن عدم وجود تأثيرات أو حتى تأثيرات سلبية. | فان دير هايدن وهورتون (2009) |
| نقل العناصر الغذائية بين النباتات | تمت ملاحظة كميات كبيرة من النقل في النباتات المتطفلة على الفطريات وبراعم الأوركيد. إن الأهمية البيئية للنقل بين النباتات الخضراء محل نقاش وتحتاج إلى مزيد من الدراسة. | سيمارد وآخرون (1997)؛ كلاين وآخرون (2016)؛ هينريكستون وآخرون (2023)؛ كارست وآخرون (2023)؛ ميركس (2023) |
وآخرون، 1997؛ وو وآخرون، 2001؛ كلاين وآخرون، 2016؛ سيلوس وآخرون، 2016؛ كاهانوفيتش وآخرون، 2022). ومع ذلك، فإن الآليات الكامنة وراء هذه التحويلات معقدة، ومن الصعب تمييز الدور المحدد لشبكات الفطريات المشتركة مقابل مسارات أخرى مثل حركة المغذيات والإشارات من خلال محلول التربة (بابيكوفا وآخرون، 2013).
العديد من الفطريات الميكوريزية هي فطريات عامة ولها نطاق مضيف واسع، ويمكن أن تكون عدة نباتات متصلة ببعضها البعض من خلال شبكات الهيفات. على سبيل المثال، قدمت التجارب في البيوت الزجاجية أدلة على أن
في الفطريات المتكافلة مع الجذور، يمكن للأفراد الجينية (الجينات) أن تستعمر في الوقت نفسه عدة أشجار (Beiler et al., 2010). وقد حددت دراسات بصمة الحمض النووي جينات كبيرة في عدة أنواع من Boletales (FioreDonno & Martin, 2001؛ Hirose et al., 2004؛ Beiler et al., 2010). على الرغم من أن هذه الدراسات قد قدمت أدلة على وجود أفراد جينية كبيرة جدًا (
نتيجة لذلك، كانت الأهمية البيئية لشبكات الفطريات الجذرية المشتركة (CMNs) في دورة المغذيات موضوع تدقيق ونقاش (روبنسون وفيتر، 1999؛ هينريكستون وآخرون، 2023؛ كارست وآخرون، 2023؛ روبنسون وآخرون، 2024). لذلك، هناك حاجة ملحة لتحديد الأدوار الفسيولوجية والبيئية لشبكات الفطريات الجذرية (بما في ذلك CMNs) في النظم البيئية الطبيعية، وأهميتها النسبية في دعم نمو النباتات وتأسيس الشتلات. يجب تطوير أدوات التصوير لتتبع واستكشاف الهيفات الفردية في الشبكات الفطرية المتداخلة في التربة لاختبار ما إذا كانت النوى، والنصوص، والبروتينات، والمواد الأيضية يمكن أن تهاجر من نبات فردي إلى آخر عبر CMNs. يتم حاليًا تطوير نهج متكامل يجمع بين تسلسل الجينوم متعدد المواقع لتقييم التوزيع المكاني وحجم الأنماط الجينية، ونشاطها من خلال الميتا ترانسكريبتوميات/الميتا بروتينوميات، وقياس تدفقات الأيض بين النباتات الفطرية المختلفة في البيئات الطبيعية، وسيقدم معلومات قيمة حول وظيفة شبكات الفطريات الجذرية.
رابعاً. رسم تنوع الفطريات الجذرية
في السنوات الأخيرة، كان هناك تركيز قوي على توثيق التنوع البيولوجي تحت الأرض وإنشاء خرائط عالمية لمختلف الكائنات الحية في التربة. تم بذل جهود لتوفير خرائط عالمية للكتلة الحيوية الميكروبية (Xu et al., 2013)، الديدان الأرضية (Phillips et al., 2019)، البروتستات (Oliverio et al., 2020)، البكتيريا (Bahram et al., 2018؛ Delgado-Baquerizo et al., 2018)، الفطريات (Tedersoo et al., 2014؛ Větrovský et al., 2020)، والبيئة الحيوية للتربة بشكل عام (DelgadoBaquerizo et al., 2020). بالإضافة إلى ذلك، تم نشر خرائط توزيع عالمية للنباتات المرتبطة بالفطريات الجذرية، والفطريات الميكوريزية، والفطريات ECM (Öpik et al., 2010؛ Tedersoo et al., 2014؛ Davison et al., 2015؛ Soudzilovskaia et al., 2019؛ الشكل 5). ومع ذلك، لا تزال خرائط عالمية لفطريات ORC وERM، بالإضافة إلى الفطريات الجذرية الدقيقة من نوع Mucoromycota، مفقودة. علاوة على ذلك، هناك حاجة إلى خرائط توثق التوزيع العالمي للميكروبات المرتبطة بالفطريات الجذرية، مثل البكتيريا المساعدة للفطريات الجذرية، والفطريات، والبروتستات، لفهم مدى أنظمة الفطريات الجذرية بشكل كامل (Frey-Klett et al., 2007؛ Emmett
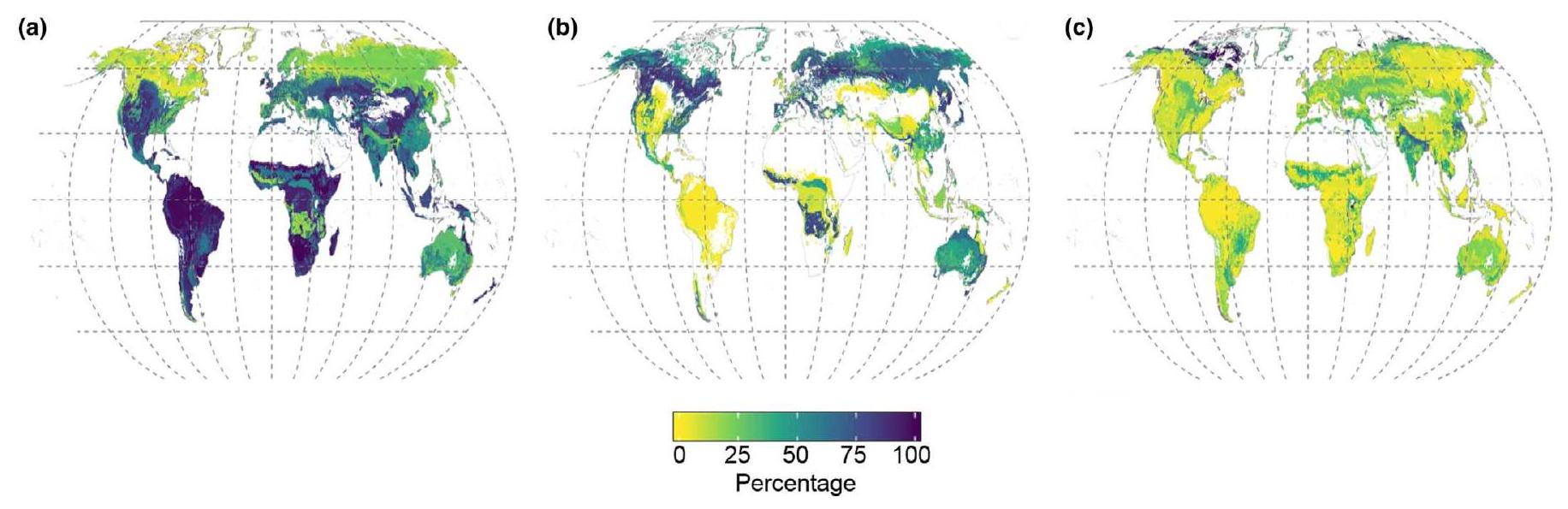
الشكل 5 خريطة عالمية لنسبة إجمالي الكتلة الحيوية للنباتات فوق الأرض المرتبطة بفئات الميكوريزا المختلفة. (أ) نباتات ميكوريزا كروية؛ (ب) نباتات ميكوريزا خارجية؛ (ج) نباتات غير ميكوريزية. كانت دقة الخريطة 10 دقائق قوسية. نُشر بواسطة سوزديلوفسكايا وآخرون (2019).
et al.، 2021؛ يانغ وآخرون، 2023؛ تشانغ وآخرون، 2023). يعد تحديد التنوع الجيني لهذه الكائنات الدقيقة المرتبطة بالفطريات الجذرية وتحليل دينامياتها المكانية والزمانية أمرًا بالغ الأهمية، حيث اقترحت الدراسات الناشئة مشاركتها في تسهيل امتصاص العناصر الغذائية والخدمات الأخرى التي تقدمها خيوط الفطريات الجذرية (تشانغ وآخرون، 2022؛ لي وآخرون، 2023؛ وانغ وآخرون، 2023أ، ب). يبدو أن قدرة الفطريات الجذرية على اكتساب العناصر الغذائية تتأثر بتكوين البكتيريا المرتبطة بخيوط الفطريات الجذرية. على سبيل المثال، أظهر تشانغ وآخرون (2023) أن البكتيريا المرتبطة بخيوط الفطريات الجذرية الشائعة تعزز نمو النباتات وتكوين الفطريات الجذرية. تم اقتراح مفهوم “التربة المثبطة للفطريات الجذرية” للتربة التي تعيق المجتمعات الميكروبية امتصاص العناصر الغذائية الذي تسهله خيوط الفطريات الجذرية (سفينينغسن وآخرون، 2018). قد تسهم التغيرات في المجتمعات الميكروبية المرتبطة بالخيوط في التحديات التي تواجه عند تكرار تجارب الفطريات الجذرية. أظهرت الدراسات الحديثة أن البروتوزوا التي تتغذى على البكتيريا المرتبطة بالخيوط يمكن أن تؤثر على امتصاص العناصر الغذائية بواسطة الفطريات الجذرية، مما يبرز الطبيعة المعقدة لهذه التفاعلات متعددة التغذية (روزموش وآخرون، 2022). بالإضافة إلى ذلك، تم اقتراح أن الفطريات الجذرية تحتوي على سلالة قديمة من الفيروسات الفطرية (إزاوا وآخرون، 2023)، وأشارت دراسة حديثة إلى إصابة حديثة واحدة فقط لجينوم فيروس dsDNA كبير في جينوم سلالة فطرية جذرية (تشاو وآخرون، 2023). ومع ذلك، لا يزال دور الفيروسات في تنظيم وظيفة الفطريات الجذرية، بما في ذلك تأثيرها على التركيب الجيني للميكوبايوما الفطري الجذري، غير مفهوم جيدًا ويتطلب مزيدًا من البحث. بشكل عام، تؤكد هذه الدراسات على الحاجة إلى مراعاة مفهوم “الهولوبيونت” عند محاولة فهم وظيفة الفطريات الجذرية (فاندنكوورنهايس وآخرون، 2015؛ وانغ وآخرون، 2023ب).
V. التعايش الفطري في النظم البيئية
1. تحديد الأهمية النسبية للفطريات الجذرية
لقد أكدت العديد من الدراسات على أهمية الفطريات الميكوريزية في تعزيز نمو النباتات ووظيفة النظام البيئي. ومع ذلك، هناك نقص في الأبحاث التي تحدد بوضوح علاقتها النسبية.

الشكل 6 الأهمية النسبية للارتباطات الفطرية الجذرية لإنتاجية النباتات وأداء النظام البيئي في أنواع النظم البيئية المختلفة: الغابات، المراعي، والأنظمة الزراعية. تهيمن أنواع النظم البيئية المختلفة على ارتباطات فطرية جذرية مختلفة. AM، الفطريات الجذرية الكيسية؛ EM، الفطريات الجذرية الخارجية؛ NM، غير الفطرية الجذرية.
الأهمية مقارنةً مع مجموعات الميكروبات الأساسية الأخرى، بما في ذلك البكتيريا، والفطريات الداخلية والفطريات المتحللة، والطلائعيات، والعتائق، والفيروسات، والميكروبيات والميكروبيات المتوسطة في التربة، بالإضافة إلى تفاعلاتها. تعتمد الأهمية النسبية للفطريات الجذرية على إنتاجية النبات وأداء النظام البيئي على نوع النظام البيئي (الشكل 6). نقترح أن الروابط الفطرية، بما في ذلك بكتيريا المساعدة الفطرية، تلعب دورًا رئيسيًا في الغابات، تليها المراعي والأراضي الزراعية (الشكل 6). تدعم هذه الفرضية الملاحظات التي تشير إلى أن الفطريات الجذرية الخارجية مسؤولة عن جزء كبير من اكتساب النيتروجين في العديد من الغابات، خاصة في التربة الفقيرة بالمغذيات (هوببي وهوببي، 2006). كشفت الدراسات الحديثة عن وجود ارتباط قوي بين إنتاجية الأشجار، وتركيب الفطريات الجذرية الخارجية، وتنوع بيتا (أنتوني وآخرون، 2022؛ س. لو وآخرون، 2023)، مما يعزز هذه الملاحظة. ومع ذلك، من الضروري الاعتراف بأن أهمية الروابط الفطرية تعتمد على توفر المغذيات، وتتناقص أهميتها في التربة الغنية بالمغذيات. كما تعتمد التأثيرات أيضًا على النبات.
هوية الأنواع، حيث أن بعض النباتات، مثل مجموعة واسعة من أعضاء عائلة الكرنب أو عائلة البروتي، لا تشارك في ارتباطات مع الفطريات الميكوريزية.
هوية الأنواع، حيث أن بعض النباتات، مثل مجموعة واسعة من أعضاء عائلة الكرنب أو عائلة البروتي، لا تشارك في ارتباطات مع الفطريات الميكوريزية.
في المراعي، تظهر الارتباطات الفطرية تنوعًا أكبر. الفطريات الميكوريزية المتكافلة (AMF)، التي تعد الفطريات الميكوريزية السائدة في المراعي، لها تأثير مهم على وظيفة المراعي من خلال تغيير التفاعلات التنافسية بين الأنواع النباتية، مما يؤدي إلى زيادة أو انخفاض تنوع النباتات (فان دير هايدن وآخرون، 1998؛ هارتنيت وآخرون، 1999). ومع ذلك، فإن الأهمية النسبية للميكورايزا في إنتاج الكتلة الحيوية في المراعي أقل، حيث يبقى إنتاج الكتلة الحيوية غالبًا مشابهًا مع أو بدون الميكورايزا، مما يدل على تغييرات في تركيب الأنواع النباتية (فان دير هايدن وآخرون، 1998؛ هارتنيت وآخرون، 1999). في الأنظمة الزراعية والمضطربة بشدة، من المتوقع أن تكون تكافؤ الميكورايزا ذات الأهمية النسبية الأقل. عادةً ما تحتوي هذه الأنظمة على وفرة أقل أو غياب للارتباطات الميكوريزية، بينما تكون مسببات الأمراض الفطرية والبكتيرية أكثر وفرة (لابويري وآخرون، 2023). غالبًا ما تهيمن الأنواع النباتية غير الميكوريزية والنباتات الروديرالية على هذه الأنظمة (ريد، 1991). ومع ذلك، من الضروري الاعتراف بأن الأنواع النباتية المتزامنة داخل نفس النظام البيئي قد تظهر استجابة ميكوريزية مختلفة، وأن بعض الأنواع النباتية والمحاصيل في الحقول الزراعية تستفيد من تكافؤ الميكورايزا. علاوة على ذلك، تم ملاحظة استثناءات، مثل الأنواع النباتية في التربة المتآكلة بشدة والفقيرة بالمغذيات في أستراليا، التي تمتلك جذور عنقودية ولا تشكل ارتباطات مع الفطريات الميكوريزية (لامبرز وآخرون، 2008). من الجدير بالذكر أن النباتات غير الميكوريزية ليست دائمًا مقيدة بالتربة منخفضة المغذيات؛ على سبيل المثال، تتواجد عائلة البروتياسية في معظم نصف الكرة الجنوبي. باختصار، تتقلب الأهمية النسبية لتكافؤ الميكورايزا في النظم البيئية اعتمادًا على نوع النظام البيئي المحدد، وتوافر المغذيات، وتركيب الأنواع النباتية. من المهم الاعتراف بالتحيز الجغرافي الحالي في أبحاث الميكورايزا، التي تركز بشكل أساسي على المناطق المعتدلة والشمالية وقد تتجاهل التنوع الأكبر لاستراتيجيات الميكورايزا وغير الميكورايزا القابلة للحياة في النظم البيئية شبه الاستوائية والاستوائية. معالجة هذه الفجوة أمر ضروري للحصول على فهم أكثر شمولاً للارتباطات الميكوريزية على مستوى العالم. بالإضافة إلى ذلك، هناك اتجاه نحو تفسير البيانات بشكل مفرط، مما يمكن أن يعيق التقدم في بعض مجالات البحث (كويبر وجانسا، 2023).
2. ربط الجينات بالخصائص البيئية
تقدم توافر جينومات الفطريات الميكوريزية المتزايد (Lebreton et al., 2021) فرصًا غير مسبوقة للتحقيق في التنوع الوظيفي لمجتمعات الفطريات في التربة. يمكن تحقيق ذلك من خلال التوصيف الدقيق والخبراء للبيانات البيئية الميتاباركويدية (Anthony et al., 2022) والميتابيونوميات (HaghDoust et al., 2023) والميتا ترانسكريبتوميات (Auer et al., 2023; Zeng et al., 2023). تم استخدام النهج الأخير لفحص تعبير الجينات في أطراف جذور الفطريات الميكوريزية في البيئات الطبيعية، بما في ذلك تأثيرات ترسيب النيتروجين الناتج عن الأنشطة البشرية على استقلاب الأشجار الميكوريزية (Liao et al., 2018; Law et al., 2022; Rivera Pérez et al., 2022). أظهر Anthony et al. (2022) أن نمو الأشجار كان مرتبطًا بعدد نسخ الجينات المعنية بامتصاص النيتروجين واستيعابه في الفطريات الميكوريزية المرتبطة. من خلال دمج الميتاباركويد العالي الإنتاجية لأنواع الفطريات المقيمة في منطقة معينة
يمكن تقييم التعبير الجيني الدقيق لكل شبكة فطرية تمتد تحت نوع شجرة من خلال موقع الغابة التجريبية مع تسلسل مستخلصات RNA حقيقية النواة من التربة (Auer et al., 2023; Zeng et al., 2023). سيساعد استخدام الميتا ترانسكرپتوميات أيضًا في فك شفرة الأهمية النسبية لكل نوع فطري ضمن الكائنات الحية المختلفة في التربة في قياس الجينات المعنية في تغذية النباتات، ودورة الكربون، ومقاومة الأمراض، والتكيف مع الضغوط.
يمكن تقييم التعبير الجيني الدقيق لكل شبكة فطرية تمتد تحت نوع شجرة من خلال موقع الغابة التجريبية مع تسلسل مستخلصات RNA حقيقية النواة من التربة (Auer et al., 2023; Zeng et al., 2023). سيساعد استخدام الميتا ترانسكرپتوميات أيضًا في فك شفرة الأهمية النسبية لكل نوع فطري ضمن الكائنات الحية المختلفة في التربة في قياس الجينات المعنية في تغذية النباتات، ودورة الكربون، ومقاومة الأمراض، والتكيف مع الضغوط.
VI. التعايش الفطري الجذري وديناميات مجتمع النباتات
يتم التعرف بشكل متزايد على أن ميكروبات التربة، بما في ذلك الفطريات الميكوريزية، يمكن أن تتوسط في تفاعلات النباتات وبالتالي قد تساهم في علاقات التنوع البيولوجي والإنتاجية، مما يشكل مجتمعات النباتات (كليرونوموس، 2002؛ فان دير هايدن وآخرون، 2008؛ شنيتزر وآخرون، 2011). لقد أظهرت التجارب التلاعبية التي تستهدف وفرة ووجود الفطريات الميكوريزية المتكافلة والفطريات الميكوريزية الإكليلية تأثيراتها العميقة على تنوع النباتات وبنية المجتمع بشكل متسق (ليانغ وآخرون، 2019). ومن الجدير بالذكر أن قلة الدراسات التي تتلاعب بأنواع أخرى من التعايشات الميكوريزية، مثل الفطريات الميكوريزية الجذرية والفطريات الميكوريزية الخارجية، للتحقيق في تأثيراتها على نمو النباتات وتأسيسها تستدعي مزيدًا من التحقيق والاهتمام البحثي. ومن الجدير بالذكر أن الغالبية العظمى من الدراسات في هذا المجال قصيرة الأجل نسبيًا، وعادة ما تمتد
لقد أبرزت مجموعة متزايدة من الأدلة من الاستطلاعات العالمية أهمية الارتباطات الفطرية في تشكيل هيكل مجتمع النباتات، وتنوعها، وإنتاجيتها. وقد قدمت جردات الغابات على نطاق واسع رؤى قيمة حول دور أنواع الفطريات الميكوريزية في هيكلة النظم البيئية الغابية وتنوع الأشجار. على سبيل المثال، قام كارترون وآخرون (2022) بتحليل 82000 قطعة غابية عبر الولايات المتحدة ووجدوا أن الغابات التي تهيمن عليها الفطريات الميكوريزية الخارجية أو الفطريات الميكوريزية الداخلية تميل إلى إظهار تنوع أقل في الأشجار. بالمقابل، دعمت الغابات التي تحتوي على مزيج من الاستراتيجيات الميكوريزية عددًا أكبر من أنواع الأشجار. مؤخرًا، أظهر ي. هـ. لو وآخرون (2023)، باستخدام نفس قاعدة البيانات، أن القطع الميكوريزية المختلطة أظهرت إنتاجية أكبر من القطع التي كانت فيها الأنواع الشجرية المرتبطة بالفطريات الميكوريزية الداخلية أو الخارجية مهيمنة. تشير هذه النتائج إلى أن التعايش بين كلا النوعين من الفطريات الميكوريزية يمكن أن يكمل بعضهما البعض ويعزز وظيفة النظام البيئي. بالإضافة إلى ذلك، كانت الآثار الإيجابية لاستراتيجيات الفطريات الميكوريزية المختلطة على إنتاجية الغابات أكثر وضوحًا في القطع ذات غنى الأنواع الشجرية المنخفض (ي. هـ. لو وآخرون، 2023). ومع ذلك، يلعب نوع الفطريات الميكوريزية للأنواع النباتية المهيمنة أيضًا دورًا حاسمًا في تسهيل أو قمع الأنواع الفرعية أو الأنواع تحت الشجر. على سبيل المثال، وجدت دراسة أجريت في جبال سموكي الكبرى في الولايات المتحدة علاقة إيجابية بين غنى الأنواع العشبية وعدد الأنواع الشجرية المرتبطة بالفطريات الميكوريزية الداخلية (نيو مان & ريديل، 1988). أجرى دينغ وآخرون (2023) تجربة واسعة النطاق لتنوع الأشجار في الصين ولاحظوا أن العلاقة بين غنى الأنواع الشجرية والإنتاجية تعتمد على نوع الفطريات الميكوريزية. على وجه التحديد،
تمت ملاحظة علاقة إيجابية بين غنى أشجار AM والإنتاجية، في حين لم يتم ملاحظة مثل هذه العلاقة لأشجار ECM. على النقيض من ذلك، أظهر S. Luo وآخرون (2023) علاقة إيجابية بين تنوع الفطريات الجذرية والإنتاجية فوق الأرض في نظام بيئي للغابات دائمة الخضرة ذات الأوراق العريضة في المناطق شبه الاستوائية. يمكن أن تساعد أساليب الميتا ترانسكرپتوميات والميتا بروتيوميات في تحديد الآليات والشبكات الجينية الرئيسية المعنية في الوساطة بين التنوع البيولوجي وعمل النظام البيئي. علاوة على ذلك، يمكن أن يسهل وجود أو غياب المتعايشات الفطرية أو يعيق إقامة الأنواع الغازية (مثل Pringle وآخرون، 2009؛ Dickie وآخرون، 2010).
تمت ملاحظة علاقة إيجابية بين غنى أشجار AM والإنتاجية، في حين لم يتم ملاحظة مثل هذه العلاقة لأشجار ECM. على النقيض من ذلك، أظهر S. Luo وآخرون (2023) علاقة إيجابية بين تنوع الفطريات الجذرية والإنتاجية فوق الأرض في نظام بيئي للغابات دائمة الخضرة ذات الأوراق العريضة في المناطق شبه الاستوائية. يمكن أن تساعد أساليب الميتا ترانسكرپتوميات والميتا بروتيوميات في تحديد الآليات والشبكات الجينية الرئيسية المعنية في الوساطة بين التنوع البيولوجي وعمل النظام البيئي. علاوة على ذلك، يمكن أن يسهل وجود أو غياب المتعايشات الفطرية أو يعيق إقامة الأنواع الغازية (مثل Pringle وآخرون، 2009؛ Dickie وآخرون، 2010).
VII. تطبيقات الفطريات الجذرية
هناك اهتمام كبير في تعزيز الاستدامة الزراعية واستخدام الأساليب البيولوجية لتعزيز إنتاجية المحاصيل، فضلاً عن تسهيل استعادة النظام البيئي واستدامته. تعتبر الفطريات الميكوريزية ذات إمكانيات مهمة حيث يُعرف عنها تعزيز نمو النباتات وتوفير الحماية ضد الضغوط الحيوية وغير الحيوية في الزراعة والغابات. أظهرت دراسة تحليلية حديثة أجراها أفيريل وآخرون (2022) أن استعادة الميكروبيوم التربة الأصلي يمكن أن تؤدي إلى تسريع متوسط لـ
تم إجراء العديد من الدراسات التي تقيم استجابات نمو النباتات للتطعيم بالفطريات الجذرية في ظروف خاضعة للتحكم بشكل كبير في البيوت الزجاجية، وغالبًا ما تستخدم تربة معقمة وذات مغذيات قليلة. يمكن أن يكون من الصعب مقارنة نتائج هذه الدراسات بالظروف الميدانية. لجعل التطعيم الميداني عمليًا ومقبولًا على نطاق واسع، من الضروري إجراء تجارب تطعيم ميدانية واستخدام تقنيات تطعيم مناسبة يمكن تنفيذها بسهولة من قبل المزارعين أو الحراجيين. يمكن تغليف الفطريات الجذرية على البذور، أو تطبيقها كطبقة أو كحبوب “سماد”، أو زراعتها مع (أو تحت) البذور (فوساتكا وآخرون، 2012؛ أوليفيرا وآخرون، 2019؛ روتشا وآخرون، 2019). كما تم التوصية بإضافة تربة طبيعية تحتوي على تجمعات محلية من الفطريات الجذرية (وغيرها من الميكروبات) إلى المواقع المجاورة أو مشاتل الأشجار، حيث قد تكون هذه اللقاحات أكثر تكيفًا مع الظروف المحلية. علاوة على ذلك، يمكن أن يغير التطعيم الميداني مجتمعات الفطريات الجذرية الأصلية (باسيرو وهيجري، 2022)، وهناك مخاوف بشأن العواقب غير المقصودة المحتملة لاستخدام
المستعمرات غير الأصلية، التي قد تشكل تهديدًا لتنوع التربة والنباتات ووظائف النظام البيئي (هارت وآخرون، 2018). قبل إدخال المستعمرات إلى السوق، من الضروري الالتزام بمجموعة من معايير الجودة الأساسية من أجل تقييم صلاحية وفعالية المستعمرات تحت ظروف محكومة (سالومون وآخرون، 2022). بالإضافة إلى ذلك، يجب تطوير أدوات تمكّن المزارعين أو الحراجيين من التنبؤ بالظروف التي تكون فيها التلقيح مفيدًا وقابلًا للتطبيق اقتصاديًا (نوع المحصول، جينوم المحصول، نوع التربة، توفر المغذيات، وظروف الطقس). على سبيل المثال، وجدت دراسة حديثة للتلقيح الميداني في 54 تربة زراعية جيدة التسميد في سويسرا أن معدل نجاح تلقيح الفطريات الميكوريزية كان أفضل ما يمكن التنبؤ به من خلال وجود جينات مسببات الأمراض الفطرية في التربة، مما يشير إلى أن التلقيح يمكن أن يساعد أيضًا في تقليل الضغط البيولوجي (لوتز وآخرون، 2023). بشكل عام، تمتلك الفطريات الميكوريزية إمكانات كبيرة كعوامل بيولوجية للإنتاج المستدام. ومع ذلك، من الضروري توضيح الظروف المحددة التي يكون فيها التلقيح أكثر فعالية. سيكون الجمع بين تقنيات تسلسل الحمض النووي الريبوزي عالي الإنتاجية، وتسلسل الحمض النووي الريبوزي، والميتابروتيوميات نهجًا قويًا لتقييم نجاح تلقيح الفطريات الميكوريزية وكشف ما إذا كان التلقيح الميداني يعزز وظائف النظام البيئي المحددة. على سبيل المثال، يمكن ربط مستوى النسخ (أو البروتينات) التي تشفر ناقلات المغذيات (أو السمات الوظيفية الأخرى التي يمكن تتبعها إلى الفطريات الميكوريزية) بوظائف النظام البيئي المحددة، مثل إنتاجية النبات، ومحتوى المغذيات، وتحلل المواد العضوية، أو مقاومة الأمراض/الضغط. يمكن أن تساعد نهج المولتي أوميكس أيضًا في كشف الأهمية النسبية لسمات الفطريات الميكوريزية مقارنةً بسمات أخرى (مثل امتصاص المغذيات بواسطة جذور النباتات أو تحلل المواد العضوية بواسطة الكائنات الحية الدقيقة غير الميكوريزية في التربة). ستعتمد مثل هذه الدراسات على مزيج من الأساليب التقليدية في علم البيئة/الزراعة (مثل تقييم إنتاجية النبات، والتحلل باستخدام الأوراق أو أكياس الشاي، واختبارات قمع الأمراض، والأقسام الهيكلية التي يمكن الوصول إليها فقط بواسطة خيوط الفطريات، أو تضمين بقع المغذيات العضوية) والأساليب الجينومية البيئية. على سبيل المثال، كشف ليو وآخرون (2023) أن تطبيق المبيدات مرتبط بجينات مسؤولة عن تثبيت النيتروجين، ونقل الحديد، واستقلاب الفوسفور، مما يمكّن من توضيح كيفية تأثير الضغوط البيئية على وظائف التربة. ومع ذلك، لا تزال المنهجية التحليلية المطلوبة لدمج أساليب علم البيئة/الزراعة مع بيانات الجينوميات، والترانسكريبتوميات، والبروتيوميات، والميتابولوميات، والفينوميات في مراحلها الأولى. نظرًا لطبيعتها، فإن بيانات -أوميكس متغيرة وعشوائية بشكل جوهري، مما يؤدي إلى عدة مشكلات عند محاولة مقارنتها أو إعادة إنتاجها في البيئات الطبيعية. من المحتمل أن يعتمد دمج بيانات المولتي أوميكس على نهج الشبكات المشتركة أو الشبكات المتزامنة، مثل تلك التي تم تطويرها في الأساليب المعتمدة على الغابات العشوائية (هويينغ-ثو وآخرون، 2010). في هذا النهج، يمكن دمج أنواع متعددة من بيانات -أوميكس والبيانات البيئية من خلال تحديد البروتينات والنسخ التي تتغير وفرتها بطريقة منسقة عبر مجموعة من الظروف التجريبية (مثل إنتاجية المحاصيل) أو العينات.
المستعمرات غير الأصلية، التي قد تشكل تهديدًا لتنوع التربة والنباتات ووظائف النظام البيئي (هارت وآخرون، 2018). قبل إدخال المستعمرات إلى السوق، من الضروري الالتزام بمجموعة من معايير الجودة الأساسية من أجل تقييم صلاحية وفعالية المستعمرات تحت ظروف محكومة (سالومون وآخرون، 2022). بالإضافة إلى ذلك، يجب تطوير أدوات تمكّن المزارعين أو الحراجيين من التنبؤ بالظروف التي تكون فيها التلقيح مفيدًا وقابلًا للتطبيق اقتصاديًا (نوع المحصول، جينوم المحصول، نوع التربة، توفر المغذيات، وظروف الطقس). على سبيل المثال، وجدت دراسة حديثة للتلقيح الميداني في 54 تربة زراعية جيدة التسميد في سويسرا أن معدل نجاح تلقيح الفطريات الميكوريزية كان أفضل ما يمكن التنبؤ به من خلال وجود جينات مسببات الأمراض الفطرية في التربة، مما يشير إلى أن التلقيح يمكن أن يساعد أيضًا في تقليل الضغط البيولوجي (لوتز وآخرون، 2023). بشكل عام، تمتلك الفطريات الميكوريزية إمكانات كبيرة كعوامل بيولوجية للإنتاج المستدام. ومع ذلك، من الضروري توضيح الظروف المحددة التي يكون فيها التلقيح أكثر فعالية. سيكون الجمع بين تقنيات تسلسل الحمض النووي الريبوزي عالي الإنتاجية، وتسلسل الحمض النووي الريبوزي، والميتابروتيوميات نهجًا قويًا لتقييم نجاح تلقيح الفطريات الميكوريزية وكشف ما إذا كان التلقيح الميداني يعزز وظائف النظام البيئي المحددة. على سبيل المثال، يمكن ربط مستوى النسخ (أو البروتينات) التي تشفر ناقلات المغذيات (أو السمات الوظيفية الأخرى التي يمكن تتبعها إلى الفطريات الميكوريزية) بوظائف النظام البيئي المحددة، مثل إنتاجية النبات، ومحتوى المغذيات، وتحلل المواد العضوية، أو مقاومة الأمراض/الضغط. يمكن أن تساعد نهج المولتي أوميكس أيضًا في كشف الأهمية النسبية لسمات الفطريات الميكوريزية مقارنةً بسمات أخرى (مثل امتصاص المغذيات بواسطة جذور النباتات أو تحلل المواد العضوية بواسطة الكائنات الحية الدقيقة غير الميكوريزية في التربة). ستعتمد مثل هذه الدراسات على مزيج من الأساليب التقليدية في علم البيئة/الزراعة (مثل تقييم إنتاجية النبات، والتحلل باستخدام الأوراق أو أكياس الشاي، واختبارات قمع الأمراض، والأقسام الهيكلية التي يمكن الوصول إليها فقط بواسطة خيوط الفطريات، أو تضمين بقع المغذيات العضوية) والأساليب الجينومية البيئية. على سبيل المثال، كشف ليو وآخرون (2023) أن تطبيق المبيدات مرتبط بجينات مسؤولة عن تثبيت النيتروجين، ونقل الحديد، واستقلاب الفوسفور، مما يمكّن من توضيح كيفية تأثير الضغوط البيئية على وظائف التربة. ومع ذلك، لا تزال المنهجية التحليلية المطلوبة لدمج أساليب علم البيئة/الزراعة مع بيانات الجينوميات، والترانسكريبتوميات، والبروتيوميات، والميتابولوميات، والفينوميات في مراحلها الأولى. نظرًا لطبيعتها، فإن بيانات -أوميكس متغيرة وعشوائية بشكل جوهري، مما يؤدي إلى عدة مشكلات عند محاولة مقارنتها أو إعادة إنتاجها في البيئات الطبيعية. من المحتمل أن يعتمد دمج بيانات المولتي أوميكس على نهج الشبكات المشتركة أو الشبكات المتزامنة، مثل تلك التي تم تطويرها في الأساليب المعتمدة على الغابات العشوائية (هويينغ-ثو وآخرون، 2010). في هذا النهج، يمكن دمج أنواع متعددة من بيانات -أوميكس والبيانات البيئية من خلال تحديد البروتينات والنسخ التي تتغير وفرتها بطريقة منسقة عبر مجموعة من الظروف التجريبية (مثل إنتاجية المحاصيل) أو العينات.
ثامناً. الاتجاهات المستقبلية
من خلال تطبيق علم الجينوم وغيرها من الأساليب -وميكس على دراسة التعايش الفطري، حصلنا على فهم أعمق للعمليات الجزيئية والفسيولوجية والبيئية التي تكمن وراء هذه.
الارتباطات التبادلية. يجب أن تُترجم هذه المعرفة إلى تطبيقات عملية تعزز من وظائف النظام البيئي والحفاظ عليه، وتساهم في التخفيف العام من تغير المناخ، وتضمن الأراضي الزراعية المستدامة. لا تزال هناك العديد من التحديات طويلة الأمد في استغلال علم الجينوم وأدوات الأوميكس الأخرى لتعميق فهمنا للتعايشات الفطرية، بما في ذلك تطورها، وتطورها، ووظيفتها، وبيئتها (الجدول 1).
الارتباطات التبادلية. يجب أن تُترجم هذه المعرفة إلى تطبيقات عملية تعزز من وظائف النظام البيئي والحفاظ عليه، وتساهم في التخفيف العام من تغير المناخ، وتضمن الأراضي الزراعية المستدامة. لا تزال هناك العديد من التحديات طويلة الأمد في استغلال علم الجينوم وأدوات الأوميكس الأخرى لتعميق فهمنا للتعايشات الفطرية، بما في ذلك تطورها، وتطورها، ووظيفتها، وبيئتها (الجدول 1).
1. توسيع مجموعة الجينومات الفطرية الميكوريزية
تمثل المجموعة الحالية من الجينومات المتسلسلة للفطريات الجذرية فقط جزءًا (حوالي
2. تعزيز فهمنا للعمليات البيولوجية الأساسية
يمكن أن تساعد الأساليب مثل تحليل التعبير الجيني المكاني ثلاثي الأبعاد للخلايا المفردة الجذور الميكوريزية، وتحليل التعبير المشترك القائم على النسخ، والبروتيوميات، ودراسات تفاعل البروتينات في ربط الجينات بالعمليات البيولوجية المعروفة، والتنبؤ بالعلاقات التطورية وكذلك التكيف البيئي (انظر الصندوق 1). يمكن أن تساعد الأساليب التي تتضمن حسابات طي البروتين عالية الإنتاجية (مثل AlphaFold؛ Jumper وآخرون، 2021) في تحديد هياكل البروتينات المشفرة بواسطة الجينات المنظمة للتعايش ذات الوظائف غير المعروفة، مثل البروتينات المفرزة الشبيهة بالعوامل (Teulet وآخرون، 2023). فهم أصل هذه البروتينات الفعالة أمر بالغ الأهمية، سواء تطورت من أسلاف سابروتروفية أو ظهرت حديثًا لتسهيل الاستعمار داخل أنسجة النبات. من الضروري معالجة مسارات الإشارة والجزيئات المحددة المعنية في التطور التعايشي والتواصل الجزيئي المعقد بين الشركاء الميكوريزيين. حاليًا، الجينات الرئيسية التي تحفز تطوير التعايش الميكوريزي الخارجي في كل من الشركاء الفطريين والنباتيين (إن وجدت) غير معروفة. علاوة على ذلك، من المهم التحقيق في العوامل الكامنة وراء اختلاف نطاقات المضيفين بين الأنواع المختلفة من الميكوريزا (Lebreton وآخرون، 2021؛ Tang وآخرون، 2021). لماذا يمكن لبعض الأنواع الفطرية الميكوريزية استعمار مجموعة واسعة من المضيفين، بينما تظهر أنواع أخرى تفضيلات أكثر تقييدًا، لا يزال جانبًا مثيرًا يتطلب مزيدًا من الاستكشاف.
3. اكتشاف تنوع الفطريات الجذرية
على الرغم من تحقيق تقدم، خاصة في النظم البيئية المعتدلة والقطبية (Peay et al.، 2016)، لا تزال هناك مناطق شاسعة من
الصندوق 1. أسئلة موضوعية في جينوميات الارتباطات الفطرية.
ما هي الآليات الجينومية التي تكمن وراء التخصص والتعرف على مضيفي النباتات والفطريات الميكوريزية وكيف تساهم هذه الآليات في إقامة وصيانة التعايش؟
كيف تؤثر الخصائص الفريدة في مجموعة الجينات للفطريات الميكوريزية على قدرتها على التفاعل مع أنواع نباتية مختلفة، وما هي الآثار المترتبة على تنوع النباتات ووظيفة النظام البيئي؟
كيف تُسهم الجينوميات في فهمنا للشبكات التنظيمية المعقدة المشاركة في التعايش الفطري، بما في ذلك الجينات والمسارات المسؤولة عن الإشارات، ونقل المغذيات، وإقامة التبادلية؟
ما هو الأساس الجينومي لاستجابة الفطريات الميكوريزية للضغوط البيئية، مثل التغيرات في درجة حموضة التربة، والجفاف، والمبيدات الحشرية، والبلاستيك، وتوافر المغذيات، وتقلبات المناخ، وكيف يؤثر ذلك على تفاعلاتها التبادلية مع النباتات؟
كيف يمكن أن تسهم الجينوميات في فهم تطور التعايش الفطري والتغيرات الجينية التي حدثت على مر الزمن، مما أدى إلى تنوع واسع من الارتباطات الفطرية التي لوحظت في الطبيعة؟
ما هي الاستراتيجيات المعتمدة على الجينوم لهندسة أو تعزيز التعايش الفطري في المحاصيل لتحسين كفاءة امتصاص العناصر الغذائية، وتحمل الإجهاد، والأداء العام للنبات؟
ما هو دور التعديلات الوراثية فوق الجينية في التوسط في تفاعلات الفطريات الميكوريزية النباتية، وكيف تؤثر التغيرات الوراثية فوق الجينية على نتائج العلاقة التبادلية؟
هل يمكن أن تسلط الجينوميات المقارنة الضوء على الأساس الجيني للاختلافات الوظيفية بين أنواع الفطريات الميكوريزية المختلفة، وكيف يمكن تطبيق هذه المعرفة لتحسين الشركاء الفطريين لأهداف زراعية أو بيئية محددة؟
كيف يمكن أن تسهم الجينوميات، والميتاترنسكريبتوميات، والميتابروتيوميات في فهم دور الفطريات الميكوريزية في دورة الكربون، وصحة التربة، والخدمات البيئية بشكل عام، وكيف يمكن أن تُستخدم هذه المعرفة في توجيه استراتيجيات إدارة البيئة؟
كوكبنا الذي لا يزال غير مستكشف إلى حد كبير. وقد ظهر توافق في الآراء بأن معظم هذه التنوعات، المعروفة وغير المعروفة، توجد في المناطق الاستوائية. لا يزال تنوع الفطريات في الصحاري والمناطق القطبية غير موصوف، لكن تجمعاتها تتغير بسرعة، مدفوعة بتغير المناخ الناتج عن الأنشطة البشرية (بالدريان وآخرون، 2023). نحن نحث الباحثين على دراسة الجغرافيا الحيوية لمختلف الارتباطات الفطرية في هذه البيئات. التهديدات الأخرى لبيئات التربة، مثل الميكروبلاستيك، يمكن أن تغير أو حتى تخلق موائل جديدة للفطريات الميكوريزية (لايفهايت وآخرون، 2021). بفضل مبادرات مثل جمعية حماية الشبكات تحت الأرض، سيسمح لنا فهم أفضل لهذه الفطريات الميكوريزية بتحسين التنبؤات حول التأثيرات البشرية على تركيب الميكروبيوم، والقدرة على التحمل، والتطور، والإنتاجية (أفيريل وآخرون، 2022؛ بالدريان وآخرون، 2023) (الصندوق 1). يجب أن يرافق الجرد العالمي للفطريات الميكوريزية، مثل GlobalFungi (فيتروفسكي وآخرون، 2020)، أطلس مصمم لاستكشاف الوظائف الميكروبية في الزمان والمكان (سيتم وصفه لاحقًا). نظرًا لزيادة عدد التهديدات على بيئة التربة، هناك حاجة ملحة لوصف الميكروبيومات الصحية في النظم البيئية التي لم تتأثر حاليًا بالتهديدات البيئية (إن وجدت). وهذا يوفر قاعدة لفهم كيف أن هذه الميكروبات
قد تستجيب المجتمعات للتغيرات العالمية. كما سيساعد ذلك في تحديد نقاط التحول التي يمكن أن تغير بشكل دائم تكوين ووظيفة الميكروبيوم التربة. على سبيل المثال، أظهرت الجرد الكبيرة للغابات الأوروبية أن ترسيب النيتروجين يسبب نقاط تحول في تكوين ووظيفة الفطريات الميكوريزية (فان دير ليندي وآخرون، 2018). على الرغم من أن المئات من الدراسات قد وصفت تكوين الميكوبيوما الميكوريزي في أنظمة بيئية مختلفة، إلا أن القليل معروف عن الترابط بين المكونات المختلفة وتحولات المجتمع الميكوريزي خلال التعاقب وعلى طول تدرج المناطق التي تهيمن عليها المراعي والشجيرات والأشجار.
ما هي الآليات الجينومية التي تكمن وراء التخصص والتعرف على مضيفي النباتات والفطريات الميكوريزية وكيف تساهم هذه الآليات في إقامة وصيانة التعايش؟
كيف تؤثر الخصائص الفريدة في مجموعة الجينات للفطريات الميكوريزية على قدرتها على التفاعل مع أنواع نباتية مختلفة، وما هي الآثار المترتبة على تنوع النباتات ووظيفة النظام البيئي؟
كيف تُسهم الجينوميات في فهمنا للشبكات التنظيمية المعقدة المشاركة في التعايش الفطري، بما في ذلك الجينات والمسارات المسؤولة عن الإشارات، ونقل المغذيات، وإقامة التبادلية؟
ما هو الأساس الجينومي لاستجابة الفطريات الميكوريزية للضغوط البيئية، مثل التغيرات في درجة حموضة التربة، والجفاف، والمبيدات الحشرية، والبلاستيك، وتوافر المغذيات، وتقلبات المناخ، وكيف يؤثر ذلك على تفاعلاتها التبادلية مع النباتات؟
كيف يمكن أن تسهم الجينوميات في فهم تطور التعايش الفطري والتغيرات الجينية التي حدثت على مر الزمن، مما أدى إلى تنوع واسع من الارتباطات الفطرية التي لوحظت في الطبيعة؟
ما هي الاستراتيجيات المعتمدة على الجينوم لهندسة أو تعزيز التعايش الفطري في المحاصيل لتحسين كفاءة امتصاص العناصر الغذائية، وتحمل الإجهاد، والأداء العام للنبات؟
ما هو دور التعديلات الوراثية فوق الجينية في التوسط في تفاعلات الفطريات الميكوريزية النباتية، وكيف تؤثر التغيرات الوراثية فوق الجينية على نتائج العلاقة التبادلية؟
هل يمكن أن تسلط الجينوميات المقارنة الضوء على الأساس الجيني للاختلافات الوظيفية بين أنواع الفطريات الميكوريزية المختلفة، وكيف يمكن تطبيق هذه المعرفة لتحسين الشركاء الفطريين لأهداف زراعية أو بيئية محددة؟
كيف يمكن أن تسهم الجينوميات، والميتاترنسكريبتوميات، والميتابروتيوميات في فهم دور الفطريات الميكوريزية في دورة الكربون، وصحة التربة، والخدمات البيئية بشكل عام، وكيف يمكن أن تُستخدم هذه المعرفة في توجيه استراتيجيات إدارة البيئة؟
كوكبنا الذي لا يزال غير مستكشف إلى حد كبير. وقد ظهر توافق في الآراء بأن معظم هذه التنوعات، المعروفة وغير المعروفة، توجد في المناطق الاستوائية. لا يزال تنوع الفطريات في الصحاري والمناطق القطبية غير موصوف، لكن تجمعاتها تتغير بسرعة، مدفوعة بتغير المناخ الناتج عن الأنشطة البشرية (بالدريان وآخرون، 2023). نحن نحث الباحثين على دراسة الجغرافيا الحيوية لمختلف الارتباطات الفطرية في هذه البيئات. التهديدات الأخرى لبيئات التربة، مثل الميكروبلاستيك، يمكن أن تغير أو حتى تخلق موائل جديدة للفطريات الميكوريزية (لايفهايت وآخرون، 2021). بفضل مبادرات مثل جمعية حماية الشبكات تحت الأرض، سيسمح لنا فهم أفضل لهذه الفطريات الميكوريزية بتحسين التنبؤات حول التأثيرات البشرية على تركيب الميكروبيوم، والقدرة على التحمل، والتطور، والإنتاجية (أفيريل وآخرون، 2022؛ بالدريان وآخرون، 2023) (الصندوق 1). يجب أن يرافق الجرد العالمي للفطريات الميكوريزية، مثل GlobalFungi (فيتروفسكي وآخرون، 2020)، أطلس مصمم لاستكشاف الوظائف الميكروبية في الزمان والمكان (سيتم وصفه لاحقًا). نظرًا لزيادة عدد التهديدات على بيئة التربة، هناك حاجة ملحة لوصف الميكروبيومات الصحية في النظم البيئية التي لم تتأثر حاليًا بالتهديدات البيئية (إن وجدت). وهذا يوفر قاعدة لفهم كيف أن هذه الميكروبات
قد تستجيب المجتمعات للتغيرات العالمية. كما سيساعد ذلك في تحديد نقاط التحول التي يمكن أن تغير بشكل دائم تكوين ووظيفة الميكروبيوم التربة. على سبيل المثال، أظهرت الجرد الكبيرة للغابات الأوروبية أن ترسيب النيتروجين يسبب نقاط تحول في تكوين ووظيفة الفطريات الميكوريزية (فان دير ليندي وآخرون، 2018). على الرغم من أن المئات من الدراسات قد وصفت تكوين الميكوبيوما الميكوريزي في أنظمة بيئية مختلفة، إلا أن القليل معروف عن الترابط بين المكونات المختلفة وتحولات المجتمع الميكوريزي خلال التعاقب وعلى طول تدرج المناطق التي تهيمن عليها المراعي والشجيرات والأشجار.
4. الجينوميات البيئية
فهم العمليات التي تؤدي إلى التغيرات في المجتمعات الميكروبية الطبيعية عبر الزمان والمكان هو تحدٍ حاسم لعلماء البيئة. مع تزايد عدد الجينومات الفطرية المتاحة من الأنواع ذات الأهمية البيئية (انظر القسم الثاني)، أصبح من الممكن الآن رسم خرائط لقراءات الحمض النووي وRNA المجهولة من التربة وجذور الفطريات الجذرية لتحديد الأنواع الفطرية الجذرية وأنماط التعبير الجيني الخاصة بها، بما في ذلك استهداف الجينات الفطرية الجذرية المسؤولة عن امتصاص المغذيات، وتطوير التعايش، والإشارات، وتحلل المواد العضوية في التربة. ومع ذلك، لا تزال الأساليب الميتاجينومية تمثل تحديًا في تجميع الجينومات الفطرية الكاملة من عينات التربة لأنها عادةً ما تمثل نسبة منخفضة جدًا من الحمض النووي في التربة. يمكن أن تساعد تقنيات التسلسل عالية الإنتاجية في معالجة مشكلة عمق التغطية، ولكن هذا قد يؤدي إلى مشاكل أخرى تتعلق بالمتطلبات الحاسوبية العالية لمجموعات البيانات الكبيرة. على النقيض من ذلك، باستخدام تحليل RNA لجذور الفطريات الجذرية ونهايات الجذور، يمكننا الآن تقييم ما إذا كانت الأنواع التعايشية المتعددة يمكن أن تتواجد على (أو تحت) نفس الشجرة المضيفة من خلال استغلال نفس الموارد وأداء وظائف بيئية متطابقة (أي إظهار تشابه وظيفي عالٍ) أو من خلال استغلال موارد مختلفة وتقديم وظائف متميزة (أي إظهار تشابه وظيفي محدود؛ انظر القسم 5.2). يمكن استخدام الميتا ترانسكريبتوميات لمجتمعات الفطريات في التربة (Auer et al., 2023; Zeng et al., 2023) لفهم دور الشبكات الفطرية الجذرية، بما في ذلك CMN، لأنها يمكن أن تساعد في تحديد ما إذا كانت الأفراد المختلفة من الأشجار داخل قطعة معينة مستعمرة حقًا بنفس الفطر، وما إذا كانت نفس مجموعات الجينات معبرة في أفراد الأشجار المختلفة. ومع ذلك، هناك العديد من التحديات التي تحد من التطبيق الواسع للميتا ترانسكريبتوميات، بما في ذلك الاستقرار المنخفض لـ RNA مقارنة بـ DNA، وصعوبة أكبر في استخراج RNA، وصعوبة في الحصول على نسخ RNA الرسول المفيدة التي تقدم رؤى حول دورها الوظيفي (Auer et al., 2023). من الصعب التأكد مما إذا كانت النوى، والنسخ، والبروتينات، والمواد الأيضية يمكن أن تنتقل من نبات إلى آخر عبر CMNs في البيئات الطبيعية. إن غياب أدوات التصوير التي يمكن أن تتبع وتحقق في الفطريات الفردية داخل الشبكات الفطرية المتداخلة في التربة يمثل عقبة كبيرة في فهم هذه الظاهرة. ومع ذلك، فإن الأبحاث الجارية تطور طرقًا متكاملة تجمع بين تحليل rDNA لتقييم توزيع الوحدات التصنيفية التشغيلية، وتقدير حجم الجينوم الفطري من خلال علم الجينوم السكاني eDNA متعدد المواقع، والميتا ترانسكريبتوميات، والميتا بروتيوميات، وقياس تدفقات الأيض بين
مكونات مختلفة من قطع الغابات أو المراعي. ستقدم هذه الأساليب المتكاملة رؤى قيمة حول وظيفة مجتمعات الفطريات/النباتات. سيكون اعتماد هذه الاستراتيجية من الجينوم إلى النظام البيئي أمرًا حاسمًا في فك رموز مساهمات النقابات الفطرية التي تزدهر في التربة والجذور داخل النظم البيئية الأرضية المعقدة (الصندوق 1).
مكونات مختلفة من قطع الغابات أو المراعي. ستقدم هذه الأساليب المتكاملة رؤى قيمة حول وظيفة مجتمعات الفطريات/النباتات. سيكون اعتماد هذه الاستراتيجية من الجينوم إلى النظام البيئي أمرًا حاسمًا في فك رموز مساهمات النقابات الفطرية التي تزدهر في التربة والجذور داخل النظم البيئية الأرضية المعقدة (الصندوق 1).
5. تعزيز الزراعة المستدامة والغابات
الحصول على رؤى حول الأساس الجيني للخصائص المفيدة في مجتمعات الفطريات الجذرية والسكان أمر بالغ الأهمية لتطوير استراتيجيات تهدف إلى تحسين استخدامها كسماد حيوي أو عوامل تحكم حيوية، مما يقلل من الاعتماد على المدخلات الكيميائية ويعزز ممارسات إدارة الأراضي المستدامة. لتحقيق الزراعة المستدامة والغابات، من الضروري استغلال إمكانيات التعايش الفطري واستغلال الخصائص الوظيفية للسماد الفطري الجذري. لإدارة مجتمعات الفطريات الجذرية بنجاح وتعزيز الزراعة المستدامة بيئيًا أو مبادرات الاستعادة، من الضروري تحديد نطاق الأنواع الفطرية الموجودة والعوامل التي تؤثر على تنوعها وتوزيعها على المستويات الإقليمية والقارية. يمكن أن تعزز استقصاءات تحليل الحمض النووي التطبيقات وتوقع الظروف التي تكون فيها التلقيح مفيدًا للمزارعين ومديري الأراضي (مثل انخفاض وفرة أو تنوع الفطريات الجذرية) (الصندوق 1). يمكن أن تحدد هذه الأساليب الجينية أيضًا ما إذا كانت الميكروبيومات المحصولية المحددة تتميز بأنظمة ذات أداء جيد (عالية الإنتاجية وكفاءة استخدام المغذيات العالية) لتمييزها (وظيفيًا) عن الأنظمة ذات الأداء الضعيف (منخفضة الإنتاجية وفقدان كبير للمغذيات). علاوة على ذلك، يمكن استخدام مثل هذه الأدوات لتحديد كيف يمكن أن يخفف التلقيح الفطري من الضغط البيولوجي (مثل ارتفاع وفرة مسببات الأمراض). علاوة على ذلك، قد توفر الميتا ترانسكريبتوميات، والميتا جينوميات، واستقصاءات النظائر المستقرة رؤى وظيفية وتحدد دور بكتيريا المساعدة الفطرية وغيرها من التجمعات الميكروبية التي تساهم في نمو النباتات. يمكن أن تسهل الفطريات الجذرية أيضًا إنشاء ونمو النباتات في المواطن المتدهورة. ومع ذلك، من الضروري التحقيق في الظروف المحددة التي يكون فيها تطبيق الفطريات الجذرية مفيدًا، والنباتات المضيفة المتوافقة (أو حتى الأنماط الجينية)، وما إذا كانت الأنواع المحددة من الفطريات الجذرية يمكن تطبيقها بمفردها أو في تجمعات ميكروبية، جنبًا إلى جنب مع ميكروبات أخرى مثل بكتيريا المساعدة الفطرية والطلائعيات، أو باستخدام تلقيح ميداني معقد (Allsup et al., 2023). على غرار الميكروبيوم البشري، هناك إمكانيات مهمة لهندسة الميكروبيوم (Silverstein et al., 2023). العديد من المنتجات الفطرية الجذرية المتاحة تجاريًا إما غير قابلة للحياة أو غير فعالة (Salomon et al., 2022)، وغالبًا ما يتم تجاهل ما إذا كانت الفطريات الموجودة في هذه التلقيحات التجارية قد تم تأسيسها بنجاح في الميدان. لذلك، من الضروري تحليل الحمض النووي للسماد، وتتبع تأسيس وبقاء عزلة الفطريات الجذرية في الميدان، وتحديد الجدوى الاقتصادية والبيئية لتطبيقها.
6. تعزيز
للتعايشات الفطرية القدرة على المساهمة في التخفيف من تغير المناخ من خلال تعزيز
(Hawkins et al., 2023). تساهم الشبكات الفطرية في حوالي 20
(Hawkins et al., 2023). تساهم الشبكات الفطرية في حوالي 20
IX. الخاتمة
في الختام، إن دمج علم الجينوم وأدوات الأوميكس الأخرى يحمل وعدًا كبيرًا لتعزيز استخدام العلاقات الفطرية في إدارة النظم البيئية المستدامة. من خلال فك الديناميات المعقدة لهذه التفاعلات التبادلية، يمكن الحصول على رؤى قيمة تعتبر حاسمة لتعزيز الاستدامة البيئية ومعالجة القضايا البيئية الملحة. ستساهم هذه المقاربة الشاملة، التي تجمع بين تقنيات الجينوم المتقدمة واستطلاعات التنوع الجزيئي العالمية الشاملة، بلا شك في الاستخدام الفعال والمسؤول للفطريات الفطرية في إدارة النظم البيئية في المستقبل.
الشكر والتقدير
تستند هذه المقالة إلى محاضرات قدمها المؤلفون في المؤتمر الدولي لعام 2021 حول الفطريات الفطرية في بكين. نعتذر مسبقًا لجميع الباحثين الذين لم يتمكنوا من الإشارة إلى أبحاثهم بشكل مناسب بسبب قيود المساحة. نود أن نشكر المحرر إيان ديكي، إيدو روج، روجر فينلاي، ومراجعين اثنين على تعليقاتهم المفيدة. شكر خاص لمارك بوي، مايرا دي فريتاس بيريرا، روزماري هونجر، فاسيليس كوكوريس، جين مكغالي، أوتا باسكوسكي، سيلفيا بيروتو، ويانيس روجينموسر لمشاركتهم المواد الفوتوغرافية وفلوريان غادين لتوضيح الفطريات الفطرية
الشبكات (الشكل 4). تم تمويل FMM من قبل مختبر التميز ARBRE (ANR-12-LABX-ARBRE-01)، وزارة الطاقة الأمريكية JGI (مكتب العلوم التابع لوزارة الطاقة الأمريكية بموجب العقد رقم DE-AC02-05CH11231)، مركز بكين للابتكار المتقدم لتربية الأشجار من خلال التصميم الجزيئي (جامعة بكين للغابات)، وجامعة هوازونغ الزراعية. يعترف MvdH بالتمويل من المؤسسة الوطنية السويسرية للعلوم (المنح 310030_188799 و40IN40_215832) ومؤسسة جيبرت ريف.
الشبكات (الشكل 4). تم تمويل FMM من قبل مختبر التميز ARBRE (ANR-12-LABX-ARBRE-01)، وزارة الطاقة الأمريكية JGI (مكتب العلوم التابع لوزارة الطاقة الأمريكية بموجب العقد رقم DE-AC02-05CH11231)، مركز بكين للابتكار المتقدم لتربية الأشجار من خلال التصميم الجزيئي (جامعة بكين للغابات)، وجامعة هوازونغ الزراعية. يعترف MvdH بالتمويل من المؤسسة الوطنية السويسرية للعلوم (المنح 310030_188799 و40IN40_215832) ومؤسسة جيبرت ريف.
المصالح المتنافسة
لا توجد مصالح معلنة.
ORCID
مارسيل ج. أ. فان دير هايدن (D) https://orcid.org/0000-0001-7040-1924
فرانسيس م. مارتن (D) https://orcid.org/0000-0002-4737-3715
فرانسيس م. مارتن (D) https://orcid.org/0000-0002-4737-3715
References
Ahonen-Jonnarth U, Göransson A, Finlay RD. 2003. Growth and nutrient uptake of ectomycorrhizal Pinus sylvestris seedlings treated with elevated Al concentrations. Tree Physiology 23: 157-167.
Albornoz FE, Ryan MH, Bending GD, Hilton S, Dickie IA, Gleeson DB, Standish RJ. 2022. Agricultural land-use favours Mucoromycotinian, but not Glomeromycotinian, arbuscular mycorrhizal fungi across ten biomes. New Phytologist 233: 1369-1382.
Allsup CM, George I, Lankau RA. 2023. Shifting microbial communities can enhance tree tolerance to changing climates. Science 380: 835-840.
An GH, Kobayashi S, Enoki H, Sonobe K, Muraki M, Karasawa T, Ezawa T. 2010. How does arbuscular mycorrhizal colonization vary with host plant genotype? An example based on maize (Zea mays) germplasms. Plant and Soil 327: 441-453.
Angulo V, Beriot N, Garcia-Hernandez E, Li E, Masteling R, Lau JA. 2022. Plantmicrobe eco-evolutionary dynamics in a changing world. New Phytologist 234: 1919-1928.
Anthony MA, Crowther TW, Van Der Linde S, Suz LM, Bidartondo MI, Cox F, Schaub M, Rautio P, Ferretti M, Vesterdal L et al. 2022. Forest tree growth is linked to mycorrhizal fungal composition and function across Europe. ISME Journal 16: 1327-1336.
Aparicio Chacón MV, Van Dingenen J, Goormachtig S. 2023. Characterization of arbuscular mycorrhizal effector proteins. International Journal of Molecular Sciences 24: 9125.
Arnebrant K, Ek H, Finlay RD, Söderström B. 1993. Nitrogen translocation between Alnus glutinosa (L.) Gaertn. seedlings inoculated with Frankia sp. and Pinus contorta Doug, ex Loud seedlings connected by a common ectomycorrhizal mycelium. New Phytologist 124: 231-242.
Auer L, Buée M, Fauchery L, Lombard V, Barry K, Clum A, Copeland A, Daum C, Foster B, LaButti K et al. 2023. Metatranscriptomics sheds light on the links between the functional traits of fungal guilds and ecological processes in forest soil ecosystems. New Phytologist. doi: 10.1111/nph.19471.
Augé RM. 2001. Water relations, drought and vesicular-arbuscular mycorrhizal symbiosis. Mycorrhiza 11: 3-42.
Averill C, Anthony MA, Baldrian P, Finkbeiner F, van den Hoogen J, Kiers T, Kohout P, Hirt E, Smith GR, Crowther TW. 2022. Defending Earth’s terrestrial microbiome. Nature Microbiology 7: 1717-1725.
Babikova Z, Gilbert L, Bruce TJ, Birkett M, Caulfield JC, Woodcock C, Pickett JA, Johnson D. 2013. Underground signals carried through common mycelial networks warn neighbouring plants of aphid attack. Ecology Letters 16: 835-843.
Albornoz FE, Ryan MH, Bending GD, Hilton S, Dickie IA, Gleeson DB, Standish RJ. 2022. Agricultural land-use favours Mucoromycotinian, but not Glomeromycotinian, arbuscular mycorrhizal fungi across ten biomes. New Phytologist 233: 1369-1382.
Allsup CM, George I, Lankau RA. 2023. Shifting microbial communities can enhance tree tolerance to changing climates. Science 380: 835-840.
An GH, Kobayashi S, Enoki H, Sonobe K, Muraki M, Karasawa T, Ezawa T. 2010. How does arbuscular mycorrhizal colonization vary with host plant genotype? An example based on maize (Zea mays) germplasms. Plant and Soil 327: 441-453.
Angulo V, Beriot N, Garcia-Hernandez E, Li E, Masteling R, Lau JA. 2022. Plantmicrobe eco-evolutionary dynamics in a changing world. New Phytologist 234: 1919-1928.
Anthony MA, Crowther TW, Van Der Linde S, Suz LM, Bidartondo MI, Cox F, Schaub M, Rautio P, Ferretti M, Vesterdal L et al. 2022. Forest tree growth is linked to mycorrhizal fungal composition and function across Europe. ISME Journal 16: 1327-1336.
Aparicio Chacón MV, Van Dingenen J, Goormachtig S. 2023. Characterization of arbuscular mycorrhizal effector proteins. International Journal of Molecular Sciences 24: 9125.
Arnebrant K, Ek H, Finlay RD, Söderström B. 1993. Nitrogen translocation between Alnus glutinosa (L.) Gaertn. seedlings inoculated with Frankia sp. and Pinus contorta Doug, ex Loud seedlings connected by a common ectomycorrhizal mycelium. New Phytologist 124: 231-242.
Auer L, Buée M, Fauchery L, Lombard V, Barry K, Clum A, Copeland A, Daum C, Foster B, LaButti K et al. 2023. Metatranscriptomics sheds light on the links between the functional traits of fungal guilds and ecological processes in forest soil ecosystems. New Phytologist. doi: 10.1111/nph.19471.
Augé RM. 2001. Water relations, drought and vesicular-arbuscular mycorrhizal symbiosis. Mycorrhiza 11: 3-42.
Averill C, Anthony MA, Baldrian P, Finkbeiner F, van den Hoogen J, Kiers T, Kohout P, Hirt E, Smith GR, Crowther TW. 2022. Defending Earth’s terrestrial microbiome. Nature Microbiology 7: 1717-1725.
Babikova Z, Gilbert L, Bruce TJ, Birkett M, Caulfield JC, Woodcock C, Pickett JA, Johnson D. 2013. Underground signals carried through common mycelial networks warn neighbouring plants of aphid attack. Ecology Letters 16: 835-843.
Bahram M, Hildebrand F, Forslund SK, Anderson JL, Soudzilovska NA, Bodegom PM, Bengtsson-Palme J, Anslan S, Coelho LP, Harend H et al. 2018. Structure and function of the global topsoil microbiome. Nature 560: 233-237.
Baldrian P, López-Mondéjar R, Kohout P. 2023. Forest microbiome and global change. Nature Reviews Microbiology 21: 487-501.
Balestrini R, Nerva L, Sillo F, Girlanda M, Perotto S. 2014. Plant and fungal gene expression in mycorrhizal protocorms of the orchid Serapias vomeracea colonized by Tulasnella calospora. Plant Signaling & Behavior 9: e977707.
Basiru S, Hijri M. 2022. The potential applications of commercial arbuscular mycorrhizal fungal inoculants and their ecological consequences. Microorganisms 10: 1897.
Beiler KJ, Durall DM, Simard SW, Maxwell SA, Kretzer AM. 2010. Architecture of the wood-wide web: Rhizopogon spp.genets link multiple Douglas-fir cohorts. New Phytologist 185: 543-553.
Bödeker ITM, Clemmensen KE, De Boer W, Martin F, Olson Å, Lindahl B. 2014. Ectomycorrhizal Cortinarius species participate in enzymatic oxidation of humus in northern forest ecosystems. New Phytologist 203: 245-256.
Bogar LM. 2023. Modified source-sink dynamics govern resource exchange in ectomycorrhizal symbiosis. New Phytologist. doi: 10.1111/nph.19259.
Bouffaud ML, Herrmann S, Tarkka MT, Bönn M, Feldhahn L, Buscot F. 2020. Oak displays common local but specific distant gene regulation responses to different mycorrhizal fungi. BMC Genomics 21: 399.
Brundrett MC, Tedersoo L. 2018. Evolutionary history of mycorrhizal symbioses and global host plant diversity. New Phytologist 220: 1108-1115.
Cahanovitc R, Livne-Luzon S, Angel R, Klein T. 2022. Ectomycorrhizal fungi mediate belowground carbon transfer between pines and oaks. ISME Journal 16: 1420-1429.
Cai J, Liu X, Vanneste K, Proost S, Tsai WC, Liu KW, Chen LJ, He Y, Xu Q, Bian C et al. 2015. The genome sequence of the orchid Phalaenopsis equestris. Nature Genetics 47: 65-72.
Carteron A, Vellend M, Laliberté E. 2022. Mycorrhizal dominance reduces local tree species diversity across US forests. Nature Ecology & Evolution 6: 370-374.
Cavagnaro TR, Bender SF, Asghari HR, van der Heijden MGA. 2015. The role of arbuscular mycorrhizas in reducing soil nutrient loss. Trends in Plant Science 20: 283-290.
Chang Y, Desirò A, Na H, Sandor L, Lipzen A, Clum A, Barry K, Grigoriev IV, Martin FM, Stajich JE et al. 2019. Phylogenomics of Endogonaceae and evolution of mycorrhizas within Mucoromycota. New Phytologist 222: 511-525.
Chaudhary VB, Rúa MA, Antoninka A, Bever JD, Cannon J, Craig A, Duchicela J, Frame A, Gardes M, Gehring C et al. 2016. MycoDB, a global database of plant response to mycorrhizal fungi. Scientific Data 3: 160028.
Chen J, Liu SS, Kohler A, Yan B, Luo HM, Chen XM, Guo SX. 2017. iTRAQ and RNA-seq analyses provide new insights into regulation mechanism of symbiotic germination of Dendrobium officinale seeds (Orchidaceae). Journal of Proteome Research 16: 2174-2187.
Chen J, Tang Y, Kohler A, Lebreton A, Xing Y, Zhou D, Li Y, Martin FM, Guo S. 2022. Comparative transcriptomics analysis of the symbiotic germination of
Clemmensen KE, Durling MB, Michelsen A, Hallin S, Finlay RD, Lindahl BD. 2021. A tipping point in carbon storage when forest expands into tundra is related to mycorrhizal recycling of nitrogen. Ecology Letters 24: 1193-1204.
Cope KR, Bascaules A, Irving TB, Venkateshwaran M, Maeda J, Garcia K, Rush TA, Ma C, Labbé J, Jawdy S et al. 2019. The ectomycorrhizal fungus Laccaria bicolor produces lipochitooligosaccharides and uses the common symbiosis pathway to colonize Populus roots. Plant Cell 10: 2386-2410.
Davison J, Moora M, Öpik M, Adholeya A, Ainsaar L, Bâ A, Burla S, Diedhiou AG, Hiiesalu I, Jairus T et al. 2015. Fungal Symbionts. Global assessment of arbuscular mycorrhizal fungus diversity reveals very low endemism. Science 349: 970-973.
De La Providencia IE, De Souza FA, Fernández F, Delmas NS, Declerck S. 2005. Arbuscular mycorrhizal fungi reveal distinct patterns of anastomosis formation and hyphal healing mechanisms between different phylogenic groups. New Phytologist 165: 261-271.
Delaux PM, Radhakrishnan GV, Jayaraman D, Cheema J, Malbreil M, Volkening JD, Sekimoto H, Nishiyama T, Melkonian M, Pokorny L et al. 2015. Algal
ancestor of land plants was preadapted for symbiosis. Proceedings of the National Academy of Sciences, USA 112: 13390-13395.
Delgado-Baquerizo M, Oliverio AM, Brewer TE, Benavent-González A, Eldridge DJ, Bardgett RD, Maestre FT, Singh BK, Fierer N. 2018. A global atlas of the dominant bacteria found in soil. Science 359: 320-325.
Delgado-Baquerizo M, Reich PB, Trivedi C, Eldridge DJ, Abades S, Alfaro FD, Bastida F, Berhe AA, Cutler NA, Gallardo A et al. 2020. Multiple elements of soil biodiversity drive ecosystem functions across biomes. Nature Ecology & Evolution 4: 210-220.
Deng M, Hu S, Guo L, Jiang L, Huang Y, Schmid B, Liu C, Chang P, Li S, Liu X et al. 2023. Tree mycorrhizal association types control biodiversity-productivity relationship in a subtropical forest. Science Advances 9: eadd4468.
Desirò A, Rimington WR, Jacob A, Vande Pol N, Smith ME, Trappe JM, Bidartondo MI, Bonito G. 2017. Multigene phylogeny of Endogonales, an early diverging lineage of fungi associated with plants. IMA Fungus 8: 245-257.
Dickie IA, Bolstridge N, Cooper JA, Peltzer DA. 2010. Co-invasion by Pinusand its mycorrhizal fungi. New Phytologist 187: 475-484.
Drula E, Garron ML, Dogan S, Lombard V, Henrissat B, Terrapon N. 2022. The carbohydrate-active enzyme database: functions and literature. Nucleic Acids Research 50: D571-D577.
Edlinger A, Garland G, Hartman K, Banerjee S, Degrune F, García-Palacios P, Hallin S, Valzano-Held A, Herzog C, Jansa J et al. 2022. Agricultural management and pesticide use reduce the functioning of beneficial plant symbionts. Nature Ecology & Evolution 6: 1145-1154.
Emmett BD, Lévesque-Tremblay V, Harrison MJ. 2021. Conserved and reproducible bacterial communities associate with extraradical hyphae of arbuscular mycorrhizal fungi. ISME Journal 15: 2276-2288.
Ezawa T, Silvestri A, Maruyama H, Tawaraya K, Suzuki M, Duan Y. 2023. Structurally distinct mitoviruses: are they an ancestral lineage of the Mitoviridae exclusive to arbuscular mycorrhizal fungi (Glomeromycotina)? MBio 14: e0024023.
Baldrian P, López-Mondéjar R, Kohout P. 2023. Forest microbiome and global change. Nature Reviews Microbiology 21: 487-501.
Balestrini R, Nerva L, Sillo F, Girlanda M, Perotto S. 2014. Plant and fungal gene expression in mycorrhizal protocorms of the orchid Serapias vomeracea colonized by Tulasnella calospora. Plant Signaling & Behavior 9: e977707.
Basiru S, Hijri M. 2022. The potential applications of commercial arbuscular mycorrhizal fungal inoculants and their ecological consequences. Microorganisms 10: 1897.
Beiler KJ, Durall DM, Simard SW, Maxwell SA, Kretzer AM. 2010. Architecture of the wood-wide web: Rhizopogon spp.genets link multiple Douglas-fir cohorts. New Phytologist 185: 543-553.
Bödeker ITM, Clemmensen KE, De Boer W, Martin F, Olson Å, Lindahl B. 2014. Ectomycorrhizal Cortinarius species participate in enzymatic oxidation of humus in northern forest ecosystems. New Phytologist 203: 245-256.
Bogar LM. 2023. Modified source-sink dynamics govern resource exchange in ectomycorrhizal symbiosis. New Phytologist. doi: 10.1111/nph.19259.
Bouffaud ML, Herrmann S, Tarkka MT, Bönn M, Feldhahn L, Buscot F. 2020. Oak displays common local but specific distant gene regulation responses to different mycorrhizal fungi. BMC Genomics 21: 399.
Brundrett MC, Tedersoo L. 2018. Evolutionary history of mycorrhizal symbioses and global host plant diversity. New Phytologist 220: 1108-1115.
Cahanovitc R, Livne-Luzon S, Angel R, Klein T. 2022. Ectomycorrhizal fungi mediate belowground carbon transfer between pines and oaks. ISME Journal 16: 1420-1429.
Cai J, Liu X, Vanneste K, Proost S, Tsai WC, Liu KW, Chen LJ, He Y, Xu Q, Bian C et al. 2015. The genome sequence of the orchid Phalaenopsis equestris. Nature Genetics 47: 65-72.
Carteron A, Vellend M, Laliberté E. 2022. Mycorrhizal dominance reduces local tree species diversity across US forests. Nature Ecology & Evolution 6: 370-374.
Cavagnaro TR, Bender SF, Asghari HR, van der Heijden MGA. 2015. The role of arbuscular mycorrhizas in reducing soil nutrient loss. Trends in Plant Science 20: 283-290.
Chang Y, Desirò A, Na H, Sandor L, Lipzen A, Clum A, Barry K, Grigoriev IV, Martin FM, Stajich JE et al. 2019. Phylogenomics of Endogonaceae and evolution of mycorrhizas within Mucoromycota. New Phytologist 222: 511-525.
Chaudhary VB, Rúa MA, Antoninka A, Bever JD, Cannon J, Craig A, Duchicela J, Frame A, Gardes M, Gehring C et al. 2016. MycoDB, a global database of plant response to mycorrhizal fungi. Scientific Data 3: 160028.
Chen J, Liu SS, Kohler A, Yan B, Luo HM, Chen XM, Guo SX. 2017. iTRAQ and RNA-seq analyses provide new insights into regulation mechanism of symbiotic germination of Dendrobium officinale seeds (Orchidaceae). Journal of Proteome Research 16: 2174-2187.
Chen J, Tang Y, Kohler A, Lebreton A, Xing Y, Zhou D, Li Y, Martin FM, Guo S. 2022. Comparative transcriptomics analysis of the symbiotic germination of
Clemmensen KE, Durling MB, Michelsen A, Hallin S, Finlay RD, Lindahl BD. 2021. A tipping point in carbon storage when forest expands into tundra is related to mycorrhizal recycling of nitrogen. Ecology Letters 24: 1193-1204.
Cope KR, Bascaules A, Irving TB, Venkateshwaran M, Maeda J, Garcia K, Rush TA, Ma C, Labbé J, Jawdy S et al. 2019. The ectomycorrhizal fungus Laccaria bicolor produces lipochitooligosaccharides and uses the common symbiosis pathway to colonize Populus roots. Plant Cell 10: 2386-2410.
Davison J, Moora M, Öpik M, Adholeya A, Ainsaar L, Bâ A, Burla S, Diedhiou AG, Hiiesalu I, Jairus T et al. 2015. Fungal Symbionts. Global assessment of arbuscular mycorrhizal fungus diversity reveals very low endemism. Science 349: 970-973.
De La Providencia IE, De Souza FA, Fernández F, Delmas NS, Declerck S. 2005. Arbuscular mycorrhizal fungi reveal distinct patterns of anastomosis formation and hyphal healing mechanisms between different phylogenic groups. New Phytologist 165: 261-271.
Delaux PM, Radhakrishnan GV, Jayaraman D, Cheema J, Malbreil M, Volkening JD, Sekimoto H, Nishiyama T, Melkonian M, Pokorny L et al. 2015. Algal
ancestor of land plants was preadapted for symbiosis. Proceedings of the National Academy of Sciences, USA 112: 13390-13395.
Delgado-Baquerizo M, Oliverio AM, Brewer TE, Benavent-González A, Eldridge DJ, Bardgett RD, Maestre FT, Singh BK, Fierer N. 2018. A global atlas of the dominant bacteria found in soil. Science 359: 320-325.
Delgado-Baquerizo M, Reich PB, Trivedi C, Eldridge DJ, Abades S, Alfaro FD, Bastida F, Berhe AA, Cutler NA, Gallardo A et al. 2020. Multiple elements of soil biodiversity drive ecosystem functions across biomes. Nature Ecology & Evolution 4: 210-220.
Deng M, Hu S, Guo L, Jiang L, Huang Y, Schmid B, Liu C, Chang P, Li S, Liu X et al. 2023. Tree mycorrhizal association types control biodiversity-productivity relationship in a subtropical forest. Science Advances 9: eadd4468.
Desirò A, Rimington WR, Jacob A, Vande Pol N, Smith ME, Trappe JM, Bidartondo MI, Bonito G. 2017. Multigene phylogeny of Endogonales, an early diverging lineage of fungi associated with plants. IMA Fungus 8: 245-257.
Dickie IA, Bolstridge N, Cooper JA, Peltzer DA. 2010. Co-invasion by Pinusand its mycorrhizal fungi. New Phytologist 187: 475-484.
Drula E, Garron ML, Dogan S, Lombard V, Henrissat B, Terrapon N. 2022. The carbohydrate-active enzyme database: functions and literature. Nucleic Acids Research 50: D571-D577.
Edlinger A, Garland G, Hartman K, Banerjee S, Degrune F, García-Palacios P, Hallin S, Valzano-Held A, Herzog C, Jansa J et al. 2022. Agricultural management and pesticide use reduce the functioning of beneficial plant symbionts. Nature Ecology & Evolution 6: 1145-1154.
Emmett BD, Lévesque-Tremblay V, Harrison MJ. 2021. Conserved and reproducible bacterial communities associate with extraradical hyphae of arbuscular mycorrhizal fungi. ISME Journal 15: 2276-2288.
Ezawa T, Silvestri A, Maruyama H, Tawaraya K, Suzuki M, Duan Y. 2023. Structurally distinct mitoviruses: are they an ancestral lineage of the Mitoviridae exclusive to arbuscular mycorrhizal fungi (Glomeromycotina)? MBio 14: e0024023.
Finlay RD, Clemmensen KE. 2017. Immobilization of carbon in mycorrhizal mycelial biomass and secretions. In: Johnson NC, Gehring K, Jansa J, eds. Mycorrhizal mediation of soil: fertility, structure and carbon storage. Amsterdam, the Netherlands: Elsevier, 413-440.
Finlay RD, Read DJ. 1986. The structure and function of the vegetative mycelium of ectomycorrhizal plants. New Phytologist 103: 143-156.
Fiore-Donno AM, Martin F. 2001. Populations of ectomycorrhizal Laccaria amethystina and Xerocomusspp. show contrasting colonization patterns in a mixed forest. New Phytologist 152: 533-542.
Floudas D, Bentzer J, Ahrén D, Johansson T, Persson P, Tunlid A. 2020. Uncovering the hidden diversity of litter-decomposition mechanisms in mushroom-forming fungi. ISME Journal 14: 2046-2059.
Floudas D, Binder M, Riley R, Barry K, Blanchette RA, Henrissat B, Martinez AT, Otillar R, Spatafora JW, Yadav JS et al. 2012. The Paleozoic origin of enzymatic lignin decomposition reconstructed from 31 fungal genomes. Science 336: 17151719.
Finlay RD, Read DJ. 1986. The structure and function of the vegetative mycelium of ectomycorrhizal plants. New Phytologist 103: 143-156.
Fiore-Donno AM, Martin F. 2001. Populations of ectomycorrhizal Laccaria amethystina and Xerocomusspp. show contrasting colonization patterns in a mixed forest. New Phytologist 152: 533-542.
Floudas D, Bentzer J, Ahrén D, Johansson T, Persson P, Tunlid A. 2020. Uncovering the hidden diversity of litter-decomposition mechanisms in mushroom-forming fungi. ISME Journal 14: 2046-2059.
Floudas D, Binder M, Riley R, Barry K, Blanchette RA, Henrissat B, Martinez AT, Otillar R, Spatafora JW, Yadav JS et al. 2012. The Paleozoic origin of enzymatic lignin decomposition reconstructed from 31 fungal genomes. Science 336: 17151719.
Frey-Klett P, Garbaye J, Tarkka M. 2007. The mycorrhiza helper bacteria revisited. New Phytologist 176: 22-36.
Garcia K, Delaux PM, Cope KR, Ané J-M. 2015. Molecular signals required for the establishment and maintenance of ectomycorrhizal symbioses. New Phytologist 208: 79-87.
Gaude N, Bortfeld S, Duensing N, Lohse M, Krajinski F. 2012. Arbusculecontaining and non-colonized cortical cells of mycorrhizal roots undergo extensive and specific reprogramming during arbuscular mycorrhizal development. The Plant Journal 69: 510-528.
Ghirardo A, Fochi V, Lange B, Witting M, Schnitzler JP, Perotto S, Balestrini R. 2020. Metabolomic adjustments in the orchid mycorrhizal fungus Tulasnella calospora during symbiosis with Serapias vomeracea. New Phytologist 228: 19391952.
Garcia K, Delaux PM, Cope KR, Ané J-M. 2015. Molecular signals required for the establishment and maintenance of ectomycorrhizal symbioses. New Phytologist 208: 79-87.
Gaude N, Bortfeld S, Duensing N, Lohse M, Krajinski F. 2012. Arbusculecontaining and non-colonized cortical cells of mycorrhizal roots undergo extensive and specific reprogramming during arbuscular mycorrhizal development. The Plant Journal 69: 510-528.
Ghirardo A, Fochi V, Lange B, Witting M, Schnitzler JP, Perotto S, Balestrini R. 2020. Metabolomic adjustments in the orchid mycorrhizal fungus Tulasnella calospora during symbiosis with Serapias vomeracea. New Phytologist 228: 19391952.
Giovannetti M, Fortuna P, Citernesi AS, Morini S, Nuti MP. 2001. The occurrence of anastomosis formation and nuclear exchange in intact arbuscular mycorrhizal networks. New Phytologist 151: 717-724.
Givnish TJ, Spalink D, Ames M, Lyon SP, Hunter SJ, Zuluaga A, Iles WJ, Clements MA, Arroyo MT, Leebens-Mack J et al. 2015. Orchid phylogenomics and multiple drivers of their extraordinary diversification. Proceedings of the Royal Society B: Biological Sciences 282: 20151553.
Givnish TJ, Spalink D, Ames M, Lyon SP, Hunter SJ, Zuluaga A, Iles WJ, Clements MA, Arroyo MT, Leebens-Mack J et al. 2015. Orchid phylogenomics and multiple drivers of their extraordinary diversification. Proceedings of the Royal Society B: Biological Sciences 282: 20151553.
Grelet G, Martino E, Dickie IA, Tajuddin R, Artz R. 2016. Ecology of ericoid mycorrhizal fungi: what insight have we gained with molecular tools and what’s missing? In: Martin F, ed. Molecular mycorrhizal symbiosis. Hoboken, NJ, USA: John Wiley & Sons, 405-419.
Grelet GA, Johnson D, Vralstad T, Alexander IJ, Anderson IC. 2010. New insights into the mycorrhizal Rhizoscyphus ericae aggregate: spatial structure and co-colonization of ectomycorrhizal and ericoid roots. New Phytologist 188: 210-222.
Grigoriev IV, Nikitin R, Haridas S, Kuo A, Ohm R, Otillar R, Riley R, Salamov A, Zhao X, Korzeniewski F et al. 2014. MycoCosm portal: gearing up for 1000 fungal genomes. Nucleic Acids Research 42: D699-D704.
Grunwald U, Guo W, Fischer K, Isayenkov S, Ludwig-Müller J, Hause B, Yan X, Küster H, Franken P. 2009. Overlapping expression patterns and differential transcript levels of phosphate transporter genes in arbuscular mycorrhizal, Pifertilised and phytohormone-treated Medicago truncatula roots. Planta 229: 1023-1034.
Gryta H, Debaud JC, Marmeisse R. 2000. Population dynamics of the symbiotic mushroom Hebeloma cylindrosporum: mycelial persistence and inbreeding. Heredity 84: 294-302.
Hagh-Doust N, Mikryukov V, Anslan S, Bahram M, Puusepp R, Dulya O, Tedersoo L. 2023. Effects of nitrogen deposition on carbon and nutrient cycling along a natural soil acidity gradient as revealed by metagenomics. New Phytologist 238: 2607-2620.
Hart MM, Antunes PM, Chaudhary VB, Abbott LK. 2018. Fungal inoculants in the field: is the reward greater than the risk? Functional Ecology 32: 126-135.
Hartnett DC, Wilson GW. 1999. Mycorrhizae influence plant community structure and diversity in tallgrass prairie. Ecology 80: 1187-1195.
Hawkins HJ, Cargill RI, Van Nuland ME, Hagen SC, Field KJ, Sheldrake M, Soudzilovskaia NA, Kiers ET. 2023. Mycorrhizal mycelium as a global carbon pool. Current Biology 33: R560-R573.
van der Heijden MG. 2004. Arbuscular mycorrhizal fungi as support systems for seedling establishment in grassland. Ecology Letters 7: 293-303.
van der Heijden MGA, Bardgett RD, van Straalen NM. 2008. The unseen majority: soil microbes as drivers of plant diversity and productivity in terrestrial ecosystems. Ecology Letters 11: 296-310.
van der Heijden MGA, Horton TR. 2009. Socialism in soil? The importance of mycorrhizal fungal networks for facilitation in natural ecosystems. Journal of Ecology 97: 1139-1150.
van der Heijden MGA, Klironomos JN, Ursic M, Moutoglis P, StreitwolfEngel R, Boller T, Wiemken A, Sanders IR. 1998. Mycorrhizal fungal diversity determines plant biodiversity, ecosystem variability and productivity. Nature 396: 69-72.
van der Heijden MGA, Martin F, Selosse MA, Sanders IR. 2015. Mycorrhizal ecology and evolution: the past, the present, and the future. New Phytologist 205: 1406-1423.
Henriksson N, Marshall J, Högberg MN, Högberg P, Polle A, Franklin O, Näsholm T. 2023. Re-examining the evidence for the mother tree hypothesisresource sharing among trees via ectomycorrhizal networks. New Phytologist 239: 19-28.
Hicks Pries CE, Lankau R, Ingham GA, Legge E, Krol O, Forrester J, Fitch A, Wurzburger N. 2023. Differences in soil organic matter between EcM-and AMdominated forests depend on tree and fungal identity. Ecology 104: e3929.
Hirose D, Kikuchi J, Kanzaki N, Futai K. 2004. Genet distribution of sporocarps and ectomycorrhizas of Suillus pictus in a Japanese white pine plantation. New Phytologist 164: 527-541.
Hobbie JE, Hobbie EA. 2006.
Hoeksema JD, Chaudhary VB, Gehring CA, Johnson NC, Karst J, Koide RT, Umbanhowar J. 2010. A meta-analysis of context-dependency in plant response to inoculation with mycorrhizal fungi. Ecology Letters 13: 394-407.
Hogekamp C, Küster H. 2013. A roadmap of cell-type specific gene expression during sequential stages of the arbuscular mycorrhiza symbiosis. BMC Genomics 14: 306.
Hohnjec N, Vieweg MF, Pühler A, Becker A, Küster H. 2005. Overlaps in the transcriptional profiles of Medicago truncatula roots inoculated with two different glomus fungi provide insights into the genetic program activated during arbuscular mycorrhiza. Plant Physiology 137: 1283-1301.
Grelet GA, Johnson D, Vralstad T, Alexander IJ, Anderson IC. 2010. New insights into the mycorrhizal Rhizoscyphus ericae aggregate: spatial structure and co-colonization of ectomycorrhizal and ericoid roots. New Phytologist 188: 210-222.
Grigoriev IV, Nikitin R, Haridas S, Kuo A, Ohm R, Otillar R, Riley R, Salamov A, Zhao X, Korzeniewski F et al. 2014. MycoCosm portal: gearing up for 1000 fungal genomes. Nucleic Acids Research 42: D699-D704.
Grunwald U, Guo W, Fischer K, Isayenkov S, Ludwig-Müller J, Hause B, Yan X, Küster H, Franken P. 2009. Overlapping expression patterns and differential transcript levels of phosphate transporter genes in arbuscular mycorrhizal, Pifertilised and phytohormone-treated Medicago truncatula roots. Planta 229: 1023-1034.
Gryta H, Debaud JC, Marmeisse R. 2000. Population dynamics of the symbiotic mushroom Hebeloma cylindrosporum: mycelial persistence and inbreeding. Heredity 84: 294-302.
Hagh-Doust N, Mikryukov V, Anslan S, Bahram M, Puusepp R, Dulya O, Tedersoo L. 2023. Effects of nitrogen deposition on carbon and nutrient cycling along a natural soil acidity gradient as revealed by metagenomics. New Phytologist 238: 2607-2620.
Hart MM, Antunes PM, Chaudhary VB, Abbott LK. 2018. Fungal inoculants in the field: is the reward greater than the risk? Functional Ecology 32: 126-135.
Hartnett DC, Wilson GW. 1999. Mycorrhizae influence plant community structure and diversity in tallgrass prairie. Ecology 80: 1187-1195.
Hawkins HJ, Cargill RI, Van Nuland ME, Hagen SC, Field KJ, Sheldrake M, Soudzilovskaia NA, Kiers ET. 2023. Mycorrhizal mycelium as a global carbon pool. Current Biology 33: R560-R573.
van der Heijden MG. 2004. Arbuscular mycorrhizal fungi as support systems for seedling establishment in grassland. Ecology Letters 7: 293-303.
van der Heijden MGA, Bardgett RD, van Straalen NM. 2008. The unseen majority: soil microbes as drivers of plant diversity and productivity in terrestrial ecosystems. Ecology Letters 11: 296-310.
van der Heijden MGA, Horton TR. 2009. Socialism in soil? The importance of mycorrhizal fungal networks for facilitation in natural ecosystems. Journal of Ecology 97: 1139-1150.
van der Heijden MGA, Klironomos JN, Ursic M, Moutoglis P, StreitwolfEngel R, Boller T, Wiemken A, Sanders IR. 1998. Mycorrhizal fungal diversity determines plant biodiversity, ecosystem variability and productivity. Nature 396: 69-72.
van der Heijden MGA, Martin F, Selosse MA, Sanders IR. 2015. Mycorrhizal ecology and evolution: the past, the present, and the future. New Phytologist 205: 1406-1423.
Henriksson N, Marshall J, Högberg MN, Högberg P, Polle A, Franklin O, Näsholm T. 2023. Re-examining the evidence for the mother tree hypothesisresource sharing among trees via ectomycorrhizal networks. New Phytologist 239: 19-28.
Hicks Pries CE, Lankau R, Ingham GA, Legge E, Krol O, Forrester J, Fitch A, Wurzburger N. 2023. Differences in soil organic matter between EcM-and AMdominated forests depend on tree and fungal identity. Ecology 104: e3929.
Hirose D, Kikuchi J, Kanzaki N, Futai K. 2004. Genet distribution of sporocarps and ectomycorrhizas of Suillus pictus in a Japanese white pine plantation. New Phytologist 164: 527-541.
Hobbie JE, Hobbie EA. 2006.
Hoeksema JD, Chaudhary VB, Gehring CA, Johnson NC, Karst J, Koide RT, Umbanhowar J. 2010. A meta-analysis of context-dependency in plant response to inoculation with mycorrhizal fungi. Ecology Letters 13: 394-407.
Hogekamp C, Küster H. 2013. A roadmap of cell-type specific gene expression during sequential stages of the arbuscular mycorrhiza symbiosis. BMC Genomics 14: 306.
Hohnjec N, Vieweg MF, Pühler A, Becker A, Küster H. 2005. Overlaps in the transcriptional profiles of Medicago truncatula roots inoculated with two different glomus fungi provide insights into the genetic program activated during arbuscular mycorrhiza. Plant Physiology 137: 1283-1301.
Horsch CC, Antunes PM, Fahey C, Grandy AS, Kallenbach CM. 2023. Traitbased assembly of arbuscular mycorrhizal fungal communities determines soil carbon formation and retention. New Phytologist 239: 311-324.
Hortal S, Plett KL, Plett JM, Cresswell T, Johansen M, Pendall E, Anderson IC. 2017. Role of plant-fungal nutrient trading and host control in determining the competitive success of ectomycorrhizal fungi. ISME Journal 11: 2666-2676.
Hortal S, Trocha LK, Murat C, Chybicki IJ, Buée M, Trojankiewicz M, Burczyk J, Martin F. 2012. Beech roots are simultaneously colonized by multiple genets of the ectomycorrhizal fungus Laccaria amethystina clustered in two genetic groups. Molecular Ecology 21: 2116-2129.
Hoysted GA, Field KJ, Sinanaj B, Bell CA, Bidartondo MI, Pressel S. 2023. Direct nitrogen, phosphorus and carbon exchanges between Mucoromycotina ‘fine root endophyte’ fungi and a flowering plant in novel monoxenic cultures. New Phytologist 238: 70-79.
Huynh-Thu VA, Irrthum A, Wehenkel L, Geurts P. 2010. Inferring regulatory networks from expression data using treebased methods. PLoS ONE 5: e12776.
Jastrow JD, Miller RM, Lussenhop J. 1998. Contributions of interacting biological mechanisms to soil aggregate stabilization in restored prairie. Soil Biology and Biochemistry 30: 905-916.
Jia Y, van der Heijden MGA, Wagg C, Feng G, Walder F. 2021. Symbiotic soil fungi enhance resistance and resilience of an experimental grassland to drought and nitrogen deposition. Journal of Ecology 109: 3171-3181.
Jiang L, Lin M, Wang H, Song H, Zhang L, Huang Q, Chen R, Song C, Li G, Cao Y. 2022. Haplotype-resolved genome assembly of Bletilla striata to elucidate medicinal value. The Plant Journal 111: 1340-1353.
Jumper J, Evans R, Pritzel A, Green T, Figurnov M, Ronneberger O, Tunyasuvunakool K, Bates R, Źídek A, Potapenko A et al. 2021. Highly accurate protein structure prediction with AlphaFold. Nature 596: 583-589.
Kakouridis A, Hagen JA, Kan MP, Mambelli S, Feldman LJ, Herman DJ, Weber PK, Pett-Ridge J, Firestone MK. 2022. Routes to roots: direct evidence of water transport by arbuscular mycorrhizal fungi to host plants. New Phytologist 236: 210-221.
Kameoka H, Gutjahr C. 2022. Functions of lipids in development and reproduction of arbuscular mycorrhizal fungi. Plant Cell Physiology 63: 13561365.
Hortal S, Plett KL, Plett JM, Cresswell T, Johansen M, Pendall E, Anderson IC. 2017. Role of plant-fungal nutrient trading and host control in determining the competitive success of ectomycorrhizal fungi. ISME Journal 11: 2666-2676.
Hortal S, Trocha LK, Murat C, Chybicki IJ, Buée M, Trojankiewicz M, Burczyk J, Martin F. 2012. Beech roots are simultaneously colonized by multiple genets of the ectomycorrhizal fungus Laccaria amethystina clustered in two genetic groups. Molecular Ecology 21: 2116-2129.
Hoysted GA, Field KJ, Sinanaj B, Bell CA, Bidartondo MI, Pressel S. 2023. Direct nitrogen, phosphorus and carbon exchanges between Mucoromycotina ‘fine root endophyte’ fungi and a flowering plant in novel monoxenic cultures. New Phytologist 238: 70-79.
Huynh-Thu VA, Irrthum A, Wehenkel L, Geurts P. 2010. Inferring regulatory networks from expression data using treebased methods. PLoS ONE 5: e12776.
Jastrow JD, Miller RM, Lussenhop J. 1998. Contributions of interacting biological mechanisms to soil aggregate stabilization in restored prairie. Soil Biology and Biochemistry 30: 905-916.
Jia Y, van der Heijden MGA, Wagg C, Feng G, Walder F. 2021. Symbiotic soil fungi enhance resistance and resilience of an experimental grassland to drought and nitrogen deposition. Journal of Ecology 109: 3171-3181.
Jiang L, Lin M, Wang H, Song H, Zhang L, Huang Q, Chen R, Song C, Li G, Cao Y. 2022. Haplotype-resolved genome assembly of Bletilla striata to elucidate medicinal value. The Plant Journal 111: 1340-1353.
Jumper J, Evans R, Pritzel A, Green T, Figurnov M, Ronneberger O, Tunyasuvunakool K, Bates R, Źídek A, Potapenko A et al. 2021. Highly accurate protein structure prediction with AlphaFold. Nature 596: 583-589.
Kakouridis A, Hagen JA, Kan MP, Mambelli S, Feldman LJ, Herman DJ, Weber PK, Pett-Ridge J, Firestone MK. 2022. Routes to roots: direct evidence of water transport by arbuscular mycorrhizal fungi to host plants. New Phytologist 236: 210-221.
Kameoka H, Gutjahr C. 2022. Functions of lipids in development and reproduction of arbuscular mycorrhizal fungi. Plant Cell Physiology 63: 13561365.
Kang H, Chen X, Kempannien M, Pardo AG, Veneault-Fourrey C, Kohler A, Martin F. 2020. The small secreted effector protein MiSSP7.6 of Laccaria bicolor is required for the establishment of ectomycorrhizal symbiosis. Environmental Microbiology 22: 1435-1446.
Karst J, Jones MD, Hoeksema JD. 2023. Positive citation bias and overinterpreted results lead to misinformation on common mycorrhizal networks in forests. Nature Ecology & Evolution 7: 501-511.
Kivlin SN. 2020. Global mycorrhizal fungal range sizes vary within and among mycorrhizal guilds but are not correlated with dispersal traits. Journal of Biogeography 47: 1994-2001.
Klein T, Siegwolf RTW, Körner C. 2016. Belowground carbon trade among tall trees in a temperate forest. Science 352: 342-344.
Klironomos JN. 2002. Feedback with soil biota contributes to plant rarity and invasiveness in communities. Nature 417: 67-70.
Kloppholz S, Kuhn H, Requena N. 2011. A secreted fungal effector of Glomus intraradices promotes symbiotic biotrophy. Current Biology 21: 1204-1209.
Kobayashi Y, Maeda T, Yamaguchi K, Kameoka H, Tanaka S, Ezawa T, Shigenobu S, Kawaguchi M. 2018. The genome of Rhizophagus clarus HR1 reveals a common genetic basis for auxotrophy among arbuscular mycorrhizal fungi. BMC Genomics 2018: 465.
Kobayashi Y, Shibata TF, Hirakawa H, Nishiyama T, Yamada A, Hasebe M, Shigenobu S, Kawaguchi M. 2023. The genome of Lyophyllum shimeji provides insight into the initial evolution of ectomycorrhizal fungal genomes.
Kohler A, Kuo A, Nagy LG, Morin E, Barry KW, Buscot F, Canbäck B, Choi C, Cichocki N, Clum A et al. 2015. Convergent losses of decay mechanisms and rapid turnover of symbiosis genes in mycorrhizal mutualists. Nature Genetics 47: 410-415.
Kokkoris V, Chagnon PL, Yildirir G, Clarke K, Goh D, MacLean AM, Dettman J, Stefani F, Corradi N. 2021. Host identity influences nuclear dynamics in arbuscular mycorrhizal fungi. Current Biology 31: 1531-1538.
Karst J, Jones MD, Hoeksema JD. 2023. Positive citation bias and overinterpreted results lead to misinformation on common mycorrhizal networks in forests. Nature Ecology & Evolution 7: 501-511.
Kivlin SN. 2020. Global mycorrhizal fungal range sizes vary within and among mycorrhizal guilds but are not correlated with dispersal traits. Journal of Biogeography 47: 1994-2001.
Klein T, Siegwolf RTW, Körner C. 2016. Belowground carbon trade among tall trees in a temperate forest. Science 352: 342-344.
Klironomos JN. 2002. Feedback with soil biota contributes to plant rarity and invasiveness in communities. Nature 417: 67-70.
Kloppholz S, Kuhn H, Requena N. 2011. A secreted fungal effector of Glomus intraradices promotes symbiotic biotrophy. Current Biology 21: 1204-1209.
Kobayashi Y, Maeda T, Yamaguchi K, Kameoka H, Tanaka S, Ezawa T, Shigenobu S, Kawaguchi M. 2018. The genome of Rhizophagus clarus HR1 reveals a common genetic basis for auxotrophy among arbuscular mycorrhizal fungi. BMC Genomics 2018: 465.
Kobayashi Y, Shibata TF, Hirakawa H, Nishiyama T, Yamada A, Hasebe M, Shigenobu S, Kawaguchi M. 2023. The genome of Lyophyllum shimeji provides insight into the initial evolution of ectomycorrhizal fungal genomes.
Kohler A, Kuo A, Nagy LG, Morin E, Barry KW, Buscot F, Canbäck B, Choi C, Cichocki N, Clum A et al. 2015. Convergent losses of decay mechanisms and rapid turnover of symbiosis genes in mycorrhizal mutualists. Nature Genetics 47: 410-415.
Kokkoris V, Chagnon PL, Yildirir G, Clarke K, Goh D, MacLean AM, Dettman J, Stefani F, Corradi N. 2021. Host identity influences nuclear dynamics in arbuscular mycorrhizal fungi. Current Biology 31: 1531-1538.
Kuyper TW, Jansa J. 2023. Arbuscular mycorrhiza: advances and retreats in our understanding of the ecological functioning of the mother of all root symbioses. Plant and Soil 489: 41-88.
Labbé J, Muchero W, Czarnecki O, Wang J, Wang X, Bryan AC, Zheng K, Yang Y, Xie M, Zhang J et al. 2019. Mediation of plant-mycorrhizal interaction by a lectin receptor-like kinase. Nature Plants 5: 676-680.
Labouyrie M, Ballabio C, Romero F, Panagos P, Jones A, Schmid MW, Mikryukov V, Dulya O, Tedersoo L, Bahram M et al. 2023. Patterns in soil microbial diversity across Europe. Nature Communications 14: 3311.
Lambers H, Raven JA, Shaver GR, Smith SE. 2008. Plant nutrient-acquisition strategies change with soil age. Trends in Ecology & Evolution 23: 95-103.
Lanfranco L, Bonfante P. 2023. Lessons from arbuscular mycorrhizal fungal genomes. Current Opinion in Microbiology 75: 102357.
Law SR, Serrano AR, Daguerre Y, Sundh J, Schneider AN, Stangl ZR, Castro D, Grabherr M, Näsholm T, Street NR et al. 2022. Metatranscriptomics captures dynamic shifts in mycorrhizal coordination in boreal forests. Proceedings of the National Academy of Sciences, USA 119: e2118852119.
Leake J, Johnson D, Donnelly D, Muckle G, Boddy L, Read D. 2004. Networks of power and influence: the role of mycorrhizal mycelium in controlling plant communities and agroecosystem functioning. Canadian Journal of Botany 82: 1016-1045.
Lebreton A, Tang N, Kuo A, LaButti K, Andreopoulos W, Drula E, Miyauchi S, Barry K, Clum A, Lipzen A et al. 2022. Comparative genomics reveals a dynamic genome evolution in the ectomycorrhizal milk-cap (Lactarius) mushrooms. New Phytologist 235: 306-319.
Lebreton A, Zeng Q, Miyauchi S, Kohler A, Dai Y-C, Martin FM. 2021. Evolution of the mode of nutrition in symbiotic and saprotrophic fungi in forest ecosystems. Annual Review of Ecology, Evolution, and Systematics 52: 385-404.
Ledford WC, Silvestri A, Fiorilli V, Roth R, Rubio-Somoza I, Lanfranco L. 2023. A journey into the world of small RNAs in the arbuscular mycorrhizal symbiosis. New Phytologist. doi: 10.1111/nph.19394.
Leifheit EF, Lehmann A, Rillig MC. 2021. Potential effects of microplastic on arbuscular mycorrhizal fungi. Frontiers in Plant Science 12: 626709.
Lekberg Y, Koide RT. 2005. Is plant performance limited by abundance of arbuscular mycorrhizal fungi? A meta-analysis of studies published between 1988 and 2003. New Phytologist 168: 189-204.
Li MH, Liu KW, Li Z, Lu HC, Ye QL, Zhang D, Wang JY, Li YF, Zhong ZM, Liu X et al. 2022. Genomes of leafy and leafless Platanthera orchids illuminate the evolution of mycoheterotrophy. Nature Plants 8: 373-388.
Li X, Zhao R, Li D, Wang G, Bei S, Ju X, An R, Li L, Kuyper TW, Christie P et al. 2023. Mycorrhiza-mediated recruitment of complete denitrifying Pseudomonas reduces
Liang M, Liu X, Parker IM, Johnson D, Zheng Y, Luo S, Gilbert GS, Yu S. 2019. Soil microbes drive phylogenetic diversity-productivity relationships in a subtropical forest. Science Advances 5: eaax5088.
Liao HL, Chen Y, Vilgalys R. 2018. Metatranscriptomic study of common and host-specific patterns of gene expression between pines and their symbiotic ectomycorrhizal fungi in the genus Suillus. PLoS Genetics 14: e1007742.
van der Linde S, Suz LM, Orme CDL, Cox F, Andreae H, Asi E, Atkinson B, Benham S, Carroll C, Cools N et al. 2018. Author correction: environment and host as large-scale controls of ectomycorrhizal fungi. Nature 561: E42.
Liu YR, van der Heijden MGA, Riedo J, Sanz-Lazaro C, Eldridge DJ, Bastida F, Moreno-Jiménez E, Zhou X-Q, Hu H-W, He J-Z et al. 2023. Soil contamination in nearby natural areas mirrors that in urban greenspaces worldwide. Nature Communications 14: 1706.
Lofgren LA, Nguyen NH, Vilgalys R, Ruytinx J, Liao H-L, Branco S, Kuo A, LaButti K, Lipzen A, Andreopoulos W et al. 2021. Comparative genomics reveals dynamic genome evolution in host specialist ectomycorrhizal fungi. New Phytologist 230: 774-792.
Looney B, Miyauchi S, Morin E, Drula E, Courty PE, Kohler A, Kuo A, LaButti K, Pangilinan J, Lipzen A et al. 2022. Evolutionary transition to the ectomycorrhizal habit in the genomes of a hyperdiverse lineage of mushroom-forming fungi. New Phytologist 233: 2294-2309.
Luo S, Phillips RP, Jo I, Fei S, Liang J, Schmid B, Eisenhauer N. 2023. Higher productivity in forests with mixed mycorrhizal strategies. Nature Communications 14: 1377.
Labbé J, Muchero W, Czarnecki O, Wang J, Wang X, Bryan AC, Zheng K, Yang Y, Xie M, Zhang J et al. 2019. Mediation of plant-mycorrhizal interaction by a lectin receptor-like kinase. Nature Plants 5: 676-680.
Labouyrie M, Ballabio C, Romero F, Panagos P, Jones A, Schmid MW, Mikryukov V, Dulya O, Tedersoo L, Bahram M et al. 2023. Patterns in soil microbial diversity across Europe. Nature Communications 14: 3311.
Lambers H, Raven JA, Shaver GR, Smith SE. 2008. Plant nutrient-acquisition strategies change with soil age. Trends in Ecology & Evolution 23: 95-103.
Lanfranco L, Bonfante P. 2023. Lessons from arbuscular mycorrhizal fungal genomes. Current Opinion in Microbiology 75: 102357.
Law SR, Serrano AR, Daguerre Y, Sundh J, Schneider AN, Stangl ZR, Castro D, Grabherr M, Näsholm T, Street NR et al. 2022. Metatranscriptomics captures dynamic shifts in mycorrhizal coordination in boreal forests. Proceedings of the National Academy of Sciences, USA 119: e2118852119.
Leake J, Johnson D, Donnelly D, Muckle G, Boddy L, Read D. 2004. Networks of power and influence: the role of mycorrhizal mycelium in controlling plant communities and agroecosystem functioning. Canadian Journal of Botany 82: 1016-1045.
Lebreton A, Tang N, Kuo A, LaButti K, Andreopoulos W, Drula E, Miyauchi S, Barry K, Clum A, Lipzen A et al. 2022. Comparative genomics reveals a dynamic genome evolution in the ectomycorrhizal milk-cap (Lactarius) mushrooms. New Phytologist 235: 306-319.
Lebreton A, Zeng Q, Miyauchi S, Kohler A, Dai Y-C, Martin FM. 2021. Evolution of the mode of nutrition in symbiotic and saprotrophic fungi in forest ecosystems. Annual Review of Ecology, Evolution, and Systematics 52: 385-404.
Ledford WC, Silvestri A, Fiorilli V, Roth R, Rubio-Somoza I, Lanfranco L. 2023. A journey into the world of small RNAs in the arbuscular mycorrhizal symbiosis. New Phytologist. doi: 10.1111/nph.19394.
Leifheit EF, Lehmann A, Rillig MC. 2021. Potential effects of microplastic on arbuscular mycorrhizal fungi. Frontiers in Plant Science 12: 626709.
Lekberg Y, Koide RT. 2005. Is plant performance limited by abundance of arbuscular mycorrhizal fungi? A meta-analysis of studies published between 1988 and 2003. New Phytologist 168: 189-204.
Li MH, Liu KW, Li Z, Lu HC, Ye QL, Zhang D, Wang JY, Li YF, Zhong ZM, Liu X et al. 2022. Genomes of leafy and leafless Platanthera orchids illuminate the evolution of mycoheterotrophy. Nature Plants 8: 373-388.
Li X, Zhao R, Li D, Wang G, Bei S, Ju X, An R, Li L, Kuyper TW, Christie P et al. 2023. Mycorrhiza-mediated recruitment of complete denitrifying Pseudomonas reduces
Liang M, Liu X, Parker IM, Johnson D, Zheng Y, Luo S, Gilbert GS, Yu S. 2019. Soil microbes drive phylogenetic diversity-productivity relationships in a subtropical forest. Science Advances 5: eaax5088.
Liao HL, Chen Y, Vilgalys R. 2018. Metatranscriptomic study of common and host-specific patterns of gene expression between pines and their symbiotic ectomycorrhizal fungi in the genus Suillus. PLoS Genetics 14: e1007742.
van der Linde S, Suz LM, Orme CDL, Cox F, Andreae H, Asi E, Atkinson B, Benham S, Carroll C, Cools N et al. 2018. Author correction: environment and host as large-scale controls of ectomycorrhizal fungi. Nature 561: E42.
Liu YR, van der Heijden MGA, Riedo J, Sanz-Lazaro C, Eldridge DJ, Bastida F, Moreno-Jiménez E, Zhou X-Q, Hu H-W, He J-Z et al. 2023. Soil contamination in nearby natural areas mirrors that in urban greenspaces worldwide. Nature Communications 14: 1706.
Lofgren LA, Nguyen NH, Vilgalys R, Ruytinx J, Liao H-L, Branco S, Kuo A, LaButti K, Lipzen A, Andreopoulos W et al. 2021. Comparative genomics reveals dynamic genome evolution in host specialist ectomycorrhizal fungi. New Phytologist 230: 774-792.
Looney B, Miyauchi S, Morin E, Drula E, Courty PE, Kohler A, Kuo A, LaButti K, Pangilinan J, Lipzen A et al. 2022. Evolutionary transition to the ectomycorrhizal habit in the genomes of a hyperdiverse lineage of mushroom-forming fungi. New Phytologist 233: 2294-2309.
Luo S, Phillips RP, Jo I, Fei S, Liang J, Schmid B, Eisenhauer N. 2023. Higher productivity in forests with mixed mycorrhizal strategies. Nature Communications 14: 1377.
Luo Y-H, Ma L-L, Seibold S, Cadotte MW, Burgess KS, Tan S-L, Ye L-J, Zheng W, Zou J-Y, Chen Z-F et al. 2023. The diversity of mycorrhiza-associated fungi and trees shapes subtropical mountain forest ecosystem functioning. Journal of Biogeography 50: 715-729.
Lutz S, Bodenhausen N, Hess J, Valzano-Held A, Waelchli J, Deslandes-Hérold G, Schlaeppi K, van der Heijden MGA. 2023. Soil microbiome indicators can predict crop growth response to large-scale inoculation with arbuscular mycorrhizal fungi. Nature Microbiology 8: 2277-2289.
Mahmood S, Fahad Z, Bolou-Bi EB, King K, Köhler SJ, Bishop K, Ekblad A, Finlay RD. 2023. Ectomycorrhizal fungi integrate nitrogen mobilisation and mineral weathering in boreal forest soil. New Phytologist. doi: 10.1111/nph. 19260.
Lutz S, Bodenhausen N, Hess J, Valzano-Held A, Waelchli J, Deslandes-Hérold G, Schlaeppi K, van der Heijden MGA. 2023. Soil microbiome indicators can predict crop growth response to large-scale inoculation with arbuscular mycorrhizal fungi. Nature Microbiology 8: 2277-2289.
Mahmood S, Fahad Z, Bolou-Bi EB, King K, Köhler SJ, Bishop K, Ekblad A, Finlay RD. 2023. Ectomycorrhizal fungi integrate nitrogen mobilisation and mineral weathering in boreal forest soil. New Phytologist. doi: 10.1111/nph. 19260.
Maillard F, Kohler A, Morin E, Hossann C, Miyauchi S, Ziegler-Devin I, Gérant D, Angeli N, Lipzen A, Keymanesh K et al. 2023. Functional genomics gives new insights into the ectomycorrhizal degradation of chitin. New Phytologist 238: 845858.
Malar CM, Krüger M, Krüger C, Wang Y, Stajich JE, Keller J, Chen ECH, Yildirir G, Villeneuve-Laroche M, Roux C et al. 2021. The genome of Geosiphon pyriformis reveals ancestral traits linked to the emergence of the arbuscular mycorrhizal symbiosis. Current Biology 31: 1570-1577.
Malar CM, Wang Y, Stajich JE, Kokkoris V, Villeneuve-Laroche M, Yildirir G, Corradi N. 2022. Early branching arbuscular mycorrhizal fungus Paraglomus occultum carries a small and repeat-poor genome compared to relatives in the Glomeromycotina. Microbial Genomics 8: 810.
Marqués-Gálvez JE, Miyauchi S, Paolocci F, Navarro-Ródenas A, Arenas F, PérezGilabert M, Morin E, Auer L, Barry KW, Kuo A et al. 2021. Desert truffle genomes reveal their reproductive modes and new insights into plant-fungal interaction and ectendomycorrhizal lifestyle. New Phytologist 229: 2917-2932.
Martin F, Aerts A, Ahrén D, Brun A, Danchin EGJ, Duchaussoy F, Gibon J, Kohler A, Lindquist E, Pereda V et al. 2008. The genome of Laccaria bicolor provides insights into mycorrhizal symbiosis. Nature 452: 88-92.
Martin F, Cullen D, Hibbett D, Pisabarro A, Spatafora JW, Baker SE, Grigoriev IV. 2011. Sequencing the fungal tree of life. New Phytologist 190: 818-821.
Malar CM, Wang Y, Stajich JE, Kokkoris V, Villeneuve-Laroche M, Yildirir G, Corradi N. 2022. Early branching arbuscular mycorrhizal fungus Paraglomus occultum carries a small and repeat-poor genome compared to relatives in the Glomeromycotina. Microbial Genomics 8: 810.
Marqués-Gálvez JE, Miyauchi S, Paolocci F, Navarro-Ródenas A, Arenas F, PérezGilabert M, Morin E, Auer L, Barry KW, Kuo A et al. 2021. Desert truffle genomes reveal their reproductive modes and new insights into plant-fungal interaction and ectendomycorrhizal lifestyle. New Phytologist 229: 2917-2932.
Martin F, Aerts A, Ahrén D, Brun A, Danchin EGJ, Duchaussoy F, Gibon J, Kohler A, Lindquist E, Pereda V et al. 2008. The genome of Laccaria bicolor provides insights into mycorrhizal symbiosis. Nature 452: 88-92.
Martin F, Cullen D, Hibbett D, Pisabarro A, Spatafora JW, Baker SE, Grigoriev IV. 2011. Sequencing the fungal tree of life. New Phytologist 190: 818-821.
Martin F, Kohler A, Murat C, Balestrini R, Coutinho PM, Jaillon O, Montanini B, Morin E, Noel B, Percudani R et al. 2010. Périgord black truffle genome uncovers evolutionary origins and mechanisms of symbiosis. Nature 464: 10331038.
Martin F, Kohler A, Murat C, Veneault-Fourrey C, Hibbett DS. 2016. Unearthing the roots of ectomycorrhizal symbioses. Nature Reviews Microbiology 14: 760773.
Martin F, Selosse MA. 2008. The Laccaria genome: a symbiont blueprint decoded. New Phytologist 180: 296-310.
Martin F, Uroz S, Barker D. 2017. Ancestral alliances: plant mutualistic symbioses with fungi and bacteria. Science 356: 819.
Martino E, Morin E, Grelet GA, Kuo A, Kohler A, Daghino S, Barry KW, Cichocki N, Clum A, Dockter RB et al. 2018. Comparative genomics and transcriptomics depict ericoid mycorrhizal fungi as versatile saprotrophs and plant mutualists. New Phytologist 217: 1213-1229.
Martín-Robles N, Lehmann A, Seco E, Aroca R, Rillig MC, Milla R. 2018. Impacts of domestication on the arbuscular mycorrhizal symbiosis of 27 crop species. New Phytologist 218: 322-334.
Mansfield TM, Albornoz FE, Ryan MH, Bending GD, Standish RJ. 2023. Niche differentiation of Mucoromycotinian and Glomeromycotinian arbuscular mycorrhizal fungi along a 2-million-year soil chronosequence. Mycorrhiza 33: 1-14.
Mateus ID, Masclaux FG, Aletti C, Rojas EC, Savary R, Dupuis C, Sanders IR. 2019. Dual RNA-seq reveals large-scale non-conserved genotype
Merckx V. 2023. Mycoheterotrophy. The biology of plants living on fungi. New York, NY, USA: Springer Science & Business Media, 356.
Miyauchi S, Kiss E, Kuo A, Drula E, Kohler A, Sánchez-García M, Morin E, Andreopoulos B, Barry KW, Bonito G et al. 2020. Large-scale genome sequencing of mycorrhizal fungi provides insights into the early evolution of symbiotic traits. Nature Communications 11: 5125.
Morin E, Miyauchi S, San Clemente H, Chen ECH, Pelin A, de la Providencia I, Ndikumana S, Beaudet D, Hainaut M, Drula E et al. 2019. Comparative
genomics of Rhizophagus irregularis, R. cerebriforme, R. diaphanus and Gigaspora rosea highlights specific genetic features in Glomeromycotina. New Phytologist 222: 1584-1598.
Murat C, Payen T, Noel B, Kuo A, Morin E, Chen J, Kohler A, Krizsán K, Balestrini R, Da Silva C et al. 2018. Pezizomycetes genomes reveal the molecular basis of ectomycorrhizal truffle lifestyle. Nature Ecology & Evolution 2: 19561965.
Martin F, Uroz S, Barker D. 2017. Ancestral alliances: plant mutualistic symbioses with fungi and bacteria. Science 356: 819.
Martino E, Morin E, Grelet GA, Kuo A, Kohler A, Daghino S, Barry KW, Cichocki N, Clum A, Dockter RB et al. 2018. Comparative genomics and transcriptomics depict ericoid mycorrhizal fungi as versatile saprotrophs and plant mutualists. New Phytologist 217: 1213-1229.
Martín-Robles N, Lehmann A, Seco E, Aroca R, Rillig MC, Milla R. 2018. Impacts of domestication on the arbuscular mycorrhizal symbiosis of 27 crop species. New Phytologist 218: 322-334.
Mansfield TM, Albornoz FE, Ryan MH, Bending GD, Standish RJ. 2023. Niche differentiation of Mucoromycotinian and Glomeromycotinian arbuscular mycorrhizal fungi along a 2-million-year soil chronosequence. Mycorrhiza 33: 1-14.
Mateus ID, Masclaux FG, Aletti C, Rojas EC, Savary R, Dupuis C, Sanders IR. 2019. Dual RNA-seq reveals large-scale non-conserved genotype
Merckx V. 2023. Mycoheterotrophy. The biology of plants living on fungi. New York, NY, USA: Springer Science & Business Media, 356.
Miyauchi S, Kiss E, Kuo A, Drula E, Kohler A, Sánchez-García M, Morin E, Andreopoulos B, Barry KW, Bonito G et al. 2020. Large-scale genome sequencing of mycorrhizal fungi provides insights into the early evolution of symbiotic traits. Nature Communications 11: 5125.
Morin E, Miyauchi S, San Clemente H, Chen ECH, Pelin A, de la Providencia I, Ndikumana S, Beaudet D, Hainaut M, Drula E et al. 2019. Comparative
genomics of Rhizophagus irregularis, R. cerebriforme, R. diaphanus and Gigaspora rosea highlights specific genetic features in Glomeromycotina. New Phytologist 222: 1584-1598.
Murat C, Payen T, Noel B, Kuo A, Morin E, Chen J, Kohler A, Krizsán K, Balestrini R, Da Silva C et al. 2018. Pezizomycetes genomes reveal the molecular basis of ectomycorrhizal truffle lifestyle. Nature Ecology & Evolution 2: 19561965.
Murat C, Rubini A, Riccioni C, De la Varga H, Akroume E, Belfiori B, Guaragno M, Le Tacon F, Robin C, Halkett F et al. 2013. Fine-scale spatial genetic structure of the black truffle (Tuber melanosporum) investigated with neutral microsatellites and functional mating type genes. New Phytologist 199: 176-187.
Nehls U, Plassard C. 2018. Nitrogen and phosphate metabolism in ectomycorrhizas. New Phytologist 220: 1047-1058.
Newman EI, Reddell P. 1988. Relationship between mycorrhizal infection and diversity in vegetation: evidence from the Great Smoky Mountains. Functional Ecology 2: 259-262.
Nicolás C, Martin-Bertelsen T, Floudas D, Bentzer J, Smits M, Johansson T, Troein C, Persson P, Tunlid A. 2019. The soil organic matter decomposition mechanisms in ectomycorrhizal fungi are tuned for liberating soil organic nitrogen. ISME Journal 13: 977-988.
Oliveira RS, Rocha I, Ma Y, Vosátka M, Freitas H. 2019. Seed coating with arbuscular mycorrhizal fungi for improved field production of chickpea. Agronomy 9: 471.
Oliverio AM, Geisen S, Delgado-Baquerizo M, Maestre FT, Turner BL, Fierer N. 2020. The global-scale distributions of soil protists and their contributions to belowground systems. Science. Advances 6: eaax8787.
Op De Beeck M, Troein C, Peterson C, Persson P, Tunlid A. 2018. Fenton reaction facilitates organic nitrogen acquisition by an ectomycorrhizal fungus. New Phytologist 218: 335-343.
Öpik M, Vanatoa A, Vanatoa E, Moora M, Davison J, Kalwij JM, Zobel M. 2010. The online database MaarjAM reveals global and ecosystemic distribution patterns in arbuscular mycorrhizal fungi Glomeromycota. New Phytologist 188: 223-241.
Paoletti M. 2016. Vegetative incompatibility in fungi: from recognition to cell death, whatever does the trick. Fungal Biology Reviews 30: 152-162.
Peay KG, Kennedy PG, Talbot JM. 2016. Dimensions of biodiversity in the earth mycobiome. Nature Reviews Microbiology 14: 434-447.
Pellegrin C, Morin E, Martin F, Veneault-Fourrey C. 2015. Comparative analysis of secretomes from ectomycorrhizal fungi with an emphasis on small-secreted proteins. Frontiers in Microbiology 6: e1278.
Pellitier PT, Zak DR. 2018. Ectomycorrhizal fungi and the enzymatic liberation of nitrogen from soil organic matter: why evolutionary history matters. New Phytologist 217: 68-73.
Perotto S, Balestrini R. 2023. At the core of the endomycorrhizal symbioses: intracellular fungal structures in orchid and arbuscular mycorrhiza. New Phytologist. doi: 10.1111/nph. 19338.
Peter M, Kohler A, Ohm RA, Kuo A, Krützmann J, Morin E, Arend M, Barry KW, Binder M, Choi C et al. 2016. Ectomycorrhizal ecology is imprinted in the genome of the dominant symbiotic fungus Cenococcum geophilum. Nature Communications 7: 12662.
Phillips HR, Guerra CA, Bartz ML, Briones MJ, Brown G, Crowther TW, Orgiazzi A. 2019. Global distribution of earthworm diversity. Science 366: 480485.
Nehls U, Plassard C. 2018. Nitrogen and phosphate metabolism in ectomycorrhizas. New Phytologist 220: 1047-1058.
Newman EI, Reddell P. 1988. Relationship between mycorrhizal infection and diversity in vegetation: evidence from the Great Smoky Mountains. Functional Ecology 2: 259-262.
Nicolás C, Martin-Bertelsen T, Floudas D, Bentzer J, Smits M, Johansson T, Troein C, Persson P, Tunlid A. 2019. The soil organic matter decomposition mechanisms in ectomycorrhizal fungi are tuned for liberating soil organic nitrogen. ISME Journal 13: 977-988.
Oliveira RS, Rocha I, Ma Y, Vosátka M, Freitas H. 2019. Seed coating with arbuscular mycorrhizal fungi for improved field production of chickpea. Agronomy 9: 471.
Oliverio AM, Geisen S, Delgado-Baquerizo M, Maestre FT, Turner BL, Fierer N. 2020. The global-scale distributions of soil protists and their contributions to belowground systems. Science. Advances 6: eaax8787.
Op De Beeck M, Troein C, Peterson C, Persson P, Tunlid A. 2018. Fenton reaction facilitates organic nitrogen acquisition by an ectomycorrhizal fungus. New Phytologist 218: 335-343.
Öpik M, Vanatoa A, Vanatoa E, Moora M, Davison J, Kalwij JM, Zobel M. 2010. The online database MaarjAM reveals global and ecosystemic distribution patterns in arbuscular mycorrhizal fungi Glomeromycota. New Phytologist 188: 223-241.
Paoletti M. 2016. Vegetative incompatibility in fungi: from recognition to cell death, whatever does the trick. Fungal Biology Reviews 30: 152-162.
Peay KG, Kennedy PG, Talbot JM. 2016. Dimensions of biodiversity in the earth mycobiome. Nature Reviews Microbiology 14: 434-447.
Pellegrin C, Morin E, Martin F, Veneault-Fourrey C. 2015. Comparative analysis of secretomes from ectomycorrhizal fungi with an emphasis on small-secreted proteins. Frontiers in Microbiology 6: e1278.
Pellitier PT, Zak DR. 2018. Ectomycorrhizal fungi and the enzymatic liberation of nitrogen from soil organic matter: why evolutionary history matters. New Phytologist 217: 68-73.
Perotto S, Balestrini R. 2023. At the core of the endomycorrhizal symbioses: intracellular fungal structures in orchid and arbuscular mycorrhiza. New Phytologist. doi: 10.1111/nph. 19338.
Peter M, Kohler A, Ohm RA, Kuo A, Krützmann J, Morin E, Arend M, Barry KW, Binder M, Choi C et al. 2016. Ectomycorrhizal ecology is imprinted in the genome of the dominant symbiotic fungus Cenococcum geophilum. Nature Communications 7: 12662.
Phillips HR, Guerra CA, Bartz ML, Briones MJ, Brown G, Crowther TW, Orgiazzi A. 2019. Global distribution of earthworm diversity. Science 366: 480485.
Plett JM, Daguerre Y, Wittulsky S, Vayssieres A, Deveau A, Melton SJ, Kohler A, Morrel-Falvey JL, Brun A, Veneault-Fourrey C et al. 2014. Effector MiSSP7 of the mutualistic fungus Laccaria bicolor stabilizes the Populus JAZ6 protein and represses jasmonic acid-responsive genes. Proceedings of the National Academy of Sciences, USA 111: 8299-8304.
Plett JM, Kemppainen M, Kale SD, Kohler A, Legué V, Brun A, Tyler B, Pardo A, Martin F. 2011. A secreted effector protein of Laccaria bicolor is required for symbiosis development. Current Biology 21: 1197-1203.
Plett JM, Martin F. 2011. Blurred boundaries: lifestyle lessons from ectomycorrhizal fungal genomes. Trends in Genetics 27: 14-22.
Plett JM, Miyauchi S, Morin E, Plett K, Wong-Bajracharya J, de Freitas PM, Kuo A, Henrissat B, Drula E, Wojtalewicz D et al. 2023. Speciation underpinned by
unexpected molecular diversity in the mycorrhizal fungal genus Pisolithus. Molecular Biology and Evolution 40: msad045.
Plett KL, Kohler A, Lebel T, Singan VR, Bauer D, He G, Ng V, Grigoriev IV, Martin F, Plett JM et al. 2021. Intra-species genetic variability drives carbon metabolism and symbiotic host interactions in the ectomycorrhizal fungus Pisolithus microcarpus. Environmental Microbiology 23: 2004-2020.
Plett KL, Raposo AE, Anderson IC, Piller SC, Plett JM. 2019. Protein arginine methyltransferase expression affects ectomycorrhizal symbiosis and the regulation of hormone signaling pathways. Molecular Plant-Microbe Interactions 32: 12911302.
Plett JM, Kemppainen M, Kale SD, Kohler A, Legué V, Brun A, Tyler B, Pardo A, Martin F. 2011. A secreted effector protein of Laccaria bicolor is required for symbiosis development. Current Biology 21: 1197-1203.
Plett JM, Martin F. 2011. Blurred boundaries: lifestyle lessons from ectomycorrhizal fungal genomes. Trends in Genetics 27: 14-22.
Plett JM, Miyauchi S, Morin E, Plett K, Wong-Bajracharya J, de Freitas PM, Kuo A, Henrissat B, Drula E, Wojtalewicz D et al. 2023. Speciation underpinned by
unexpected molecular diversity in the mycorrhizal fungal genus Pisolithus. Molecular Biology and Evolution 40: msad045.
Plett KL, Kohler A, Lebel T, Singan VR, Bauer D, He G, Ng V, Grigoriev IV, Martin F, Plett JM et al. 2021. Intra-species genetic variability drives carbon metabolism and symbiotic host interactions in the ectomycorrhizal fungus Pisolithus microcarpus. Environmental Microbiology 23: 2004-2020.
Plett KL, Raposo AE, Anderson IC, Piller SC, Plett JM. 2019. Protein arginine methyltransferase expression affects ectomycorrhizal symbiosis and the regulation of hormone signaling pathways. Molecular Plant-Microbe Interactions 32: 12911302.
Plett KL, Wojtalewicz D, Anderson IC, Plett JM. 2023. Fungal metabolism and free amino acid content may predict nitrogen transfer to the host plant in the ectomycorrhizal relationship between Pisolithus spp. and Eucalyptus grandis. New Phytologist. doi: 10.1111/nph.19400.
Pringle A, Bever JD, Gardes M, Parrent JL, Rillig MC, Klironomos JN. 2009. Mycorrhizal symbioses and plant invasions. Annual Review of Ecology, Evolution, and Systematics 40: 699-715.
Querejeta JI, Schlaeppi K, López-García Á, Ondoño S, Prieto I, van Der Heijden MG, del Mar Alguacil M. 2021. Lower relative abundance of ectomycorrhizal fungi under a warmer and drier climate is linked to enhanced soil organic matter decomposition. New Phytologist 232: 1399-1413.
Rasmussen HN, Rasmussen FN. 2014. Seedling mycorrhiza: a discussion of origin and evolution in Orchidaceae. Botanical Journal of the Linnean Society 175: 313327.
Pringle A, Bever JD, Gardes M, Parrent JL, Rillig MC, Klironomos JN. 2009. Mycorrhizal symbioses and plant invasions. Annual Review of Ecology, Evolution, and Systematics 40: 699-715.
Querejeta JI, Schlaeppi K, López-García Á, Ondoño S, Prieto I, van Der Heijden MG, del Mar Alguacil M. 2021. Lower relative abundance of ectomycorrhizal fungi under a warmer and drier climate is linked to enhanced soil organic matter decomposition. New Phytologist 232: 1399-1413.
Rasmussen HN, Rasmussen FN. 2014. Seedling mycorrhiza: a discussion of origin and evolution in Orchidaceae. Botanical Journal of the Linnean Society 175: 313327.
Read DJ. 1991. Mycorrhizas in ecosystems. Experientia 47: 376-391.
Reinhardt D, Roux C, Corradi N, Di Pietro A. 2021. Lineage-specific genes and cryptic sex: parallels and differences between arbuscular mycorrhizal fungi and fungal pathogens. Trends in Plant Science 26: 111-123.
Rillig MC, Aguilar-Trigueros CA, Camenzind T, Cavagnaro TR, Degrune F, Hohmann P, Lammel DR, Mansour I, Roy J, van der Heijden MGA et al. 2019a. Why farmers should manage the arbuscular mycorrhizal symbiosis. New Phytologist 222: 1171-1175.
Rillig MC, Ryo M, Lehmann A, Aguilar-Trigueros CA, Buchert S, Wulf A, Yang G, Roy J, Yang G. 2019b. The role of multiple global change factors in driving soil functions and microbial biodiversity. Science 366: 886-890.
Rillig MC, van der Heijden M, Berdugo M, Liu YR, Riedo J, Sanz-Lazaro C, Delgado-Baquerizo M. 2023. Increasing the number of stressors reduces soil ecosystem services worldwide. Nature Climate Change 13: 478-483.
Rivera Pérez CA, Janz D, Schneider D, Daniel R, Polle A. 2022. Transcriptional landscape of ectomycorrhizal fungi and their host provides insight into N uptake from forest soil. mSystems 7: e0095721.
Robinson D, Fitter A. 1999. The magnitude and control of carbon transfer between plants linked by a common mycorrhizal network. Journal of Experimental Botany 50: 9-13.
Robinson DG, Ammer C, Polle A, Bauhus J, Aloni R, Annighöfer P, Baskin TI, Blatt MR, Bolte A, Bugmann H et al. 2024. Mother trees, altruistic fungi, and the perils of plant personification. Trends in Plant Sciences 29: 20-31.
Rocha I, Ma Y, Souza-Alonso P, Vosátka M, Freitas H, Oliveira RS. 2019. Seed coating: a tool for delivering beneficial microbes to agricultural crops. Frontiers in Plant Science 10: 1357.
Rog I, Rosenstock NP, Körner C, Klein T. 2020. Share the wealth: trees with greater ectomycorrhizal species overlap share more carbon. Molecular Ecology 29: 23212333.
Reinhardt D, Roux C, Corradi N, Di Pietro A. 2021. Lineage-specific genes and cryptic sex: parallels and differences between arbuscular mycorrhizal fungi and fungal pathogens. Trends in Plant Science 26: 111-123.
Rillig MC, Aguilar-Trigueros CA, Camenzind T, Cavagnaro TR, Degrune F, Hohmann P, Lammel DR, Mansour I, Roy J, van der Heijden MGA et al. 2019a. Why farmers should manage the arbuscular mycorrhizal symbiosis. New Phytologist 222: 1171-1175.
Rillig MC, Ryo M, Lehmann A, Aguilar-Trigueros CA, Buchert S, Wulf A, Yang G, Roy J, Yang G. 2019b. The role of multiple global change factors in driving soil functions and microbial biodiversity. Science 366: 886-890.
Rillig MC, van der Heijden M, Berdugo M, Liu YR, Riedo J, Sanz-Lazaro C, Delgado-Baquerizo M. 2023. Increasing the number of stressors reduces soil ecosystem services worldwide. Nature Climate Change 13: 478-483.
Rivera Pérez CA, Janz D, Schneider D, Daniel R, Polle A. 2022. Transcriptional landscape of ectomycorrhizal fungi and their host provides insight into N uptake from forest soil. mSystems 7: e0095721.
Robinson D, Fitter A. 1999. The magnitude and control of carbon transfer between plants linked by a common mycorrhizal network. Journal of Experimental Botany 50: 9-13.
Robinson DG, Ammer C, Polle A, Bauhus J, Aloni R, Annighöfer P, Baskin TI, Blatt MR, Bolte A, Bugmann H et al. 2024. Mother trees, altruistic fungi, and the perils of plant personification. Trends in Plant Sciences 29: 20-31.
Rocha I, Ma Y, Souza-Alonso P, Vosátka M, Freitas H, Oliveira RS. 2019. Seed coating: a tool for delivering beneficial microbes to agricultural crops. Frontiers in Plant Science 10: 1357.
Rog I, Rosenstock NP, Körner C, Klein T. 2020. Share the wealth: trees with greater ectomycorrhizal species overlap share more carbon. Molecular Ecology 29: 23212333.
Ropars J, Toro KS, Noel J, Pelin A, Charron P, Farinelli L, Marton T, Krüger M, Fuchs J, Brachmann A et al. 2016. Evidence for the sexual origin of heterokaryosis in arbuscular mycorrhizal fungi. Nature Microbiology 1: 16033.
Rozmoš M, Bukovská P, Hršelová H, Kotianová M, Dudáš M, Gančarčíková K, Jansa J. 2022. Organic nitrogen utilisation by an arbuscular mycorrhizal fungus is mediated by specific soil bacteria and a protist. ISME Journal 16: 676-685.
Ruytinx J, Miyauchi S, Hartmann-Wittulsky S, de Freitas PM, Guinet F, Churin JL, Put C, Le Tacon F, Veneault-Fourrey C, Martin F et al. 2021. A transcriptomic atlas of the ectomycorrhizal fungus Laccaria bicolor. Microorganisms 9: 2612.
Ryan MH, Graham JH. 2018. Little evidence that farmers should consider abundance or diversity of arbuscular mycorrhizal fungi when managing crops. New Phytologist 220: 1092-1107.
Rozmoš M, Bukovská P, Hršelová H, Kotianová M, Dudáš M, Gančarčíková K, Jansa J. 2022. Organic nitrogen utilisation by an arbuscular mycorrhizal fungus is mediated by specific soil bacteria and a protist. ISME Journal 16: 676-685.
Ruytinx J, Miyauchi S, Hartmann-Wittulsky S, de Freitas PM, Guinet F, Churin JL, Put C, Le Tacon F, Veneault-Fourrey C, Martin F et al. 2021. A transcriptomic atlas of the ectomycorrhizal fungus Laccaria bicolor. Microorganisms 9: 2612.
Ryan MH, Graham JH. 2018. Little evidence that farmers should consider abundance or diversity of arbuscular mycorrhizal fungi when managing crops. New Phytologist 220: 1092-1107.
Saito K, Ezawa T. 2016. Phosphorus metabolism and transport in arbuscular mycorrhizal symbiosis. Molecular mycorrhizal symbiosis: 197-216.
Säle V, Palenzuela J, Azcón-Aguilar C, Sánchez-Castro I, da Silva GA, Seitz B, Sieverding E, van der Heijden MGA, Oehl F. 2021. Ancient lineages of arbuscular mycorrhizal fungi provide little plant benefit. Mycorrhiza 31: 559576.
Säle V, Palenzuela J, Azcón-Aguilar C, Sánchez-Castro I, da Silva GA, Seitz B, Sieverding E, van der Heijden MGA, Oehl F. 2021. Ancient lineages of arbuscular mycorrhizal fungi provide little plant benefit. Mycorrhiza 31: 559576.
Salomon MJ, Watts-Williams SJ, McLaughlin MJ, Bücking H, Singh BK, Hutter I, Schneider C, Martin FM, Vosatka M, Guo L et al. 2022. Establishing a quality management framework for commercial inoculants containing arbuscular mycorrhizal fungi. iScience 25: 7.
Sato H. 2023. The evolution of ectomycorrhizal symbiosis in the Late Cretaceous is a key driver of explosive diversification in Agaricomycetes. New Phytologist 241: 444460.
Sato H. 2023. The evolution of ectomycorrhizal symbiosis in the Late Cretaceous is a key driver of explosive diversification in Agaricomycetes. New Phytologist 241: 444460.
Schnitzer SA, Klironomos JN, HilleRisLambers J, Kinkel LL, Reich PB, Xiao K, Rillig MC, Sikes B, Callaway RM, Mangan SA et al. 2011. Soil microbes drive the classic plant diversity-productivity pattern. Ecology 92: 296-303.
Schwery O, Onstein RE, Bouchenak-Khelladi Y, Xing Y, Carter RJ, Linder HP. 2015. As old as the mountains: the radiations of the Ericaceae. New Phytologist 207: 355-367.
Selosse MA, Bocayuva MF, Kasuya MCM, Courty PE. 2016. Mixotrophy in mycorrhizal plants: extracting carbon from mycorrhizal networks. Molecular mycorrhizal symbiosis: 451-471.
Selosse MA, Le Tacon F. 1998. The land flora: a phototroph-fungus partnership? Trends in Ecology & Evolution 13: 15-20.
Serrano K, Bezrutczyk M, Goudeau D, Dao T, O’Malley R, Malmstrom RR, Visel A, Scheller H, Cole B. 2023. Spatial co-transcriptomics reveals discrete stages of the arbuscular mycorrhizal symbiosis. BioRxiv. doi: 10.1101/2023.08.02. 551648.
Schwery O, Onstein RE, Bouchenak-Khelladi Y, Xing Y, Carter RJ, Linder HP. 2015. As old as the mountains: the radiations of the Ericaceae. New Phytologist 207: 355-367.
Selosse MA, Bocayuva MF, Kasuya MCM, Courty PE. 2016. Mixotrophy in mycorrhizal plants: extracting carbon from mycorrhizal networks. Molecular mycorrhizal symbiosis: 451-471.
Selosse MA, Le Tacon F. 1998. The land flora: a phototroph-fungus partnership? Trends in Ecology & Evolution 13: 15-20.
Serrano K, Bezrutczyk M, Goudeau D, Dao T, O’Malley R, Malmstrom RR, Visel A, Scheller H, Cole B. 2023. Spatial co-transcriptomics reveals discrete stages of the arbuscular mycorrhizal symbiosis. BioRxiv. doi: 10.1101/2023.08.02. 551648.
Silverstein MR, Segrè D, Bhatnagar JM. 2023. Environmental microbiome engineering for the mitigation of climate change. Global Change Biology 29: 20502066.
Simard SW, Perry DA, Jones MD, Myrold DD, Durall DM, Molina R. 1997. Net transfer of carbon between ectomycorrhizal tree species in the field. Nature 388: 579-582.
Simon L, Bousquet J, Lévesque R, Lalonde M. 1993. Origin and diversification of endomycorrhizal fungi and coincidence with vascular land plants. Nature 363: 67-69.
Song C, Wang Y, Manzoor MA, Mao D, Wei P, Cao Y, Zhu F. 2022. In-depth analysis of genomes and functional genomics of orchid using cutting-edge highthroughput sequencing. Frontiers in Plant Science 13: 1018029.
Soudzilovskaia NA, van Bodegom PM, Terrer C, Zelfde MVT, McCallum I, Luke McCormack M, Tedersoo L. 2019. Global mycorrhizal plant distribution linked to terrestrial carbon stocks. Nature Communications 10: 5077.
Spatafora JW, Chang Y, Benny GL, Lazarus K, Smith ME, Berbee ML, Bonito G, Corradi N, Grigoriev I, Gryganskyi A et al. 2016. A phylum-level phylogenetic classification of zygomycete fungi based on genome-scale data. Mycologia 108: 1028-1046.
Sperschneider J, Yildirir G, Rizzi Y, Malar MC, Mayrand A, Sorwar E, Chen ECH, Iwasaki W, Brauer EK, Bosnich W et al. 2023. Arbuscular mycorrhizal fungi heterokaryons have two nuclear populations with distinct roles in host-plant interactions. Nature Microbiology 8: 2142-2153.
Strassert JF, Monaghan MT. 2022. Phylogenomic insights into the early diversification of fungi. Current Biology 32: 3628-3635.
Strullu-Derrien C, Selosse M-A, Kenrick P, Martin FM. 2018. The origin and evolution of mycorrhizal symbioses: from palaeomycology to phylogenomics. New Phytologist 220: 1012-1030.
Stuart EK, Singan V, Amirebrahimi M, Na H, Ng V, Grigoriev I, Martin F, Anderson IC, Plett JM, Plett KL. 2023. Acquisition of host-derived carbon in biomass of the ectomycorrhizal fungus Pisolithus microcarpus is correlated to fungal carbon demand and plant defences. FEMS Microbiology Ecology 99: fiad037.
Svenningsen NB, Watts-Williams SJ, Joner EJ, Battini F, Efthymiou A, Cruz-Paredes C, Nybroe O, Jakobsen I. 2018. Suppression of the activity of arbuscular mycorrhizal fungi by the soil microbiota. ISME Journal 12: 1296-1307.
Tagu D, Bastien C, Faivre-Rampant P, Garbaye J, Vion P, Villar M, Martin F. 2005. Genetic analysis of phenotypic variation for ectomycorrhiza
formation in an interspecific F1 poplar full-sib family. Mycorrhiza 15: 87-91.
Tang N, Lebreton A, Xu W, Dai Y-C, Yu F, Martin FM. 2021. Transcriptome profiling reveals differential gene expression of secreted proteases and highly specific gene repertoires involved in Lactarius-Pinus symbioses. Frontiers in Plant Science 12: 1775.
Tedersoo L, Bahram M, Põlme S, Kõljalg U, Yorou NS, Wijesundera R, Villarreal Ruiz L, Vasco-Palacios AM, Thu PQ, Suija A et al. 2014. Global diversity and geography of soil fungi. Science 346: 6213.
Tedersoo L, May TW, Smith ME. 2010. Ectomycorrhizal lifestyle in fungi: global diversity, distribution, and evolution of phylogenetic lineages. Mycorrhiza 20: 217-263.
Teulet A, Quan C, Evangelisti E, Wanke A, Yang W, Schornack S. 2023. A pathogen effector FOLD diversified in symbiotic fungi. New Phytologist 239: 1127-1139.
Thirkell TJ, Grimmer M, James L, Pastok D, Allary T, Elliott A, Paveley N, Daniell T, Field KJ. 2022. Variation in mycorrhizal growth response among a spring wheat mapping population shows potential to breed for symbiotic benefit. Food and Energy Security 11: e370.
Tisserant E, Malbreil M, Kuo A, Kohler A, Symeonidi A, Balestrini R, Charron P, Duensing N, Frei Dit Frey N, Gianinazzi-Pearson V et al. 2013. Genome of an arbuscular mycorrhizal fungus provides insight into the oldest plant symbiosis. Proceedings of the National Academy of Sciences, USA 110: 20117-20122.
Trivedi P, Batista BD, Bazany KE, Singh BK. 2022. Plant-microbiome interactions under a changing world: responses, consequences and perspectives. New Phytologist 234: 1951-1959.
Tsuzuki S, Handa Y, Takeda N, Kawaguchi M. 2016. Strigolactone-induced putative secreted protein 1 is required for the establishment of symbiosis by the arbuscular mycorrhizal fungus Rhizophagus irregularis. Molecular Plant-Microbe Interactions 29: 277-286.
Vandenkoornhuyse P, Quaiser A, Duhamel M, Le Van A, Dufresne A. 2015. The importance of the microbiome of the plant holobiont. New Phytologist 206: 11961206.
Simon L, Bousquet J, Lévesque R, Lalonde M. 1993. Origin and diversification of endomycorrhizal fungi and coincidence with vascular land plants. Nature 363: 67-69.
Song C, Wang Y, Manzoor MA, Mao D, Wei P, Cao Y, Zhu F. 2022. In-depth analysis of genomes and functional genomics of orchid using cutting-edge highthroughput sequencing. Frontiers in Plant Science 13: 1018029.
Soudzilovskaia NA, van Bodegom PM, Terrer C, Zelfde MVT, McCallum I, Luke McCormack M, Tedersoo L. 2019. Global mycorrhizal plant distribution linked to terrestrial carbon stocks. Nature Communications 10: 5077.
Spatafora JW, Chang Y, Benny GL, Lazarus K, Smith ME, Berbee ML, Bonito G, Corradi N, Grigoriev I, Gryganskyi A et al. 2016. A phylum-level phylogenetic classification of zygomycete fungi based on genome-scale data. Mycologia 108: 1028-1046.
Sperschneider J, Yildirir G, Rizzi Y, Malar MC, Mayrand A, Sorwar E, Chen ECH, Iwasaki W, Brauer EK, Bosnich W et al. 2023. Arbuscular mycorrhizal fungi heterokaryons have two nuclear populations with distinct roles in host-plant interactions. Nature Microbiology 8: 2142-2153.
Strassert JF, Monaghan MT. 2022. Phylogenomic insights into the early diversification of fungi. Current Biology 32: 3628-3635.
Strullu-Derrien C, Selosse M-A, Kenrick P, Martin FM. 2018. The origin and evolution of mycorrhizal symbioses: from palaeomycology to phylogenomics. New Phytologist 220: 1012-1030.
Stuart EK, Singan V, Amirebrahimi M, Na H, Ng V, Grigoriev I, Martin F, Anderson IC, Plett JM, Plett KL. 2023. Acquisition of host-derived carbon in biomass of the ectomycorrhizal fungus Pisolithus microcarpus is correlated to fungal carbon demand and plant defences. FEMS Microbiology Ecology 99: fiad037.
Svenningsen NB, Watts-Williams SJ, Joner EJ, Battini F, Efthymiou A, Cruz-Paredes C, Nybroe O, Jakobsen I. 2018. Suppression of the activity of arbuscular mycorrhizal fungi by the soil microbiota. ISME Journal 12: 1296-1307.
Tagu D, Bastien C, Faivre-Rampant P, Garbaye J, Vion P, Villar M, Martin F. 2005. Genetic analysis of phenotypic variation for ectomycorrhiza
formation in an interspecific F1 poplar full-sib family. Mycorrhiza 15: 87-91.
Tang N, Lebreton A, Xu W, Dai Y-C, Yu F, Martin FM. 2021. Transcriptome profiling reveals differential gene expression of secreted proteases and highly specific gene repertoires involved in Lactarius-Pinus symbioses. Frontiers in Plant Science 12: 1775.
Tedersoo L, Bahram M, Põlme S, Kõljalg U, Yorou NS, Wijesundera R, Villarreal Ruiz L, Vasco-Palacios AM, Thu PQ, Suija A et al. 2014. Global diversity and geography of soil fungi. Science 346: 6213.
Tedersoo L, May TW, Smith ME. 2010. Ectomycorrhizal lifestyle in fungi: global diversity, distribution, and evolution of phylogenetic lineages. Mycorrhiza 20: 217-263.
Teulet A, Quan C, Evangelisti E, Wanke A, Yang W, Schornack S. 2023. A pathogen effector FOLD diversified in symbiotic fungi. New Phytologist 239: 1127-1139.
Thirkell TJ, Grimmer M, James L, Pastok D, Allary T, Elliott A, Paveley N, Daniell T, Field KJ. 2022. Variation in mycorrhizal growth response among a spring wheat mapping population shows potential to breed for symbiotic benefit. Food and Energy Security 11: e370.
Tisserant E, Malbreil M, Kuo A, Kohler A, Symeonidi A, Balestrini R, Charron P, Duensing N, Frei Dit Frey N, Gianinazzi-Pearson V et al. 2013. Genome of an arbuscular mycorrhizal fungus provides insight into the oldest plant symbiosis. Proceedings of the National Academy of Sciences, USA 110: 20117-20122.
Trivedi P, Batista BD, Bazany KE, Singh BK. 2022. Plant-microbiome interactions under a changing world: responses, consequences and perspectives. New Phytologist 234: 1951-1959.
Tsuzuki S, Handa Y, Takeda N, Kawaguchi M. 2016. Strigolactone-induced putative secreted protein 1 is required for the establishment of symbiosis by the arbuscular mycorrhizal fungus Rhizophagus irregularis. Molecular Plant-Microbe Interactions 29: 277-286.
Vandenkoornhuyse P, Quaiser A, Duhamel M, Le Van A, Dufresne A. 2015. The importance of the microbiome of the plant holobiont. New Phytologist 206: 11961206.
Veneault-Fourrey C, Commun C, Kohler A, Morin E, Balestrini R, Plett J, Danchin E, Coutinho P, Wiebenga A, de Vries RP et al. 2014. Genomic and transcriptomic analysis of Laccaria bicolor CAZome reveals insights into polysaccharides remodeling during symbiosis establishment. Fungal Genetics and Biology 72: 168-181.
Venice F, Ghignone S, Salvioli A, Amselem J, Novero M, Xianan X, Sedzielewska Toro K, Morin E, Lipzen A, Grigoriev IV et al. 2020. At the nexus of three kingdoms: the genome of the mycorrhizal fungus Gigaspora margarita provides insights into plant, endobacterial and fungal interactions. Environmental Microbiology 22: 122-141.
Větrovský T, Morais D, Kohout P, Lepinay C, Algora C, Awokunle Hollá S, Bahnmann BD, Bílohnědá K, Brabcová V, D’Alò F et al. 2020. GlobalFungi, a global database of fungal occurrences from high-throughput-sequencing metabarcoding studies. Scientific Data 13: 228.
Vosátka M, Látr A, Gianinazzi S, Albrechtová J. 2012. Development of arbuscular mycorrhizal biotechnology and industry: current achievements and bottlenecks. Symbiosis 58: 29-37.
Voß S, Betz R, Heidt S, Corradi N, Requena N. 2018. RiCRN1, a crinkler effector from the arbuscular mycorrhizal fungus Rhizophagus irregularis, functions in arbuscule development. Frontiers in Microbiology 9: 2068.
Wang J, Hu A, Meng F, Zhao W, Yang Y, Soininen J, Shen J, Zhou J. 2022. Embracing mountain microbiome and ecosystem functions under global change. New Phytologist 234: 1987-2002.
Wang L, George TS, Feng G. 2023a. Concepts and consequences of the hyphosphere core microbiome for arbuscular mycorrhizal fungal fitness and function. New Phytologist. doi: 10.1111/nph. 19396.
Wang L, Zhang L, George TS, Feng G. 2023b. A core microbiome in the hyphosphere of arbuscular mycorrhizal fungi has functional significance in organic phosphorus mineralization. New Phytologist 238: 859-873.
Wang P, Jiang H, Boeren S, Dings H, Kulikova O, Bisseling T, Limpens EA. 2021. Nuclear-targeted effector of Rhizophagus irregularis interferes with histone 2B mono-ubiquitination to promote arbuscular mycorrhization. New Phytologist 230: 1142-1155.
Venice F, Ghignone S, Salvioli A, Amselem J, Novero M, Xianan X, Sedzielewska Toro K, Morin E, Lipzen A, Grigoriev IV et al. 2020. At the nexus of three kingdoms: the genome of the mycorrhizal fungus Gigaspora margarita provides insights into plant, endobacterial and fungal interactions. Environmental Microbiology 22: 122-141.
Větrovský T, Morais D, Kohout P, Lepinay C, Algora C, Awokunle Hollá S, Bahnmann BD, Bílohnědá K, Brabcová V, D’Alò F et al. 2020. GlobalFungi, a global database of fungal occurrences from high-throughput-sequencing metabarcoding studies. Scientific Data 13: 228.
Vosátka M, Látr A, Gianinazzi S, Albrechtová J. 2012. Development of arbuscular mycorrhizal biotechnology and industry: current achievements and bottlenecks. Symbiosis 58: 29-37.
Voß S, Betz R, Heidt S, Corradi N, Requena N. 2018. RiCRN1, a crinkler effector from the arbuscular mycorrhizal fungus Rhizophagus irregularis, functions in arbuscule development. Frontiers in Microbiology 9: 2068.
Wang J, Hu A, Meng F, Zhao W, Yang Y, Soininen J, Shen J, Zhou J. 2022. Embracing mountain microbiome and ecosystem functions under global change. New Phytologist 234: 1987-2002.
Wang L, George TS, Feng G. 2023a. Concepts and consequences of the hyphosphere core microbiome for arbuscular mycorrhizal fungal fitness and function. New Phytologist. doi: 10.1111/nph. 19396.
Wang L, Zhang L, George TS, Feng G. 2023b. A core microbiome in the hyphosphere of arbuscular mycorrhizal fungi has functional significance in organic phosphorus mineralization. New Phytologist 238: 859-873.
Wang P, Jiang H, Boeren S, Dings H, Kulikova O, Bisseling T, Limpens EA. 2021. Nuclear-targeted effector of Rhizophagus irregularis interferes with histone 2B mono-ubiquitination to promote arbuscular mycorrhization. New Phytologist 230: 1142-1155.
Wolfe BE,Tulloss RR,Pringle A.2012.The irreversible loss of a decomposition pathway marks the single origin of an ectomycorrhizal symbiosis.PLoS ONE 7: e39597.
Wong-Bajracharya J,Singan VR,Monti R,Plett KL,Ng V,Grigoriev IV,Martin FM,Anderson IC,Plett JM.2022.The ectomycorrhizal fungus Pisolithus microcarpus encodes a microRNA involved in cross-kingdom gene silencing during symbiosis.Proceedings of the National Academy of Sciences,USA 119: e2103527119.
Wu B,Nara K,Hogetsu T.2001.Can
Wu G,Miyauchi S,Morin E,Kuo A,Drula E,Varga T,Kohler A,Feng B,Cao Y, Lipzen A et al.2022.Evolutionary innovations through gain and loss of genes in the ectomycorrhizal Boletales.New Phytologist 233:1383-1400.
Wu S,Fu W,Rillig MC,Chen B,Zhu Y-G,Huang L.2023.Soil organic matter dynamics mediated by arbuscular mycorrhizal fungi-an updated conceptual framework.New Phytologist.doi:10.1111/nph. 19178.
Xu X,Thornton PE,Post WM.2013.A global analysis of soil microbial biomass carbon,nitrogen and phosphorus in terrestrial ecosystems.Global Ecology and Biogeography 22:737-749.
Yang FX,Gao J,Wei YL,Ren R,Zhang GQ,Lu CQ,Jin JP,Ai Y,Wang YQ,Chen LJ et al.2021.The genome of Cymbidium sinense revealed the evolution of orchid traits.Plant Biotechnology Journal 19:2501-2516.
Yang Z,Dong H,Zhang S,Jiang J,Zhu H,Yang H,Li L.2023.Isolation and identification of mycorrhizal helper bacteria of Vaccinium uliginosum and their interaction with mycorrhizal fungi.Frontiers in Microbiology 14: 1180319.
Yildirir G,Sperschneider J,Malar CM,Chen ECH,Iwasaki W,Cornell C,Corradi N.2022.Long reads and Hi-C sequencing illuminate the two-compartment genome of the model arbuscular mycorrhizal symbiont Rhizophagus irregularis. New Phytologist 233:1097-1107.
Wong-Bajracharya J,Singan VR,Monti R,Plett KL,Ng V,Grigoriev IV,Martin FM,Anderson IC,Plett JM.2022.The ectomycorrhizal fungus Pisolithus microcarpus encodes a microRNA involved in cross-kingdom gene silencing during symbiosis.Proceedings of the National Academy of Sciences,USA 119: e2103527119.
Wu B,Nara K,Hogetsu T.2001.Can
Wu G,Miyauchi S,Morin E,Kuo A,Drula E,Varga T,Kohler A,Feng B,Cao Y, Lipzen A et al.2022.Evolutionary innovations through gain and loss of genes in the ectomycorrhizal Boletales.New Phytologist 233:1383-1400.
Wu S,Fu W,Rillig MC,Chen B,Zhu Y-G,Huang L.2023.Soil organic matter dynamics mediated by arbuscular mycorrhizal fungi-an updated conceptual framework.New Phytologist.doi:10.1111/nph. 19178.
Xu X,Thornton PE,Post WM.2013.A global analysis of soil microbial biomass carbon,nitrogen and phosphorus in terrestrial ecosystems.Global Ecology and Biogeography 22:737-749.
Yang FX,Gao J,Wei YL,Ren R,Zhang GQ,Lu CQ,Jin JP,Ai Y,Wang YQ,Chen LJ et al.2021.The genome of Cymbidium sinense revealed the evolution of orchid traits.Plant Biotechnology Journal 19:2501-2516.
Yang Z,Dong H,Zhang S,Jiang J,Zhu H,Yang H,Li L.2023.Isolation and identification of mycorrhizal helper bacteria of Vaccinium uliginosum and their interaction with mycorrhizal fungi.Frontiers in Microbiology 14: 1180319.
Yildirir G,Sperschneider J,Malar CM,Chen ECH,Iwasaki W,Cornell C,Corradi N.2022.Long reads and Hi-C sequencing illuminate the two-compartment genome of the model arbuscular mycorrhizal symbiont Rhizophagus irregularis. New Phytologist 233:1097-1107.
Zeng Q,Lebreton A,Auer L,Man X,Jia L,Wang G,Gong S,Lombard V,Buée M, Wu G et al.2023.Stable functional structure despite high taxonomic variability across fungal communities in soils of old-growth montane forests.Microbiome 11: 217.
Zeng T,Holmer R,Hontelez J,Te Lintel-Hekkert B,Marufu L,de Zeeuw T,Wu F,Schijlen E,Bisseling T,Limpens E.2018.Host-and stage-dependent secretome of the arbuscular mycorrhizal fungus Rhizophagus irregularis.The Plant Journal 94:411-425.
Zeng T,Rodriguez-Moreno L,Mansurkhodzaev A,Wang P,van den Berg W, Gasciolli V,Cottaz S,Fort S,Thomma BPHJ,Bono J-J et al.2020.A lysin motif effector subverts chitin-triggered immunity to facilitate arbuscular mycorrhizal symbiosis.New Phytologist 225:448-460.
Zhang C,van der Heijden MG,Dodds BK,Nguyen TB,Spooren J,Held A,Cosme M,Berendsen R.2023.A tripartite bacterial-fungal-plant symbiosis in the mycorrhiza-shaped microbiome drives plant growth and mycorrhization. Microbiome,in press.
Zhang GQ,Liu KW,Li Z,Lohaus R,Hsiao YY,Niu SC,Wang JY,Lin YC,Xu Q, Chen LJ et al.2017.The Apostasia genome and the evolution of orchids.Nature 549:379-383.
Zhang GQ,Xu Q,Bian C,Tsai WC,Yeh CM,Liu KW,Yoshida K, Zhang LS,Chang SB,Chen F et al.2016.The Dendrobium catenatum Lindl.genome sequence provides insights into polysaccharide synthase,floral development and adaptive evolution.Scientific Reports 6: 19029.
Zeng T,Rodriguez-Moreno L,Mansurkhodzaev A,Wang P,van den Berg W, Gasciolli V,Cottaz S,Fort S,Thomma BPHJ,Bono J-J et al.2020.A lysin motif effector subverts chitin-triggered immunity to facilitate arbuscular mycorrhizal symbiosis.New Phytologist 225:448-460.
Zhang C,van der Heijden MG,Dodds BK,Nguyen TB,Spooren J,Held A,Cosme M,Berendsen R.2023.A tripartite bacterial-fungal-plant symbiosis in the mycorrhiza-shaped microbiome drives plant growth and mycorrhization. Microbiome,in press.
Zhang GQ,Liu KW,Li Z,Lohaus R,Hsiao YY,Niu SC,Wang JY,Lin YC,Xu Q, Chen LJ et al.2017.The Apostasia genome and the evolution of orchids.Nature 549:379-383.
Zhang GQ,Xu Q,Bian C,Tsai WC,Yeh CM,Liu KW,Yoshida K, Zhang LS,Chang SB,Chen F et al.2016.The Dendrobium catenatum Lindl.genome sequence provides insights into polysaccharide synthase,floral development and adaptive evolution.Scientific Reports 6: 19029.
Zhang L,Zhou J,George TS,Limpens E,Feng G.2022.Arbuscular mycorrhizal fungi conducting the hyphosphere bacterial orchestra.Trends in Plant Science 27: 402-411.
Zhao H,Zhang R,Wu J,Meng L,Okazaki Y,Hikida H,Ogata H.2023.A 1.5 Mb continuous endogenous viral region in the arbuscular mycorrhizal fungus,Rhizophagus irregularis.Virus Evaluation 9:vead064.
Zhao H,Zhang R,Wu J,Meng L,Okazaki Y,Hikida H,Ogata H.2023.A 1.5 Mb continuous endogenous viral region in the arbuscular mycorrhizal fungus,Rhizophagus irregularis.Virus Evaluation 9:vead064.
Journal: New Phytologist, Volume: 242, Issue: 4
DOI: https://doi.org/10.1111/nph.19541
PMID: https://pubmed.ncbi.nlm.nih.gov/38297461
Publication Date: 2024-01-31
DOI: https://doi.org/10.1111/nph.19541
PMID: https://pubmed.ncbi.nlm.nih.gov/38297461
Publication Date: 2024-01-31
Year: 2024
The mycorrhizal symbiosis: research frontiers in genomics, ecology, and agricultural application
DOI: https://doi.org/10.1111/nph. 19541
Tansley review
The mycorrhizal symbiosis: research frontiers in genomics, ecology, and agricultural application
Authors for correspondence:
Francis Martin
Email: francis.martin@inrae.fr
Marcel G. A. van der Heijden
Email: marcel.vanderheijden@agroscope. admin.ch
Francis Martin
Email: francis.martin@inrae.fr
Marcel G. A. van der Heijden
Email: marcel.vanderheijden@agroscope. admin.ch
Contents
| Summary | 1 | VI. Mycorrhizal symbiosis and plant community dynamics | 11 | ||
| I. Introduction | 2 | VII. Mycorrhiza applications | 12 | ||
| II. Harnessing genomics to enhance our understanding of the development, evolution, and functioning of mycorrhizal symbioses | 3 |
|
12 | ||
| III. Mycorrhizal networks | 8 | Acknowledgements | 15 | ||
| IV. Mapping mycorrhizal diversity | 9 | References | 15 | ||
| V. Mycorrhizal symbiosis in ecosystems | 10 |
New Phytologist (2024)
doi: 10.1111/nph. 19541
doi: 10.1111/nph. 19541
Key words: arbuscular mycorrhizal fungi, biodiversity, ectomycorrhizal fungi, ericoid mycorrhizal fungi, mutualism, orchid mycorrhizal fungi, plant-microbe interactions, population genetics.
Summary
Mycorrhizal symbioses between plants and fungi are vital for the soil structure, nutrient cycling, plant diversity, and ecosystem sustainability. More than 250000 plant species are associated with mycorrhizal fungi. Recent advances in genomics and related approaches have revolutionized our understanding of the biology and ecology of mycorrhizal associations. The genomes of 250+ mycorrhizal fungi have been released and hundreds of genes that play pivotal roles in regulating symbiosis development and metabolism have been characterized. rDNA metabarcoding and metatranscriptomics provide novel insights into the ecological cues driving mycorrhizal communities and functions expressed by these associations, linking genes to ecological traits such as nutrient acquisition and soil organic matter decomposition. Here, we review genomic studies that have revealed genes involved in nutrient uptake and symbiosis development, and discuss adaptations that are fundamental to the evolution of mycorrhizal lifestyles. We also evaluated the ecosystem services provided by mycorrhizal networks and discuss how mycorrhizal symbioses hold promise for sustainable agriculture and forestry by enhancing nutrient acquisition and stress tolerance. Overall, unraveling the intricate dynamics of mycorrhizal symbioses is paramount
for promoting ecological sustainability and addressing current pressing environmental concerns. This review ends with major frontiers for further research.
for promoting ecological sustainability and addressing current pressing environmental concerns. This review ends with major frontiers for further research.
I. Introduction
It is widely acknowledged that mycorrhizal associations are prevalent in almost all terrestrial ecosystems, ranging from deserts and tropical forests to savannahs and arable land (Tedersoo et al., 2014; van der Heijden et al., 2015). Over
phosphorus (P), as well as a wide range of micro-elements (e.g. Fe,
phosphorus (P), as well as a wide range of micro-elements (e.g. Fe,
Mycorrhizal symbioses are of immense significance for biologists and ecologists because of their profound influence on plant and fungal growth, fitness, and diversity (van der Heijden et al., 2015; Brundrett & Tedersoo, 2018). Moreover, these mutualistic relationships play a pivotal role in the N and P cycles within ecosystems (Saito & Ezawa, 2016; Nehls & Plassard, 2018; Pellitier & Zak, 2018). Numerous studies have demonstrated that AMF and ECM mycorrhizal symbionts can drain a substantial proportion of host C compounds, accounting for 5-30% of C flow. Globally, the flux of

Fig. 1 Typical structures of various types of mycorrhizal symbioses. (a) Arbuscular mycorrhizal fungi (AMF) spores of Rhizophagus irregularis containing hundreds of stained nuclei; photo courtesy of Vasilis Kokkoris (Vrije Universeit, Amsterdam). Nuclei are visible due to staining with SYTO 13 Green Fluorescent nucleic acid dye. Each image is the result of
plant-fixed C entering the living mycelium is equivalent to
Over the past two decades, genomics, as a tool for analyzing the genetic information of organisms, has emerged as a fundamental approach for understanding the biology, evolution, and ecology of mycorrhizal symbioses. In addition to providing critical mechanistic insights, genetic information also identifies targets that can be prioritized for the application of mycorrhizal symbioses in agriculture and forestry. In this review, we discuss recent studies that illustrate the impact of genomics and related approaches (e.g. DNA metabarcoding and transcriptomics) on our understanding of mycorrhizal associations. First, we summarize the current knowledge on mycorrhizal fungal genomes and their implications for understanding the transition from saprotrophism to symbiosis across the fungal tree of life. Second, we examine how the genomes of mycorrhizal fungi and their host trees have been utilized to understand the molecular mechanisms underlying the establishment and function of symbiosis. These resources have facilitated the identification of the genes and pathways involved in symbiotic development, nutrient exchange, and stress tolerance. Third, we discuss how high-throughput metabarcoding tools have unraveled the remarkable biodiversity of soil fungi, revealing their distribution from small soil cores to the entire continent. In our discussion, we emphasize the significance of mycorrhizal networks in natural ecosystems and highlight their roles in providing various ecosystem services. We also briefly discuss the potential applications of mycorrhizal symbiosis in sustainable agriculture and ecosystem restoration, underscoring the importance of understanding the functional traits and ecological roles of mycorrhizal fungi. By synthesizing the latest research findings, this review aims to identify knowledge gaps and propose future directions for mycorrhizal research to address the global challenges in agriculture, forestry, and environmental sustainability.
II. Harnessing genomics to enhance our understanding of the development, evolution, and functioning of mycorrhizal symbioses
The Joint Genome Institute (JGI) MycoCosm database currently contains 2701 fungal genomes (as of November 2023), including 220+ mycorrhizal fungi, and the National Center for Biotechnology Information GenBank has annotations for 4770 genomes (as of November 2023), demonstrating the enormous progress made in the field (Martin et al., 2011; Grigoriev et al., 2014). The fungal species that have been sequenced are highly diverse and comprise major clades of Mycota. Among the genomes available in the MycoCosm database, 1693 belonged to Ascomycota, 696 to
Basidiomycota, and 181 to Mucoromycota (including 13 to Glomeromycotina).
The release of the genomes of the ECM fungi Laccaria bicolor and Tuber melanosporum (Martin et al., 2008, 2010) and the AM fungi Rhizophagus irregularis (Tisserant et al., 2013) has opened up new avenues for geneticists to compare the mycorrhizal genomic blueprint with those of saprotrophic and pathogenic species, thereby advancing our understanding of mycorrhizal symbiont evolution. The current selection of sequenced mycorrhizal fungi is based on their ability to establish different mycorrhizal associations, their ecological significance, phylogenetic relationships, and practical considerations. This array of available genome sequences provides opportunities to investigate the evolutionary patterns and functional attributes of mycorrhizal symbioses across dozens of fungal families (Wolfe et al., 2012; Kohler et al., 2015; Peter et al., 2016; Murat et al., 2018; Miyauchi et al., 2020; MarquésGálvez et al., 2021; Lebreton et al., 2022; Looney et al., 2022; Kobayashi et al., 2023; Plett et al., 2023). For several families, such as Boletales, the data highlighted not only which areas of the genomes were similar between the different species but also when their sequences diverged on a scale of millions of years (Miyauchi et al., 2020; Lofgren et al., 2021; Wu et al., 2022). The primary objectives of these genome-based studies were to (1) monitor the various independent lifestyle transitions from saprotrophism to mutualism in fungal lineages (e.g. Agaricales, Boletales, and Russulales), (2) establish connections between genome characteristics and important ecological traits, such as nutrient uptake and soil organic matter (SOM), and (3) identify gene sets involved in the development and functioning of mycorrhizal associations. In the following sections, we provide a summary of the key findings of these studies regarding the major types of mycorrhizal symbioses.
1. Arbuscular mycorrhizal fungi
Arbuscular mycorrhizal fungi (AMF) have captivated evolutionary biologists because of their ancient origins, which date back to
Arbuscular mycorrhizal fungi possess coenocytic hyphae that contain thousands of nuclei and reproduce as large multinucleated spores (Fig. 1). To date, nuclear genome assemblies have been released from representative species of different AMF orders, from Glomerales to Diversisporales, and Archaeosporales. They have unusually large genomes: c. 150 Mb for Rhizophagus species to 784 Mb for Gigaspora species (Tisserant et al., 2013; Kobayashi et al., 2018; Morin et al., 2019; Venice et al., 2020), which are rich in repetitive sequences. The early diverging AMF Paraglomus

Fig. 2 Emergence of key symbiotic genomic traits related to mycorrhizal symbiosis evolution (left) based on molecular clock estimates along the geological time scale/geological epochs from the Cambrian to the Paleogene (middle) with oldest known fossils (right). The orange asterisk represents the formation of the Rhynie chert at 407 Ma . Illustrations of early land plants (liverwort-like plants, Horneophyton), early relatives of conifers (Cordaites), conifers (Telemachus, Pinus), and Angiosperms (Quercus, Medicago) are shown. CAZymes, carbohydrate-active enzymes; GH, glycosyl hydrolase; MiSSPs, mycorrhiza-induced small secreted proteins; PCWDEs, plant cell wall-degrading enzymes. Adapted from Strullu-Derrien et al. (2018).
occultum (Paraglomerales) possesses a much smaller genome size
The notion that AMF are ancient asexual organisms has been challenged by genomic analysis (Ropars et al., 2016). Genomic regions and nuclear organizations associated with sexual reproduction have been identified, including meiosis-specific genes, putative mating-type loci, and homokaryotic-dikaryotic life stages (Kokkoris et al., 2021; Reinhardt et al., 2021; Yildirir et al., 2022).
Various Mucoromycotina fungi can establish AMF-like symbiotic associations with liverworts, hornworts, and other land plants. However, the genomes of these symbionts have not yet been sequenced. These so-called fine-root endophytes can enhance plant growth and nutrition (Hoysted et al., 2023) and are often more abundant in agricultural fields than in native biomes (Albornoz et al., 2022). Niche differentiation between the Mucoromycotinian and Glomeromycotinian AMF has been suggested (Mansfield et al., 2023), pointing to their agronomic and ecological relevance, as they may have complementary functions in ecosystems. Genome sequencing of fine-root endophytes will highlight their evolution, biology, and ecology.
The development of symbiotic structures and metabolic activity in symbiotic tissues requires integrated coordination and communication between symbionts, leading to changes in gene expression and activation of signaling pathways. Conservation of the ‘common symbiotic signaling pathway’ (CSSP) among various angiosperm species is highly noteworthy (Fig. 2; Delaux et al., 2015). This underscores the shared evolutionary origin of this ancient symbiotic signaling pathway. Phylogenomic studies have suggested that certain features of this pathway may have predated the evolution of the first terrestrial plants in the Middle to Late Ordovician and contributed to the establishment of the earliest AM associations with ancestral land plants (Delaux et al., 2015). To date, only a few studies have measured fungal transcriptional responses to different environmental conditions (Grunwald et al., 2009; Hohnjec et al., 2005; Zeng et al., 2018). Serrano et al. (2023) utilized both single-
nucleus and spatial RNA sequencing techniques to explore the transcriptomes of AM symbiosis at the cellular and spatial resolution level. They identified cell type-specific responses to symbiosis, in agreement with previous studies using laser capture microdissection-based transcriptomic analyses (Gaude et al., 2012; Hogekamp & Küster, 2013). These RNA profiling studies have identified a set of robust symbiosis-responsive genes, including many characterized marker genes that are critical to symbiosis, such as those involved in P metabolism (e.g. MtPT4) and those encoding effector-like proteins (Fig. 2). Although the identity and function of most transcripts differentially expressed by plants and fungi during symbiotic interactions have not yet been characterized, these studies provide valuable insights into the molecular mechanisms underlying AM symbiosis (see Lanfranco & Bonfante, 2023 for a review).
nucleus and spatial RNA sequencing techniques to explore the transcriptomes of AM symbiosis at the cellular and spatial resolution level. They identified cell type-specific responses to symbiosis, in agreement with previous studies using laser capture microdissection-based transcriptomic analyses (Gaude et al., 2012; Hogekamp & Küster, 2013). These RNA profiling studies have identified a set of robust symbiosis-responsive genes, including many characterized marker genes that are critical to symbiosis, such as those involved in P metabolism (e.g. MtPT4) and those encoding effector-like proteins (Fig. 2). Although the identity and function of most transcripts differentially expressed by plants and fungi during symbiotic interactions have not yet been characterized, these studies provide valuable insights into the molecular mechanisms underlying AM symbiosis (see Lanfranco & Bonfante, 2023 for a review).
As effector-like small secreted proteins and small RNAs play a key role in plant-pathogen interactions, they have also been investigated in AM associations (Ledford et al., 2023). Aparicio Chacón et al. (2023) identified a set of mycorrhiza-induced secreted effector-like proteins, some of which regulate the expression of host genes. Teulet et al. (2023) found proteins with structural similarity to known Fusarium oxysporum dual domain effectors. Of the hundreds of predicted effector proteins from the AM fungus
There is limited information on how plant or fungal genetic variations affect plant transcriptional responses to fungi (Mateus et al., 2019). The impact of AMF endobacteria and virome and hyphal-associated microbial communities on the regulation of gene expression and physiological capabilities of the association remain unclear and require further investigation (Lanfranco & Bonfante, 2023).
2. Ectomycorrhizal fungi
The transition from saprotrophism to ECM symbiosis is a key evolutionary event that has occurred multiple times during the evolution of Fungi (Tedersoo et al., 2010; Strullu-Derrien et al., 2018; Fig. 2). Although the exact mechanisms driving these transitions remain unclear, comparative genomic and phylogenomic studies have provided hypotheses and patterns for further investigations (Kohler et al., 2015; Miyauchi et al., 2020; Lebreton et al., 2021). The transition to an ECM lifestyle likely occurred through a series of steps, starting with soil/litter saprotrophic fungi colonizing plant roots and gradually forming a mutualistic relationship with the host. Ecological factors such as easy access to soluble carbohydrates may have driven this transition. In nutrient-poor environments, the formation of mutualistic associations with plants allows ECM fungi to access soluble carbohydrates and vitamins, thereby expanding their ecological niche. The ability of ECM fungi to form symbiotic associations with a diverse range of tree species suggests adaptive radiation, specific symbiotic mechanisms, and niche specialization during their evolutionary
history (Martin et al., 2016, 2017; Sato, 2023; Fig. 2). ECM fungal lineages diverging in the Late Cretaceous might have had opportunities to co-evolve with ECM angiosperms (e.g. through the development of host recognition systems; Tang et al., 2021; Lebreton et al., 2022; Looney et al., 2022) and repression of plant defenses (Plett et al., 2011, 2014; Kang et al., 2020), resulting in their expansion into novel niche spaces and subsequent rapid diversification (Martin et al., 2016; Sato, 2023). The repeated evolution of the ECM lifestyle (and the absence of reversion to the ancestral state) and convergent evolution in hundreds of fungal lineages highlight its adaptive significance and underscore the selective pressures driving this evolutionary development (Wolfe et al., 2012; Lebreton et al., 2021).
history (Martin et al., 2016, 2017; Sato, 2023; Fig. 2). ECM fungal lineages diverging in the Late Cretaceous might have had opportunities to co-evolve with ECM angiosperms (e.g. through the development of host recognition systems; Tang et al., 2021; Lebreton et al., 2022; Looney et al., 2022) and repression of plant defenses (Plett et al., 2011, 2014; Kang et al., 2020), resulting in their expansion into novel niche spaces and subsequent rapid diversification (Martin et al., 2016; Sato, 2023). The repeated evolution of the ECM lifestyle (and the absence of reversion to the ancestral state) and convergent evolution in hundreds of fungal lineages highlight its adaptive significance and underscore the selective pressures driving this evolutionary development (Wolfe et al., 2012; Lebreton et al., 2021).
Gene copy numbers of PCWDEs tend to show distinct patterns associated with specific ecological niches in fungi (Floudas et al., 2012, 2020; Kohler et al., 2015) because of their role in the degradation of plant cell wall material (Fig. 2). A drastic contraction in the gene families encoding PCWDEs is a hallmark of ECM and AM fungi (Fig. 3). Most lack ligninolytic class II peroxidases (PODs), invertases, endocellulases, cellobiohydrolases, and cellulose-binding motifs, which restrict their ability to degrade SOM and the plant cell walls of their hosts. As ancestral ECM lineages shifted toward symbiotic relationships, the need for these degradative enzymes diminished, resulting in their gradual loss over evolutionary time (Martin & Selosse, 2008; Wolfe et al., 2012;
Lebreton et al., 2021). This makes mycorrhizal fungi dependent on their host plants for C. Nevertheless, many of the sequenced fungi have maintained a unique array of PCWDEs, including endoglucanases and oxidoreductases/laccases, suggesting that several ECM fungi possess diverse abilities to scavenge plant and microbial detritus (necromass) from soil and litter (Bödeker et al., 2014; VeneaultFourrey et al., 2014; Op De Beeck et al., 2018; Pellitier & Zak, 2018; Nicolás et al., 2019; Floudas et al., 2020; Maillard et al., 2023). Finetuned regulation of these PCWDE genes is necessary to avoid triggering plant defense responses (Miyauchi et al., 2020).
In Mucoromycota, Endogonales is an enigmatic group of fungi that establish ectomycorrhizal symbiosis and biotrophic associations with ferns, lycophytes, hornworts, and liverworts, as well as comprising of putative saprotrophs associated with decayed wood and leaf litter (Desirò et al., 2017). To better understand the evolution of mycorrhizal symbioses and trophic ecology in Endogonales, the genomes of four collections of Endogonaceae, including three ECM collections and one putative saprotroph, were sequenced (Chang et al., 2019). The genomes of ECM isolates exhibit typical characteristics of ECM fungi, including a restricted set of PCWDE genes. Furthermore, dating analysis suggested that Endogonaceae originated in the Permian-Triassic boundary, indicating that the mycorrhizal association between Endogonales and plants may have played a critical role in the colonization of land by plants.

Fig. 3 Differential distribution of the gene copy numbers for CAZyme genes acting on microbial, fungal, and plant polysaccharides, and lignin derivatives in the genomes of 217 species of arbuscular mycorrhizal fungi, ectomycorrhizal fungi, orchid mycorrhizal fungi or ericoid mycorrhizal fungi with contrasting ecologies. Principal coordinate analysis of the total CAZyme gene copy numbers identified in arbuscular mycorrhizal fungi, ectomycorrhizal fungi, ericoid mycorrhizal fungi, and orchid mycorrhizal fungi. Each symbol corresponds to a genome of the mycorrhizal species available in the JGI MycoCosm database (mycocosm.jgi.doe.gov). The taxonomic order of the sequenced species is indicated by different colored symbols as shown in the legend (right panel). Note the divergent distributions in CAZyme gene sets in various mycorrhizal lifestyles with an increasing repertoire of CAZyme from arbuscular mycorrhizal fungi (lower) to ericoid mycorrhizal fungi (higher; from left to right). Principal component analysis (PCA) was performed using the MycoCosm PCA tool (mycocosm.jgi.doe.gov). CAZyme gene repertoires were obtained after semi-manual curation of protein sequences by the CAZy team (www.cazy.org) (Drula et al., 2022).
The field of mycorrhizal genetics has strayed beyond mere genome comparison and gene discovery to the realm of systems biology, exploring the cellular and molecular bases of symbiosis. This evolution was driven by the availability of large-scale transcriptomic datasets (Plett et al., 2014, 2019; Liao et al., 2018; Bouffaud et al., 2020; Ruytinx et al., 2021). They have facilitated the identification of gene networks and hubs involved in key biological processes such as nutrient uptake, metabolism, and signaling in ECM symbiosis. Various ECM symbioses are characterized by the upregulation of genes from overlapping functional categories, such as information storage and processing, transport, and metabolism. However, each association expressed a distinct set of symbiosis-associated genes involved in redox reactions, nutrient transport, and metabolism. A significant number of these genes have orthologs in saprotrophic species, indicating that they are not unique to ECM fungi but are associated with essential core metabolic pathways (Lebreton et al., 2022; Looney et al., 2022; Wu et al., 2022). Because of these alterations in gene expression, ectomycorrhizal interactions lead to changes in the levels of fungal and host plant metabolites, including marked shifts in the metabolism and transport of carbohydrates, amino acids, aromatic acids, organic acids, fatty acids, and water (Hortal et al., 2017; Plett et al., 2021, 2023; Tang et al., 2021; Bogar, 2023; Stuart et al., 2023).
Novel genes specific to the ECM lifestyle, such as those coding for mycorrhiza-induced small secreted proteins (Plett et al., 2011, 2014; Plett & Martin, 2011; Pellegrin et al., 2015) and small RNAs (Wong-Bajracharya et al., 2022), have also emerged, enhancing the symbiotic capabilities of ECM fungi and controlling host plant immunity as well as the ingress of other microbes colonizing the host plant. Several genes that possibly mediate compatibility between plants and fungi have been identified (Lofgren et al., 2021; Tang et al., 2021; Lebreton et al., 2022).
Identifying plant genes that contribute to and are required for ECM symbiosis is a daunting task, and a limited number of genes have been identified and characterized (Tagu et al., 2005; Labbé et al., 2019). Notably, Cope et al. (2019) have demonstrated that the ECM fungus
3. Orchid mycorrhizal fungi
With nearly 30000 species, the orchid family offers valuable resources for studying plant evolution and environmental adaptation, and serves as an economically and culturally important ornamental plant in human society. Orchids appeared to have emerged c. 112 million years ago (Ma; Fig. 2). The subfamilies Orchidoideae and Epidendroideae diverged from each other at the end of the Cretaceous, and the tribes of the upper epidendroids diverged rapidly between 37.9 and 30.8 Ma (Givnish et al., 2015; Zhang et al., 2017). Orchids are renowned for their reliance on mycorrhizal fungi during germination, seedling establishment, and nutrient acquisition, particularly during the early stages of their life cycle. The protocorms of germinating orchids are fully dependent
on colonization by mycorrhizal fungi for their establishment and growth (Perotto & Balestrini, 2023). Thus, orchids cannot survive without mycorrhizal fungi (Rasmussen & Rasmussen, 2014). In recent years, there has been a growing interest in the genomics of orchids and ORM fungi to unravel the genetic mechanisms underlying orchid-fungal interactions and the specificity of these symbiotic relationships (Chen et al., 2022; Song et al., 2022). The genomes of several orchid species (Cai et al., 2015; Zhang et al., 2016, 2017; Yang et al., 2021; Jiang et al., 2022; Li et al., 2022) have been sequenced. Comparison of chromosomescale genome assemblies of two closely related orchid species, the partially mycoheterotrophic
on colonization by mycorrhizal fungi for their establishment and growth (Perotto & Balestrini, 2023). Thus, orchids cannot survive without mycorrhizal fungi (Rasmussen & Rasmussen, 2014). In recent years, there has been a growing interest in the genomics of orchids and ORM fungi to unravel the genetic mechanisms underlying orchid-fungal interactions and the specificity of these symbiotic relationships (Chen et al., 2022; Song et al., 2022). The genomes of several orchid species (Cai et al., 2015; Zhang et al., 2016, 2017; Yang et al., 2021; Jiang et al., 2022; Li et al., 2022) have been sequenced. Comparison of chromosomescale genome assemblies of two closely related orchid species, the partially mycoheterotrophic
Genome sequencing efforts have targeted specific ORM fungi to gain a deeper understanding of their genetic repertoires and symbiotic capabilities. The genomes of Tulasnella calospora, Sebacina vermifera, and Ceratobasidium spp. (Miyauchi et al., 2020) have been published, and a dozen additional ORM fungi in Tulasnellaceae, Serendipitaceae, and Ceratobasidiaceae have been sequenced at the JGI. In contrast to ECM and AMF, ORM fungi possess a large set of PCWDEs that are expressed during their interactions (Balestrini et al., 2014; Chen et al., 2017, 2022; Ghirardo et al., 2020; Fig. 3). Similar to ERM fungi, ORM species often have a higher set of PCWDEs than wood decayers and soil/litter decomposers (Miyauchi et al., 2020), which supports their dual saprotrophic/symbiotic lifestyles. We hypothesized that the size of the PCWDE repertoire is reflective of the age of the mycorrhizal symbiosis along the saprotrophy to symbiosis continuum, with AMF symbiosis emerging in the Early Devonian (
4. Ericoid fungi
Ericoid mycorrhiza fungi form mutualistic symbioses with a wide range of Ericaceae plants, including heathers, blueberries, and rhododendrons, and play crucial roles in nutrient acquisition and stress tolerance in nutrient-poor and acidic soils. Genome sequencing has focused on several key ERM species, including the archetypal ERM fungi Rhizoscyphus ericae and Oidiodendron maius (Kohler et al., 2015; Grelet et al., 2016; Martino et al., 2018). The availability of these genomes has provided insights into the genetic
repertoire and functional attributes of ERM fungi. According to phylogenomic analysis, the most recent common ancestor of the sequenced ERM fungi, including
repertoire and functional attributes of ERM fungi. According to phylogenomic analysis, the most recent common ancestor of the sequenced ERM fungi, including
III. Mycorrhizal networks
Underground mycorrhizal networks are crucial for nutrient acquisition and transport by plants. They consist of interconnected hyphae that extend from the mycorrhizal roots to the surrounding soil, forming extraradical mycelial threads (Fig. 4). In natural soils, numerous intermingled mycorrhizal networks of varying sizes develop as each host plant is colonized by multiple fungal symbionts, up to several dozen per adult tree (Rog et al., 2020). They play a vital role in efficiently prospecting soil, scavenging nutrients, transporting them to the host roots, and contributing to essential ecosystem services, including soil structure enhancement and water uptake (Table 1). Furthermore, mycorrhizal networks play a crucial role in C cycling and storage (Hawkins et al., 2023; Table 1). The establishment and activity of these hyphal networks relies on a continuous supply of plant C compounds.
Within hyphal networks established by mycorrhizal fungi, a specific subset called common mycorrhizal networks (CMNs) is formed when the mycobiont establishes physical connections between the roots of two or more plant species. The transfer of sugars and other nutrients among interconnected plants, often involving saplings, through CMNs has been suggested in several studies (Finlay & Read, 1986; Arnebrant et al. 1993; Simard

Fig. 4 View of the soil mycorrhizal community in a hypothetical forest dominated by oak species associated with different types of mycorrhizal fungi that form separate underground networks (MN) that may possibly be used to transport amino acids, carbohydrates, fatty acids, signaling microRNAs or peptides and hormones. An evergreen oak (Quercus ilex) (1) forms ectomycorrhizal roots and hyphal networks with Xerocomellus chrysentheron (red cracking bolete) (2) and Laccaria amethystina (amethyst deceiver) (3), whereas a sessile oak (Quercus petrea) (4) establishes ectomycorrhiza (ECM) and hyphal networks with Xerocomellus chrysentheron (2) and fly agaric (Amanita muscaria) (5). ECM rootlets are shown as colored dots on root systems. X. chrysentheron hyphal networks link Q. ilex and Q. petrea through a common mycorrhizal network (CMN). The nonphotosynthetic bird’s nest orchid Neottia nidus-avis and its orchid mycorrhiza (ORM) fungal network (6) parasitize the evergreen oak mycorrhizal network. The lesser periwinkle (Vinca minor) is colonized by AMF, forming an independent mycorrhizal network (7). Different colors represent different mycorrhizal fungal networks, with ECM rootlets as dots of the same color. Note that other combinations are possible (e.g. boreal forests with ECM trees often harbor an understory of shrubs (e.g. Vaccinium) that form ericoid mycorrhiza (ERM) associations; Illustration courtesy of Florian Gadenne).
Table 1 Ecosystem services provide by mycorrhizal mycelia and networks.
| Ecosystem service | Ecological importance | Selected references |
| Nutrient uptake and plant growth stimulation | Mycorrhizal fungi can promote plant growth and nutrient uptake. Up to 90% of plant P and N can be acquired by mycorrhizal fungi. Some plants are fully dependent on nutrients acquired by mycorrhizal fungi. | van der Heijden et al. (2008) |
| Belowground C storage and C cycling | Plants allocate 5-30% of their photoassimilates to mycorrhizal symbionts. Up to 13 Gt of
|
van der Heijden et al. (2008); Hawkins et al. (2023) |
| Soil structure formation and aggregation | Fungal hyphae and hyphal exudates enmesh soil particles together into larger aggregates and contribute substantially to the promotion of soil structure. | Jastrow et al. (1998) |
| Water uptake and reduction in nutrient leaching losses | Reduction in nutrient leaching by mycorrhizas. Water uptake by fungal hyphae. Evidence from natural systems is largely missing. | Ahonen-Jonnarth et al. (2003); Cavagnaro et al. (2015); Kakouridis et al. (2022) |
| Promotion of seedling establishment | CMN can quickly colonize the roots of emerging seedlings and can have strong effects on the success of seedling establishment. No effects or even negative effects are also regularly reported. | van der Heijden & Horton (2009) |
| Interplant C and nutrient transfer | High amounts of transfer have been observed in mycoheterotrophic plants and orchid protocorms. The ecological significance of transfer among green plants is debated and needs further study. | Simard et al. (1997); Klein et al. (2016); Henriksson et al. (2023); Karst et al. (2023); Merckx (2023) |
et al., 1997; Wu et al., 2001; Klein et al., 2016; Selosse et al., 2016; Cahanovitc et al., 2022). However, the mechanisms underlying these transfers are complex, and it is challenging to discern the specific role of CMNs vs other pathways such as nutrient and signal movement through the soil solution (Babikova et al., 2013).
Many mycorrhizal fungi are generalists and have a broad host range, and several plants can be interconnected by hyphal networks. For instance, glasshouse experiments have provided evidence that
In ECM fungi, genetic individuals (genets) can simultaneously colonize multiple trees (Beiler et al., 2010). DNA fingerprinting studies have identified large genets in several Boletales species (FioreDonno & Martin, 2001; Hirose et al., 2004; Beiler et al., 2010). Although these studies have provided evidence of very large genetic individuals (
Consequently, the ecological significance of CMNs in nutrient cycling has been the subject of scrutiny and debate (Robinson & Fitter, 1999; Henriksson et al., 2023; Karst et al., 2023; Robinson et al., 2024). Therefore, there is an urgent need to identify the physiological and ecological roles of mycorrhizal networks (including CMNs) in natural ecosystems, and their relative importance in supporting plant growth and seedling establishment. Imaging tools for tracking and probing individual hyphae in intermingled mycelial networks in soils should be developed to test whether nuclei, transcripts, proteins, and metabolites can migrate from one plant individual to another via CMNs. Integrated approaches combining multi-locus genomic sequencing to assess the spatial distribution and size of genotypes, their activity by metatranscriptomics/metaproteomics, and the measurement of metabolic fluxes between different mycorrhizal plants in natural settings are currently being developed and will provide valuable information on the functioning of mycorrhizal networks.
IV. Mapping mycorrhizal diversity
In recent years, there has been strong focus on documenting underground biodiversity and creating global maps of various soil organisms. Efforts have been made to provide global maps of microbial biomass (Xu et al., 2013), earthworms (Phillips et al., 2019), protists (Oliverio et al., 2020), bacteria (Bahram et al., 2018; Delgado-Baquerizo et al., 2018), fungi (Tedersoo et al., 2014; Větrovský et al., 2020), and overall soil biota (DelgadoBaquerizo et al., 2020). In addition, global distribution maps of mycorrhizal plants, AM, and ECM fungi have been published (Öpik et al., 2010; Tedersoo et al., 2014; Davison et al., 2015; Soudzilovskaia et al., 2019; Fig. 5). However, global maps of ORC and ERM fungi, as well as ‘mycorrhizal’ Mucoromycota fine-root endophytes, are still lacking. Furthermore, maps documenting the world-wide distribution of mycorrhiza-associated microbes, such as mycorrhizahelper bacteria, fungi, and protists, are required to fully understand the breadth of mycorrhizal systems (Frey-Klett et al., 2007; Emmett

Fig. 5 Global map of the percentage of total aboveground plant biomass linked to the different mycorrhizal categories. (a) arbuscular mycorrhizal plants; (b) ectomycorrhizal plants; (c) nonmycorrhizal plants. Map resolution was 10 arcmin. Published by Soudzilovskaia et al. (2019).
et al., 2021; Yang et al., 2023; Zhang et al., 2023). Identifying the genetic diversity of these mycorrhiza-associated microorganisms and analyzing their spatiotemporal dynamics is crucial, as emerging studies have suggested their involvement in nutrient uptake facilitation and other services provided by mycorrhizal hyphae (Zhang et al., 2022; Li et al., 2023; Wang et al., 2023a,b). The ability of mycorrhizal fungi to acquire nutrients appears to be influenced by the composition of the bacteria associated with mycorrhizal hyphae. For instance, Zhang et al. (2023) demonstrated that common AM hyphal-associated bacteria promote plant growth and mycorrhizal formation. The concept of ‘mycorrhiza suppressive soil’ has been proposed for soils where microbial communities impede the nutrient uptake facilitated by mycorrhizal hyphae (Svenningsen et al., 2018). Variations in hyphae-associated microbial communities may contribute to the challenges encountered when replicating mycorrhizal experiments. Recent studies have demonstrated that protists feeding on hyphae-associated bacteria can affect mycorrhizal nutrient uptake, thereby highlighting the intricate nature of these multitrophic interactions (Rozmoš et al., 2022). Additionally, it has been suggested that AMF harbor an ancient lineage of mycoviruses (Ezawa et al., 2023), and a recent study suggested a recent and single infection of a large dsDNA viral genome in the genome of an AM fungal strain (Zhao et al., 2023). However, the role of viruses in regulating mycorrhizal function, including their effect on the genetic structure of the mycorrhizal mycobiome, remains poorly understood and requires further investigation. Overall, these studies emphasize the need to consider the ‘holobiont’ concept when trying to understand mycorrhizal functioning (Vandenkoornhuyse et al., 2015; Wang et al., 2023b).
V. Mycorrhizal symbiosis in ecosystems
1. Determining the relative importance of mycorrhizas
Numerous studies have emphasized the significance of mycorrhizal fungi in promoting plant growth and ecosystem functioning. However, there is a lack of research that clearly defines their relative

Fig. 6 Relative importance of mycorrhizal associations for plant productivity and ecosystem performance in different ecosystem types. forests, grasslands, and arable ecosystems. Different ecosystem types are dominated by different mycorrhizal associations. AM, arbuscular mycorrhizas; EM, ectomycorrhizas; NM, nonmycorrhizal.
importance compared with other essential microbial groups, including bacteria, endophytic and saprotrophic fungi, protists, archaea, viruses, and soil micro- and mesofauna, as well as their interactions. The relative importance of mycorrhizas for plant productivity and ecosystem performance depends on the ecosystem type (Fig. 6). We propose that mycorrhizal associations, including mycorrhizal helper bacteria, play a paramount role in forests, followed by grasslands and arable land (Fig. 6). This contention is supported by observations that ECM fungi are responsible for a substantial part of N acquisition in many forests, especially in nutrient-poor soils (Hobbie & Hobbie, 2006). Recent studies have revealed a strong correlation among tree productivity, ECM composition, and beta diversity (Anthony et al., 2022; S. Luo et al., 2023), further reinforcing this observation. However, it is crucial to acknowledge that the significance of mycorrhizal associations depends on nutrient availability, and their importance diminishes in nutrient-rich soils. The effects also depend on plant
species identity, as some plants, such as a wide range of members of Brassicaceae or Proteaceae, do not engage in associations with mycorrhizal fungi.
species identity, as some plants, such as a wide range of members of Brassicaceae or Proteaceae, do not engage in associations with mycorrhizal fungi.
In grasslands, mycorrhizal associations exhibit greater variability. AMF, the dominant mycorrhizal fungi in grasslands, have an important impact on grassland functioning by altering competitive interactions among plant species, leading to either increased or decreased plant diversity (van der Heijden et al., 1998; Hartnett et al., 1999). However, the relative importance of mycorrhizas for biomass production in grasslands is lower, as biomass production often remains similar with or without mycorrhizas, signifying changes in plant species composition (van der Heijden et al., 1998; Hartnett et al., 1999). In arable and highly disturbed systems, mycorrhizal symbiosis is expected to have the lowest relative importance. These systems usually have a lower abundance or an absence of mycorrhizal associations, whereas fungal and bacterial pathogens are more abundant (Labouyrie et al., 2023). Nonmycorrhizal and ruderal plant species often dominate these systems (Read, 1991). Nonetheless, it is crucial to acknowledge that cooccurring plant species within the same ecosystem may exhibit different mycorrhizal responsiveness, and that some plant species and crops in arable fields benefit from mycorrhizal symbiosis. Moreover, exceptions have been observed, such as plant species in extremely weathered and nutrient-poor soils in Australia, which have cluster roots and do not form associations with mycorrhizal fungi (Lambers et al., 2008). Notably, nonmycorrhizal plants are not always restricted to low-nutrient soils; for example, Proteaceae are present throughout most of the Southern Hemisphere. In brief, the relative importance of mycorrhizal symbiosis in ecosystems fluctuates depending on the specific ecosystem type, nutrient availability, and plant species composition. It is important to acknowledge the current geographical bias in mycorrhizal research, which predominantly focuses on temperate and boreal regions and potentially overlooks the greater diversity of viable mycorrhizal and nonmycorrhizal strategies in subtropical and tropical ecosystems. Addressing this gap is essential for obtaining a more comprehensive understanding of mycorrhizal associations world-wide. Additionally, there is a trend toward overinterpreting data, which can hinder progress in certain research fields (Kuyper & Jansa, 2023).
2. Linking genes to ecological traits
The growing availability of mycorrhizal fungal genomes (Lebreton et al., 2021) presents unprecedented opportunities to investigate the functional diversity of soil fungal communities. This can be achieved through expert and accurate annotation of environmental metabarcoding (Anthony et al., 2022), metagenomics (HaghDoust et al., 2023), and metatranscriptomic (Auer et al., 2023; Zeng et al., 2023) datasets. The latter approach has been used to examine gene expression in ECM root tips in natural environments, including the effects of anthropogenic N deposition on ECM tree metabolism (Liao et al., 2018; Law et al., 2022; Rivera Pérez et al., 2022). Anthony et al. (2022) showed that tree growth was linked to the gene copy number of genes involved N uptake and assimilation in associated ECM symbionts. By combining highthroughput metabarcoding of fungal species residing in a specific
experimental forest site with the sequencing of soil eukaryotic RNA extracts, the precise gene expression of each mycelial network extending beneath a tree species can be evaluated (Auer et al., 2023; Zeng et al., 2023). The use of metatranscriptomics will also help decipher the relative importance of each fungal species within the different soil biota in measuring genes involved in plant nutrition, C cycling, disease resistance, and stress adaptation.
experimental forest site with the sequencing of soil eukaryotic RNA extracts, the precise gene expression of each mycelial network extending beneath a tree species can be evaluated (Auer et al., 2023; Zeng et al., 2023). The use of metatranscriptomics will also help decipher the relative importance of each fungal species within the different soil biota in measuring genes involved in plant nutrition, C cycling, disease resistance, and stress adaptation.
VI. Mycorrhizal symbiosis and plant community dynamics
It is increasingly recognized that soil microbes, including mycorrhizal fungi, can mediate plant interactions and thus may contribute to biodiversity-productivity relationships, shaping plant communities (Klironomos, 2002; Van der Heijden et al., 2008; Schnitzer et al., 2011). Manipulative experiments targeting the abundance and presence of AMF and ECM have consistently demonstrated their profound effects on plant diversity and community structure (Liang et al., 2019). Notably, the paucity of studies that manipulate other types of mycorrhizal symbioses, such as ERM and ORM, to investigate their effects on plant growth and establishment warrants further investigation and research attention. It is worth noting that the majority of studies in this field are relatively short-term, typically spanning
An increasing body of evidence from global surveys has highlighted the significance of mycorrhizal associations in shaping plant community structure, diversity, and productivity. Large-scale forest inventories have provided valuable insights into the role of mycorrhizal types in structuring forest ecosystems and tree diversity. Carteron et al. (2022), for instance, conducted an analysis of 82000 forest plots across the United States and found that forests dominated by ECM fungi or AMF tend to exhibit lower tree diversity. By contrast, forests with a mixture of mycorrhizal strategies supported a higher number of tree species. More recently, Y. H. Luo et al. (2023), using the same database, demonstrated that mixed mycorrhizal plots displayed greater productivity than plots in which either AMF- or ECM-associated tree species were dominant. These findings suggest that the coexistence of both mycorrhizal types can complement each other and enhance ecosystem function. Additionally, the positive effects of mixed mycorrhizal strategies on forest productivity were more pronounced in plots with lower tree species richness (Y. H. Luo et al., 2023). However, the mycorrhizal type of the dominant plant species also plays a crucial role in facilitating or suppressing subordinate or understory species. For example, a study conducted in the Great Smoky Mountains of the United States found a positive relationship between herb species richness and the number of AM tree species (Newman & Reddell, 1988). Deng et al. (2023) conducted a large-scale tree biodiversity experiment in China and observed that the relationship between tree species richness and productivity is dependent on the mycorrhizal type. Specifically,
a positive relationship was observed between the richness of AM trees and productivity, whereas no such relationship was observed for ECM trees. By contrast, S. Luo et al. (2023) showed a positive relationship between mycorrhizal diversity and aboveground productivity in a subtropical evergreen broad-leaved forest ecosystem. Metatranscriptomics and metaproteomics approaches can help identify the mechanisms and key gene networks involved in mediating the observed biodiversity-ecosystem functioning relationships. Moreover, the presence or absence of mycorrhizal symbionts can facilitate or impair the establishment of invasive species (e.g. Pringle et al., 2009; Dickie et al., 2010).
a positive relationship was observed between the richness of AM trees and productivity, whereas no such relationship was observed for ECM trees. By contrast, S. Luo et al. (2023) showed a positive relationship between mycorrhizal diversity and aboveground productivity in a subtropical evergreen broad-leaved forest ecosystem. Metatranscriptomics and metaproteomics approaches can help identify the mechanisms and key gene networks involved in mediating the observed biodiversity-ecosystem functioning relationships. Moreover, the presence or absence of mycorrhizal symbionts can facilitate or impair the establishment of invasive species (e.g. Pringle et al., 2009; Dickie et al., 2010).
VII. Mycorrhiza applications
There is considerable interest in enhancing agricultural sustainability and utilizing biological approaches to promote crop plant yield, as well as facilitate ecosystem restoration and sustainability. Mycorrhizal fungi have important potential as they are known to promote plant growth and provide protection against biotic and abiotic stresses in agriculture and forestry. A recent meta-analysis by Averill et al. (2022) indicated that restoring the native soil microbiome could lead to an average acceleration of
Many studies evaluating plant growth responses to mycorrhizal inoculation have been conducted under highly controlled conditions in glasshouses, often using sterilized and nutrient-poor soils. Comparing the results of such studies to field conditions can be challenging. To make field inoculation practical and widely adopted, it is essential to conduct field inoculation trials and employ appropriate inoculation techniques that can easily be implemented by farmers or foresters. Mycorrhizal fungi can be coated onto seeds, applied as dressing or as ‘fertilizer’ grains, or sown together with (or below) seeds (Vosátka et al., 2012; Oliveira et al., 2019; Rocha et al., 2019). The addition of natural soils containing indigenous populations of mycorrhizal fungi (and other microbes) to neighboring sites or tree nurseries has also been advocated, as these inoculants may be better adapted to local conditions. Furthermore, field inoculation can change native AMF communities (Basiru & Hijri, 2022), and there are concerns regarding the potential unintended consequences of using a
nonnative inoculum, which may pose a threat to soil and plant biodiversity and ecosystem functioning (Hart et al., 2018). Before introducing an inoculant to the market, it is crucial to adhere to a range of basic quality criteria in order to assess inoculum viability and efficacy under controlled conditions (Salomon et al., 2022). Additionally, tools need to be developed to enable farmers or foresters to predict under which conditions (crop type, crop genotype, soil type, nutrient availability, and weather conditions) inoculation is beneficial and economically viable. For example, a recent field inoculation study in 54 well-fertilized arable soils in Switzerland found that the success rate of AMF inoculation was best predicted by the presence of fungal pathogen genes in the soil, suggesting that inoculation can also help reduce biotic stress (Lutz et al., 2023). Overall, mycorrhizal fungi have a great potential as biological agents for sustainable production. However, it is essential to elucidate the specific conditions under which inoculation is most effective. A combination of high-throughput rDNA metabarcoding, RNA sequencing, and metaproteomics would be a powerful approach to assess mycorrhizal inoculation success and to reveal whether field inoculation enhances specific ecosystem functions. For instance, the level of transcripts (or proteins) coding for nutrient transporters (or other functional traits that can be traced back to mycorrhizal fungi) can be linked to particular ecosystem functions, such as plant yield, nutrient content, SOM decomposition, or disease/stress resistance. Multiomics approaches can also help to reveal the relative importance of mycorrhizal traits in comparison with other traits (e.g. nutrient uptake by plant roots or organic matter decomposition by nonmycorrhizal soil biota). Such studies will rely on a combination of traditional ecology/agronomy approaches (e.g. assessing plant yield, decomposition with litter or tea bags, disease suppression assays, hyphal compartments only accessible to fungal hyphae, or the inclusion of organic nutrient patches) and environmental genomics approaches. For instance, Liu et al. (2023) revealed that the application of pesticides is linked to genes responsible for N fixation, iron transport, and P metabolism, enabling the elucidation of how environmental stressors can influence soil functioning. However, the analytical methodology required to integrate ecology/agronomy approaches with genomics, transcriptomics, proteomics, metabolomics, and phenomics data remains nascent. Owing to their nature, -omics data are intrinsically highly variable and noisy, leading to several issues when trying to compare or reproduce them in natural settings. Integrating multi-omic data will likely rely on co-expression or co-abundance network approaches, such as those developed in random forest-based methods (Huynh-Thu et al., 2010). In this approach, multiple types of -omics and ecological data can be integrated by identifying proteins and transcripts that shift their abundance in a coordinated manner across a range of experimental conditions (e.g. crop yield) or samples.
nonnative inoculum, which may pose a threat to soil and plant biodiversity and ecosystem functioning (Hart et al., 2018). Before introducing an inoculant to the market, it is crucial to adhere to a range of basic quality criteria in order to assess inoculum viability and efficacy under controlled conditions (Salomon et al., 2022). Additionally, tools need to be developed to enable farmers or foresters to predict under which conditions (crop type, crop genotype, soil type, nutrient availability, and weather conditions) inoculation is beneficial and economically viable. For example, a recent field inoculation study in 54 well-fertilized arable soils in Switzerland found that the success rate of AMF inoculation was best predicted by the presence of fungal pathogen genes in the soil, suggesting that inoculation can also help reduce biotic stress (Lutz et al., 2023). Overall, mycorrhizal fungi have a great potential as biological agents for sustainable production. However, it is essential to elucidate the specific conditions under which inoculation is most effective. A combination of high-throughput rDNA metabarcoding, RNA sequencing, and metaproteomics would be a powerful approach to assess mycorrhizal inoculation success and to reveal whether field inoculation enhances specific ecosystem functions. For instance, the level of transcripts (or proteins) coding for nutrient transporters (or other functional traits that can be traced back to mycorrhizal fungi) can be linked to particular ecosystem functions, such as plant yield, nutrient content, SOM decomposition, or disease/stress resistance. Multiomics approaches can also help to reveal the relative importance of mycorrhizal traits in comparison with other traits (e.g. nutrient uptake by plant roots or organic matter decomposition by nonmycorrhizal soil biota). Such studies will rely on a combination of traditional ecology/agronomy approaches (e.g. assessing plant yield, decomposition with litter or tea bags, disease suppression assays, hyphal compartments only accessible to fungal hyphae, or the inclusion of organic nutrient patches) and environmental genomics approaches. For instance, Liu et al. (2023) revealed that the application of pesticides is linked to genes responsible for N fixation, iron transport, and P metabolism, enabling the elucidation of how environmental stressors can influence soil functioning. However, the analytical methodology required to integrate ecology/agronomy approaches with genomics, transcriptomics, proteomics, metabolomics, and phenomics data remains nascent. Owing to their nature, -omics data are intrinsically highly variable and noisy, leading to several issues when trying to compare or reproduce them in natural settings. Integrating multi-omic data will likely rely on co-expression or co-abundance network approaches, such as those developed in random forest-based methods (Huynh-Thu et al., 2010). In this approach, multiple types of -omics and ecological data can be integrated by identifying proteins and transcripts that shift their abundance in a coordinated manner across a range of experimental conditions (e.g. crop yield) or samples.
VIII. Future directions
By applying genomics and other -omics approaches to the study of mycorrhizal symbioses, we gained deeper insight into the molecular, physiological, and ecological processes underlying these
mutualistic associations. This knowledge should be translated into practical applications that promote ecosystem functioning and conservation, contribute to the overall mitigation of climate change, and ensure sustainable arable land. Numerous long-term challenges remain in leveraging genomics and other-omics tools to deepen our understanding of mycorrhizal symbioses, including their evolution, development, function, and ecology (Table 1).
mutualistic associations. This knowledge should be translated into practical applications that promote ecosystem functioning and conservation, contribute to the overall mitigation of climate change, and ensure sustainable arable land. Numerous long-term challenges remain in leveraging genomics and other-omics tools to deepen our understanding of mycorrhizal symbioses, including their evolution, development, function, and ecology (Table 1).
1. Expanding the repertoire of mycorrhizal genomes
The current collection of sequenced mycorrhizal genomes represents only a fraction (c.
2. Advancing our understanding of basic biological processes
Utilizing approaches such as multiplexed single-cell and 3D spatial gene expression analysis of mycorrhizal roots, transcriptomic-based co-expression analysis, proteomics, and protein-protein interaction studies can aid in linking genes to established biological processes and potentially predict evolutionary relationships as well as ecological adaptation (see Box 1). Approaches involving highthroughput protein-folding computations (e.g. AlphaFold; Jumper et al., 2021) can help identify protein structures encoded by symbiosis-regulated genes with unknown functions, such as effector-like secreted proteins (Teulet et al., 2023). Understanding the origin of these effector proteins is crucial, whether they evolved from saprotrophic ancestors or emerged anew to facilitate colonization within plant tissues. Addressing the signaling pathways and specific molecules involved in symbiotic development and the intricate molecular crosstalk between mycorrhizal partners are needed. Currently, the master genes that trigger the development of ectomycorrhizal symbiosis in both fungal and plant partners (if any) are unknown. Furthermore, it is important to investigate the factors underlying the varying host ranges of different mycorrhizal species (Lebreton et al., 2021; Tang et al., 2021). Why certain mycorrhizal fungal species can colonize a wide range of hosts, whereas others exhibit more restricted preferences, remains an intriguing aspect that requires further exploration.
3. Unearthing mycorrhizal diversity
Although progress has been made, especially in temperate and boreal ecosystems (Peay et al., 2016), there are still vast regions of
Box 1. Topical questions in the genomics of mycorrhizal associations.
What are the genomic mechanisms underlying the specificity and recognition of plant hosts and mycorrhizal fungi and how do these mechanisms contribute to the establishment and maintenance of symbiosis?
How do idiosyncrasies in the gene repertoire of mycorrhizal fungi influence their ability to interact with different plant species, and what are the implications for plant diversity and ecosystem functioning?
How does genomics inform our understanding of the complex regulatory networks involved in mycorrhizal symbiosis, including the genes and pathways responsible for signaling, nutrient transport, and establishment of mutualism?
What is the genomic basis of the response of mycorrhizal fungi to environmental stressors, such as changes in soil pH , drought, pesticides, plastics, nutrient availability, and climate fluctuations, and how does this affect their symbiotic interactions with plants?
How can genomics contribute to the understanding of the evolution of mycorrhizal symbiosis and the genetic changes that have occurred over time, leading to a diverse range of mycorrhizal associations observed in nature?
What are genomics-based strategies for engineering or enhancing mycorrhizal symbiosis in crops to improve nutrient uptake efficiency, stress tolerance, and overall plant performance?
What is the role of epigenetic modifications in mediating plantmycorrhizal fungus interactions, and how do epigenetic changes influence the outcomes of the symbiotic relationship?
Can comparative genomics shed light on the genetic basis of functional differences among various mycorrhizal fungal species and how can this knowledge be applied to optimize fungal partners for specific agricultural or ecological goals?
How can genomics, metatranscriptomics, and metaproteomics contribute to our understanding of the role of mycorrhizal fungi in carbon cycling, soil health, and overall ecosystem services and how might this knowledge inform environmental management strategies?
our planet that remain largely underexplored. A consensus has emerged that most of this diversity, both known and unknown, resides in the tropics. The mycobiome diversity in deserts and polar regions remains uncharacterized, but their populations are changing rapidly, driven by anthropogenic climate change (Baldrian et al., 2023). We urge researchers to examine the biogeography of various mycorrhizal associations within these biomes. Other threats to soil habitats, such as microplastics, can also alter or even create new habitats for mycorrhizal fungi (Leifheit et al., 2021). Owing to initiatives such as the Society for the Protection of Underground Networks, a better understanding of these mycorrhizal mycobiomes would allow us to improve predictions of anthropogenic impacts on microbiome composition, resilience, evolution, and productivity (Averill et al., 2022; Baldrian et al., 2023) (Box 1). The global inventory of mycorrhizal fungi, such as GlobalFungi (Větrovský et al., 2020), must be accompanied by an atlas designed to explore microbial functions in space and time (to be described later). Given the increasing number of threats to the soil environment, there is an urgent need to characterize healthy soil microbiomes in ecosystems that are currently unaffected by environmental threats (if any). This provides a baseline for understanding how these microbial
communities might respond to global changes. It would also help to identify tipping points that could permanently change the composition and function of the soil microbiome. For instance, large-scale inventories of European forests have shown that N deposition causes tipping points in mycorrhizal composition and functioning (van der Linde et al., 2018). Although hundreds of studies have described the composition of the mycorrhizal mycobiome in various ecosystems, little is known about the connectivity between different components and mycorrhizal community shifts during succession and along the gradient of grassland-, shrub-, and tree-dominated zones.
What are the genomic mechanisms underlying the specificity and recognition of plant hosts and mycorrhizal fungi and how do these mechanisms contribute to the establishment and maintenance of symbiosis?
How do idiosyncrasies in the gene repertoire of mycorrhizal fungi influence their ability to interact with different plant species, and what are the implications for plant diversity and ecosystem functioning?
How does genomics inform our understanding of the complex regulatory networks involved in mycorrhizal symbiosis, including the genes and pathways responsible for signaling, nutrient transport, and establishment of mutualism?
What is the genomic basis of the response of mycorrhizal fungi to environmental stressors, such as changes in soil pH , drought, pesticides, plastics, nutrient availability, and climate fluctuations, and how does this affect their symbiotic interactions with plants?
How can genomics contribute to the understanding of the evolution of mycorrhizal symbiosis and the genetic changes that have occurred over time, leading to a diverse range of mycorrhizal associations observed in nature?
What are genomics-based strategies for engineering or enhancing mycorrhizal symbiosis in crops to improve nutrient uptake efficiency, stress tolerance, and overall plant performance?
What is the role of epigenetic modifications in mediating plantmycorrhizal fungus interactions, and how do epigenetic changes influence the outcomes of the symbiotic relationship?
Can comparative genomics shed light on the genetic basis of functional differences among various mycorrhizal fungal species and how can this knowledge be applied to optimize fungal partners for specific agricultural or ecological goals?
How can genomics, metatranscriptomics, and metaproteomics contribute to our understanding of the role of mycorrhizal fungi in carbon cycling, soil health, and overall ecosystem services and how might this knowledge inform environmental management strategies?
our planet that remain largely underexplored. A consensus has emerged that most of this diversity, both known and unknown, resides in the tropics. The mycobiome diversity in deserts and polar regions remains uncharacterized, but their populations are changing rapidly, driven by anthropogenic climate change (Baldrian et al., 2023). We urge researchers to examine the biogeography of various mycorrhizal associations within these biomes. Other threats to soil habitats, such as microplastics, can also alter or even create new habitats for mycorrhizal fungi (Leifheit et al., 2021). Owing to initiatives such as the Society for the Protection of Underground Networks, a better understanding of these mycorrhizal mycobiomes would allow us to improve predictions of anthropogenic impacts on microbiome composition, resilience, evolution, and productivity (Averill et al., 2022; Baldrian et al., 2023) (Box 1). The global inventory of mycorrhizal fungi, such as GlobalFungi (Větrovský et al., 2020), must be accompanied by an atlas designed to explore microbial functions in space and time (to be described later). Given the increasing number of threats to the soil environment, there is an urgent need to characterize healthy soil microbiomes in ecosystems that are currently unaffected by environmental threats (if any). This provides a baseline for understanding how these microbial
communities might respond to global changes. It would also help to identify tipping points that could permanently change the composition and function of the soil microbiome. For instance, large-scale inventories of European forests have shown that N deposition causes tipping points in mycorrhizal composition and functioning (van der Linde et al., 2018). Although hundreds of studies have described the composition of the mycorrhizal mycobiome in various ecosystems, little is known about the connectivity between different components and mycorrhizal community shifts during succession and along the gradient of grassland-, shrub-, and tree-dominated zones.
4. Environmental genomics
Understanding the processes driving variations in natural microbial communities across space and time is a critical challenge for ecologists. With an increasing number of available fungal genomes from ecologically relevant species (see Section II), it is now possible to map anonymous DNA and RNA reads from soil and mycorrhizal roots to identify mycorrhizal species and their specific gene expression patterns, including targeting the mycorrhizal genes responsible for nutrient uptake, symbiosis development and signaling, and SOM decomposition. However, metagenomic approaches are still challenging for assembling entire fungal genomes from soil samples because they usually represent a very low proportion of soil DNA. Higher-throughput sequencing can help address the depth-of-coverage issue, but this can lead to other problems related to the high computational demands for large datasets. By contrast, using RNA profiling of soil hyphae and mycorrhizal root tips, we can now assess whether multiple symbiotic species can coexist on (or beneath) the same host tree by utilizing the same resources and performing identical ecological functions (i.e. exhibiting high functional similarity) or by utilizing different resources and providing distinct functions (i.e. exhibiting limited functional similarity; see Section V.2). Metatranscriptomics of soil fungal communities (Auer et al., 2023; Zeng et al., 2023) can be used to better understand the role of mycorrhizal networks, including CMN, because they can help identify whether different tree individuals within a plot are truly colonized by the same fungus, and whether the same sets of genes are expressed in different tree individuals. However, several challenges limit the widespread application of metatranscriptomics, including the lower stability of RNA compared with that of DNA, greater difficulty in extracting RNA, and difficulty in obtaining informative messenger RNA transcripts that provide insights into its functional role (Auer et al., 2023). It is difficult to ascertain whether nuclei, transcripts, proteins, and metabolites can translocate from one plant to another through CMNs in natural settings. The absence of imaging tools that can trace and investigate individual hyphae within intermingled mycelial networks in soil presents a significant obstacle in understanding this phenomenon. Nevertheless, ongoing research is developing integrated methods that combine rDNA metabarcoding to evaluate the distribution of operational taxonomic units, estimate fungal genotype size through multi-locus eDNA population genomics, metatranscriptomics, metaproteomics, and measurement of metabolic fluxes between
various components of forest or grassland plots. These integrated approaches will offer valuable insights into the functioning of fungal/plant communities. The adoption of this genome-toecosystem strategy will be crucial in unraveling the contributions of fungal guilds that thrive in the soil and roots within intricate terrestrial ecosystems (Box 1).
various components of forest or grassland plots. These integrated approaches will offer valuable insights into the functioning of fungal/plant communities. The adoption of this genome-toecosystem strategy will be crucial in unraveling the contributions of fungal guilds that thrive in the soil and roots within intricate terrestrial ecosystems (Box 1).
5. Promoting sustainable agriculture and forestry
Gaining insights into the genetic basis of beneficial traits in mycorrhizal fungal populations and communities is crucial for the development of strategies aimed at optimizing their use as biofertilizers or biocontrol agents, thereby reducing reliance on chemical inputs and promoting sustainable land management practices. To achieve sustainable agriculture and forestry, it is imperative to harness the potential of mycorrhizal symbioses and exploit the functional traits of the mycorrhizal inoculants. To successfully manage mycorrhizal fungal communities and promote ecologically sustainable agriculture or restoration initiatives, it is crucial to identify the range of fungal taxa present and the factors that influence their diversity and distribution at both regional and continental levels. DNA metabarcoding surveys can foster applications and predict under which conditions inoculation is beneficial for farmers and land managers (e.g. low abundance or diversity of mycorrhizal fungi) (Box 1). These genotyping approaches can also identify whether specific crop microbiomes are characteristic of well-performing (high-yield and high nutrient use efficiency) systems to distinguish them (functionally) from poorly performing (low-yield and large nutrient losses) systems. Moreover, such tools can be used to identify how mycorrhizal inoculation can alleviate biotic stress (e.g. high pathogen abundance). Furthermore, metatranscriptomics, metagenomics, and stable isotope probing may provide functional insights into and identify the role of mycorrhizal helper bacteria and other microbial consortia that contribute to plant growth. Mycorrhizal fungi can also facilitate the establishment and growth of plants in degraded habitats. However, it is necessary to investigate the specific conditions under which the application of mycorrhizal fungi is beneficial, the compatible host plants (or even genotypes), and whether specific taxa of mycorrhizal fungi can be applied alone or in microbial consortia, along with other microorganisms such as mycorrhiza-helper bacteria and protists, or using a complex field inoculum (Allsup et al., 2023). Similar to the human microbiome, there is an important potential for microbiome engineering (Silverstein et al., 2023). Many commercially available mycorrhizal products are either nonviable or ineffective (Salomon et al., 2022), and it is often overlooked whether the fungi present in these commercial inoculants have been successfully established in the field. Therefore, it is essential to DNA barcode the inoculum, trace the establishment and survival of mycorrhizal fungal isolate(s) in the field, and determine the economic and ecological feasibility of their application.
6. Promoting
Mycorrhizal symbioses have the potential to contribute to climate change mitigation by promoting
(Hawkins et al., 2023). Mycorrhizal networks contribute to c. 20
(Hawkins et al., 2023). Mycorrhizal networks contribute to c. 20
IX. Conclusion
In conclusion, the integration of genomics and other -omics tools hold great promise for enhancing the utilization of mycorrhizal associations in sustainable ecosystem management. By unraveling the intricate dynamics of these symbiotic interactions, valuable insights that are crucial for promoting ecological sustainability and addressing pressing environmental concerns can be gained. This holistic approach, combining advanced genomic techniques with comprehensive global molecular diversity surveys, will undoubtedly contribute to the effective and responsible use of mycorrhizal fungi in ecosystem management in the future.
Acknowledgements
This article is based on lectures given by the authors at the 2021 International Conference on Mycorrhizas in Beijing. We apologize in advance to all investigators whose research could not be appropriately cited owing to space limitations. We would like to thank the editor Ian Dickie, Ido Rog, Roger Finlay, and two reviewers for their helpful comments. Special thanks to Marc Buée, Maira de Freitas Pereira, Rosmarie Honegger, Vasilis Kokkoris, Jen McGaley, Uta Paszkowski, Silvia Perotto, and Janis Rogenmoser for sharing photographic material and Florian Gadenne for illustrating the mycorrhizal
networks (Fig. 4). FMM was funded by the Laboratory of Excellence ARBRE (ANR-12-LABX-ARBRE-01), the U.S. Department of Energy JGI (Office of Science of the US Department of Energy under Contract No. DE-AC02-05CH11231), the Beijing Advanced Innovation Center for Tree Breeding by Molecular Design (Beijing Forestry University), and Huazhong Agricultural University. MvdH acknowledges funding from the Swiss National Science Foundation (grants 310030_188799 and 40IN40_215832) and the Gebert Rüf Foundation.
networks (Fig. 4). FMM was funded by the Laboratory of Excellence ARBRE (ANR-12-LABX-ARBRE-01), the U.S. Department of Energy JGI (Office of Science of the US Department of Energy under Contract No. DE-AC02-05CH11231), the Beijing Advanced Innovation Center for Tree Breeding by Molecular Design (Beijing Forestry University), and Huazhong Agricultural University. MvdH acknowledges funding from the Swiss National Science Foundation (grants 310030_188799 and 40IN40_215832) and the Gebert Rüf Foundation.
Competing interests
None declared.
ORCID
Marcel G. A. van der Heijden (D) https://orcid.org/0000-0001-7040-1924
Francis M. Martin (D) https://orcid.org/0000-0002-4737-3715
Francis M. Martin (D) https://orcid.org/0000-0002-4737-3715
References
Ahonen-Jonnarth U, Göransson A, Finlay RD. 2003. Growth and nutrient uptake of ectomycorrhizal Pinus sylvestris seedlings treated with elevated Al concentrations. Tree Physiology 23: 157-167.
Albornoz FE, Ryan MH, Bending GD, Hilton S, Dickie IA, Gleeson DB, Standish RJ. 2022. Agricultural land-use favours Mucoromycotinian, but not Glomeromycotinian, arbuscular mycorrhizal fungi across ten biomes. New Phytologist 233: 1369-1382.
Allsup CM, George I, Lankau RA. 2023. Shifting microbial communities can enhance tree tolerance to changing climates. Science 380: 835-840.
An GH, Kobayashi S, Enoki H, Sonobe K, Muraki M, Karasawa T, Ezawa T. 2010. How does arbuscular mycorrhizal colonization vary with host plant genotype? An example based on maize (Zea mays) germplasms. Plant and Soil 327: 441-453.
Angulo V, Beriot N, Garcia-Hernandez E, Li E, Masteling R, Lau JA. 2022. Plantmicrobe eco-evolutionary dynamics in a changing world. New Phytologist 234: 1919-1928.
Anthony MA, Crowther TW, Van Der Linde S, Suz LM, Bidartondo MI, Cox F, Schaub M, Rautio P, Ferretti M, Vesterdal L et al. 2022. Forest tree growth is linked to mycorrhizal fungal composition and function across Europe. ISME Journal 16: 1327-1336.
Aparicio Chacón MV, Van Dingenen J, Goormachtig S. 2023. Characterization of arbuscular mycorrhizal effector proteins. International Journal of Molecular Sciences 24: 9125.
Arnebrant K, Ek H, Finlay RD, Söderström B. 1993. Nitrogen translocation between Alnus glutinosa (L.) Gaertn. seedlings inoculated with Frankia sp. and Pinus contorta Doug, ex Loud seedlings connected by a common ectomycorrhizal mycelium. New Phytologist 124: 231-242.
Auer L, Buée M, Fauchery L, Lombard V, Barry K, Clum A, Copeland A, Daum C, Foster B, LaButti K et al. 2023. Metatranscriptomics sheds light on the links between the functional traits of fungal guilds and ecological processes in forest soil ecosystems. New Phytologist. doi: 10.1111/nph.19471.
Augé RM. 2001. Water relations, drought and vesicular-arbuscular mycorrhizal symbiosis. Mycorrhiza 11: 3-42.
Averill C, Anthony MA, Baldrian P, Finkbeiner F, van den Hoogen J, Kiers T, Kohout P, Hirt E, Smith GR, Crowther TW. 2022. Defending Earth’s terrestrial microbiome. Nature Microbiology 7: 1717-1725.
Babikova Z, Gilbert L, Bruce TJ, Birkett M, Caulfield JC, Woodcock C, Pickett JA, Johnson D. 2013. Underground signals carried through common mycelial networks warn neighbouring plants of aphid attack. Ecology Letters 16: 835-843.
Albornoz FE, Ryan MH, Bending GD, Hilton S, Dickie IA, Gleeson DB, Standish RJ. 2022. Agricultural land-use favours Mucoromycotinian, but not Glomeromycotinian, arbuscular mycorrhizal fungi across ten biomes. New Phytologist 233: 1369-1382.
Allsup CM, George I, Lankau RA. 2023. Shifting microbial communities can enhance tree tolerance to changing climates. Science 380: 835-840.
An GH, Kobayashi S, Enoki H, Sonobe K, Muraki M, Karasawa T, Ezawa T. 2010. How does arbuscular mycorrhizal colonization vary with host plant genotype? An example based on maize (Zea mays) germplasms. Plant and Soil 327: 441-453.
Angulo V, Beriot N, Garcia-Hernandez E, Li E, Masteling R, Lau JA. 2022. Plantmicrobe eco-evolutionary dynamics in a changing world. New Phytologist 234: 1919-1928.
Anthony MA, Crowther TW, Van Der Linde S, Suz LM, Bidartondo MI, Cox F, Schaub M, Rautio P, Ferretti M, Vesterdal L et al. 2022. Forest tree growth is linked to mycorrhizal fungal composition and function across Europe. ISME Journal 16: 1327-1336.
Aparicio Chacón MV, Van Dingenen J, Goormachtig S. 2023. Characterization of arbuscular mycorrhizal effector proteins. International Journal of Molecular Sciences 24: 9125.
Arnebrant K, Ek H, Finlay RD, Söderström B. 1993. Nitrogen translocation between Alnus glutinosa (L.) Gaertn. seedlings inoculated with Frankia sp. and Pinus contorta Doug, ex Loud seedlings connected by a common ectomycorrhizal mycelium. New Phytologist 124: 231-242.
Auer L, Buée M, Fauchery L, Lombard V, Barry K, Clum A, Copeland A, Daum C, Foster B, LaButti K et al. 2023. Metatranscriptomics sheds light on the links between the functional traits of fungal guilds and ecological processes in forest soil ecosystems. New Phytologist. doi: 10.1111/nph.19471.
Augé RM. 2001. Water relations, drought and vesicular-arbuscular mycorrhizal symbiosis. Mycorrhiza 11: 3-42.
Averill C, Anthony MA, Baldrian P, Finkbeiner F, van den Hoogen J, Kiers T, Kohout P, Hirt E, Smith GR, Crowther TW. 2022. Defending Earth’s terrestrial microbiome. Nature Microbiology 7: 1717-1725.
Babikova Z, Gilbert L, Bruce TJ, Birkett M, Caulfield JC, Woodcock C, Pickett JA, Johnson D. 2013. Underground signals carried through common mycelial networks warn neighbouring plants of aphid attack. Ecology Letters 16: 835-843.
Bahram M, Hildebrand F, Forslund SK, Anderson JL, Soudzilovska NA, Bodegom PM, Bengtsson-Palme J, Anslan S, Coelho LP, Harend H et al. 2018. Structure and function of the global topsoil microbiome. Nature 560: 233-237.
Baldrian P, López-Mondéjar R, Kohout P. 2023. Forest microbiome and global change. Nature Reviews Microbiology 21: 487-501.
Balestrini R, Nerva L, Sillo F, Girlanda M, Perotto S. 2014. Plant and fungal gene expression in mycorrhizal protocorms of the orchid Serapias vomeracea colonized by Tulasnella calospora. Plant Signaling & Behavior 9: e977707.
Basiru S, Hijri M. 2022. The potential applications of commercial arbuscular mycorrhizal fungal inoculants and their ecological consequences. Microorganisms 10: 1897.
Beiler KJ, Durall DM, Simard SW, Maxwell SA, Kretzer AM. 2010. Architecture of the wood-wide web: Rhizopogon spp.genets link multiple Douglas-fir cohorts. New Phytologist 185: 543-553.
Bödeker ITM, Clemmensen KE, De Boer W, Martin F, Olson Å, Lindahl B. 2014. Ectomycorrhizal Cortinarius species participate in enzymatic oxidation of humus in northern forest ecosystems. New Phytologist 203: 245-256.
Bogar LM. 2023. Modified source-sink dynamics govern resource exchange in ectomycorrhizal symbiosis. New Phytologist. doi: 10.1111/nph.19259.
Bouffaud ML, Herrmann S, Tarkka MT, Bönn M, Feldhahn L, Buscot F. 2020. Oak displays common local but specific distant gene regulation responses to different mycorrhizal fungi. BMC Genomics 21: 399.
Brundrett MC, Tedersoo L. 2018. Evolutionary history of mycorrhizal symbioses and global host plant diversity. New Phytologist 220: 1108-1115.
Cahanovitc R, Livne-Luzon S, Angel R, Klein T. 2022. Ectomycorrhizal fungi mediate belowground carbon transfer between pines and oaks. ISME Journal 16: 1420-1429.
Cai J, Liu X, Vanneste K, Proost S, Tsai WC, Liu KW, Chen LJ, He Y, Xu Q, Bian C et al. 2015. The genome sequence of the orchid Phalaenopsis equestris. Nature Genetics 47: 65-72.
Carteron A, Vellend M, Laliberté E. 2022. Mycorrhizal dominance reduces local tree species diversity across US forests. Nature Ecology & Evolution 6: 370-374.
Cavagnaro TR, Bender SF, Asghari HR, van der Heijden MGA. 2015. The role of arbuscular mycorrhizas in reducing soil nutrient loss. Trends in Plant Science 20: 283-290.
Chang Y, Desirò A, Na H, Sandor L, Lipzen A, Clum A, Barry K, Grigoriev IV, Martin FM, Stajich JE et al. 2019. Phylogenomics of Endogonaceae and evolution of mycorrhizas within Mucoromycota. New Phytologist 222: 511-525.
Chaudhary VB, Rúa MA, Antoninka A, Bever JD, Cannon J, Craig A, Duchicela J, Frame A, Gardes M, Gehring C et al. 2016. MycoDB, a global database of plant response to mycorrhizal fungi. Scientific Data 3: 160028.
Chen J, Liu SS, Kohler A, Yan B, Luo HM, Chen XM, Guo SX. 2017. iTRAQ and RNA-seq analyses provide new insights into regulation mechanism of symbiotic germination of Dendrobium officinale seeds (Orchidaceae). Journal of Proteome Research 16: 2174-2187.
Chen J, Tang Y, Kohler A, Lebreton A, Xing Y, Zhou D, Li Y, Martin FM, Guo S. 2022. Comparative transcriptomics analysis of the symbiotic germination of
Clemmensen KE, Durling MB, Michelsen A, Hallin S, Finlay RD, Lindahl BD. 2021. A tipping point in carbon storage when forest expands into tundra is related to mycorrhizal recycling of nitrogen. Ecology Letters 24: 1193-1204.
Cope KR, Bascaules A, Irving TB, Venkateshwaran M, Maeda J, Garcia K, Rush TA, Ma C, Labbé J, Jawdy S et al. 2019. The ectomycorrhizal fungus Laccaria bicolor produces lipochitooligosaccharides and uses the common symbiosis pathway to colonize Populus roots. Plant Cell 10: 2386-2410.
Davison J, Moora M, Öpik M, Adholeya A, Ainsaar L, Bâ A, Burla S, Diedhiou AG, Hiiesalu I, Jairus T et al. 2015. Fungal Symbionts. Global assessment of arbuscular mycorrhizal fungus diversity reveals very low endemism. Science 349: 970-973.
De La Providencia IE, De Souza FA, Fernández F, Delmas NS, Declerck S. 2005. Arbuscular mycorrhizal fungi reveal distinct patterns of anastomosis formation and hyphal healing mechanisms between different phylogenic groups. New Phytologist 165: 261-271.
Delaux PM, Radhakrishnan GV, Jayaraman D, Cheema J, Malbreil M, Volkening JD, Sekimoto H, Nishiyama T, Melkonian M, Pokorny L et al. 2015. Algal
ancestor of land plants was preadapted for symbiosis. Proceedings of the National Academy of Sciences, USA 112: 13390-13395.
Delgado-Baquerizo M, Oliverio AM, Brewer TE, Benavent-González A, Eldridge DJ, Bardgett RD, Maestre FT, Singh BK, Fierer N. 2018. A global atlas of the dominant bacteria found in soil. Science 359: 320-325.
Delgado-Baquerizo M, Reich PB, Trivedi C, Eldridge DJ, Abades S, Alfaro FD, Bastida F, Berhe AA, Cutler NA, Gallardo A et al. 2020. Multiple elements of soil biodiversity drive ecosystem functions across biomes. Nature Ecology & Evolution 4: 210-220.
Deng M, Hu S, Guo L, Jiang L, Huang Y, Schmid B, Liu C, Chang P, Li S, Liu X et al. 2023. Tree mycorrhizal association types control biodiversity-productivity relationship in a subtropical forest. Science Advances 9: eadd4468.
Desirò A, Rimington WR, Jacob A, Vande Pol N, Smith ME, Trappe JM, Bidartondo MI, Bonito G. 2017. Multigene phylogeny of Endogonales, an early diverging lineage of fungi associated with plants. IMA Fungus 8: 245-257.
Dickie IA, Bolstridge N, Cooper JA, Peltzer DA. 2010. Co-invasion by Pinusand its mycorrhizal fungi. New Phytologist 187: 475-484.
Drula E, Garron ML, Dogan S, Lombard V, Henrissat B, Terrapon N. 2022. The carbohydrate-active enzyme database: functions and literature. Nucleic Acids Research 50: D571-D577.
Edlinger A, Garland G, Hartman K, Banerjee S, Degrune F, García-Palacios P, Hallin S, Valzano-Held A, Herzog C, Jansa J et al. 2022. Agricultural management and pesticide use reduce the functioning of beneficial plant symbionts. Nature Ecology & Evolution 6: 1145-1154.
Emmett BD, Lévesque-Tremblay V, Harrison MJ. 2021. Conserved and reproducible bacterial communities associate with extraradical hyphae of arbuscular mycorrhizal fungi. ISME Journal 15: 2276-2288.
Ezawa T, Silvestri A, Maruyama H, Tawaraya K, Suzuki M, Duan Y. 2023. Structurally distinct mitoviruses: are they an ancestral lineage of the Mitoviridae exclusive to arbuscular mycorrhizal fungi (Glomeromycotina)? MBio 14: e0024023.
Baldrian P, López-Mondéjar R, Kohout P. 2023. Forest microbiome and global change. Nature Reviews Microbiology 21: 487-501.
Balestrini R, Nerva L, Sillo F, Girlanda M, Perotto S. 2014. Plant and fungal gene expression in mycorrhizal protocorms of the orchid Serapias vomeracea colonized by Tulasnella calospora. Plant Signaling & Behavior 9: e977707.
Basiru S, Hijri M. 2022. The potential applications of commercial arbuscular mycorrhizal fungal inoculants and their ecological consequences. Microorganisms 10: 1897.
Beiler KJ, Durall DM, Simard SW, Maxwell SA, Kretzer AM. 2010. Architecture of the wood-wide web: Rhizopogon spp.genets link multiple Douglas-fir cohorts. New Phytologist 185: 543-553.
Bödeker ITM, Clemmensen KE, De Boer W, Martin F, Olson Å, Lindahl B. 2014. Ectomycorrhizal Cortinarius species participate in enzymatic oxidation of humus in northern forest ecosystems. New Phytologist 203: 245-256.
Bogar LM. 2023. Modified source-sink dynamics govern resource exchange in ectomycorrhizal symbiosis. New Phytologist. doi: 10.1111/nph.19259.
Bouffaud ML, Herrmann S, Tarkka MT, Bönn M, Feldhahn L, Buscot F. 2020. Oak displays common local but specific distant gene regulation responses to different mycorrhizal fungi. BMC Genomics 21: 399.
Brundrett MC, Tedersoo L. 2018. Evolutionary history of mycorrhizal symbioses and global host plant diversity. New Phytologist 220: 1108-1115.
Cahanovitc R, Livne-Luzon S, Angel R, Klein T. 2022. Ectomycorrhizal fungi mediate belowground carbon transfer between pines and oaks. ISME Journal 16: 1420-1429.
Cai J, Liu X, Vanneste K, Proost S, Tsai WC, Liu KW, Chen LJ, He Y, Xu Q, Bian C et al. 2015. The genome sequence of the orchid Phalaenopsis equestris. Nature Genetics 47: 65-72.
Carteron A, Vellend M, Laliberté E. 2022. Mycorrhizal dominance reduces local tree species diversity across US forests. Nature Ecology & Evolution 6: 370-374.
Cavagnaro TR, Bender SF, Asghari HR, van der Heijden MGA. 2015. The role of arbuscular mycorrhizas in reducing soil nutrient loss. Trends in Plant Science 20: 283-290.
Chang Y, Desirò A, Na H, Sandor L, Lipzen A, Clum A, Barry K, Grigoriev IV, Martin FM, Stajich JE et al. 2019. Phylogenomics of Endogonaceae and evolution of mycorrhizas within Mucoromycota. New Phytologist 222: 511-525.
Chaudhary VB, Rúa MA, Antoninka A, Bever JD, Cannon J, Craig A, Duchicela J, Frame A, Gardes M, Gehring C et al. 2016. MycoDB, a global database of plant response to mycorrhizal fungi. Scientific Data 3: 160028.
Chen J, Liu SS, Kohler A, Yan B, Luo HM, Chen XM, Guo SX. 2017. iTRAQ and RNA-seq analyses provide new insights into regulation mechanism of symbiotic germination of Dendrobium officinale seeds (Orchidaceae). Journal of Proteome Research 16: 2174-2187.
Chen J, Tang Y, Kohler A, Lebreton A, Xing Y, Zhou D, Li Y, Martin FM, Guo S. 2022. Comparative transcriptomics analysis of the symbiotic germination of
Clemmensen KE, Durling MB, Michelsen A, Hallin S, Finlay RD, Lindahl BD. 2021. A tipping point in carbon storage when forest expands into tundra is related to mycorrhizal recycling of nitrogen. Ecology Letters 24: 1193-1204.
Cope KR, Bascaules A, Irving TB, Venkateshwaran M, Maeda J, Garcia K, Rush TA, Ma C, Labbé J, Jawdy S et al. 2019. The ectomycorrhizal fungus Laccaria bicolor produces lipochitooligosaccharides and uses the common symbiosis pathway to colonize Populus roots. Plant Cell 10: 2386-2410.
Davison J, Moora M, Öpik M, Adholeya A, Ainsaar L, Bâ A, Burla S, Diedhiou AG, Hiiesalu I, Jairus T et al. 2015. Fungal Symbionts. Global assessment of arbuscular mycorrhizal fungus diversity reveals very low endemism. Science 349: 970-973.
De La Providencia IE, De Souza FA, Fernández F, Delmas NS, Declerck S. 2005. Arbuscular mycorrhizal fungi reveal distinct patterns of anastomosis formation and hyphal healing mechanisms between different phylogenic groups. New Phytologist 165: 261-271.
Delaux PM, Radhakrishnan GV, Jayaraman D, Cheema J, Malbreil M, Volkening JD, Sekimoto H, Nishiyama T, Melkonian M, Pokorny L et al. 2015. Algal
ancestor of land plants was preadapted for symbiosis. Proceedings of the National Academy of Sciences, USA 112: 13390-13395.
Delgado-Baquerizo M, Oliverio AM, Brewer TE, Benavent-González A, Eldridge DJ, Bardgett RD, Maestre FT, Singh BK, Fierer N. 2018. A global atlas of the dominant bacteria found in soil. Science 359: 320-325.
Delgado-Baquerizo M, Reich PB, Trivedi C, Eldridge DJ, Abades S, Alfaro FD, Bastida F, Berhe AA, Cutler NA, Gallardo A et al. 2020. Multiple elements of soil biodiversity drive ecosystem functions across biomes. Nature Ecology & Evolution 4: 210-220.
Deng M, Hu S, Guo L, Jiang L, Huang Y, Schmid B, Liu C, Chang P, Li S, Liu X et al. 2023. Tree mycorrhizal association types control biodiversity-productivity relationship in a subtropical forest. Science Advances 9: eadd4468.
Desirò A, Rimington WR, Jacob A, Vande Pol N, Smith ME, Trappe JM, Bidartondo MI, Bonito G. 2017. Multigene phylogeny of Endogonales, an early diverging lineage of fungi associated with plants. IMA Fungus 8: 245-257.
Dickie IA, Bolstridge N, Cooper JA, Peltzer DA. 2010. Co-invasion by Pinusand its mycorrhizal fungi. New Phytologist 187: 475-484.
Drula E, Garron ML, Dogan S, Lombard V, Henrissat B, Terrapon N. 2022. The carbohydrate-active enzyme database: functions and literature. Nucleic Acids Research 50: D571-D577.
Edlinger A, Garland G, Hartman K, Banerjee S, Degrune F, García-Palacios P, Hallin S, Valzano-Held A, Herzog C, Jansa J et al. 2022. Agricultural management and pesticide use reduce the functioning of beneficial plant symbionts. Nature Ecology & Evolution 6: 1145-1154.
Emmett BD, Lévesque-Tremblay V, Harrison MJ. 2021. Conserved and reproducible bacterial communities associate with extraradical hyphae of arbuscular mycorrhizal fungi. ISME Journal 15: 2276-2288.
Ezawa T, Silvestri A, Maruyama H, Tawaraya K, Suzuki M, Duan Y. 2023. Structurally distinct mitoviruses: are they an ancestral lineage of the Mitoviridae exclusive to arbuscular mycorrhizal fungi (Glomeromycotina)? MBio 14: e0024023.
Finlay RD, Clemmensen KE. 2017. Immobilization of carbon in mycorrhizal mycelial biomass and secretions. In: Johnson NC, Gehring K, Jansa J, eds. Mycorrhizal mediation of soil: fertility, structure and carbon storage. Amsterdam, the Netherlands: Elsevier, 413-440.
Finlay RD, Read DJ. 1986. The structure and function of the vegetative mycelium of ectomycorrhizal plants. New Phytologist 103: 143-156.
Fiore-Donno AM, Martin F. 2001. Populations of ectomycorrhizal Laccaria amethystina and Xerocomusspp. show contrasting colonization patterns in a mixed forest. New Phytologist 152: 533-542.
Floudas D, Bentzer J, Ahrén D, Johansson T, Persson P, Tunlid A. 2020. Uncovering the hidden diversity of litter-decomposition mechanisms in mushroom-forming fungi. ISME Journal 14: 2046-2059.
Floudas D, Binder M, Riley R, Barry K, Blanchette RA, Henrissat B, Martinez AT, Otillar R, Spatafora JW, Yadav JS et al. 2012. The Paleozoic origin of enzymatic lignin decomposition reconstructed from 31 fungal genomes. Science 336: 17151719.
Finlay RD, Read DJ. 1986. The structure and function of the vegetative mycelium of ectomycorrhizal plants. New Phytologist 103: 143-156.
Fiore-Donno AM, Martin F. 2001. Populations of ectomycorrhizal Laccaria amethystina and Xerocomusspp. show contrasting colonization patterns in a mixed forest. New Phytologist 152: 533-542.
Floudas D, Bentzer J, Ahrén D, Johansson T, Persson P, Tunlid A. 2020. Uncovering the hidden diversity of litter-decomposition mechanisms in mushroom-forming fungi. ISME Journal 14: 2046-2059.
Floudas D, Binder M, Riley R, Barry K, Blanchette RA, Henrissat B, Martinez AT, Otillar R, Spatafora JW, Yadav JS et al. 2012. The Paleozoic origin of enzymatic lignin decomposition reconstructed from 31 fungal genomes. Science 336: 17151719.
Frey-Klett P, Garbaye J, Tarkka M. 2007. The mycorrhiza helper bacteria revisited. New Phytologist 176: 22-36.
Garcia K, Delaux PM, Cope KR, Ané J-M. 2015. Molecular signals required for the establishment and maintenance of ectomycorrhizal symbioses. New Phytologist 208: 79-87.
Gaude N, Bortfeld S, Duensing N, Lohse M, Krajinski F. 2012. Arbusculecontaining and non-colonized cortical cells of mycorrhizal roots undergo extensive and specific reprogramming during arbuscular mycorrhizal development. The Plant Journal 69: 510-528.
Ghirardo A, Fochi V, Lange B, Witting M, Schnitzler JP, Perotto S, Balestrini R. 2020. Metabolomic adjustments in the orchid mycorrhizal fungus Tulasnella calospora during symbiosis with Serapias vomeracea. New Phytologist 228: 19391952.
Garcia K, Delaux PM, Cope KR, Ané J-M. 2015. Molecular signals required for the establishment and maintenance of ectomycorrhizal symbioses. New Phytologist 208: 79-87.
Gaude N, Bortfeld S, Duensing N, Lohse M, Krajinski F. 2012. Arbusculecontaining and non-colonized cortical cells of mycorrhizal roots undergo extensive and specific reprogramming during arbuscular mycorrhizal development. The Plant Journal 69: 510-528.
Ghirardo A, Fochi V, Lange B, Witting M, Schnitzler JP, Perotto S, Balestrini R. 2020. Metabolomic adjustments in the orchid mycorrhizal fungus Tulasnella calospora during symbiosis with Serapias vomeracea. New Phytologist 228: 19391952.
Giovannetti M, Fortuna P, Citernesi AS, Morini S, Nuti MP. 2001. The occurrence of anastomosis formation and nuclear exchange in intact arbuscular mycorrhizal networks. New Phytologist 151: 717-724.
Givnish TJ, Spalink D, Ames M, Lyon SP, Hunter SJ, Zuluaga A, Iles WJ, Clements MA, Arroyo MT, Leebens-Mack J et al. 2015. Orchid phylogenomics and multiple drivers of their extraordinary diversification. Proceedings of the Royal Society B: Biological Sciences 282: 20151553.
Givnish TJ, Spalink D, Ames M, Lyon SP, Hunter SJ, Zuluaga A, Iles WJ, Clements MA, Arroyo MT, Leebens-Mack J et al. 2015. Orchid phylogenomics and multiple drivers of their extraordinary diversification. Proceedings of the Royal Society B: Biological Sciences 282: 20151553.
Grelet G, Martino E, Dickie IA, Tajuddin R, Artz R. 2016. Ecology of ericoid mycorrhizal fungi: what insight have we gained with molecular tools and what’s missing? In: Martin F, ed. Molecular mycorrhizal symbiosis. Hoboken, NJ, USA: John Wiley & Sons, 405-419.
Grelet GA, Johnson D, Vralstad T, Alexander IJ, Anderson IC. 2010. New insights into the mycorrhizal Rhizoscyphus ericae aggregate: spatial structure and co-colonization of ectomycorrhizal and ericoid roots. New Phytologist 188: 210-222.
Grigoriev IV, Nikitin R, Haridas S, Kuo A, Ohm R, Otillar R, Riley R, Salamov A, Zhao X, Korzeniewski F et al. 2014. MycoCosm portal: gearing up for 1000 fungal genomes. Nucleic Acids Research 42: D699-D704.
Grunwald U, Guo W, Fischer K, Isayenkov S, Ludwig-Müller J, Hause B, Yan X, Küster H, Franken P. 2009. Overlapping expression patterns and differential transcript levels of phosphate transporter genes in arbuscular mycorrhizal, Pifertilised and phytohormone-treated Medicago truncatula roots. Planta 229: 1023-1034.
Gryta H, Debaud JC, Marmeisse R. 2000. Population dynamics of the symbiotic mushroom Hebeloma cylindrosporum: mycelial persistence and inbreeding. Heredity 84: 294-302.
Hagh-Doust N, Mikryukov V, Anslan S, Bahram M, Puusepp R, Dulya O, Tedersoo L. 2023. Effects of nitrogen deposition on carbon and nutrient cycling along a natural soil acidity gradient as revealed by metagenomics. New Phytologist 238: 2607-2620.
Hart MM, Antunes PM, Chaudhary VB, Abbott LK. 2018. Fungal inoculants in the field: is the reward greater than the risk? Functional Ecology 32: 126-135.
Hartnett DC, Wilson GW. 1999. Mycorrhizae influence plant community structure and diversity in tallgrass prairie. Ecology 80: 1187-1195.
Hawkins HJ, Cargill RI, Van Nuland ME, Hagen SC, Field KJ, Sheldrake M, Soudzilovskaia NA, Kiers ET. 2023. Mycorrhizal mycelium as a global carbon pool. Current Biology 33: R560-R573.
van der Heijden MG. 2004. Arbuscular mycorrhizal fungi as support systems for seedling establishment in grassland. Ecology Letters 7: 293-303.
van der Heijden MGA, Bardgett RD, van Straalen NM. 2008. The unseen majority: soil microbes as drivers of plant diversity and productivity in terrestrial ecosystems. Ecology Letters 11: 296-310.
van der Heijden MGA, Horton TR. 2009. Socialism in soil? The importance of mycorrhizal fungal networks for facilitation in natural ecosystems. Journal of Ecology 97: 1139-1150.
van der Heijden MGA, Klironomos JN, Ursic M, Moutoglis P, StreitwolfEngel R, Boller T, Wiemken A, Sanders IR. 1998. Mycorrhizal fungal diversity determines plant biodiversity, ecosystem variability and productivity. Nature 396: 69-72.
van der Heijden MGA, Martin F, Selosse MA, Sanders IR. 2015. Mycorrhizal ecology and evolution: the past, the present, and the future. New Phytologist 205: 1406-1423.
Henriksson N, Marshall J, Högberg MN, Högberg P, Polle A, Franklin O, Näsholm T. 2023. Re-examining the evidence for the mother tree hypothesisresource sharing among trees via ectomycorrhizal networks. New Phytologist 239: 19-28.
Hicks Pries CE, Lankau R, Ingham GA, Legge E, Krol O, Forrester J, Fitch A, Wurzburger N. 2023. Differences in soil organic matter between EcM-and AMdominated forests depend on tree and fungal identity. Ecology 104: e3929.
Hirose D, Kikuchi J, Kanzaki N, Futai K. 2004. Genet distribution of sporocarps and ectomycorrhizas of Suillus pictus in a Japanese white pine plantation. New Phytologist 164: 527-541.
Hobbie JE, Hobbie EA. 2006.
Hoeksema JD, Chaudhary VB, Gehring CA, Johnson NC, Karst J, Koide RT, Umbanhowar J. 2010. A meta-analysis of context-dependency in plant response to inoculation with mycorrhizal fungi. Ecology Letters 13: 394-407.
Hogekamp C, Küster H. 2013. A roadmap of cell-type specific gene expression during sequential stages of the arbuscular mycorrhiza symbiosis. BMC Genomics 14: 306.
Hohnjec N, Vieweg MF, Pühler A, Becker A, Küster H. 2005. Overlaps in the transcriptional profiles of Medicago truncatula roots inoculated with two different glomus fungi provide insights into the genetic program activated during arbuscular mycorrhiza. Plant Physiology 137: 1283-1301.
Grelet GA, Johnson D, Vralstad T, Alexander IJ, Anderson IC. 2010. New insights into the mycorrhizal Rhizoscyphus ericae aggregate: spatial structure and co-colonization of ectomycorrhizal and ericoid roots. New Phytologist 188: 210-222.
Grigoriev IV, Nikitin R, Haridas S, Kuo A, Ohm R, Otillar R, Riley R, Salamov A, Zhao X, Korzeniewski F et al. 2014. MycoCosm portal: gearing up for 1000 fungal genomes. Nucleic Acids Research 42: D699-D704.
Grunwald U, Guo W, Fischer K, Isayenkov S, Ludwig-Müller J, Hause B, Yan X, Küster H, Franken P. 2009. Overlapping expression patterns and differential transcript levels of phosphate transporter genes in arbuscular mycorrhizal, Pifertilised and phytohormone-treated Medicago truncatula roots. Planta 229: 1023-1034.
Gryta H, Debaud JC, Marmeisse R. 2000. Population dynamics of the symbiotic mushroom Hebeloma cylindrosporum: mycelial persistence and inbreeding. Heredity 84: 294-302.
Hagh-Doust N, Mikryukov V, Anslan S, Bahram M, Puusepp R, Dulya O, Tedersoo L. 2023. Effects of nitrogen deposition on carbon and nutrient cycling along a natural soil acidity gradient as revealed by metagenomics. New Phytologist 238: 2607-2620.
Hart MM, Antunes PM, Chaudhary VB, Abbott LK. 2018. Fungal inoculants in the field: is the reward greater than the risk? Functional Ecology 32: 126-135.
Hartnett DC, Wilson GW. 1999. Mycorrhizae influence plant community structure and diversity in tallgrass prairie. Ecology 80: 1187-1195.
Hawkins HJ, Cargill RI, Van Nuland ME, Hagen SC, Field KJ, Sheldrake M, Soudzilovskaia NA, Kiers ET. 2023. Mycorrhizal mycelium as a global carbon pool. Current Biology 33: R560-R573.
van der Heijden MG. 2004. Arbuscular mycorrhizal fungi as support systems for seedling establishment in grassland. Ecology Letters 7: 293-303.
van der Heijden MGA, Bardgett RD, van Straalen NM. 2008. The unseen majority: soil microbes as drivers of plant diversity and productivity in terrestrial ecosystems. Ecology Letters 11: 296-310.
van der Heijden MGA, Horton TR. 2009. Socialism in soil? The importance of mycorrhizal fungal networks for facilitation in natural ecosystems. Journal of Ecology 97: 1139-1150.
van der Heijden MGA, Klironomos JN, Ursic M, Moutoglis P, StreitwolfEngel R, Boller T, Wiemken A, Sanders IR. 1998. Mycorrhizal fungal diversity determines plant biodiversity, ecosystem variability and productivity. Nature 396: 69-72.
van der Heijden MGA, Martin F, Selosse MA, Sanders IR. 2015. Mycorrhizal ecology and evolution: the past, the present, and the future. New Phytologist 205: 1406-1423.
Henriksson N, Marshall J, Högberg MN, Högberg P, Polle A, Franklin O, Näsholm T. 2023. Re-examining the evidence for the mother tree hypothesisresource sharing among trees via ectomycorrhizal networks. New Phytologist 239: 19-28.
Hicks Pries CE, Lankau R, Ingham GA, Legge E, Krol O, Forrester J, Fitch A, Wurzburger N. 2023. Differences in soil organic matter between EcM-and AMdominated forests depend on tree and fungal identity. Ecology 104: e3929.
Hirose D, Kikuchi J, Kanzaki N, Futai K. 2004. Genet distribution of sporocarps and ectomycorrhizas of Suillus pictus in a Japanese white pine plantation. New Phytologist 164: 527-541.
Hobbie JE, Hobbie EA. 2006.
Hoeksema JD, Chaudhary VB, Gehring CA, Johnson NC, Karst J, Koide RT, Umbanhowar J. 2010. A meta-analysis of context-dependency in plant response to inoculation with mycorrhizal fungi. Ecology Letters 13: 394-407.
Hogekamp C, Küster H. 2013. A roadmap of cell-type specific gene expression during sequential stages of the arbuscular mycorrhiza symbiosis. BMC Genomics 14: 306.
Hohnjec N, Vieweg MF, Pühler A, Becker A, Küster H. 2005. Overlaps in the transcriptional profiles of Medicago truncatula roots inoculated with two different glomus fungi provide insights into the genetic program activated during arbuscular mycorrhiza. Plant Physiology 137: 1283-1301.
Horsch CC, Antunes PM, Fahey C, Grandy AS, Kallenbach CM. 2023. Traitbased assembly of arbuscular mycorrhizal fungal communities determines soil carbon formation and retention. New Phytologist 239: 311-324.
Hortal S, Plett KL, Plett JM, Cresswell T, Johansen M, Pendall E, Anderson IC. 2017. Role of plant-fungal nutrient trading and host control in determining the competitive success of ectomycorrhizal fungi. ISME Journal 11: 2666-2676.
Hortal S, Trocha LK, Murat C, Chybicki IJ, Buée M, Trojankiewicz M, Burczyk J, Martin F. 2012. Beech roots are simultaneously colonized by multiple genets of the ectomycorrhizal fungus Laccaria amethystina clustered in two genetic groups. Molecular Ecology 21: 2116-2129.
Hoysted GA, Field KJ, Sinanaj B, Bell CA, Bidartondo MI, Pressel S. 2023. Direct nitrogen, phosphorus and carbon exchanges between Mucoromycotina ‘fine root endophyte’ fungi and a flowering plant in novel monoxenic cultures. New Phytologist 238: 70-79.
Huynh-Thu VA, Irrthum A, Wehenkel L, Geurts P. 2010. Inferring regulatory networks from expression data using treebased methods. PLoS ONE 5: e12776.
Jastrow JD, Miller RM, Lussenhop J. 1998. Contributions of interacting biological mechanisms to soil aggregate stabilization in restored prairie. Soil Biology and Biochemistry 30: 905-916.
Jia Y, van der Heijden MGA, Wagg C, Feng G, Walder F. 2021. Symbiotic soil fungi enhance resistance and resilience of an experimental grassland to drought and nitrogen deposition. Journal of Ecology 109: 3171-3181.
Jiang L, Lin M, Wang H, Song H, Zhang L, Huang Q, Chen R, Song C, Li G, Cao Y. 2022. Haplotype-resolved genome assembly of Bletilla striata to elucidate medicinal value. The Plant Journal 111: 1340-1353.
Jumper J, Evans R, Pritzel A, Green T, Figurnov M, Ronneberger O, Tunyasuvunakool K, Bates R, Źídek A, Potapenko A et al. 2021. Highly accurate protein structure prediction with AlphaFold. Nature 596: 583-589.
Kakouridis A, Hagen JA, Kan MP, Mambelli S, Feldman LJ, Herman DJ, Weber PK, Pett-Ridge J, Firestone MK. 2022. Routes to roots: direct evidence of water transport by arbuscular mycorrhizal fungi to host plants. New Phytologist 236: 210-221.
Kameoka H, Gutjahr C. 2022. Functions of lipids in development and reproduction of arbuscular mycorrhizal fungi. Plant Cell Physiology 63: 13561365.
Hortal S, Plett KL, Plett JM, Cresswell T, Johansen M, Pendall E, Anderson IC. 2017. Role of plant-fungal nutrient trading and host control in determining the competitive success of ectomycorrhizal fungi. ISME Journal 11: 2666-2676.
Hortal S, Trocha LK, Murat C, Chybicki IJ, Buée M, Trojankiewicz M, Burczyk J, Martin F. 2012. Beech roots are simultaneously colonized by multiple genets of the ectomycorrhizal fungus Laccaria amethystina clustered in two genetic groups. Molecular Ecology 21: 2116-2129.
Hoysted GA, Field KJ, Sinanaj B, Bell CA, Bidartondo MI, Pressel S. 2023. Direct nitrogen, phosphorus and carbon exchanges between Mucoromycotina ‘fine root endophyte’ fungi and a flowering plant in novel monoxenic cultures. New Phytologist 238: 70-79.
Huynh-Thu VA, Irrthum A, Wehenkel L, Geurts P. 2010. Inferring regulatory networks from expression data using treebased methods. PLoS ONE 5: e12776.
Jastrow JD, Miller RM, Lussenhop J. 1998. Contributions of interacting biological mechanisms to soil aggregate stabilization in restored prairie. Soil Biology and Biochemistry 30: 905-916.
Jia Y, van der Heijden MGA, Wagg C, Feng G, Walder F. 2021. Symbiotic soil fungi enhance resistance and resilience of an experimental grassland to drought and nitrogen deposition. Journal of Ecology 109: 3171-3181.
Jiang L, Lin M, Wang H, Song H, Zhang L, Huang Q, Chen R, Song C, Li G, Cao Y. 2022. Haplotype-resolved genome assembly of Bletilla striata to elucidate medicinal value. The Plant Journal 111: 1340-1353.
Jumper J, Evans R, Pritzel A, Green T, Figurnov M, Ronneberger O, Tunyasuvunakool K, Bates R, Źídek A, Potapenko A et al. 2021. Highly accurate protein structure prediction with AlphaFold. Nature 596: 583-589.
Kakouridis A, Hagen JA, Kan MP, Mambelli S, Feldman LJ, Herman DJ, Weber PK, Pett-Ridge J, Firestone MK. 2022. Routes to roots: direct evidence of water transport by arbuscular mycorrhizal fungi to host plants. New Phytologist 236: 210-221.
Kameoka H, Gutjahr C. 2022. Functions of lipids in development and reproduction of arbuscular mycorrhizal fungi. Plant Cell Physiology 63: 13561365.
Kang H, Chen X, Kempannien M, Pardo AG, Veneault-Fourrey C, Kohler A, Martin F. 2020. The small secreted effector protein MiSSP7.6 of Laccaria bicolor is required for the establishment of ectomycorrhizal symbiosis. Environmental Microbiology 22: 1435-1446.
Karst J, Jones MD, Hoeksema JD. 2023. Positive citation bias and overinterpreted results lead to misinformation on common mycorrhizal networks in forests. Nature Ecology & Evolution 7: 501-511.
Kivlin SN. 2020. Global mycorrhizal fungal range sizes vary within and among mycorrhizal guilds but are not correlated with dispersal traits. Journal of Biogeography 47: 1994-2001.
Klein T, Siegwolf RTW, Körner C. 2016. Belowground carbon trade among tall trees in a temperate forest. Science 352: 342-344.
Klironomos JN. 2002. Feedback with soil biota contributes to plant rarity and invasiveness in communities. Nature 417: 67-70.
Kloppholz S, Kuhn H, Requena N. 2011. A secreted fungal effector of Glomus intraradices promotes symbiotic biotrophy. Current Biology 21: 1204-1209.
Kobayashi Y, Maeda T, Yamaguchi K, Kameoka H, Tanaka S, Ezawa T, Shigenobu S, Kawaguchi M. 2018. The genome of Rhizophagus clarus HR1 reveals a common genetic basis for auxotrophy among arbuscular mycorrhizal fungi. BMC Genomics 2018: 465.
Kobayashi Y, Shibata TF, Hirakawa H, Nishiyama T, Yamada A, Hasebe M, Shigenobu S, Kawaguchi M. 2023. The genome of Lyophyllum shimeji provides insight into the initial evolution of ectomycorrhizal fungal genomes.
Kohler A, Kuo A, Nagy LG, Morin E, Barry KW, Buscot F, Canbäck B, Choi C, Cichocki N, Clum A et al. 2015. Convergent losses of decay mechanisms and rapid turnover of symbiosis genes in mycorrhizal mutualists. Nature Genetics 47: 410-415.
Kokkoris V, Chagnon PL, Yildirir G, Clarke K, Goh D, MacLean AM, Dettman J, Stefani F, Corradi N. 2021. Host identity influences nuclear dynamics in arbuscular mycorrhizal fungi. Current Biology 31: 1531-1538.
Karst J, Jones MD, Hoeksema JD. 2023. Positive citation bias and overinterpreted results lead to misinformation on common mycorrhizal networks in forests. Nature Ecology & Evolution 7: 501-511.
Kivlin SN. 2020. Global mycorrhizal fungal range sizes vary within and among mycorrhizal guilds but are not correlated with dispersal traits. Journal of Biogeography 47: 1994-2001.
Klein T, Siegwolf RTW, Körner C. 2016. Belowground carbon trade among tall trees in a temperate forest. Science 352: 342-344.
Klironomos JN. 2002. Feedback with soil biota contributes to plant rarity and invasiveness in communities. Nature 417: 67-70.
Kloppholz S, Kuhn H, Requena N. 2011. A secreted fungal effector of Glomus intraradices promotes symbiotic biotrophy. Current Biology 21: 1204-1209.
Kobayashi Y, Maeda T, Yamaguchi K, Kameoka H, Tanaka S, Ezawa T, Shigenobu S, Kawaguchi M. 2018. The genome of Rhizophagus clarus HR1 reveals a common genetic basis for auxotrophy among arbuscular mycorrhizal fungi. BMC Genomics 2018: 465.
Kobayashi Y, Shibata TF, Hirakawa H, Nishiyama T, Yamada A, Hasebe M, Shigenobu S, Kawaguchi M. 2023. The genome of Lyophyllum shimeji provides insight into the initial evolution of ectomycorrhizal fungal genomes.
Kohler A, Kuo A, Nagy LG, Morin E, Barry KW, Buscot F, Canbäck B, Choi C, Cichocki N, Clum A et al. 2015. Convergent losses of decay mechanisms and rapid turnover of symbiosis genes in mycorrhizal mutualists. Nature Genetics 47: 410-415.
Kokkoris V, Chagnon PL, Yildirir G, Clarke K, Goh D, MacLean AM, Dettman J, Stefani F, Corradi N. 2021. Host identity influences nuclear dynamics in arbuscular mycorrhizal fungi. Current Biology 31: 1531-1538.
Kuyper TW, Jansa J. 2023. Arbuscular mycorrhiza: advances and retreats in our understanding of the ecological functioning of the mother of all root symbioses. Plant and Soil 489: 41-88.
Labbé J, Muchero W, Czarnecki O, Wang J, Wang X, Bryan AC, Zheng K, Yang Y, Xie M, Zhang J et al. 2019. Mediation of plant-mycorrhizal interaction by a lectin receptor-like kinase. Nature Plants 5: 676-680.
Labouyrie M, Ballabio C, Romero F, Panagos P, Jones A, Schmid MW, Mikryukov V, Dulya O, Tedersoo L, Bahram M et al. 2023. Patterns in soil microbial diversity across Europe. Nature Communications 14: 3311.
Lambers H, Raven JA, Shaver GR, Smith SE. 2008. Plant nutrient-acquisition strategies change with soil age. Trends in Ecology & Evolution 23: 95-103.
Lanfranco L, Bonfante P. 2023. Lessons from arbuscular mycorrhizal fungal genomes. Current Opinion in Microbiology 75: 102357.
Law SR, Serrano AR, Daguerre Y, Sundh J, Schneider AN, Stangl ZR, Castro D, Grabherr M, Näsholm T, Street NR et al. 2022. Metatranscriptomics captures dynamic shifts in mycorrhizal coordination in boreal forests. Proceedings of the National Academy of Sciences, USA 119: e2118852119.
Leake J, Johnson D, Donnelly D, Muckle G, Boddy L, Read D. 2004. Networks of power and influence: the role of mycorrhizal mycelium in controlling plant communities and agroecosystem functioning. Canadian Journal of Botany 82: 1016-1045.
Lebreton A, Tang N, Kuo A, LaButti K, Andreopoulos W, Drula E, Miyauchi S, Barry K, Clum A, Lipzen A et al. 2022. Comparative genomics reveals a dynamic genome evolution in the ectomycorrhizal milk-cap (Lactarius) mushrooms. New Phytologist 235: 306-319.
Lebreton A, Zeng Q, Miyauchi S, Kohler A, Dai Y-C, Martin FM. 2021. Evolution of the mode of nutrition in symbiotic and saprotrophic fungi in forest ecosystems. Annual Review of Ecology, Evolution, and Systematics 52: 385-404.
Ledford WC, Silvestri A, Fiorilli V, Roth R, Rubio-Somoza I, Lanfranco L. 2023. A journey into the world of small RNAs in the arbuscular mycorrhizal symbiosis. New Phytologist. doi: 10.1111/nph.19394.
Leifheit EF, Lehmann A, Rillig MC. 2021. Potential effects of microplastic on arbuscular mycorrhizal fungi. Frontiers in Plant Science 12: 626709.
Lekberg Y, Koide RT. 2005. Is plant performance limited by abundance of arbuscular mycorrhizal fungi? A meta-analysis of studies published between 1988 and 2003. New Phytologist 168: 189-204.
Li MH, Liu KW, Li Z, Lu HC, Ye QL, Zhang D, Wang JY, Li YF, Zhong ZM, Liu X et al. 2022. Genomes of leafy and leafless Platanthera orchids illuminate the evolution of mycoheterotrophy. Nature Plants 8: 373-388.
Li X, Zhao R, Li D, Wang G, Bei S, Ju X, An R, Li L, Kuyper TW, Christie P et al. 2023. Mycorrhiza-mediated recruitment of complete denitrifying Pseudomonas reduces
Liang M, Liu X, Parker IM, Johnson D, Zheng Y, Luo S, Gilbert GS, Yu S. 2019. Soil microbes drive phylogenetic diversity-productivity relationships in a subtropical forest. Science Advances 5: eaax5088.
Liao HL, Chen Y, Vilgalys R. 2018. Metatranscriptomic study of common and host-specific patterns of gene expression between pines and their symbiotic ectomycorrhizal fungi in the genus Suillus. PLoS Genetics 14: e1007742.
van der Linde S, Suz LM, Orme CDL, Cox F, Andreae H, Asi E, Atkinson B, Benham S, Carroll C, Cools N et al. 2018. Author correction: environment and host as large-scale controls of ectomycorrhizal fungi. Nature 561: E42.
Liu YR, van der Heijden MGA, Riedo J, Sanz-Lazaro C, Eldridge DJ, Bastida F, Moreno-Jiménez E, Zhou X-Q, Hu H-W, He J-Z et al. 2023. Soil contamination in nearby natural areas mirrors that in urban greenspaces worldwide. Nature Communications 14: 1706.
Lofgren LA, Nguyen NH, Vilgalys R, Ruytinx J, Liao H-L, Branco S, Kuo A, LaButti K, Lipzen A, Andreopoulos W et al. 2021. Comparative genomics reveals dynamic genome evolution in host specialist ectomycorrhizal fungi. New Phytologist 230: 774-792.
Looney B, Miyauchi S, Morin E, Drula E, Courty PE, Kohler A, Kuo A, LaButti K, Pangilinan J, Lipzen A et al. 2022. Evolutionary transition to the ectomycorrhizal habit in the genomes of a hyperdiverse lineage of mushroom-forming fungi. New Phytologist 233: 2294-2309.
Luo S, Phillips RP, Jo I, Fei S, Liang J, Schmid B, Eisenhauer N. 2023. Higher productivity in forests with mixed mycorrhizal strategies. Nature Communications 14: 1377.
Labbé J, Muchero W, Czarnecki O, Wang J, Wang X, Bryan AC, Zheng K, Yang Y, Xie M, Zhang J et al. 2019. Mediation of plant-mycorrhizal interaction by a lectin receptor-like kinase. Nature Plants 5: 676-680.
Labouyrie M, Ballabio C, Romero F, Panagos P, Jones A, Schmid MW, Mikryukov V, Dulya O, Tedersoo L, Bahram M et al. 2023. Patterns in soil microbial diversity across Europe. Nature Communications 14: 3311.
Lambers H, Raven JA, Shaver GR, Smith SE. 2008. Plant nutrient-acquisition strategies change with soil age. Trends in Ecology & Evolution 23: 95-103.
Lanfranco L, Bonfante P. 2023. Lessons from arbuscular mycorrhizal fungal genomes. Current Opinion in Microbiology 75: 102357.
Law SR, Serrano AR, Daguerre Y, Sundh J, Schneider AN, Stangl ZR, Castro D, Grabherr M, Näsholm T, Street NR et al. 2022. Metatranscriptomics captures dynamic shifts in mycorrhizal coordination in boreal forests. Proceedings of the National Academy of Sciences, USA 119: e2118852119.
Leake J, Johnson D, Donnelly D, Muckle G, Boddy L, Read D. 2004. Networks of power and influence: the role of mycorrhizal mycelium in controlling plant communities and agroecosystem functioning. Canadian Journal of Botany 82: 1016-1045.
Lebreton A, Tang N, Kuo A, LaButti K, Andreopoulos W, Drula E, Miyauchi S, Barry K, Clum A, Lipzen A et al. 2022. Comparative genomics reveals a dynamic genome evolution in the ectomycorrhizal milk-cap (Lactarius) mushrooms. New Phytologist 235: 306-319.
Lebreton A, Zeng Q, Miyauchi S, Kohler A, Dai Y-C, Martin FM. 2021. Evolution of the mode of nutrition in symbiotic and saprotrophic fungi in forest ecosystems. Annual Review of Ecology, Evolution, and Systematics 52: 385-404.
Ledford WC, Silvestri A, Fiorilli V, Roth R, Rubio-Somoza I, Lanfranco L. 2023. A journey into the world of small RNAs in the arbuscular mycorrhizal symbiosis. New Phytologist. doi: 10.1111/nph.19394.
Leifheit EF, Lehmann A, Rillig MC. 2021. Potential effects of microplastic on arbuscular mycorrhizal fungi. Frontiers in Plant Science 12: 626709.
Lekberg Y, Koide RT. 2005. Is plant performance limited by abundance of arbuscular mycorrhizal fungi? A meta-analysis of studies published between 1988 and 2003. New Phytologist 168: 189-204.
Li MH, Liu KW, Li Z, Lu HC, Ye QL, Zhang D, Wang JY, Li YF, Zhong ZM, Liu X et al. 2022. Genomes of leafy and leafless Platanthera orchids illuminate the evolution of mycoheterotrophy. Nature Plants 8: 373-388.
Li X, Zhao R, Li D, Wang G, Bei S, Ju X, An R, Li L, Kuyper TW, Christie P et al. 2023. Mycorrhiza-mediated recruitment of complete denitrifying Pseudomonas reduces
Liang M, Liu X, Parker IM, Johnson D, Zheng Y, Luo S, Gilbert GS, Yu S. 2019. Soil microbes drive phylogenetic diversity-productivity relationships in a subtropical forest. Science Advances 5: eaax5088.
Liao HL, Chen Y, Vilgalys R. 2018. Metatranscriptomic study of common and host-specific patterns of gene expression between pines and their symbiotic ectomycorrhizal fungi in the genus Suillus. PLoS Genetics 14: e1007742.
van der Linde S, Suz LM, Orme CDL, Cox F, Andreae H, Asi E, Atkinson B, Benham S, Carroll C, Cools N et al. 2018. Author correction: environment and host as large-scale controls of ectomycorrhizal fungi. Nature 561: E42.
Liu YR, van der Heijden MGA, Riedo J, Sanz-Lazaro C, Eldridge DJ, Bastida F, Moreno-Jiménez E, Zhou X-Q, Hu H-W, He J-Z et al. 2023. Soil contamination in nearby natural areas mirrors that in urban greenspaces worldwide. Nature Communications 14: 1706.
Lofgren LA, Nguyen NH, Vilgalys R, Ruytinx J, Liao H-L, Branco S, Kuo A, LaButti K, Lipzen A, Andreopoulos W et al. 2021. Comparative genomics reveals dynamic genome evolution in host specialist ectomycorrhizal fungi. New Phytologist 230: 774-792.
Looney B, Miyauchi S, Morin E, Drula E, Courty PE, Kohler A, Kuo A, LaButti K, Pangilinan J, Lipzen A et al. 2022. Evolutionary transition to the ectomycorrhizal habit in the genomes of a hyperdiverse lineage of mushroom-forming fungi. New Phytologist 233: 2294-2309.
Luo S, Phillips RP, Jo I, Fei S, Liang J, Schmid B, Eisenhauer N. 2023. Higher productivity in forests with mixed mycorrhizal strategies. Nature Communications 14: 1377.
Luo Y-H, Ma L-L, Seibold S, Cadotte MW, Burgess KS, Tan S-L, Ye L-J, Zheng W, Zou J-Y, Chen Z-F et al. 2023. The diversity of mycorrhiza-associated fungi and trees shapes subtropical mountain forest ecosystem functioning. Journal of Biogeography 50: 715-729.
Lutz S, Bodenhausen N, Hess J, Valzano-Held A, Waelchli J, Deslandes-Hérold G, Schlaeppi K, van der Heijden MGA. 2023. Soil microbiome indicators can predict crop growth response to large-scale inoculation with arbuscular mycorrhizal fungi. Nature Microbiology 8: 2277-2289.
Mahmood S, Fahad Z, Bolou-Bi EB, King K, Köhler SJ, Bishop K, Ekblad A, Finlay RD. 2023. Ectomycorrhizal fungi integrate nitrogen mobilisation and mineral weathering in boreal forest soil. New Phytologist. doi: 10.1111/nph. 19260.
Lutz S, Bodenhausen N, Hess J, Valzano-Held A, Waelchli J, Deslandes-Hérold G, Schlaeppi K, van der Heijden MGA. 2023. Soil microbiome indicators can predict crop growth response to large-scale inoculation with arbuscular mycorrhizal fungi. Nature Microbiology 8: 2277-2289.
Mahmood S, Fahad Z, Bolou-Bi EB, King K, Köhler SJ, Bishop K, Ekblad A, Finlay RD. 2023. Ectomycorrhizal fungi integrate nitrogen mobilisation and mineral weathering in boreal forest soil. New Phytologist. doi: 10.1111/nph. 19260.
Maillard F, Kohler A, Morin E, Hossann C, Miyauchi S, Ziegler-Devin I, Gérant D, Angeli N, Lipzen A, Keymanesh K et al. 2023. Functional genomics gives new insights into the ectomycorrhizal degradation of chitin. New Phytologist 238: 845858.
Malar CM, Krüger M, Krüger C, Wang Y, Stajich JE, Keller J, Chen ECH, Yildirir G, Villeneuve-Laroche M, Roux C et al. 2021. The genome of Geosiphon pyriformis reveals ancestral traits linked to the emergence of the arbuscular mycorrhizal symbiosis. Current Biology 31: 1570-1577.
Malar CM, Wang Y, Stajich JE, Kokkoris V, Villeneuve-Laroche M, Yildirir G, Corradi N. 2022. Early branching arbuscular mycorrhizal fungus Paraglomus occultum carries a small and repeat-poor genome compared to relatives in the Glomeromycotina. Microbial Genomics 8: 810.
Marqués-Gálvez JE, Miyauchi S, Paolocci F, Navarro-Ródenas A, Arenas F, PérezGilabert M, Morin E, Auer L, Barry KW, Kuo A et al. 2021. Desert truffle genomes reveal their reproductive modes and new insights into plant-fungal interaction and ectendomycorrhizal lifestyle. New Phytologist 229: 2917-2932.
Martin F, Aerts A, Ahrén D, Brun A, Danchin EGJ, Duchaussoy F, Gibon J, Kohler A, Lindquist E, Pereda V et al. 2008. The genome of Laccaria bicolor provides insights into mycorrhizal symbiosis. Nature 452: 88-92.
Martin F, Cullen D, Hibbett D, Pisabarro A, Spatafora JW, Baker SE, Grigoriev IV. 2011. Sequencing the fungal tree of life. New Phytologist 190: 818-821.
Malar CM, Wang Y, Stajich JE, Kokkoris V, Villeneuve-Laroche M, Yildirir G, Corradi N. 2022. Early branching arbuscular mycorrhizal fungus Paraglomus occultum carries a small and repeat-poor genome compared to relatives in the Glomeromycotina. Microbial Genomics 8: 810.
Marqués-Gálvez JE, Miyauchi S, Paolocci F, Navarro-Ródenas A, Arenas F, PérezGilabert M, Morin E, Auer L, Barry KW, Kuo A et al. 2021. Desert truffle genomes reveal their reproductive modes and new insights into plant-fungal interaction and ectendomycorrhizal lifestyle. New Phytologist 229: 2917-2932.
Martin F, Aerts A, Ahrén D, Brun A, Danchin EGJ, Duchaussoy F, Gibon J, Kohler A, Lindquist E, Pereda V et al. 2008. The genome of Laccaria bicolor provides insights into mycorrhizal symbiosis. Nature 452: 88-92.
Martin F, Cullen D, Hibbett D, Pisabarro A, Spatafora JW, Baker SE, Grigoriev IV. 2011. Sequencing the fungal tree of life. New Phytologist 190: 818-821.
Martin F, Kohler A, Murat C, Balestrini R, Coutinho PM, Jaillon O, Montanini B, Morin E, Noel B, Percudani R et al. 2010. Périgord black truffle genome uncovers evolutionary origins and mechanisms of symbiosis. Nature 464: 10331038.
Martin F, Kohler A, Murat C, Veneault-Fourrey C, Hibbett DS. 2016. Unearthing the roots of ectomycorrhizal symbioses. Nature Reviews Microbiology 14: 760773.
Martin F, Selosse MA. 2008. The Laccaria genome: a symbiont blueprint decoded. New Phytologist 180: 296-310.
Martin F, Uroz S, Barker D. 2017. Ancestral alliances: plant mutualistic symbioses with fungi and bacteria. Science 356: 819.
Martino E, Morin E, Grelet GA, Kuo A, Kohler A, Daghino S, Barry KW, Cichocki N, Clum A, Dockter RB et al. 2018. Comparative genomics and transcriptomics depict ericoid mycorrhizal fungi as versatile saprotrophs and plant mutualists. New Phytologist 217: 1213-1229.
Martín-Robles N, Lehmann A, Seco E, Aroca R, Rillig MC, Milla R. 2018. Impacts of domestication on the arbuscular mycorrhizal symbiosis of 27 crop species. New Phytologist 218: 322-334.
Mansfield TM, Albornoz FE, Ryan MH, Bending GD, Standish RJ. 2023. Niche differentiation of Mucoromycotinian and Glomeromycotinian arbuscular mycorrhizal fungi along a 2-million-year soil chronosequence. Mycorrhiza 33: 1-14.
Mateus ID, Masclaux FG, Aletti C, Rojas EC, Savary R, Dupuis C, Sanders IR. 2019. Dual RNA-seq reveals large-scale non-conserved genotype
Merckx V. 2023. Mycoheterotrophy. The biology of plants living on fungi. New York, NY, USA: Springer Science & Business Media, 356.
Miyauchi S, Kiss E, Kuo A, Drula E, Kohler A, Sánchez-García M, Morin E, Andreopoulos B, Barry KW, Bonito G et al. 2020. Large-scale genome sequencing of mycorrhizal fungi provides insights into the early evolution of symbiotic traits. Nature Communications 11: 5125.
Morin E, Miyauchi S, San Clemente H, Chen ECH, Pelin A, de la Providencia I, Ndikumana S, Beaudet D, Hainaut M, Drula E et al. 2019. Comparative
genomics of Rhizophagus irregularis, R. cerebriforme, R. diaphanus and Gigaspora rosea highlights specific genetic features in Glomeromycotina. New Phytologist 222: 1584-1598.
Murat C, Payen T, Noel B, Kuo A, Morin E, Chen J, Kohler A, Krizsán K, Balestrini R, Da Silva C et al. 2018. Pezizomycetes genomes reveal the molecular basis of ectomycorrhizal truffle lifestyle. Nature Ecology & Evolution 2: 19561965.
Martin F, Uroz S, Barker D. 2017. Ancestral alliances: plant mutualistic symbioses with fungi and bacteria. Science 356: 819.
Martino E, Morin E, Grelet GA, Kuo A, Kohler A, Daghino S, Barry KW, Cichocki N, Clum A, Dockter RB et al. 2018. Comparative genomics and transcriptomics depict ericoid mycorrhizal fungi as versatile saprotrophs and plant mutualists. New Phytologist 217: 1213-1229.
Martín-Robles N, Lehmann A, Seco E, Aroca R, Rillig MC, Milla R. 2018. Impacts of domestication on the arbuscular mycorrhizal symbiosis of 27 crop species. New Phytologist 218: 322-334.
Mansfield TM, Albornoz FE, Ryan MH, Bending GD, Standish RJ. 2023. Niche differentiation of Mucoromycotinian and Glomeromycotinian arbuscular mycorrhizal fungi along a 2-million-year soil chronosequence. Mycorrhiza 33: 1-14.
Mateus ID, Masclaux FG, Aletti C, Rojas EC, Savary R, Dupuis C, Sanders IR. 2019. Dual RNA-seq reveals large-scale non-conserved genotype
Merckx V. 2023. Mycoheterotrophy. The biology of plants living on fungi. New York, NY, USA: Springer Science & Business Media, 356.
Miyauchi S, Kiss E, Kuo A, Drula E, Kohler A, Sánchez-García M, Morin E, Andreopoulos B, Barry KW, Bonito G et al. 2020. Large-scale genome sequencing of mycorrhizal fungi provides insights into the early evolution of symbiotic traits. Nature Communications 11: 5125.
Morin E, Miyauchi S, San Clemente H, Chen ECH, Pelin A, de la Providencia I, Ndikumana S, Beaudet D, Hainaut M, Drula E et al. 2019. Comparative
genomics of Rhizophagus irregularis, R. cerebriforme, R. diaphanus and Gigaspora rosea highlights specific genetic features in Glomeromycotina. New Phytologist 222: 1584-1598.
Murat C, Payen T, Noel B, Kuo A, Morin E, Chen J, Kohler A, Krizsán K, Balestrini R, Da Silva C et al. 2018. Pezizomycetes genomes reveal the molecular basis of ectomycorrhizal truffle lifestyle. Nature Ecology & Evolution 2: 19561965.
Murat C, Rubini A, Riccioni C, De la Varga H, Akroume E, Belfiori B, Guaragno M, Le Tacon F, Robin C, Halkett F et al. 2013. Fine-scale spatial genetic structure of the black truffle (Tuber melanosporum) investigated with neutral microsatellites and functional mating type genes. New Phytologist 199: 176-187.
Nehls U, Plassard C. 2018. Nitrogen and phosphate metabolism in ectomycorrhizas. New Phytologist 220: 1047-1058.
Newman EI, Reddell P. 1988. Relationship between mycorrhizal infection and diversity in vegetation: evidence from the Great Smoky Mountains. Functional Ecology 2: 259-262.
Nicolás C, Martin-Bertelsen T, Floudas D, Bentzer J, Smits M, Johansson T, Troein C, Persson P, Tunlid A. 2019. The soil organic matter decomposition mechanisms in ectomycorrhizal fungi are tuned for liberating soil organic nitrogen. ISME Journal 13: 977-988.
Oliveira RS, Rocha I, Ma Y, Vosátka M, Freitas H. 2019. Seed coating with arbuscular mycorrhizal fungi for improved field production of chickpea. Agronomy 9: 471.
Oliverio AM, Geisen S, Delgado-Baquerizo M, Maestre FT, Turner BL, Fierer N. 2020. The global-scale distributions of soil protists and their contributions to belowground systems. Science. Advances 6: eaax8787.
Op De Beeck M, Troein C, Peterson C, Persson P, Tunlid A. 2018. Fenton reaction facilitates organic nitrogen acquisition by an ectomycorrhizal fungus. New Phytologist 218: 335-343.
Öpik M, Vanatoa A, Vanatoa E, Moora M, Davison J, Kalwij JM, Zobel M. 2010. The online database MaarjAM reveals global and ecosystemic distribution patterns in arbuscular mycorrhizal fungi Glomeromycota. New Phytologist 188: 223-241.
Paoletti M. 2016. Vegetative incompatibility in fungi: from recognition to cell death, whatever does the trick. Fungal Biology Reviews 30: 152-162.
Peay KG, Kennedy PG, Talbot JM. 2016. Dimensions of biodiversity in the earth mycobiome. Nature Reviews Microbiology 14: 434-447.
Pellegrin C, Morin E, Martin F, Veneault-Fourrey C. 2015. Comparative analysis of secretomes from ectomycorrhizal fungi with an emphasis on small-secreted proteins. Frontiers in Microbiology 6: e1278.
Pellitier PT, Zak DR. 2018. Ectomycorrhizal fungi and the enzymatic liberation of nitrogen from soil organic matter: why evolutionary history matters. New Phytologist 217: 68-73.
Perotto S, Balestrini R. 2023. At the core of the endomycorrhizal symbioses: intracellular fungal structures in orchid and arbuscular mycorrhiza. New Phytologist. doi: 10.1111/nph. 19338.
Peter M, Kohler A, Ohm RA, Kuo A, Krützmann J, Morin E, Arend M, Barry KW, Binder M, Choi C et al. 2016. Ectomycorrhizal ecology is imprinted in the genome of the dominant symbiotic fungus Cenococcum geophilum. Nature Communications 7: 12662.
Phillips HR, Guerra CA, Bartz ML, Briones MJ, Brown G, Crowther TW, Orgiazzi A. 2019. Global distribution of earthworm diversity. Science 366: 480485.
Nehls U, Plassard C. 2018. Nitrogen and phosphate metabolism in ectomycorrhizas. New Phytologist 220: 1047-1058.
Newman EI, Reddell P. 1988. Relationship between mycorrhizal infection and diversity in vegetation: evidence from the Great Smoky Mountains. Functional Ecology 2: 259-262.
Nicolás C, Martin-Bertelsen T, Floudas D, Bentzer J, Smits M, Johansson T, Troein C, Persson P, Tunlid A. 2019. The soil organic matter decomposition mechanisms in ectomycorrhizal fungi are tuned for liberating soil organic nitrogen. ISME Journal 13: 977-988.
Oliveira RS, Rocha I, Ma Y, Vosátka M, Freitas H. 2019. Seed coating with arbuscular mycorrhizal fungi for improved field production of chickpea. Agronomy 9: 471.
Oliverio AM, Geisen S, Delgado-Baquerizo M, Maestre FT, Turner BL, Fierer N. 2020. The global-scale distributions of soil protists and their contributions to belowground systems. Science. Advances 6: eaax8787.
Op De Beeck M, Troein C, Peterson C, Persson P, Tunlid A. 2018. Fenton reaction facilitates organic nitrogen acquisition by an ectomycorrhizal fungus. New Phytologist 218: 335-343.
Öpik M, Vanatoa A, Vanatoa E, Moora M, Davison J, Kalwij JM, Zobel M. 2010. The online database MaarjAM reveals global and ecosystemic distribution patterns in arbuscular mycorrhizal fungi Glomeromycota. New Phytologist 188: 223-241.
Paoletti M. 2016. Vegetative incompatibility in fungi: from recognition to cell death, whatever does the trick. Fungal Biology Reviews 30: 152-162.
Peay KG, Kennedy PG, Talbot JM. 2016. Dimensions of biodiversity in the earth mycobiome. Nature Reviews Microbiology 14: 434-447.
Pellegrin C, Morin E, Martin F, Veneault-Fourrey C. 2015. Comparative analysis of secretomes from ectomycorrhizal fungi with an emphasis on small-secreted proteins. Frontiers in Microbiology 6: e1278.
Pellitier PT, Zak DR. 2018. Ectomycorrhizal fungi and the enzymatic liberation of nitrogen from soil organic matter: why evolutionary history matters. New Phytologist 217: 68-73.
Perotto S, Balestrini R. 2023. At the core of the endomycorrhizal symbioses: intracellular fungal structures in orchid and arbuscular mycorrhiza. New Phytologist. doi: 10.1111/nph. 19338.
Peter M, Kohler A, Ohm RA, Kuo A, Krützmann J, Morin E, Arend M, Barry KW, Binder M, Choi C et al. 2016. Ectomycorrhizal ecology is imprinted in the genome of the dominant symbiotic fungus Cenococcum geophilum. Nature Communications 7: 12662.
Phillips HR, Guerra CA, Bartz ML, Briones MJ, Brown G, Crowther TW, Orgiazzi A. 2019. Global distribution of earthworm diversity. Science 366: 480485.
Plett JM, Daguerre Y, Wittulsky S, Vayssieres A, Deveau A, Melton SJ, Kohler A, Morrel-Falvey JL, Brun A, Veneault-Fourrey C et al. 2014. Effector MiSSP7 of the mutualistic fungus Laccaria bicolor stabilizes the Populus JAZ6 protein and represses jasmonic acid-responsive genes. Proceedings of the National Academy of Sciences, USA 111: 8299-8304.
Plett JM, Kemppainen M, Kale SD, Kohler A, Legué V, Brun A, Tyler B, Pardo A, Martin F. 2011. A secreted effector protein of Laccaria bicolor is required for symbiosis development. Current Biology 21: 1197-1203.
Plett JM, Martin F. 2011. Blurred boundaries: lifestyle lessons from ectomycorrhizal fungal genomes. Trends in Genetics 27: 14-22.
Plett JM, Miyauchi S, Morin E, Plett K, Wong-Bajracharya J, de Freitas PM, Kuo A, Henrissat B, Drula E, Wojtalewicz D et al. 2023. Speciation underpinned by
unexpected molecular diversity in the mycorrhizal fungal genus Pisolithus. Molecular Biology and Evolution 40: msad045.
Plett KL, Kohler A, Lebel T, Singan VR, Bauer D, He G, Ng V, Grigoriev IV, Martin F, Plett JM et al. 2021. Intra-species genetic variability drives carbon metabolism and symbiotic host interactions in the ectomycorrhizal fungus Pisolithus microcarpus. Environmental Microbiology 23: 2004-2020.
Plett KL, Raposo AE, Anderson IC, Piller SC, Plett JM. 2019. Protein arginine methyltransferase expression affects ectomycorrhizal symbiosis and the regulation of hormone signaling pathways. Molecular Plant-Microbe Interactions 32: 12911302.
Plett JM, Kemppainen M, Kale SD, Kohler A, Legué V, Brun A, Tyler B, Pardo A, Martin F. 2011. A secreted effector protein of Laccaria bicolor is required for symbiosis development. Current Biology 21: 1197-1203.
Plett JM, Martin F. 2011. Blurred boundaries: lifestyle lessons from ectomycorrhizal fungal genomes. Trends in Genetics 27: 14-22.
Plett JM, Miyauchi S, Morin E, Plett K, Wong-Bajracharya J, de Freitas PM, Kuo A, Henrissat B, Drula E, Wojtalewicz D et al. 2023. Speciation underpinned by
unexpected molecular diversity in the mycorrhizal fungal genus Pisolithus. Molecular Biology and Evolution 40: msad045.
Plett KL, Kohler A, Lebel T, Singan VR, Bauer D, He G, Ng V, Grigoriev IV, Martin F, Plett JM et al. 2021. Intra-species genetic variability drives carbon metabolism and symbiotic host interactions in the ectomycorrhizal fungus Pisolithus microcarpus. Environmental Microbiology 23: 2004-2020.
Plett KL, Raposo AE, Anderson IC, Piller SC, Plett JM. 2019. Protein arginine methyltransferase expression affects ectomycorrhizal symbiosis and the regulation of hormone signaling pathways. Molecular Plant-Microbe Interactions 32: 12911302.
Plett KL, Wojtalewicz D, Anderson IC, Plett JM. 2023. Fungal metabolism and free amino acid content may predict nitrogen transfer to the host plant in the ectomycorrhizal relationship between Pisolithus spp. and Eucalyptus grandis. New Phytologist. doi: 10.1111/nph.19400.
Pringle A, Bever JD, Gardes M, Parrent JL, Rillig MC, Klironomos JN. 2009. Mycorrhizal symbioses and plant invasions. Annual Review of Ecology, Evolution, and Systematics 40: 699-715.
Querejeta JI, Schlaeppi K, López-García Á, Ondoño S, Prieto I, van Der Heijden MG, del Mar Alguacil M. 2021. Lower relative abundance of ectomycorrhizal fungi under a warmer and drier climate is linked to enhanced soil organic matter decomposition. New Phytologist 232: 1399-1413.
Rasmussen HN, Rasmussen FN. 2014. Seedling mycorrhiza: a discussion of origin and evolution in Orchidaceae. Botanical Journal of the Linnean Society 175: 313327.
Pringle A, Bever JD, Gardes M, Parrent JL, Rillig MC, Klironomos JN. 2009. Mycorrhizal symbioses and plant invasions. Annual Review of Ecology, Evolution, and Systematics 40: 699-715.
Querejeta JI, Schlaeppi K, López-García Á, Ondoño S, Prieto I, van Der Heijden MG, del Mar Alguacil M. 2021. Lower relative abundance of ectomycorrhizal fungi under a warmer and drier climate is linked to enhanced soil organic matter decomposition. New Phytologist 232: 1399-1413.
Rasmussen HN, Rasmussen FN. 2014. Seedling mycorrhiza: a discussion of origin and evolution in Orchidaceae. Botanical Journal of the Linnean Society 175: 313327.
Read DJ. 1991. Mycorrhizas in ecosystems. Experientia 47: 376-391.
Reinhardt D, Roux C, Corradi N, Di Pietro A. 2021. Lineage-specific genes and cryptic sex: parallels and differences between arbuscular mycorrhizal fungi and fungal pathogens. Trends in Plant Science 26: 111-123.
Rillig MC, Aguilar-Trigueros CA, Camenzind T, Cavagnaro TR, Degrune F, Hohmann P, Lammel DR, Mansour I, Roy J, van der Heijden MGA et al. 2019a. Why farmers should manage the arbuscular mycorrhizal symbiosis. New Phytologist 222: 1171-1175.
Rillig MC, Ryo M, Lehmann A, Aguilar-Trigueros CA, Buchert S, Wulf A, Yang G, Roy J, Yang G. 2019b. The role of multiple global change factors in driving soil functions and microbial biodiversity. Science 366: 886-890.
Rillig MC, van der Heijden M, Berdugo M, Liu YR, Riedo J, Sanz-Lazaro C, Delgado-Baquerizo M. 2023. Increasing the number of stressors reduces soil ecosystem services worldwide. Nature Climate Change 13: 478-483.
Rivera Pérez CA, Janz D, Schneider D, Daniel R, Polle A. 2022. Transcriptional landscape of ectomycorrhizal fungi and their host provides insight into N uptake from forest soil. mSystems 7: e0095721.
Robinson D, Fitter A. 1999. The magnitude and control of carbon transfer between plants linked by a common mycorrhizal network. Journal of Experimental Botany 50: 9-13.
Robinson DG, Ammer C, Polle A, Bauhus J, Aloni R, Annighöfer P, Baskin TI, Blatt MR, Bolte A, Bugmann H et al. 2024. Mother trees, altruistic fungi, and the perils of plant personification. Trends in Plant Sciences 29: 20-31.
Rocha I, Ma Y, Souza-Alonso P, Vosátka M, Freitas H, Oliveira RS. 2019. Seed coating: a tool for delivering beneficial microbes to agricultural crops. Frontiers in Plant Science 10: 1357.
Rog I, Rosenstock NP, Körner C, Klein T. 2020. Share the wealth: trees with greater ectomycorrhizal species overlap share more carbon. Molecular Ecology 29: 23212333.
Reinhardt D, Roux C, Corradi N, Di Pietro A. 2021. Lineage-specific genes and cryptic sex: parallels and differences between arbuscular mycorrhizal fungi and fungal pathogens. Trends in Plant Science 26: 111-123.
Rillig MC, Aguilar-Trigueros CA, Camenzind T, Cavagnaro TR, Degrune F, Hohmann P, Lammel DR, Mansour I, Roy J, van der Heijden MGA et al. 2019a. Why farmers should manage the arbuscular mycorrhizal symbiosis. New Phytologist 222: 1171-1175.
Rillig MC, Ryo M, Lehmann A, Aguilar-Trigueros CA, Buchert S, Wulf A, Yang G, Roy J, Yang G. 2019b. The role of multiple global change factors in driving soil functions and microbial biodiversity. Science 366: 886-890.
Rillig MC, van der Heijden M, Berdugo M, Liu YR, Riedo J, Sanz-Lazaro C, Delgado-Baquerizo M. 2023. Increasing the number of stressors reduces soil ecosystem services worldwide. Nature Climate Change 13: 478-483.
Rivera Pérez CA, Janz D, Schneider D, Daniel R, Polle A. 2022. Transcriptional landscape of ectomycorrhizal fungi and their host provides insight into N uptake from forest soil. mSystems 7: e0095721.
Robinson D, Fitter A. 1999. The magnitude and control of carbon transfer between plants linked by a common mycorrhizal network. Journal of Experimental Botany 50: 9-13.
Robinson DG, Ammer C, Polle A, Bauhus J, Aloni R, Annighöfer P, Baskin TI, Blatt MR, Bolte A, Bugmann H et al. 2024. Mother trees, altruistic fungi, and the perils of plant personification. Trends in Plant Sciences 29: 20-31.
Rocha I, Ma Y, Souza-Alonso P, Vosátka M, Freitas H, Oliveira RS. 2019. Seed coating: a tool for delivering beneficial microbes to agricultural crops. Frontiers in Plant Science 10: 1357.
Rog I, Rosenstock NP, Körner C, Klein T. 2020. Share the wealth: trees with greater ectomycorrhizal species overlap share more carbon. Molecular Ecology 29: 23212333.
Ropars J, Toro KS, Noel J, Pelin A, Charron P, Farinelli L, Marton T, Krüger M, Fuchs J, Brachmann A et al. 2016. Evidence for the sexual origin of heterokaryosis in arbuscular mycorrhizal fungi. Nature Microbiology 1: 16033.
Rozmoš M, Bukovská P, Hršelová H, Kotianová M, Dudáš M, Gančarčíková K, Jansa J. 2022. Organic nitrogen utilisation by an arbuscular mycorrhizal fungus is mediated by specific soil bacteria and a protist. ISME Journal 16: 676-685.
Ruytinx J, Miyauchi S, Hartmann-Wittulsky S, de Freitas PM, Guinet F, Churin JL, Put C, Le Tacon F, Veneault-Fourrey C, Martin F et al. 2021. A transcriptomic atlas of the ectomycorrhizal fungus Laccaria bicolor. Microorganisms 9: 2612.
Ryan MH, Graham JH. 2018. Little evidence that farmers should consider abundance or diversity of arbuscular mycorrhizal fungi when managing crops. New Phytologist 220: 1092-1107.
Rozmoš M, Bukovská P, Hršelová H, Kotianová M, Dudáš M, Gančarčíková K, Jansa J. 2022. Organic nitrogen utilisation by an arbuscular mycorrhizal fungus is mediated by specific soil bacteria and a protist. ISME Journal 16: 676-685.
Ruytinx J, Miyauchi S, Hartmann-Wittulsky S, de Freitas PM, Guinet F, Churin JL, Put C, Le Tacon F, Veneault-Fourrey C, Martin F et al. 2021. A transcriptomic atlas of the ectomycorrhizal fungus Laccaria bicolor. Microorganisms 9: 2612.
Ryan MH, Graham JH. 2018. Little evidence that farmers should consider abundance or diversity of arbuscular mycorrhizal fungi when managing crops. New Phytologist 220: 1092-1107.
Saito K, Ezawa T. 2016. Phosphorus metabolism and transport in arbuscular mycorrhizal symbiosis. Molecular mycorrhizal symbiosis: 197-216.
Säle V, Palenzuela J, Azcón-Aguilar C, Sánchez-Castro I, da Silva GA, Seitz B, Sieverding E, van der Heijden MGA, Oehl F. 2021. Ancient lineages of arbuscular mycorrhizal fungi provide little plant benefit. Mycorrhiza 31: 559576.
Säle V, Palenzuela J, Azcón-Aguilar C, Sánchez-Castro I, da Silva GA, Seitz B, Sieverding E, van der Heijden MGA, Oehl F. 2021. Ancient lineages of arbuscular mycorrhizal fungi provide little plant benefit. Mycorrhiza 31: 559576.
Salomon MJ, Watts-Williams SJ, McLaughlin MJ, Bücking H, Singh BK, Hutter I, Schneider C, Martin FM, Vosatka M, Guo L et al. 2022. Establishing a quality management framework for commercial inoculants containing arbuscular mycorrhizal fungi. iScience 25: 7.
Sato H. 2023. The evolution of ectomycorrhizal symbiosis in the Late Cretaceous is a key driver of explosive diversification in Agaricomycetes. New Phytologist 241: 444460.
Sato H. 2023. The evolution of ectomycorrhizal symbiosis in the Late Cretaceous is a key driver of explosive diversification in Agaricomycetes. New Phytologist 241: 444460.
Schnitzer SA, Klironomos JN, HilleRisLambers J, Kinkel LL, Reich PB, Xiao K, Rillig MC, Sikes B, Callaway RM, Mangan SA et al. 2011. Soil microbes drive the classic plant diversity-productivity pattern. Ecology 92: 296-303.
Schwery O, Onstein RE, Bouchenak-Khelladi Y, Xing Y, Carter RJ, Linder HP. 2015. As old as the mountains: the radiations of the Ericaceae. New Phytologist 207: 355-367.
Selosse MA, Bocayuva MF, Kasuya MCM, Courty PE. 2016. Mixotrophy in mycorrhizal plants: extracting carbon from mycorrhizal networks. Molecular mycorrhizal symbiosis: 451-471.
Selosse MA, Le Tacon F. 1998. The land flora: a phototroph-fungus partnership? Trends in Ecology & Evolution 13: 15-20.
Serrano K, Bezrutczyk M, Goudeau D, Dao T, O’Malley R, Malmstrom RR, Visel A, Scheller H, Cole B. 2023. Spatial co-transcriptomics reveals discrete stages of the arbuscular mycorrhizal symbiosis. BioRxiv. doi: 10.1101/2023.08.02. 551648.
Schwery O, Onstein RE, Bouchenak-Khelladi Y, Xing Y, Carter RJ, Linder HP. 2015. As old as the mountains: the radiations of the Ericaceae. New Phytologist 207: 355-367.
Selosse MA, Bocayuva MF, Kasuya MCM, Courty PE. 2016. Mixotrophy in mycorrhizal plants: extracting carbon from mycorrhizal networks. Molecular mycorrhizal symbiosis: 451-471.
Selosse MA, Le Tacon F. 1998. The land flora: a phototroph-fungus partnership? Trends in Ecology & Evolution 13: 15-20.
Serrano K, Bezrutczyk M, Goudeau D, Dao T, O’Malley R, Malmstrom RR, Visel A, Scheller H, Cole B. 2023. Spatial co-transcriptomics reveals discrete stages of the arbuscular mycorrhizal symbiosis. BioRxiv. doi: 10.1101/2023.08.02. 551648.
Silverstein MR, Segrè D, Bhatnagar JM. 2023. Environmental microbiome engineering for the mitigation of climate change. Global Change Biology 29: 20502066.
Simard SW, Perry DA, Jones MD, Myrold DD, Durall DM, Molina R. 1997. Net transfer of carbon between ectomycorrhizal tree species in the field. Nature 388: 579-582.
Simon L, Bousquet J, Lévesque R, Lalonde M. 1993. Origin and diversification of endomycorrhizal fungi and coincidence with vascular land plants. Nature 363: 67-69.
Song C, Wang Y, Manzoor MA, Mao D, Wei P, Cao Y, Zhu F. 2022. In-depth analysis of genomes and functional genomics of orchid using cutting-edge highthroughput sequencing. Frontiers in Plant Science 13: 1018029.
Soudzilovskaia NA, van Bodegom PM, Terrer C, Zelfde MVT, McCallum I, Luke McCormack M, Tedersoo L. 2019. Global mycorrhizal plant distribution linked to terrestrial carbon stocks. Nature Communications 10: 5077.
Spatafora JW, Chang Y, Benny GL, Lazarus K, Smith ME, Berbee ML, Bonito G, Corradi N, Grigoriev I, Gryganskyi A et al. 2016. A phylum-level phylogenetic classification of zygomycete fungi based on genome-scale data. Mycologia 108: 1028-1046.
Sperschneider J, Yildirir G, Rizzi Y, Malar MC, Mayrand A, Sorwar E, Chen ECH, Iwasaki W, Brauer EK, Bosnich W et al. 2023. Arbuscular mycorrhizal fungi heterokaryons have two nuclear populations with distinct roles in host-plant interactions. Nature Microbiology 8: 2142-2153.
Strassert JF, Monaghan MT. 2022. Phylogenomic insights into the early diversification of fungi. Current Biology 32: 3628-3635.
Strullu-Derrien C, Selosse M-A, Kenrick P, Martin FM. 2018. The origin and evolution of mycorrhizal symbioses: from palaeomycology to phylogenomics. New Phytologist 220: 1012-1030.
Stuart EK, Singan V, Amirebrahimi M, Na H, Ng V, Grigoriev I, Martin F, Anderson IC, Plett JM, Plett KL. 2023. Acquisition of host-derived carbon in biomass of the ectomycorrhizal fungus Pisolithus microcarpus is correlated to fungal carbon demand and plant defences. FEMS Microbiology Ecology 99: fiad037.
Svenningsen NB, Watts-Williams SJ, Joner EJ, Battini F, Efthymiou A, Cruz-Paredes C, Nybroe O, Jakobsen I. 2018. Suppression of the activity of arbuscular mycorrhizal fungi by the soil microbiota. ISME Journal 12: 1296-1307.
Tagu D, Bastien C, Faivre-Rampant P, Garbaye J, Vion P, Villar M, Martin F. 2005. Genetic analysis of phenotypic variation for ectomycorrhiza
formation in an interspecific F1 poplar full-sib family. Mycorrhiza 15: 87-91.
Tang N, Lebreton A, Xu W, Dai Y-C, Yu F, Martin FM. 2021. Transcriptome profiling reveals differential gene expression of secreted proteases and highly specific gene repertoires involved in Lactarius-Pinus symbioses. Frontiers in Plant Science 12: 1775.
Tedersoo L, Bahram M, Põlme S, Kõljalg U, Yorou NS, Wijesundera R, Villarreal Ruiz L, Vasco-Palacios AM, Thu PQ, Suija A et al. 2014. Global diversity and geography of soil fungi. Science 346: 6213.
Tedersoo L, May TW, Smith ME. 2010. Ectomycorrhizal lifestyle in fungi: global diversity, distribution, and evolution of phylogenetic lineages. Mycorrhiza 20: 217-263.
Teulet A, Quan C, Evangelisti E, Wanke A, Yang W, Schornack S. 2023. A pathogen effector FOLD diversified in symbiotic fungi. New Phytologist 239: 1127-1139.
Thirkell TJ, Grimmer M, James L, Pastok D, Allary T, Elliott A, Paveley N, Daniell T, Field KJ. 2022. Variation in mycorrhizal growth response among a spring wheat mapping population shows potential to breed for symbiotic benefit. Food and Energy Security 11: e370.
Tisserant E, Malbreil M, Kuo A, Kohler A, Symeonidi A, Balestrini R, Charron P, Duensing N, Frei Dit Frey N, Gianinazzi-Pearson V et al. 2013. Genome of an arbuscular mycorrhizal fungus provides insight into the oldest plant symbiosis. Proceedings of the National Academy of Sciences, USA 110: 20117-20122.
Trivedi P, Batista BD, Bazany KE, Singh BK. 2022. Plant-microbiome interactions under a changing world: responses, consequences and perspectives. New Phytologist 234: 1951-1959.
Tsuzuki S, Handa Y, Takeda N, Kawaguchi M. 2016. Strigolactone-induced putative secreted protein 1 is required for the establishment of symbiosis by the arbuscular mycorrhizal fungus Rhizophagus irregularis. Molecular Plant-Microbe Interactions 29: 277-286.
Vandenkoornhuyse P, Quaiser A, Duhamel M, Le Van A, Dufresne A. 2015. The importance of the microbiome of the plant holobiont. New Phytologist 206: 11961206.
Simon L, Bousquet J, Lévesque R, Lalonde M. 1993. Origin and diversification of endomycorrhizal fungi and coincidence with vascular land plants. Nature 363: 67-69.
Song C, Wang Y, Manzoor MA, Mao D, Wei P, Cao Y, Zhu F. 2022. In-depth analysis of genomes and functional genomics of orchid using cutting-edge highthroughput sequencing. Frontiers in Plant Science 13: 1018029.
Soudzilovskaia NA, van Bodegom PM, Terrer C, Zelfde MVT, McCallum I, Luke McCormack M, Tedersoo L. 2019. Global mycorrhizal plant distribution linked to terrestrial carbon stocks. Nature Communications 10: 5077.
Spatafora JW, Chang Y, Benny GL, Lazarus K, Smith ME, Berbee ML, Bonito G, Corradi N, Grigoriev I, Gryganskyi A et al. 2016. A phylum-level phylogenetic classification of zygomycete fungi based on genome-scale data. Mycologia 108: 1028-1046.
Sperschneider J, Yildirir G, Rizzi Y, Malar MC, Mayrand A, Sorwar E, Chen ECH, Iwasaki W, Brauer EK, Bosnich W et al. 2023. Arbuscular mycorrhizal fungi heterokaryons have two nuclear populations with distinct roles in host-plant interactions. Nature Microbiology 8: 2142-2153.
Strassert JF, Monaghan MT. 2022. Phylogenomic insights into the early diversification of fungi. Current Biology 32: 3628-3635.
Strullu-Derrien C, Selosse M-A, Kenrick P, Martin FM. 2018. The origin and evolution of mycorrhizal symbioses: from palaeomycology to phylogenomics. New Phytologist 220: 1012-1030.
Stuart EK, Singan V, Amirebrahimi M, Na H, Ng V, Grigoriev I, Martin F, Anderson IC, Plett JM, Plett KL. 2023. Acquisition of host-derived carbon in biomass of the ectomycorrhizal fungus Pisolithus microcarpus is correlated to fungal carbon demand and plant defences. FEMS Microbiology Ecology 99: fiad037.
Svenningsen NB, Watts-Williams SJ, Joner EJ, Battini F, Efthymiou A, Cruz-Paredes C, Nybroe O, Jakobsen I. 2018. Suppression of the activity of arbuscular mycorrhizal fungi by the soil microbiota. ISME Journal 12: 1296-1307.
Tagu D, Bastien C, Faivre-Rampant P, Garbaye J, Vion P, Villar M, Martin F. 2005. Genetic analysis of phenotypic variation for ectomycorrhiza
formation in an interspecific F1 poplar full-sib family. Mycorrhiza 15: 87-91.
Tang N, Lebreton A, Xu W, Dai Y-C, Yu F, Martin FM. 2021. Transcriptome profiling reveals differential gene expression of secreted proteases and highly specific gene repertoires involved in Lactarius-Pinus symbioses. Frontiers in Plant Science 12: 1775.
Tedersoo L, Bahram M, Põlme S, Kõljalg U, Yorou NS, Wijesundera R, Villarreal Ruiz L, Vasco-Palacios AM, Thu PQ, Suija A et al. 2014. Global diversity and geography of soil fungi. Science 346: 6213.
Tedersoo L, May TW, Smith ME. 2010. Ectomycorrhizal lifestyle in fungi: global diversity, distribution, and evolution of phylogenetic lineages. Mycorrhiza 20: 217-263.
Teulet A, Quan C, Evangelisti E, Wanke A, Yang W, Schornack S. 2023. A pathogen effector FOLD diversified in symbiotic fungi. New Phytologist 239: 1127-1139.
Thirkell TJ, Grimmer M, James L, Pastok D, Allary T, Elliott A, Paveley N, Daniell T, Field KJ. 2022. Variation in mycorrhizal growth response among a spring wheat mapping population shows potential to breed for symbiotic benefit. Food and Energy Security 11: e370.
Tisserant E, Malbreil M, Kuo A, Kohler A, Symeonidi A, Balestrini R, Charron P, Duensing N, Frei Dit Frey N, Gianinazzi-Pearson V et al. 2013. Genome of an arbuscular mycorrhizal fungus provides insight into the oldest plant symbiosis. Proceedings of the National Academy of Sciences, USA 110: 20117-20122.
Trivedi P, Batista BD, Bazany KE, Singh BK. 2022. Plant-microbiome interactions under a changing world: responses, consequences and perspectives. New Phytologist 234: 1951-1959.
Tsuzuki S, Handa Y, Takeda N, Kawaguchi M. 2016. Strigolactone-induced putative secreted protein 1 is required for the establishment of symbiosis by the arbuscular mycorrhizal fungus Rhizophagus irregularis. Molecular Plant-Microbe Interactions 29: 277-286.
Vandenkoornhuyse P, Quaiser A, Duhamel M, Le Van A, Dufresne A. 2015. The importance of the microbiome of the plant holobiont. New Phytologist 206: 11961206.
Veneault-Fourrey C, Commun C, Kohler A, Morin E, Balestrini R, Plett J, Danchin E, Coutinho P, Wiebenga A, de Vries RP et al. 2014. Genomic and transcriptomic analysis of Laccaria bicolor CAZome reveals insights into polysaccharides remodeling during symbiosis establishment. Fungal Genetics and Biology 72: 168-181.
Venice F, Ghignone S, Salvioli A, Amselem J, Novero M, Xianan X, Sedzielewska Toro K, Morin E, Lipzen A, Grigoriev IV et al. 2020. At the nexus of three kingdoms: the genome of the mycorrhizal fungus Gigaspora margarita provides insights into plant, endobacterial and fungal interactions. Environmental Microbiology 22: 122-141.
Větrovský T, Morais D, Kohout P, Lepinay C, Algora C, Awokunle Hollá S, Bahnmann BD, Bílohnědá K, Brabcová V, D’Alò F et al. 2020. GlobalFungi, a global database of fungal occurrences from high-throughput-sequencing metabarcoding studies. Scientific Data 13: 228.
Vosátka M, Látr A, Gianinazzi S, Albrechtová J. 2012. Development of arbuscular mycorrhizal biotechnology and industry: current achievements and bottlenecks. Symbiosis 58: 29-37.
Voß S, Betz R, Heidt S, Corradi N, Requena N. 2018. RiCRN1, a crinkler effector from the arbuscular mycorrhizal fungus Rhizophagus irregularis, functions in arbuscule development. Frontiers in Microbiology 9: 2068.
Wang J, Hu A, Meng F, Zhao W, Yang Y, Soininen J, Shen J, Zhou J. 2022. Embracing mountain microbiome and ecosystem functions under global change. New Phytologist 234: 1987-2002.
Wang L, George TS, Feng G. 2023a. Concepts and consequences of the hyphosphere core microbiome for arbuscular mycorrhizal fungal fitness and function. New Phytologist. doi: 10.1111/nph. 19396.
Wang L, Zhang L, George TS, Feng G. 2023b. A core microbiome in the hyphosphere of arbuscular mycorrhizal fungi has functional significance in organic phosphorus mineralization. New Phytologist 238: 859-873.
Wang P, Jiang H, Boeren S, Dings H, Kulikova O, Bisseling T, Limpens EA. 2021. Nuclear-targeted effector of Rhizophagus irregularis interferes with histone 2B mono-ubiquitination to promote arbuscular mycorrhization. New Phytologist 230: 1142-1155.
Venice F, Ghignone S, Salvioli A, Amselem J, Novero M, Xianan X, Sedzielewska Toro K, Morin E, Lipzen A, Grigoriev IV et al. 2020. At the nexus of three kingdoms: the genome of the mycorrhizal fungus Gigaspora margarita provides insights into plant, endobacterial and fungal interactions. Environmental Microbiology 22: 122-141.
Větrovský T, Morais D, Kohout P, Lepinay C, Algora C, Awokunle Hollá S, Bahnmann BD, Bílohnědá K, Brabcová V, D’Alò F et al. 2020. GlobalFungi, a global database of fungal occurrences from high-throughput-sequencing metabarcoding studies. Scientific Data 13: 228.
Vosátka M, Látr A, Gianinazzi S, Albrechtová J. 2012. Development of arbuscular mycorrhizal biotechnology and industry: current achievements and bottlenecks. Symbiosis 58: 29-37.
Voß S, Betz R, Heidt S, Corradi N, Requena N. 2018. RiCRN1, a crinkler effector from the arbuscular mycorrhizal fungus Rhizophagus irregularis, functions in arbuscule development. Frontiers in Microbiology 9: 2068.
Wang J, Hu A, Meng F, Zhao W, Yang Y, Soininen J, Shen J, Zhou J. 2022. Embracing mountain microbiome and ecosystem functions under global change. New Phytologist 234: 1987-2002.
Wang L, George TS, Feng G. 2023a. Concepts and consequences of the hyphosphere core microbiome for arbuscular mycorrhizal fungal fitness and function. New Phytologist. doi: 10.1111/nph. 19396.
Wang L, Zhang L, George TS, Feng G. 2023b. A core microbiome in the hyphosphere of arbuscular mycorrhizal fungi has functional significance in organic phosphorus mineralization. New Phytologist 238: 859-873.
Wang P, Jiang H, Boeren S, Dings H, Kulikova O, Bisseling T, Limpens EA. 2021. Nuclear-targeted effector of Rhizophagus irregularis interferes with histone 2B mono-ubiquitination to promote arbuscular mycorrhization. New Phytologist 230: 1142-1155.
Wolfe BE,Tulloss RR,Pringle A.2012.The irreversible loss of a decomposition pathway marks the single origin of an ectomycorrhizal symbiosis.PLoS ONE 7: e39597.
Wong-Bajracharya J,Singan VR,Monti R,Plett KL,Ng V,Grigoriev IV,Martin FM,Anderson IC,Plett JM.2022.The ectomycorrhizal fungus Pisolithus microcarpus encodes a microRNA involved in cross-kingdom gene silencing during symbiosis.Proceedings of the National Academy of Sciences,USA 119: e2103527119.
Wu B,Nara K,Hogetsu T.2001.Can
Wu G,Miyauchi S,Morin E,Kuo A,Drula E,Varga T,Kohler A,Feng B,Cao Y, Lipzen A et al.2022.Evolutionary innovations through gain and loss of genes in the ectomycorrhizal Boletales.New Phytologist 233:1383-1400.
Wu S,Fu W,Rillig MC,Chen B,Zhu Y-G,Huang L.2023.Soil organic matter dynamics mediated by arbuscular mycorrhizal fungi-an updated conceptual framework.New Phytologist.doi:10.1111/nph. 19178.
Xu X,Thornton PE,Post WM.2013.A global analysis of soil microbial biomass carbon,nitrogen and phosphorus in terrestrial ecosystems.Global Ecology and Biogeography 22:737-749.
Yang FX,Gao J,Wei YL,Ren R,Zhang GQ,Lu CQ,Jin JP,Ai Y,Wang YQ,Chen LJ et al.2021.The genome of Cymbidium sinense revealed the evolution of orchid traits.Plant Biotechnology Journal 19:2501-2516.
Yang Z,Dong H,Zhang S,Jiang J,Zhu H,Yang H,Li L.2023.Isolation and identification of mycorrhizal helper bacteria of Vaccinium uliginosum and their interaction with mycorrhizal fungi.Frontiers in Microbiology 14: 1180319.
Yildirir G,Sperschneider J,Malar CM,Chen ECH,Iwasaki W,Cornell C,Corradi N.2022.Long reads and Hi-C sequencing illuminate the two-compartment genome of the model arbuscular mycorrhizal symbiont Rhizophagus irregularis. New Phytologist 233:1097-1107.
Wong-Bajracharya J,Singan VR,Monti R,Plett KL,Ng V,Grigoriev IV,Martin FM,Anderson IC,Plett JM.2022.The ectomycorrhizal fungus Pisolithus microcarpus encodes a microRNA involved in cross-kingdom gene silencing during symbiosis.Proceedings of the National Academy of Sciences,USA 119: e2103527119.
Wu B,Nara K,Hogetsu T.2001.Can
Wu G,Miyauchi S,Morin E,Kuo A,Drula E,Varga T,Kohler A,Feng B,Cao Y, Lipzen A et al.2022.Evolutionary innovations through gain and loss of genes in the ectomycorrhizal Boletales.New Phytologist 233:1383-1400.
Wu S,Fu W,Rillig MC,Chen B,Zhu Y-G,Huang L.2023.Soil organic matter dynamics mediated by arbuscular mycorrhizal fungi-an updated conceptual framework.New Phytologist.doi:10.1111/nph. 19178.
Xu X,Thornton PE,Post WM.2013.A global analysis of soil microbial biomass carbon,nitrogen and phosphorus in terrestrial ecosystems.Global Ecology and Biogeography 22:737-749.
Yang FX,Gao J,Wei YL,Ren R,Zhang GQ,Lu CQ,Jin JP,Ai Y,Wang YQ,Chen LJ et al.2021.The genome of Cymbidium sinense revealed the evolution of orchid traits.Plant Biotechnology Journal 19:2501-2516.
Yang Z,Dong H,Zhang S,Jiang J,Zhu H,Yang H,Li L.2023.Isolation and identification of mycorrhizal helper bacteria of Vaccinium uliginosum and their interaction with mycorrhizal fungi.Frontiers in Microbiology 14: 1180319.
Yildirir G,Sperschneider J,Malar CM,Chen ECH,Iwasaki W,Cornell C,Corradi N.2022.Long reads and Hi-C sequencing illuminate the two-compartment genome of the model arbuscular mycorrhizal symbiont Rhizophagus irregularis. New Phytologist 233:1097-1107.
Zeng Q,Lebreton A,Auer L,Man X,Jia L,Wang G,Gong S,Lombard V,Buée M, Wu G et al.2023.Stable functional structure despite high taxonomic variability across fungal communities in soils of old-growth montane forests.Microbiome 11: 217.
Zeng T,Holmer R,Hontelez J,Te Lintel-Hekkert B,Marufu L,de Zeeuw T,Wu F,Schijlen E,Bisseling T,Limpens E.2018.Host-and stage-dependent secretome of the arbuscular mycorrhizal fungus Rhizophagus irregularis.The Plant Journal 94:411-425.
Zeng T,Rodriguez-Moreno L,Mansurkhodzaev A,Wang P,van den Berg W, Gasciolli V,Cottaz S,Fort S,Thomma BPHJ,Bono J-J et al.2020.A lysin motif effector subverts chitin-triggered immunity to facilitate arbuscular mycorrhizal symbiosis.New Phytologist 225:448-460.
Zhang C,van der Heijden MG,Dodds BK,Nguyen TB,Spooren J,Held A,Cosme M,Berendsen R.2023.A tripartite bacterial-fungal-plant symbiosis in the mycorrhiza-shaped microbiome drives plant growth and mycorrhization. Microbiome,in press.
Zhang GQ,Liu KW,Li Z,Lohaus R,Hsiao YY,Niu SC,Wang JY,Lin YC,Xu Q, Chen LJ et al.2017.The Apostasia genome and the evolution of orchids.Nature 549:379-383.
Zhang GQ,Xu Q,Bian C,Tsai WC,Yeh CM,Liu KW,Yoshida K, Zhang LS,Chang SB,Chen F et al.2016.The Dendrobium catenatum Lindl.genome sequence provides insights into polysaccharide synthase,floral development and adaptive evolution.Scientific Reports 6: 19029.
Zeng T,Rodriguez-Moreno L,Mansurkhodzaev A,Wang P,van den Berg W, Gasciolli V,Cottaz S,Fort S,Thomma BPHJ,Bono J-J et al.2020.A lysin motif effector subverts chitin-triggered immunity to facilitate arbuscular mycorrhizal symbiosis.New Phytologist 225:448-460.
Zhang C,van der Heijden MG,Dodds BK,Nguyen TB,Spooren J,Held A,Cosme M,Berendsen R.2023.A tripartite bacterial-fungal-plant symbiosis in the mycorrhiza-shaped microbiome drives plant growth and mycorrhization. Microbiome,in press.
Zhang GQ,Liu KW,Li Z,Lohaus R,Hsiao YY,Niu SC,Wang JY,Lin YC,Xu Q, Chen LJ et al.2017.The Apostasia genome and the evolution of orchids.Nature 549:379-383.
Zhang GQ,Xu Q,Bian C,Tsai WC,Yeh CM,Liu KW,Yoshida K, Zhang LS,Chang SB,Chen F et al.2016.The Dendrobium catenatum Lindl.genome sequence provides insights into polysaccharide synthase,floral development and adaptive evolution.Scientific Reports 6: 19029.
Zhang L,Zhou J,George TS,Limpens E,Feng G.2022.Arbuscular mycorrhizal fungi conducting the hyphosphere bacterial orchestra.Trends in Plant Science 27: 402-411.
Zhao H,Zhang R,Wu J,Meng L,Okazaki Y,Hikida H,Ogata H.2023.A 1.5 Mb continuous endogenous viral region in the arbuscular mycorrhizal fungus,Rhizophagus irregularis.Virus Evaluation 9:vead064.
Zhao H,Zhang R,Wu J,Meng L,Okazaki Y,Hikida H,Ogata H.2023.A 1.5 Mb continuous endogenous viral region in the arbuscular mycorrhizal fungus,Rhizophagus irregularis.Virus Evaluation 9:vead064.
