DOI: https://doi.org/10.1186/s40168-024-01750-y
PMID: https://pubmed.ncbi.nlm.nih.gov/38504383
تاريخ النشر: 2024-03-19
المركبات الإندولية المستمدة من الميكروبيوتا تخفف الالتهاب المعوي وتعدل الميكروبيوم من خلال التغذية المتبادلة بين الميكروبات
الملخص
الخلفية: تلعب تفاعلات المضيف والميكروبات دورًا حاسمًا في الحفاظ على التوازن الداخلي وقابلية الإصابة بالأمراض، وتعتبر نواتج التمثيل الغذائي للتريبتوفان الميكروبية من المنظمات القوية لفيزيولوجيا المضيف. ومع ذلك، لا يزال غير واضح ما إذا كانت هذه النواتج وكيفية تأثيرها على تفاعلات المضيف والميكروبات، خاصة من حيث التواصل بين الميكروبات. النتائج: هنا، أظهرنا أن حمض الإندول-3-لاكتك (ILA) هو جزيء رئيسي تنتجه بكتيريا اللاكتوباسيلس في الحماية من الالتهابات المعوية وتصحيح اختلال الميكروبات. على وجه التحديد، تقوم بكتيريا اللاكتوباسيلس بتمثيل التريبتوفان إلى ILA، مما يعزز التعبير عن إنزيمات بكتيرية رئيسية مرتبطة بتمثيل التريبتوفان، مما يؤدي إلى تخليق نواتج إندول أخرى بما في ذلك حمض الإندول-3-بروبيونيك (IPA) وحمض الإندول-3-أسيتيك (IAA). من الجدير بالذكر أن ILA وIPA وIAA تمتلك القدرة على تخفيف الالتهابات المعوية وتعديل الميكروبات المعوية في نماذج التهاب القولون العفوي المستحث بـ DSS وIL-10-/-. يزيد ILA من وفرة البكتيريا الممثلة للتريبتوفان (مثل Clostridium)، بالإضافة إلى التعبير عن mRNA لإنزيمات أكيل-CoA ديهيدروجيناز وإندول لاكتات ديهيدروجيناز في الجسم الحي وفي المختبر، مما يؤدي إلى زيادة إنتاج IPA وIAA. علاوة على ذلك، فإن سلالة متحورة من بكتيريا اللاكتوباسيلس تفشل في الحماية من الالتهابات وإنتاج نواتج أخرى. كان التغذية المتبادلة الموجهة بواسطة ILA تعتمد على الميكروبات وزادت بشكل خاص من إنتاج نواتج الإندول تحت ظروف اختلال الميكروبات المستحثة بواسطة Citrobacter rodentium أو DSS، ولكن ليس من اضطراب الميكروبات بالمضادات الحيوية. الخلاصة: مجتمعة، نبرز الآليات التي من خلالها يتحكم التواصل بين الميكروبات والمضيف بشكل تعاوني في التوازن الداخلي المعوي من خلال الإندولات المستمدة من الميكروبات التي تتوسط التواصل بين الميكروبات. قد تسهم هذه النتائج في تطوير نواتج مستمدة من الميكروبات أو “ما بعد الحيوية” المستهدفة كتدخلات محتملة لعلاج أو الوقاية من الأمراض المدفوعة باختلال الميكروبات.
الخلفية
يجب توضيح الميكروبيوم أيضًا. في الدراسة الحالية، اخترنا Lactobacillus reuteri، وهو بروبيوتيك لديه القدرة على تعديل المناعة المعوية وميكروبيوتا الأمعاء، للتحقيق في دور وآليات الإندولات في الوساطة بين تفاعلات المضيف والميكروبيوتا والتواصل بين الميكروبات. وجدنا أن ILA هو جزيء رئيسي من Lactobacillus في تخفيف الالتهاب وتعديل ميكروبيوتا الأمعاء. زاد ILA من تعبير الإنزيمات ذات الصلة المشاركة في استقلاب التربتوفان عبر التغذية المتبادلة بين البكتيريا وعزز من تخليق مستقلبات التربتوفان الميكروبية بما في ذلك IPA وIAA في الجسم الحي وفي المختبر. كانت تنظيم ميكروبيوتا الأمعاء بواسطة ILA تعتمد على الميكروبيوتا وزادت بشكل خاص من مستويات الإندولات في ظل ظروف اختلال التوازن الميكروبي الناتج عن Citrobacter rodentium أو DSS، ولكن ليس في حالة تدمير الميكروبيوتا بالمضادات الحيوية. كانت التأثيرات المضادة للالتهابات وتقوية حاجز الأمعاء لـ IPA منظمة مباشرة بواسطة تنشيط PXR، بينما كانت تأثيرات IAA تعتمد على الميكروبيوتا. معًا، اقترحت هذه النتائج بشكل جماعي أن التواصل بين الميكروبات الذي يتوسطه ILA يعدل التفاعل بين الميكروبيوم والمضيف ويضبط التوازن المعوي بالتعاون مع إندولات أخرى مشتقة من الميكروبيوتا.
طرق
دراسات الحيوانات والبيان الأخلاقي
سلالات بكتيرية والتحضير
سرعة دوران 15 دقيقة، غسلت مرة واحدة، وأعيد تعليقها عند
التركيب المتجانس
زراعة الميكروبات القولونية
التهاب القولون الناتج عن DSS
علاج بالمضادات الحيوية
تقييم نفاذية الأمعاء
تم القياس عند طول موجة تحفيز قدره 490 نانومتر وطول موجة انبعاث قدره 530 نانومتر.
تحليل نسيجي وتقييم خلايا الكأس
زراعة لاهوائية للميكروبات
عزل اللمفاويات وقياس التدفق الخلوي
للكشف عن خلايا Th1 و Th2 و Th17F و Th17A، تم تحفيز اللمفاويات لمدة 4 ساعات بـ
تحليل البقعة الغربية
استخراج RNA وPCR الكمي
إليزا
تحديد نواتج استقلاب التربتوفان
استخراج الحمض النووي، تسلسل جين 16S rRNA، وتحليل البيانات
نظام Illumina MiSeq (Illumina، سان دييغو، كاليفورنيا، الولايات المتحدة الأمريكية) لقراءات الطرفين. تم إجراء تحليل التسلسل باستخدام خط أنابيب UNOISE من خلال USEARCH v10.0 [21] و VSEARCH v2.15 [22]. باختصار، تم دمج تسلسلات الطرفين، وتصفيتها من حيث الجودة، وإزالة التعقيد باستخدام VSEARCH. تم إجراء إزالة الضوضاء وإزالة الشيميرا باستخدام UNOISE3 لتصحيح أخطاء التسلسل. تم تعيين التسلسلات المجمعة مرة أخرى إلى التسلسلات الخالية من الشيميرا التي تم إزالة الضوضاء منها كـ OTUs مع
تحليل الميتا ترانسكريبتوم
تمت معالجة قراءات التسلسل الخام أولاً من خلال مراقبة الجودة باستخدام Trimmomatic [25]، ثم تم محاذاة القراءات النظيفة إلى جينوم الفأر وقاعدة بيانات سيلفا باستخدام Bowtie2 [26] لاستبعاد تلوث الحمض النووي الريبي المضيف والريبوسومي. تم تجميع القراءات المصفاة من الميتا ترانسكريبتوم بشكل ديو نو لكل عينة باستخدام Trinity (v2.2.0) [27]. تم تجميع جميع الكتل باستخدام CD-HIT (v 4.5.8) [28] للحصول على الجينات الفردية. تم تقدير وفرة الجينات الفردية في كل عينة من خلال حساب قيمة النسخ لكل مليون (TPM) بناءً على عدد القراءات المحاذاة باستخدام Salmon (v 0.9.1) [29]. تم التنبؤ بتسلسلات ترميز البروتين من الكتل باستخدام MetaGeneMark [30]. للتعليق الوظيفي، تم استخدام تسلسلات البروتين.
تمت التوصيفات مقابل تلك الخاصة بالجينات المحفوظة تطوريًا في المجموعات المتجانسة غير المشروطة (eggNOG) باستخدام DIAMOND (الإصدار 0.8.23) [31] تحت الإعدادات الافتراضية. تم تحديد الثراء التفاضلي للجينات والوظائف بواسطة DESeq2 [32]. تم محاذاة التصنيفات التصنيفية ضد قاعدة بيانات تصنيف NCBI باستخدام DIAMOND (الإصدار 0.8.23) [31] مع المعلمات الافتراضية. ثم تم تحليل نتائج blastx باستخدام خوارزمية LCA الخاصة بـ MEGAN [33].
تسلسل RNA وتحليل البيانات
تحليل شبكة الارتباط الموزون
تحليل مجموعات البيانات الميتا ترانسكريبتومية لالتهاب الأمعاء البشري
قراءات تسلسل الميتا ترانسكريبتوم في قاعدة بيانات الأمراض المعوية الالتهابية متعددة الأوميات [37]. كانت القراءات المستخدمة في الدراسة الحالية من 46 مريضًا بمرض كرون (CD)، و21 مريضًا بالتهاب القولون التقرحي (UC)، و11 شخصًا سليمًا. تم معالجة البيانات وتحليلها بطريقة مشابهة لبيانات الميتا ترانسكريبتوم الفأر كما هو موضح أعلاه. بالنسبة لمجموعة مرضى التهاب الأمعاء، تم تطبيق نماذج التأثيرات المختلطة الخطية لتحديد وفرة الجينات الميكروبية المعبر عنها بشكل مختلف كما يلي: الجين
تحليل الإحصائيات
النتائج
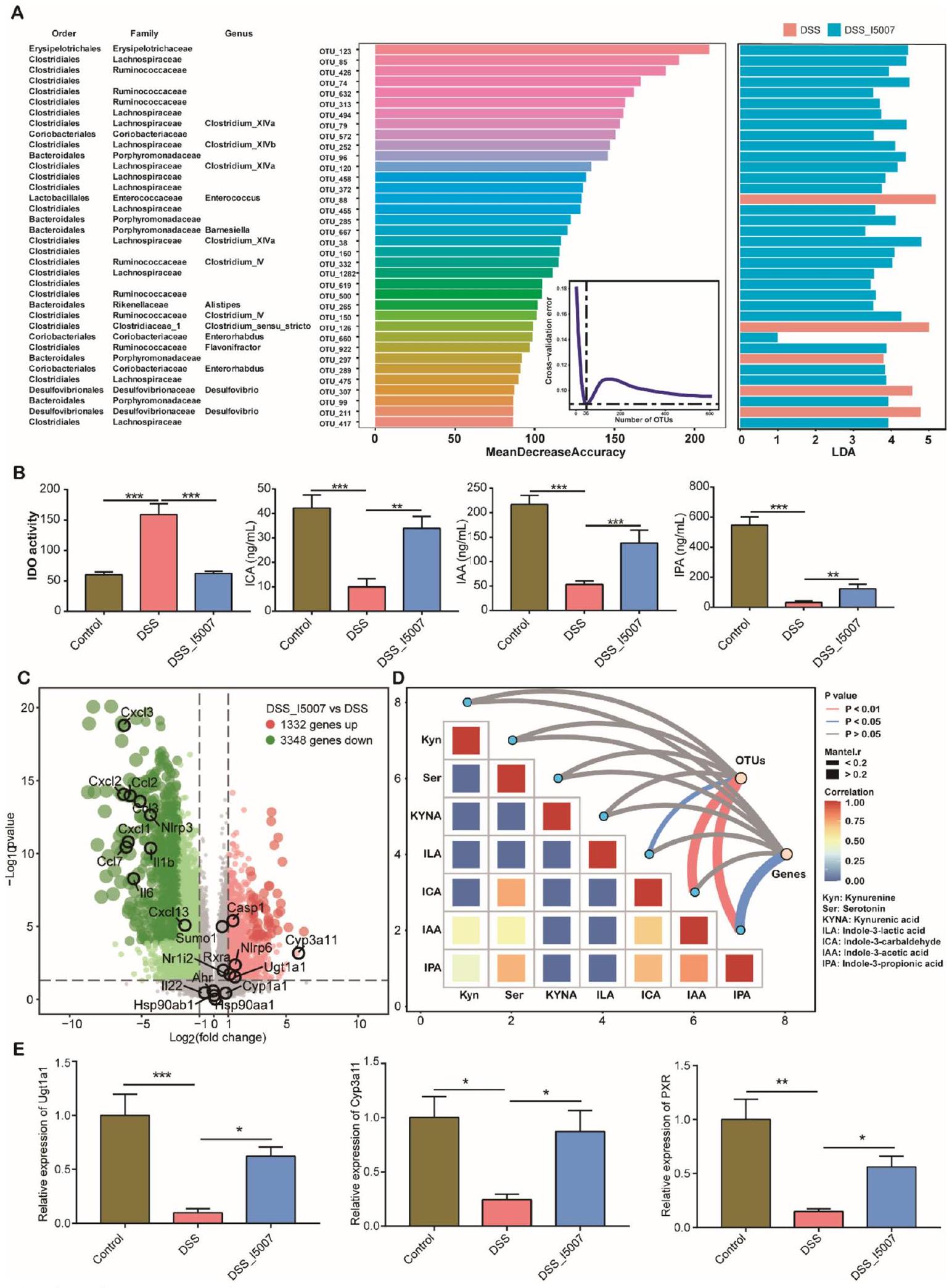
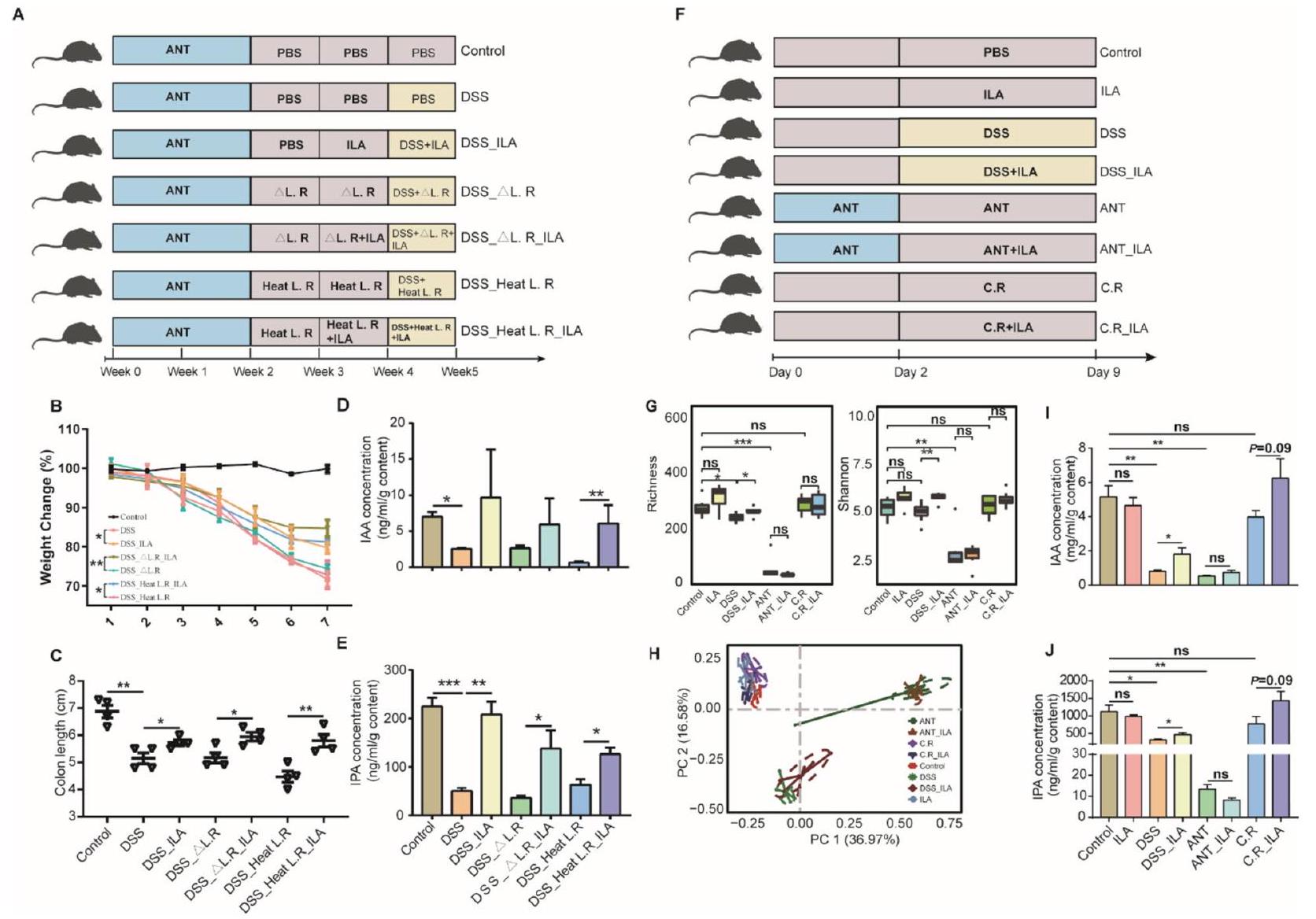
الـ L. reuteri 15007 يحمي الفئران من التهاب القولون من خلال تعديل الميكروبيوم المعوي
مجموعات من الفئران المعالجة بـ DSS (الملف الإضافي 1: الشكل S1BD). من المثير للاهتمام أن الميكروبات المعوية للفئران المعالجة بـ DSS قد استعادت بشكل كبير حالتها الطبيعية، بينما كانت تلك الخاصة بـ
يرتبط التأثير الوقائي لـ L. reuteri بإنتاج مشتقات الإندول
ل. ريوتيري 15007 في الفئران المعالجة بـ DSS (الشكل 1C). أظهرت تحليل إثراء مسار KEGG وتحليل إثراء مجموعة الجينات (GSEA) أن
كما هو متوقع، تعبير
كلا من IPA و IAA يحسنان التهاب القولون الناتج عن DSS
ريوتيري، فشلت IAA في تعزيز التعبير عن
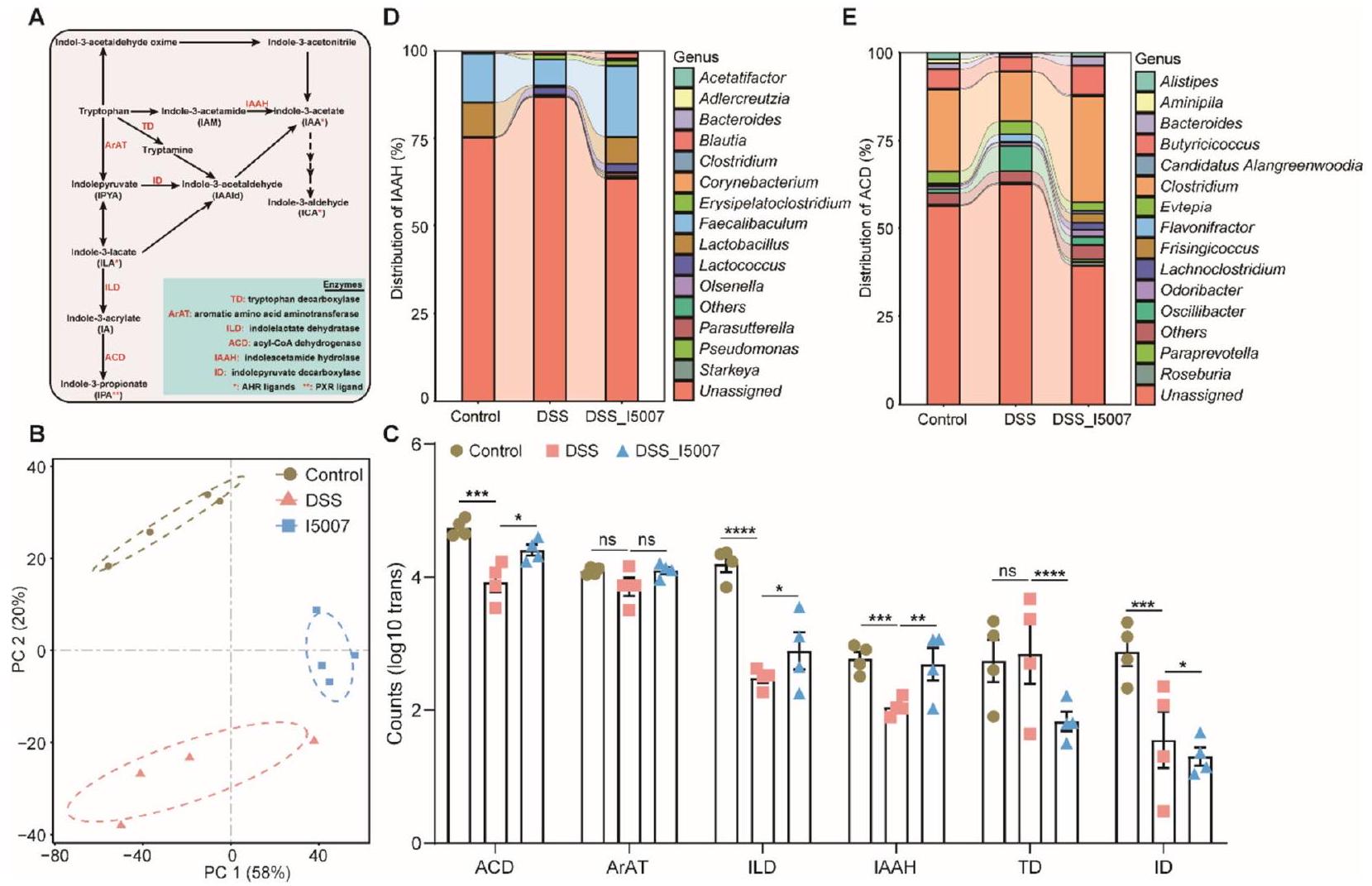
الـ L. reuteri يغير من استقلاب التريبتوفان الميكروبي من خلال تخليق ILA
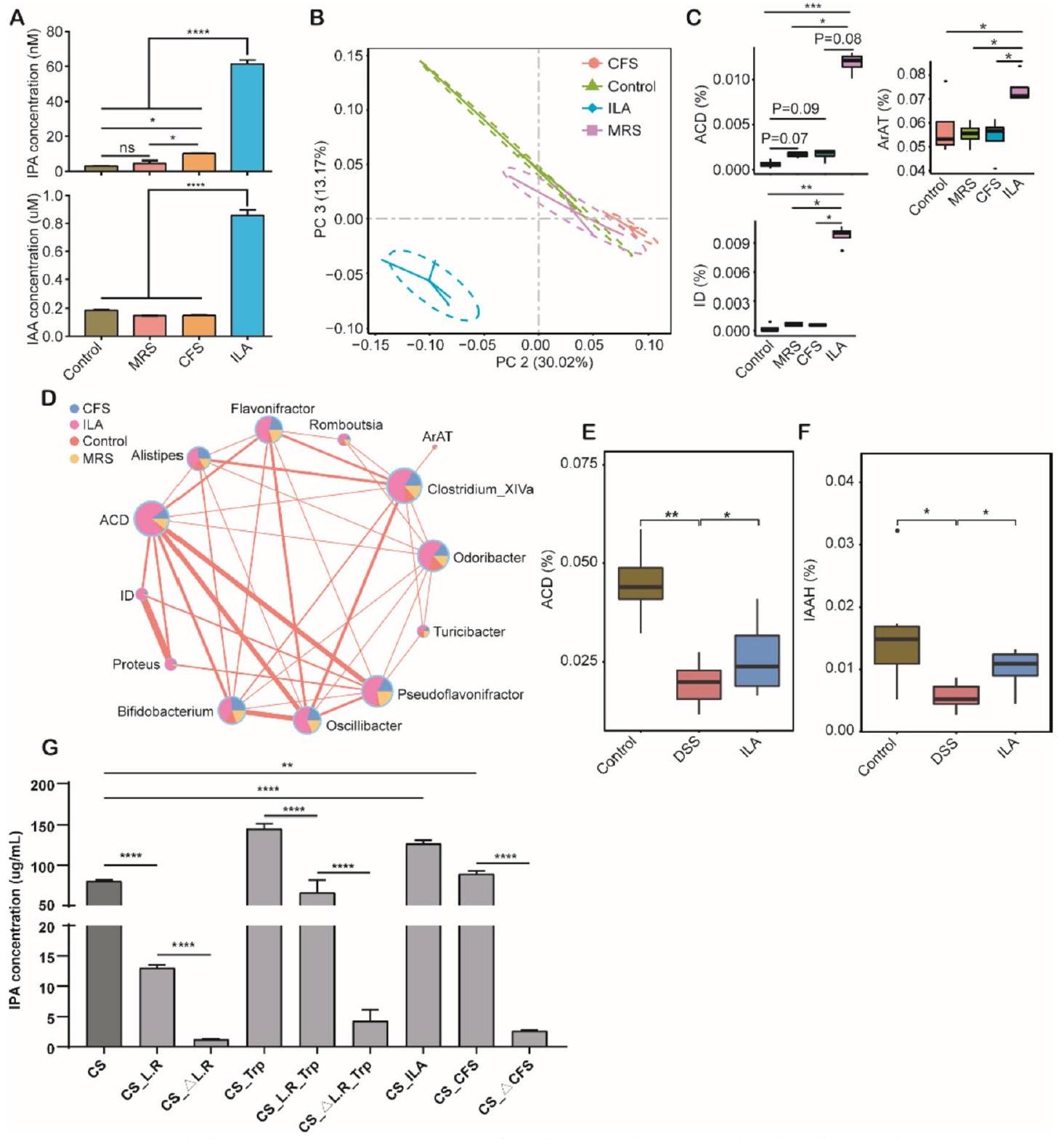
ILA يحسن التهاب القولون الناتج عن DSS ويغير تركيبة الميكروبات المعوية
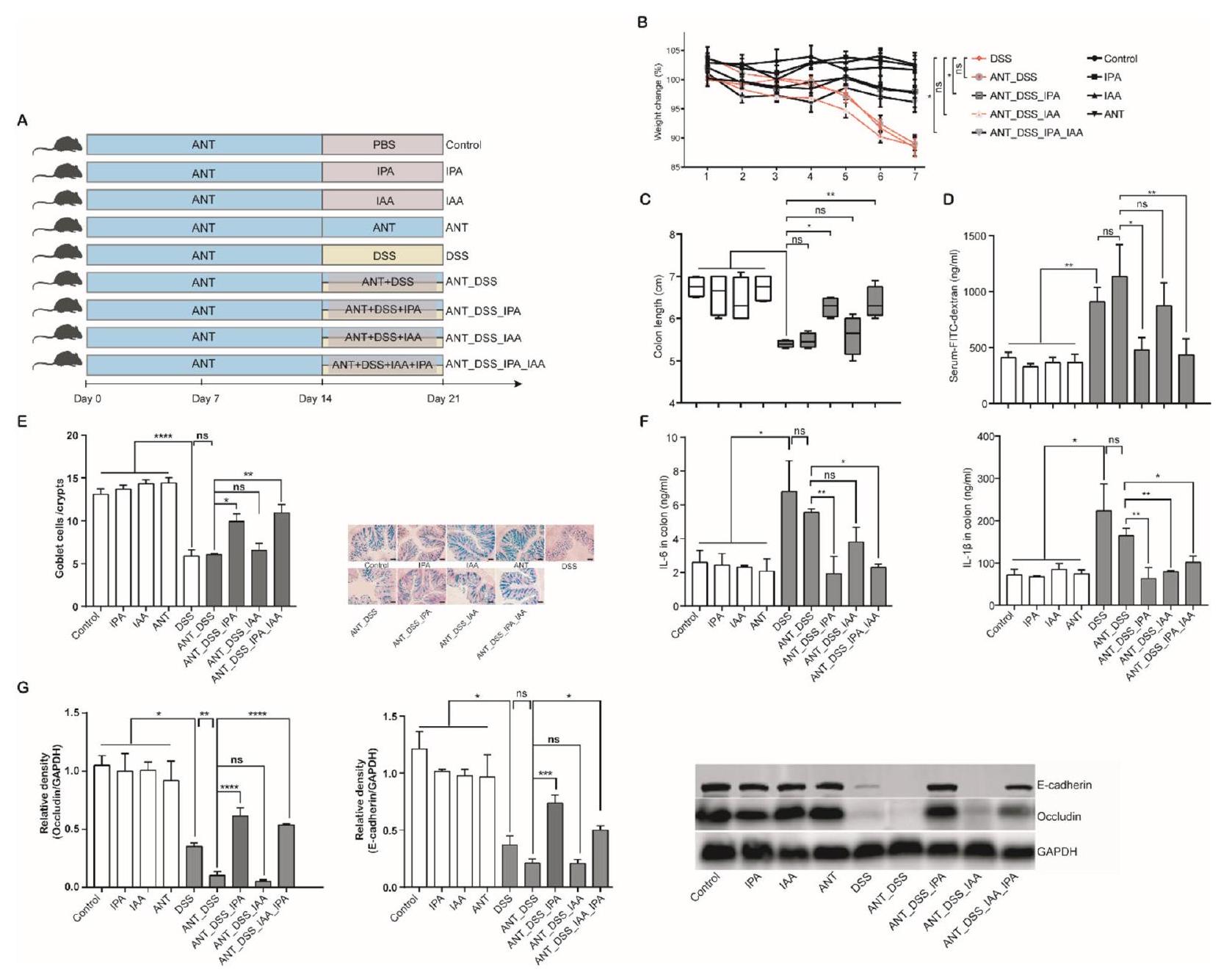
تم إعطاؤه يوميًا مع أو بدون ILA لمدة أسبوع. عكس ILA بشكل كبير فقدان الوزن الناتج عن DSS (الملف الإضافي 1: الشكل S7A) وتقليل طول القولون (الملف الإضافي 1: الشكل S7B). وبشكل متسق، قمع ILA بشكل كبير إنتاج TNF- الناتج عن DSS.
ILA و IPA و IAA تخفف الالتهاب المعوي في
(الشكل 4B). بشكل عام، تشير هذه النتائج إلى أن ILA و IPA أكثر كفاءة من IAA في حماية الفئران من الالتهاب المعوي في كل من DSS و IL-
ILA تزيد من إنتاج IPA وIAA من خلال التغذية المتبادلة الميكروبية سواء في الجسم الحي أو في المختبر
للمزيد من التحقق من أن ILA تعزز إنتاج مشتقات الإندول الأخرى، قمنا بزراعة التريبتوفان معًا،
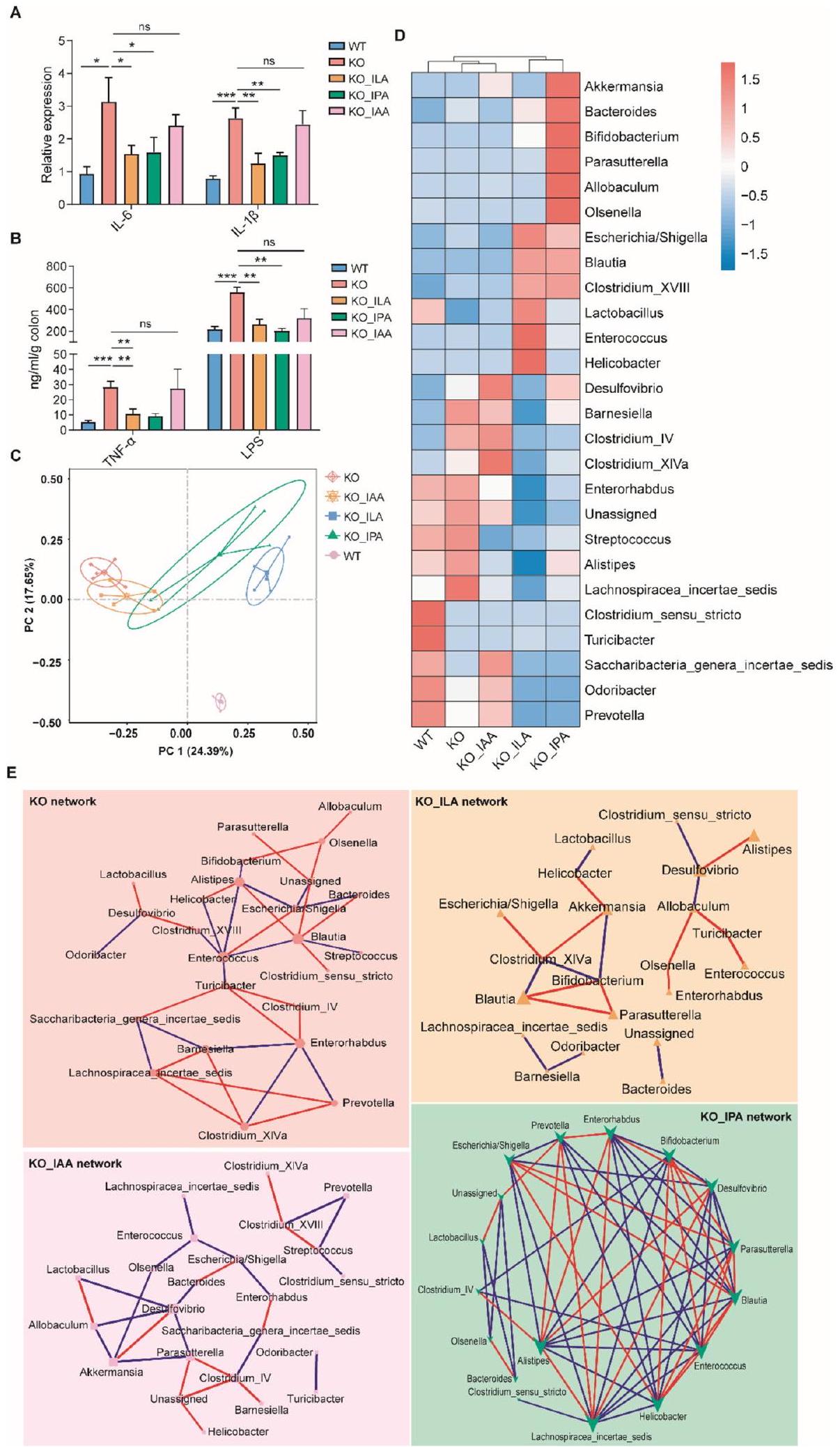
التغيرات في استقلاب التريبتوفان الميكروبي لدى مرضى التهاب الأمعاء
ومع ذلك، فإنه من غير الواضح ما إذا كان استقلاب الميكروبات للتريبتوفان مثبطًا في مرضى التهاب الأمعاء كما هو الحال في الفئران المعالجة بـ DSS. لفحص مستويات التعبير للجينات البكتيرية الرئيسية
“(21.9%)، روزبورية (20.7%)، وبريفوتيلا (7.5%) في عينات البراز، وكانت وفرتها منخفضة بشكل ملحوظ في مرضى التهاب الأمعاء (الشكل S9C و9E). على مستوى الأنواع، كانت البكتيريا الثلاثة الأكثر وفرة تشمل روزبورية سب. CAG:18_43_25 (13.2%)، فاسيكاليباكتيريوم براوزنيتزي (
نقاش
يتماشى مع دراسة سابقة [47]، وجدنا أن L. reuteri يزيد بشكل كبير من Clostridium XIVa وClostridium IV، اللذان هما المسؤولان عن تكاثر
الآلية الجزيئية الأساسية لـ ILA في الحفاظ على توازن الأمعاء غير معروفة إلى حد كبير. تشير بياناتنا إلى أن ILA يقلل من الالتهاب المعوي والأمراض المرتبطة بالتهاب القولون الناتج عن DSS. من المعروف أن ILA يثبط الالتهاب عن طريق تقليل مستويات السيتوكينات المؤيدة للالتهاب من خلال تنشيط AhR. ومع ذلك، أظهرنا أن التأثير الوقائي لـ ILA يتأثر بشكل كبير بمضاد AhR في المعالجة بـ DSS.
الفئران. كشفت تحليلات الميكروبيوتا لدينا أن ILA غيرت بشكل ملحوظ الميكروبيوتا المعوية، مع زيادة كبيرة في Clostridium XIVa، وهو بكتيريا رئيسية تقوم بتمثيل التربتوفان. بالإضافة إلى ذلك، لم تستجب الفئران المصابة بالتهاب القولون المعالجة بالمضادات الحيوية بشكل إيجابي لإدارة ILA. علاوة على ذلك، خفف ILA الالتهاب المعوي وغير تركيب الميكروبيوتا في IL-
تم الإبلاغ عن أن فعالية العلاج الكيميائي للسرطان أو الاستجابة للتدخلات البروبيوتيك تتأثر بشدة بميكروبيوتا الأمعاء لدى الأفراد. بشكل متسق، كانت تنظيم الميكروبيوم المعوي بواسطة ILA معتمدة على الميكروبيوتا وزادت بشكل خاص من مستويات الإندولات في ظروف اختلال التوازن الميكروبي مع Citrobacter rodentium أو DSS، ولكن ليس في حالة تدمير الميكروبيوتا بالمضادات الحيوية. قد يكون هذا مرتبطًا بتقليل بعض البكتيريا المحددة التي تقوم بتمثيل التربتوفان. باستخدام نهج الميتا ترانسكروم، أكدنا اختلال التوازن في بكتيريا تمثيل التربتوفان، وتم تقليل التعبير عن أسيل-CoA ديهيدروجيناز، الذي يتطلب لإنتاج IPA، بشكل كبير في مرضى التهاب الأمعاء.
لذلك، تشير دراستنا إلى أن تركيبة الميكروبيوتا ووظيفتها قد يتم تنظيمها من خلال الأساليب البروبيوتيك. قد يمكّن علاج المستقلبات الذي يتوسط المستقلبات المشتقة من الميكروبيوتا من التحكم بشكل أفضل في تفاعلات المضيف والميكروبيوتا. قد يساعد استخدام العوامل الذاتية التي تصحح الميكروبيوتا من حالة المرض إلى حالة صحية في التغلب على التباين الكبير بين الأفراد، والذي يحد حاليًا من فعالية العلاجات الدوائية أو التدخلات البروبيوتيك. قد توفر المستقلبات المشتقة من الميكروبيوتا أو “البوستبيوتيك” المستهدفة أساليب جديدة للوقاية أو العلاج من الأمراض الناتجة عن اختلال التوازن الميكروبي.
الاختصارات
| AhR | مستقبل الهيدروكربون العطري |
| ACD | ديهيدروجيناز أسيل-CoA |
| أرات | أمينوترانسفيراز الأحماض الأمينية العطرية |
| قرص مضغوط | مرض كرون |
| متلازمة التعب المزمن | السائل الطافح الخالي من الخلايا |
| CLP | سلف اللمفاويات المشترك |
| CMT | زراعة ميكروبات القولون |
| داي | مؤشر نشاط المرض |
| تحليل المسار الجيني | تحليل إثراء مجموعة الجينات |
| IAA | حمض الإندول-3-أسيتيك |
| IAAH | هيدرازين إندول أسيتاميد |
| IBD | مرض الأمعاء الالتهابي |
| ICA | إندول-3-كاربالديهيد |
| هوية | ديكربوكسيلاز إندول بيوفريت |
| إيدو | إنزيمات إندولامين 2,3-ديوكسيجيناز |
| إل | إنترلوكين |
| إلى | حمض الإندول-3-لاكتك |
| ILD | ديهيدروجيناز إندول لاكتات |
| IPA | حمض الإندول-3-بروبيونيك |
| كيج | موسوعة كيوتو للجينات والجينومات |
| ضربة قاضية | ضربة قاضية |
| ليفس | حجم تأثير تحليل التمييز الخطي |
| ل. ريوتيري | لاكتوباسيلس ريوتيري |
| MLNs | الغدد اللمفاوية المساريقية |
| PBS | محلول ملحي معزز بالفوسفات |
| تفاعل البوليميراز المتسلسل | تفاعل البوليميراز المتسلسل |
| PXR | مستقبل الحمل X |
| تسلسل RNA | تسلسل RNA |
| الرنا الريبوسومي | الرنا الريبوسومي |
| ث | خلايا ت المساعدة |
| UC | التهاب القولون التقرحي |
| WT | النوع البري |
| س. سبوروجينيس | كلوستريديوم سبوروجينيس |
معلومات إضافية
تعبير AhR وCyp1a1 وPxr mRNAs (
شكر وتقدير
مساهمات المؤلفين
تمويل
توفر البيانات والمواد
إعلانات
موافقة الأخلاقيات والموافقة على المشاركة
الموافقة على النشر
المصالح المتنافسة
تفاصيل المؤلف
تم النشر على الإنترنت: 19 مارس 2024
References
- Fang H, Fu L, Wang J. Protocol for fecal microbiota transplantation in inflammatory bowel disease: a systematic review and meta-analysis. Biomed Res Int. 2018;2018:8941340.
- Lamas B, Richard ML, Leducq V, Pham H-P, Michel M-L, Da Costa G, et al. CARD9 impacts colitis by altering gut microbiota metabolism of tryptophan into aryl hydrocarbon receptor ligands. Nat Med. 2016;22:598-605.
- Koh A, De Vadder F, Kovatcheva-Datchary P, et al. From dietary fiber to host physiology: short-chain fatty acids as key bacterial metabolites. Cell. 2016;165:1332-45.
- Tintelnot J, Xu Y, Lesker TR, et al. Microbiota-derived 3-IAA influences chemotherapy efficacy in pancreatic cancer. Nature. 2023;615:168-74.
- Wang G, Huang S, Wang Y, et al. Bridging intestinal immunity and gut microbiota by metabolites. Cell Mol Life Sci. 2019;76:3917-37.
- Roth W, Zadeh K, Vekariya R, et al. Tryptophan metabolism and gut-brain homeostasis. Int J Mol Sci. 2021;22:2973.
- Dodd D, Spitzer MH, Van Treuren W, et al. A gut bacterial pathway metabolizes aromatic amino acids into nine circulating metabolites. Nature. 2017;551:648-52.
- Zelante T, lannitti RG, Cunha C, et al. Tryptophan catabolites from microbiota engage aryl hydrocarbon receptor and balance mucosal reactivity via interleukin-22. Immunity. 2013;39:372-85.
- Krishnan S, Ding Y, Saedi N, et al. Gut microbiota-derived tryptophan metabolites modulate inflammatory response in hepatocytes and macrophages. Cell Rep. 2018;23:1099-111.
-
, et al. Rewiring the altered tryptophan metabolism as a novel therapeutic strategy in inflammatory bowel diseases. Gut. 2023;72. Available from: https://pubmed.ncbi.nlm.nih.gov/ 36270778/. Cited 2023 Nov 21. - Cervantes-Barragan L, Chai JN, Tianero MD, et al. Lactobacillus reuteri induces gut intraepithelial CD4+CD8aa+T cells. Science. 2017;357:806-10.
- Wilck N, Matus MG, Kearney SM, et al. Salt-responsive gut commensal modulates TH17 axis and disease. Nature. 2017;551:585-9.
- Laursen MF, Sakanaka M, von Burg N, et al. Bifidobacterium species associated with breastfeeding produce aromatic lactic acids in the infant gut. Nat Microbiol. 2021;6:1367-82.
- Wang G, Huang S, Cai S, et al. Lactobacillus reuteri ameliorates intestinal inflammation and modulates gut microbiota and metabolic disorders in dextran sulfate sodium-induced colitis in mice. Nutrients. 2020;12:2298.
- Zhu H, Cao C, Wu Z, et al. The probiotic L. casei Zhang slows the progression of acute and chronic kidney disease. Cell Metab. 2021;33:1926-1942.e8.
- Hu J, Ma L, Nie Y, et al. A microbiota-derived bacteriocin targets the host to confer diarrhea resistance in early-weaned piglets. Cell Host Microbe. 2018;24:817-832.e8.
- Stillie R, Stadnyk AW. Role of TNF receptors, TNFR1 and TNFR2, in dextran sodium sulfate-induced colitis. Inflamm Bowel Dis. 2009;15:1515-25.
- Suez J, Zmora N, Zilberman-Schapira G, et al. Post-antibiotic gut mucosal microbiome reconstitution is impaired by probiotics and improved by autologous FMT. Cell. 2018;174:1406-1423.e16.
- Weigmann B, Tubbe I, Seidel D, et al. Isolation and subsequent analysis of murine lamina propria mononuclear cells from colonic tissue. Nat Protoc. 2007;2:2307-11.
- Liu H, Hou C, Wang G, et al. Lactobacillus reuteri I5007 modulates intestinal host defense peptide expression in the model of IPEC-J2 cells and neonatal piglets. Nutrients. 2017;9:559.
- Edgar RC. Search and clustering orders of magnitude faster than BLAST. Bioinformatics. 2010;26:2460-1.
- Rognes T, Flouri T, Nichols B, et al. VSEARCH: a versatile open source tool for metagenomics. PeerJ. 2016;4:e2584.
- Cole JR, Wang Q, Fish JA, et al. Ribosomal Database Project: data and tools for high throughput rRNA analysis. Nucleic Acids Res. 2014;42:D633-42.
- Douglas GM, Maffei VJ, Zaneveld JR, et al. PICRUSt2 for prediction of metagenome functions. Nat Biotechnol. 2020;38:685-8.
- Bolger AM, Lohse M, Usadel B. Trimmomatic: a flexible trimmer for Illumina sequence data. Bioinformatics. 2014;30:2114-20.
- Langmead B, Salzberg SL. Fast gapped-read alignment with Bowtie 2. Nat Methods. 2012;9:357-9.
- Grabherr MG, Haas BJ, Yassour M, et al. Full-length transcriptome assembly from RNA-Seq data without a reference genome. Nat Biotechnol. 2011;29:644-52.
- Li W, Godzik A. Cd-hit: a fast program for clustering and comparing large sets of protein or nucleotide sequences. Bioinformatics. 2006;22:1658-9.
- Patro R, Duggal G, Love MI, et al. Salmon provides fast and bias-aware quantification of transcript expression. Nat Methods. 2017;14:417-9.
- Zhu W, Lomsadze A, Borodovsky M. Ab initio gene identification in metagenomic sequences. Nucleic Acids Res. 2010;38:e132-e132.
- Buchfink B, Xie C, Huson DH. Fast and sensitive protein alignment using DIAMOND. Nat Methods. 2015;12:59-60.
- Love MI, Huber W, Anders S. Moderated estimation of fold change and dispersion for RNA-seq data with DESeq2. Genome Biol. 2014;15:550.
- Huson DH, Auch AF, Qi J, et al. MEGAN analysis of metagenomic data. Genome Res. 2007;17:377-86.
- Kim D, Langmead B, Salzberg SL. HISAT: a fast spliced aligner with low memory requirements. Nat Methods. 2015;12:357-60.
- Yu G, Wang L-G, Han Y, et al. clusterProfiler: an R package for comparing biological themes among gene clusters. OMICS. 2012;16:284-7.
- Langfelder P, Zhang B, Horvath S. Defining clusters from a hierarchical cluster tree: the Dynamic Tree Cut package for R. Bioinformatics. 2008;24:719-20.
- IBDMDB Investigators, Lloyd-Price J, Arze C, et al. Multi-omics of the gut microbial ecosystem in inflammatory bowel diseases. Nature. 2019;569:655-62.
- Wang G, Wang X, Ma Y, et al. Lactobacillus reuteri improves the development and maturation of fecal microbiota in piglets through mother-to-infant microbe and metabolite vertical transmission. Microbiome. 2022;10:211.
- Qi Q, Li J, Yu B, et al. Host and gut microbial tryptophan metabolism and type 2 diabetes: an integrative analysis of host genetics, diet, gut microbiome and circulating metabolites in cohort studies. Gut. 2022;71:1095-105.
- Platten M, Wick W, Van den Eynde BJ. Tryptophan catabolism in cancer: beyond IDO and tryptophan depletion. Cancer Res. 2012;72:5435-40.
- Venkatesh M, Mukherjee S, Wang H, et al. Symbiotic bacterial metabolites regulate gastrointestinal barrier function via the xenobiotic sensor PXR and Toll-like receptor 4. Immunity. 2014;41:296-310.
- Lee JS, Cella M, McDonald KG, et al. AHR drives the development of gut ILC22 cells and postnatal lymphoid tissues via pathways dependent on and independent of Notch. Nat Immunol. 2012;13:144-51.
- Wilson MS, Ramalingam TR, Rivollier A, et al. Colitis and intestinal inflammation in IL10-/- mice results from IL-13Ra2-mediated attenuation of IL-13 activity. Gastroenterology. 2011;140:254-64.
- Nikolaus S, Schulte B, Al-Massad N, et al. Increased tryptophan metabolism is associated with activity of inflammatory bowel diseases. Gastroenterology. 2017;153:1504-1516.e2.
- Schirmer M, Franzosa EA, Lloyd-Price J, et al. Dynamics of metatranscription in the inflammatory bowel disease gut microbiome. Nat Microbiol. 2018;3:337-46.
- Investigators IBDMDB, Lloyd-Price J, Arze C, Ananthakrishnan AN, et al. Multi-omics of the gut microbial ecosystem in inflammatory bowel diseases. Nature. 2019;569:655-62.
- Jang YJ, Kim W-K, Han DH, et al. Lactobacillus fermentum species ameliorate dextran sulfate sodium-induced colitis by regulating the immune response and altering gut microbiota. Gut Microbes. 2019;10:696-711.
- Atarashi K, Tanoue T, Oshima K, et al. Treg induction by a rationally selected mixture of Clostridia strains from the human microbiota. Nature. 2013;500:232-6.
- Ehrlich AM, Pacheco AR, Henrick BM, et al. Indole-3-lactic acid associated with Bifidobacterium-dominated microbiota significantly decreases inflammation in intestinal epithelial cells. BMC Microbiol. 2020;20:357.
- Zelezniak A, Andrejev S, Ponomarova O, et al. Metabolic dependencies drive species co-occurrence in diverse microbial communities. Proc Natl Acad Sci U S A. 2015;112:6449-54.
- Meng D, Sommella E, Salviati E, et al. Indole-3-lactic acid, a metabolite of tryptophan, secreted by Bifidobacterium longum subspecies infantis is anti-inflammatory in the immature intestine. Pediatr Res. 2020;88:209-17.
- Menni C, Hernandez MM, Vital M, et al. Circulating levels of the antioxidant indoleproprionic acid are associated with higher gut microbiome diversity. Gut Microbes. 2019;10:688-95.
- Zmora N, Zilberman-Schapira G, Suez J, et al. Personalized gut mucosal colonization resistance to empiric probiotics is associated with unique host and microbiome features. Cell. 2018;174:1388-1405.e21.
ملاحظة الناشر
- (انظر الشكل في الصفحة التالية.)
الشكل 1 L. reuteri يغير تركيبة الميكروبيوم المعوي ومستقلب التريبتوفان الميكروبي. A إثراء تفاضلي للميكروبيوم القولوني للفئران استجابةً لـ. مكملات reuteri في اليوم 28 (انظر أسطورة الشكل S1A للمخطط التجريبي). يتم عرض التصنيفات التصنيفية للبكتيريا في اللوحة اليسرى. الفراغات هي تصنيفات غير محددة. يتم تصنيف 36 تصنيفًا بكتيريًا مختلفًا بناءً على تقدير الانخفاض المتوسط في الدقة استنادًا إلى تحليل الغابة العشوائية. تُظهر اللوحة اليمنى تحليل LEfSe لـ 36 تصنيفًا بكتيريًا. ب تركيزات المصل من المستقلبات في استقلاب التريبتوفان بين ثلاث مجموعات من الفئران ( ). ج مخطط البركان يظهر التعبير الجيني التفاضلي في القولون للفئران المعالجة بـ DSS مع أو بدون مكملات L. reuteri. كل نقطة حمراء تشير إلى جين تم تنظيمه بشكل كبير، بينما تمثل النقطة الزرقاء جينًا تم تنظيمه بشكل كبير لأسفل، مع كل نقطة رمادية تظهر جينًا بلا فرق كبير. د المقارنات الزوجية لمستقلبات التريبتوفان، مع تدرج لوني يدل على معامل ارتباط سبيرمان. الارتباط بين ملفات التعبير البكتيرية/ الجينية وكل مستقلب باستخدام اختبارات مانتل الجزئية. عرض الحافة يتناسب مع إحصائية R لمانتل للارتباطات المسافة المقابلة، ولون الحافة يدل على الأهمية الإحصائية. هـ اختبار PCR في الوقت الحقيقي يظهر تعبير Pxr وجيناته المستهدفة ، - و
(انظر الشكل في الصفحة التالية.)الشكل 4 مشتقات الإندول تخفف الالتهاب المعوي في الفئران. تم تغذية الفئران بعمر ستة أسابيع يوميًا عن طريق الفم مع أو بدون ILA ( )، IPA ( )، أو IAA ( ) لمدة 4 أسابيع ( ). تم أيضًا تغذية الفئران البرية المتجانسة (WT) يوميًا لمدة 4 أسابيع كضوابط سلبية. أ مستويات التعبير الجيني للـ mRNA في القولون للسيتوكينات المسببة للالتهابات (IL-6 و IL-1 ) ( ) بواسطة RT-qPCR. ب مستويات TNF-a و LPS في القولون ( ) باستخدام ELISA. ج مخطط PCoA لمسافة Bray-Curtis للميكروبيوم القولوني بين مجموعات مختلفة من الفئران. د خريطة حرارية تظهر الوفرة النسبية لأجناس بكتيرية مختلفة. هـ تحليل شبكة التواجد المشترك لأجناس بكتيرية بين مجموعات مختلفة من الفئران. الحواف التي تمثل ارتباطات SparCC الهامة تشير إلى و . كل خط أزرق فاتح يمثل ارتباطًا سلبيًا كبيرًا، بينما يمثل كل خط أحمر فاتح ارتباطًا إيجابيًا كبيرًا. حجم النقاط يمثل درجة العقدة. سمك الخط يتناسب مع درجة الارتباط. ; ns، غير مهم
DOI: https://doi.org/10.1186/s40168-024-01750-y
PMID: https://pubmed.ncbi.nlm.nih.gov/38504383
Publication Date: 2024-03-19
Microbiota-derived indoles alleviate intestinal inflammation and modulate microbiome by microbial cross-feeding
Abstract
Background The host-microbiota interaction plays a crucial role in maintaining homeostasis and disease susceptibility, and microbial tryptophan metabolites are potent modulators of host physiology. However, whether and how these metabolites mediate host-microbiota interactions, particularly in terms of inter-microbial communication, remains unclear. Results Here, we have demonstrated that indole-3-lactic acid (ILA) is a key molecule produced by Lactobacillus in protecting against intestinal inflammation and correcting microbial dysbiosis. Specifically, Lactobacillus metabolizes tryptophan into ILA, thereby augmenting the expression of key bacterial enzymes implicated in tryptophan metabolism, leading to the synthesis of other indole derivatives including indole-3-propionic acid (IPA) and indole-3-acetic acid (IAA). Notably, ILA, IPA, and IAA possess the ability to mitigate intestinal inflammation and modulate the gut microbiota in both DSS-induced and IL-10-/- spontaneous colitis models. ILA increases the abundance of tryptophanmetabolizing bacteria (e.g., Clostridium), as well as the mRNA expression of acyl-CoA dehydrogenase and indolelactate dehydrogenase in vivo and in vitro, resulting in an augmented production of IPA and IAA. Furthermore, a mutant strain of Lactobacillus fails to protect against inflammation and producing other derivatives. ILA-mediated microbial cross-feeding was microbiota-dependent and specifically enhanced indole derivatives production under conditions of dysbiosis induced by Citrobacter rodentium or DSS, but not of microbiota disruption with antibiotics. Conclusion Taken together, we highlight mechanisms by which microbiome-host crosstalk cooperatively control intestinal homoeostasis through microbiota-derived indoles mediating the inter-microbial communication. These findings may contribute to the development of microbiota-derived metabolites or targeted “postbiotic” as potential interventions for the treatment or prevention of dysbiosis-driven diseases.
Background
the microbiome likewise needs to be elucidated. In the present study, we chose Lactobacillus reuteri, a probiotic with the ability to modulate intestinal immunity and gut microbiota, to investigate the role and mechanisms of indoles in mediating host-microbiota interactions and inter-microbial communication. We found that ILA is a key molecule of Lactobacillus in the alleviation of inflammation and modulation of the gut microbiota. ILA increased the expression of relevant enzymes involved tryptophan metabolism via bacterial cross-feeding and promoted the synthesis of microbial tryptophan metabolites including IPA and IAA in vivo and in vitro. The regulation of the intestinal microbiota by ILA was micro-biota-dependent and specifically increasing indoles levels under conditions of dybiosis induced by Citrobacter rodentium or DSS, but not of microbiota disruption with antibiotics. The anti-inflammatory and intestinal bar-rier-strengthening effects of IPA were directly regulated by PXR activation, while those of IAA were microbi-ota-dependent. Together, these results collectively suggested ILA-mediated the inter-microbial communication modulates the microbiome-host crosstalk and controls intestinal homeostasis in conjunction with other micro-biota-derived indoles.
Methods
Animal studies and ethical statement
Bacterial strains and preparation
rpm for 15 min , washed once, and resuspended at
Homologous recombination
Colonic microbiota transplantation
DSS-induced colitis
Antibiotic treatment
Intestinal permeability assessment
measured at an excitation wavelength of 490 nm and an emission wavelength of 530 nm .
Histological analysis and goblet cell evaluation
Anaerobic culture of the microbiota
Isolation of lymphocytes and flow cytometry
For detection of Th1, Th2, Th17F, and Th17A cells, lymphocytes were stimulated for 4 h with
Western blot analysis
RNA extraction and quantitative PCR
ELISA
Determination of tryptophan metabolites
DNA extraction, 16S rRNA gene sequencing, and data analysis
the Illumina MiSeq system (Illumina, San Diego, CA, USA) for paired-end reads. Sequence analysis was performed using the UNOISE pipeline through USEARCH v10.0 [21] and VSEARCH v2.15 [22]. Briefly, paired-end sequences were merged, quality filtered, and decomplexed using VSEARCH. Denoising and chimera removal were conducted using UNOISE3 to correct for sequencing errors. Assembled sequences were mapped back to the denoised chimera-free sequences as OTUs with
Metatranscriptomic analysis
Raw sequencing reads first went through quality control using Trimmomatic [25], and clean reads were then aligned to the mouse genome and the Silva database with Bowtie2 [26] to exclude host and ribosomal RNA contamination. Quality-filtered metatranscriptomic reads were de novo assembled for each sample with Trinity (v2.2.0) [27]. All contigs were clustered using CD-HIT (v 4.5.8) [28] to obtain unigenes. The abundance of unigenes in each sample was estimated by calculating the transcripts per million (TPM) value based on the number of aligned reads using Salmon (v 0.9.1) [29]. Protein coding sequences were predicted from contigs using MetaGeneMark [30]. For functional annotation, protein sequences
were annotated against those of evolutionarily conserved genes in the nonsupervised orthologous groups (eggNOG) using DIAMOND (v.0.8.23) [31] under default settings. Differential enrichment of genes and functions were determined by DESeq2 [32]. Taxonomic classifications were aligned against the NCBI taxonomy database using DIAMOND (v.0.8.23) [31] with default parameters. Then, the blastx results were analyzed using the longReads LCA algorithm of MEGAN [33].
RNA sequencing and data analysis
Weighted correlation network analysis
Analysis of the human IBD metatranscriptomic datasets
metatranscriptomic sequencing reads in the Inflammatory Bowel Disease Multiomics Database [37]. The reads used in the present study were from 46 patients with Crohn’s disease (CD), 21 patients with ulcerative colitis (UC), and 11 healthy controls. The data was processed and analyzed similarly to the mouse metatranscriptomic data as described above. For the IBD cohort, linear mixed effect models were further applied to determine the abundance of diagnosed microbial DEGs as follows: gene
Statistics analysis
Results
L. reuteri 15007 protects mice from colitis through modulating the intestinal microbiota
groups of DSS-treated mice (Additional file 1: Fig. S1BD). Interestingly, the colonic microbiota of DSS-treated mice was largely restored to normal, while that of
Protective effect of L. reuteri is associated with the production of indole derivatives
L. reuteri 15007 in DSS-treated mice (Fig. 1C). KEGG pathway enrichment and gene set enrichment analysis (GSEA) showed that
As expected, the expression of
Both IPA and IAA ameliorate DSS-induced colitis
reuteri, IAA failed to enhance the expression of
L. reuteri alters the microbial tryptophan metabolism through synthesis of ILA


ILA ameliorates DSS-induced colitis and alters the intestinal microbiota composition
administered daily with or without ILA for a week. ILA significantly reversed DSS-induced weight loss (Additional file 1: Fig. S7A) and colon length shortening (Additional file 1: Fig. S7B). Consistently, ILA significantly suppressed DSS-induced production of TNF-

ILA, IPA, and IAA alleviate intestinal inflammation in
(Fig. 4B). Overall, these results suggested that ILA and IPA are more efficient than IAA in protecting mice from intestinal inflammation in both DSS and IL-
ILA increases IPA and IAA production through microbial cross-feeding both in vivo and in vitro
further verify that ILA promotes the production of other indole derivatives, we co-cultured tryptophan,


Alterations in microbial tryptophan metabolism in IBD patients
in IBD patients [44]. However, it is unclear whether microbial metabolism of tryptophan is suppressed in IBD patients as in DSS-treated mice. To examine the expression levels of the major bacterial genes

(21.9%), Roseburia (20.7%), and Prevotella (7.5%) in the stool samples, and their abundances were substantially reduced in IBD patients (Figure S9C and 9E). At the species level, three most abundant bacteria including Roseburia sp. CAG:18_43_25 (13.2%), Faecalibacterium prausnitzii (
Discussion
Consistent with an earlier study [47], we found that L. reuteri dramatically enriches Clostridium XIVa and Clostridium IV, which are responsible for the proliferation of
The underlying molecular mechanism of ILA in maintaining intestinal homeostasis is largely unknown [49]. Our data indicated that ILA reduces the intestinal inflammation and pathologies in DSS-induced colitis. ILA is known to inhibit inflammation by decreasing proinflammatory cytokine levels via activation of AhR [13, 49]. However, we showed that the protective effect of ILA is largely unaffected by an AhR antagonist in DSS-treated
mice. Our microbiota analysis further revealed that ILA markedly altered the intestinal microbiota, with a significant enrichment of Clostridium XIVa, a major tryptophan-metabolizing bacterium [39]. Additionally, antibiotic-treated colitis mice failed to respond positively to ILA administration. Furthermore, ILA alleviated the intestinal inflammation and altered the microbiota composition in IL-
The efficacy of cancer chemotherapy or the response to probiotic interventions has been reported to be strongly influenced by individuals intestinal microbiota [4, 53]. Consistently, the regulation of the intestinal microbiome by ILA was microbiota-dependent and specifically enhanced indoles levels under conditions of dybiosis with Citrobacter rodentium or DSS inducing, but not of microbiota disruption with antibiotics. This may be related to the reduction of some specific tryptophan-metabolizing bacteria. Using metatranscriptome approach, we confirmed dysbiosis of tryptophan-metabolizing bacteria and the expression of acyl-CoA dehydrogenase, which is required for producing IPA [7], is drastically diminished in IBD patients.
Therefore, our study suggests that microbiota composition and function may be regulated by probiotic approaches. The metabolite treatment mediating downstream microbiota-derived metabolites may more enable to control the host-microbiota interactions. Utilizing the endogenous factors correcting the microbiota from disease to a healthy state may help overcome the strong inter-individual variability, which currently limits the effectiveness of drug treatments or probiotic interventions. The microbiota-derived metabolites or targeted “postbiotic” may provide novel prevention or treatment approaches of dysbiosis-driven diseases.
Abbreviations
| AhR | Aryl hydrocarbon receptor |
| ACD | Acyl-CoA dehydrogenase |
| ArAT | Aromatic amino acid aminotransferase |
| CD | Crohn’s disease |
| CFS | Cell-free supernatant |
| CLP | Common lymphoid progenitor |
| CMT | Colonic microbiota transplantation |
| DAI | Disease activity index |
| GSEA | Gene set enrichment analysis |
| IAA | Indole-3-acetic acid |
| IAAH | Indoleacetamide hydrolase |
| IBD | Inflammatory bowel disease |
| ICA | Indole-3-carbaldehyde |
| ID | Indolepyruvate decarboxylase |
| IDO | Indoleamine 2,3-dioxygenases |
| IL | Interleukin |
| ILA | Indole-3-lactic acid |
| ILD | Indolelactate dehydrogenase |
| IPA | Indole-3-propionic acid |
| KEGG | Kyoto Encyclopedia of Genes and Genomes |
| KO | Knockout |
| LEfSe | Linear discriminant analysis effect size |
| L. reuteri | Lactobacillus reuteri |
| MLNs | Mesenteric lymph nodes |
| PBS | Phosphate-buffered saline |
| PCR | Polymerase chain reaction |
| PXR | Pregnane X receptor |
| RNA-seq | RNA sequencing |
| rRNA | Ribosomal RNA |
| Th | T helper |
| UC | Ulcerative colitis |
| WT | Wild-type |
| C. sporogenes | Clostridium sporogenes |
Supplementary Information
expression of the AhR, Cyp1a1 and Pxr mRNAs (
Acknowledgements
Authors’ contributions
Funding
Availability of data and materials
Declarations
Ethics approval and consent to participate
Consent for publication
Competing interests
Author details
Published online: 19 March 2024
References
- Fang H, Fu L, Wang J. Protocol for fecal microbiota transplantation in inflammatory bowel disease: a systematic review and meta-analysis. Biomed Res Int. 2018;2018:8941340.
- Lamas B, Richard ML, Leducq V, Pham H-P, Michel M-L, Da Costa G, et al. CARD9 impacts colitis by altering gut microbiota metabolism of tryptophan into aryl hydrocarbon receptor ligands. Nat Med. 2016;22:598-605.
- Koh A, De Vadder F, Kovatcheva-Datchary P, et al. From dietary fiber to host physiology: short-chain fatty acids as key bacterial metabolites. Cell. 2016;165:1332-45.
- Tintelnot J, Xu Y, Lesker TR, et al. Microbiota-derived 3-IAA influences chemotherapy efficacy in pancreatic cancer. Nature. 2023;615:168-74.
- Wang G, Huang S, Wang Y, et al. Bridging intestinal immunity and gut microbiota by metabolites. Cell Mol Life Sci. 2019;76:3917-37.
- Roth W, Zadeh K, Vekariya R, et al. Tryptophan metabolism and gut-brain homeostasis. Int J Mol Sci. 2021;22:2973.
- Dodd D, Spitzer MH, Van Treuren W, et al. A gut bacterial pathway metabolizes aromatic amino acids into nine circulating metabolites. Nature. 2017;551:648-52.
- Zelante T, lannitti RG, Cunha C, et al. Tryptophan catabolites from microbiota engage aryl hydrocarbon receptor and balance mucosal reactivity via interleukin-22. Immunity. 2013;39:372-85.
- Krishnan S, Ding Y, Saedi N, et al. Gut microbiota-derived tryptophan metabolites modulate inflammatory response in hepatocytes and macrophages. Cell Rep. 2018;23:1099-111.
-
, et al. Rewiring the altered tryptophan metabolism as a novel therapeutic strategy in inflammatory bowel diseases. Gut. 2023;72. Available from: https://pubmed.ncbi.nlm.nih.gov/ 36270778/. Cited 2023 Nov 21. - Cervantes-Barragan L, Chai JN, Tianero MD, et al. Lactobacillus reuteri induces gut intraepithelial CD4+CD8aa+T cells. Science. 2017;357:806-10.
- Wilck N, Matus MG, Kearney SM, et al. Salt-responsive gut commensal modulates TH17 axis and disease. Nature. 2017;551:585-9.
- Laursen MF, Sakanaka M, von Burg N, et al. Bifidobacterium species associated with breastfeeding produce aromatic lactic acids in the infant gut. Nat Microbiol. 2021;6:1367-82.
- Wang G, Huang S, Cai S, et al. Lactobacillus reuteri ameliorates intestinal inflammation and modulates gut microbiota and metabolic disorders in dextran sulfate sodium-induced colitis in mice. Nutrients. 2020;12:2298.
- Zhu H, Cao C, Wu Z, et al. The probiotic L. casei Zhang slows the progression of acute and chronic kidney disease. Cell Metab. 2021;33:1926-1942.e8.
- Hu J, Ma L, Nie Y, et al. A microbiota-derived bacteriocin targets the host to confer diarrhea resistance in early-weaned piglets. Cell Host Microbe. 2018;24:817-832.e8.
- Stillie R, Stadnyk AW. Role of TNF receptors, TNFR1 and TNFR2, in dextran sodium sulfate-induced colitis. Inflamm Bowel Dis. 2009;15:1515-25.
- Suez J, Zmora N, Zilberman-Schapira G, et al. Post-antibiotic gut mucosal microbiome reconstitution is impaired by probiotics and improved by autologous FMT. Cell. 2018;174:1406-1423.e16.
- Weigmann B, Tubbe I, Seidel D, et al. Isolation and subsequent analysis of murine lamina propria mononuclear cells from colonic tissue. Nat Protoc. 2007;2:2307-11.
- Liu H, Hou C, Wang G, et al. Lactobacillus reuteri I5007 modulates intestinal host defense peptide expression in the model of IPEC-J2 cells and neonatal piglets. Nutrients. 2017;9:559.
- Edgar RC. Search and clustering orders of magnitude faster than BLAST. Bioinformatics. 2010;26:2460-1.
- Rognes T, Flouri T, Nichols B, et al. VSEARCH: a versatile open source tool for metagenomics. PeerJ. 2016;4:e2584.
- Cole JR, Wang Q, Fish JA, et al. Ribosomal Database Project: data and tools for high throughput rRNA analysis. Nucleic Acids Res. 2014;42:D633-42.
- Douglas GM, Maffei VJ, Zaneveld JR, et al. PICRUSt2 for prediction of metagenome functions. Nat Biotechnol. 2020;38:685-8.
- Bolger AM, Lohse M, Usadel B. Trimmomatic: a flexible trimmer for Illumina sequence data. Bioinformatics. 2014;30:2114-20.
- Langmead B, Salzberg SL. Fast gapped-read alignment with Bowtie 2. Nat Methods. 2012;9:357-9.
- Grabherr MG, Haas BJ, Yassour M, et al. Full-length transcriptome assembly from RNA-Seq data without a reference genome. Nat Biotechnol. 2011;29:644-52.
- Li W, Godzik A. Cd-hit: a fast program for clustering and comparing large sets of protein or nucleotide sequences. Bioinformatics. 2006;22:1658-9.
- Patro R, Duggal G, Love MI, et al. Salmon provides fast and bias-aware quantification of transcript expression. Nat Methods. 2017;14:417-9.
- Zhu W, Lomsadze A, Borodovsky M. Ab initio gene identification in metagenomic sequences. Nucleic Acids Res. 2010;38:e132-e132.
- Buchfink B, Xie C, Huson DH. Fast and sensitive protein alignment using DIAMOND. Nat Methods. 2015;12:59-60.
- Love MI, Huber W, Anders S. Moderated estimation of fold change and dispersion for RNA-seq data with DESeq2. Genome Biol. 2014;15:550.
- Huson DH, Auch AF, Qi J, et al. MEGAN analysis of metagenomic data. Genome Res. 2007;17:377-86.
- Kim D, Langmead B, Salzberg SL. HISAT: a fast spliced aligner with low memory requirements. Nat Methods. 2015;12:357-60.
- Yu G, Wang L-G, Han Y, et al. clusterProfiler: an R package for comparing biological themes among gene clusters. OMICS. 2012;16:284-7.
- Langfelder P, Zhang B, Horvath S. Defining clusters from a hierarchical cluster tree: the Dynamic Tree Cut package for R. Bioinformatics. 2008;24:719-20.
- IBDMDB Investigators, Lloyd-Price J, Arze C, et al. Multi-omics of the gut microbial ecosystem in inflammatory bowel diseases. Nature. 2019;569:655-62.
- Wang G, Wang X, Ma Y, et al. Lactobacillus reuteri improves the development and maturation of fecal microbiota in piglets through mother-to-infant microbe and metabolite vertical transmission. Microbiome. 2022;10:211.
- Qi Q, Li J, Yu B, et al. Host and gut microbial tryptophan metabolism and type 2 diabetes: an integrative analysis of host genetics, diet, gut microbiome and circulating metabolites in cohort studies. Gut. 2022;71:1095-105.
- Platten M, Wick W, Van den Eynde BJ. Tryptophan catabolism in cancer: beyond IDO and tryptophan depletion. Cancer Res. 2012;72:5435-40.
- Venkatesh M, Mukherjee S, Wang H, et al. Symbiotic bacterial metabolites regulate gastrointestinal barrier function via the xenobiotic sensor PXR and Toll-like receptor 4. Immunity. 2014;41:296-310.
- Lee JS, Cella M, McDonald KG, et al. AHR drives the development of gut ILC22 cells and postnatal lymphoid tissues via pathways dependent on and independent of Notch. Nat Immunol. 2012;13:144-51.
- Wilson MS, Ramalingam TR, Rivollier A, et al. Colitis and intestinal inflammation in IL10-/- mice results from IL-13Ra2-mediated attenuation of IL-13 activity. Gastroenterology. 2011;140:254-64.
- Nikolaus S, Schulte B, Al-Massad N, et al. Increased tryptophan metabolism is associated with activity of inflammatory bowel diseases. Gastroenterology. 2017;153:1504-1516.e2.
- Schirmer M, Franzosa EA, Lloyd-Price J, et al. Dynamics of metatranscription in the inflammatory bowel disease gut microbiome. Nat Microbiol. 2018;3:337-46.
- Investigators IBDMDB, Lloyd-Price J, Arze C, Ananthakrishnan AN, et al. Multi-omics of the gut microbial ecosystem in inflammatory bowel diseases. Nature. 2019;569:655-62.
- Jang YJ, Kim W-K, Han DH, et al. Lactobacillus fermentum species ameliorate dextran sulfate sodium-induced colitis by regulating the immune response and altering gut microbiota. Gut Microbes. 2019;10:696-711.
- Atarashi K, Tanoue T, Oshima K, et al. Treg induction by a rationally selected mixture of Clostridia strains from the human microbiota. Nature. 2013;500:232-6.
- Ehrlich AM, Pacheco AR, Henrick BM, et al. Indole-3-lactic acid associated with Bifidobacterium-dominated microbiota significantly decreases inflammation in intestinal epithelial cells. BMC Microbiol. 2020;20:357.
- Zelezniak A, Andrejev S, Ponomarova O, et al. Metabolic dependencies drive species co-occurrence in diverse microbial communities. Proc Natl Acad Sci U S A. 2015;112:6449-54.
- Meng D, Sommella E, Salviati E, et al. Indole-3-lactic acid, a metabolite of tryptophan, secreted by Bifidobacterium longum subspecies infantis is anti-inflammatory in the immature intestine. Pediatr Res. 2020;88:209-17.
- Menni C, Hernandez MM, Vital M, et al. Circulating levels of the antioxidant indoleproprionic acid are associated with higher gut microbiome diversity. Gut Microbes. 2019;10:688-95.
- Zmora N, Zilberman-Schapira G, Suez J, et al. Personalized gut mucosal colonization resistance to empiric probiotics is associated with unique host and microbiome features. Cell. 2018;174:1388-1405.e21.
Publisher’s Note
- *Correspondence:
Xiangfang Zeng
ziyangzxf@163.com
Full list of author information is available at the end of the article - (See figure on next page.)
Fig. 1 L. reuteri alters the intestinal microbiota composition and microbial tryptophan metabolite. A Differential enrichment of the mouse colonic microbiota in response to. reuteri supplementation on day 28 (see the Figure S1A legend for the experimental scheme). The taxonomic classifications of the bacteria are shown in the left panel. Blanks are unassigned taxa. The 36 differentially enriched bacterial taxa are ranked by estimating the mean decrease in accuracy based on random forest analysis. The right panel shows the LEfSe analysis of the 36 bacterial taxa. B Serum concentrations of metabolites in tryptophan metabolism among three groups of mice ( ). C Volcano plot showing differential gene expression in the colon of DSS-treated mice with or without L. reuteri supplementation. Each red dot indicates a significantly upregulated gene, while a blue dot represents a significantly downregulated gene, with each gray dot showing a gene with no significant difference. D Pairwise comparisons of tryptophan metabolites, with a color gradient denoting Spearman’s correlation coefficient. The correlation between the bacterial/ gene expression profiles and each metabolite using partial Mantel tests. The edge width corresponds to Mantel’s R statistic for the corresponding distance correlations, and the edge color denotes statistical significance. E Real-time PCR assay showing the expression of Pxr and its target genes , and - (See figure on next page.)
Fig. 4 Indole derivatives alleviate intestinal inflammation inmice. Six-week-old mice were orally gavaged daily with or without ILA ( ), IPA ( ), or IAA ( ) for 4 weeks ( ). Congenic wild-type (WT) mice were also gavaged daily for 4 weeks as negative controls. A Colonic mRNA expression levels of proinflammatory cytokines (IL-6 and IL-1 ) ( ) by RT-qPCR. B The levels of TNF-a and LPS in the colon ( ) using ELISA. C PCoA plot of the Bray-Curtis distance of the colonic microbiota among different groups of mice. D Heatmap showing the relative abundance of different bacterial genera. E Co-occurrence network analysis of bacterial genera among different groups of mice. Edges representing significant SparCC correlations indicate and . Each light blue line represents a significant negative correlation, while each light red line represents a significant positive correlation. The size of the points represents the degree of the node. The thickness of the line is proportional to the degree of correlation. ; ns, not significant
