DOI: https://doi.org/10.1186/s12967-023-04840-x
PMID: https://pubmed.ncbi.nlm.nih.gov/38246999
تاريخ النشر: 2024-01-21
دمج الصيدلة الشبكية والتحقق التجريبي على الحيوانات لدراسة آلية عمل حمض الأولينوليك في السمنة
الملخص
خلفية: السمنة، وهي حالة مرتبطة بتطور أمراض القلب والأوعية الدموية واسعة الانتشار، والاضطرابات الأيضية، ومضاعفات صحية أخرى، أصبحت قضية صحية عالمية هامة. حمض الأوليانوليك (OA)، وهو مركب ثلاثي الحلقات موجود على نطاق واسع في العديد من النباتات الطبيعية، أظهر خصائص محتملة مضادة للالتهابات ومضادة للتصلب العصيدي. ومع ذلك، لم يتم دراسة الآلية التي يقاوم بها OA السمنة بشكل جيد.
الطريقة: تم استخدام الصيدلة الشبكية للبحث عن الأهداف والمسارات المحتملة لـ OA ضد السمنة. تم استخدام تقنية الربط الجزيئي والمحاكاة الديناميكية الجزيئية للتحقق من تفاعل OA مع الأهداف الأساسية، ثم تم استخدام نموذج حيواني للسمنة الناتجة عن تناول الدهون العالية لتأكيد الأهداف الأكثر مركزية. النتائج: درست دراسة الصيدلة الشبكية 42 هدفًا مهمًا لـ OA لعلاج السمنة. تم تحديد العمليات البيولوجية الرئيسية (BP) والمكونات الخلوية (CC) والوظائف الجزيئية (MF) لـ OA لمكافحة السمنة باستخدام تحليل إثراء GO، بما في ذلك إشارات مستقبلات داخل الخلايا، وإشارات مستقبلات الهرمونات الستيرويدية داخل الخلايا، والكروماتين، والنواة، ومجمع المستقبلات، وغشاء الشبكة الإندوبلازمية، ونشاط عامل النسخ RNA polymerase II. وجدت دراسة إثراء قاعدة بيانات KEGG/DAVID أن المسارات الأيضية، ومسارات إشارات PPAR، ومسارات السرطان/مسارات إشارات PPAR، ومقاومة الأنسولين، وتكوين الستيرويدات المبيضية تلعب جميعها أدوارًا أساسية في علاج السمنة وOA. تم استخدام شبكة التفاعل بين البروتينات (PPI) لتصفية تسعة أهداف رئيسية: PPARG، PPARA، MAPK3، NR3C1، PTGS2، CYP19A1، CNR1، HSD11B1، وAGTR1. باستخدام تقنية الربط الجزيئي، تم التحقق من آلية الربط المحتملة ودرجة الربط بين OA وكل هدف مهم، مما يوضح أن OA لديه قدرة ربط جيدة مع كل هدف. أظهرت محاكاة الديناميكا الجزيئية انحراف الجذر المتوسط (RMSD) ونصف قطر الدوران (Rg) أن OA لديه استقرار ربط قوي مع كل هدف. أكدت دراسات حيوانية إضافية أهمية الهدف الأساسي PPARG والمسار الأساسي لمسار إشارات PPAR في مكافحة السمنة بواسطة OA. الخلاصة: بشكل عام، استخدمت دراستنا نهجًا متعدد الأوجه للتحقيق في قيمة وآليات OA في علاج السمنة، مما يوفر أساسًا جديدًا لتحديد وتطوير العلاجات الدوائية الطبيعية.
المقدمة
حمض الأوليانوليك (OA)، وهو ثلاثي الحلقات، هو مكون نشط في الأعشاب مثل Fructus Ligustri Lucidi وFolium Camellia Sinensis، وكذلك في العديد من الأطعمة الشائعة بما في ذلك أوراق الزيتون، والتفاح، والعنب، والزنجبيل، والمانجو [12]. في الصين، تم استخدام OA كدواء واقي للكبد [13]، وهناك دليل سريري على فعاليته في علاج فرط شحميات الدم [14]. علاوة على ذلك، أظهرت دراسات سريرية متعددة إمكانيات مختلفة لمشتقات OA في منع أو علاج مجموعة من الأمراض، بما في ذلك السرطان، والسكري، والعدوى الفيروسية [13، 15، 16]. أظهرت الأبحاث السابقة أن OA يمتلك خصائص دوائية متنوعة، مثل التأثيرات المضادة للفيروسات، والمضادة للبكتيريا، والمضادة للسرطان، والمضادة للالتهابات، ومضادة للأكسدة، والواقية للكبد، والواقية للجهاز الهضمي [17-19]. لقد أظهر OA تحسين التغيرات الشاذة في معايير الدهون، وتقليل الكبد الدهني المجهري، وزيادة محتوى اللبتين، وتقليل الدهون الحشوية بشكل كبير، وتحسين تحمل الجلوكوز، وزيادة مستويات الأنسولين [20،21]. كما قلل من الالتهاب الجهازي، وعزز تكوين الدهون في الكبد، وزاد من إدراك طعم الدهون الغذائية في الفئران التي تتغذى على نظام غذائي عالي الدهون [21]. في الوقت نفسه، تظهر مشتقات OA، مثل Nano-OA، تأثيرات مشابهة في خفض الدهون [22]. على الرغم من تأكيد تأثير OA في خفض الدهون [23، 24]، إلا أن المزيد من الأبحاث مطلوبة لتوضيح
الآليات الكامنة وراء دوره المحتمل في مكافحة السمنة.
النتائج
تحليل الصيدلة الشبكية
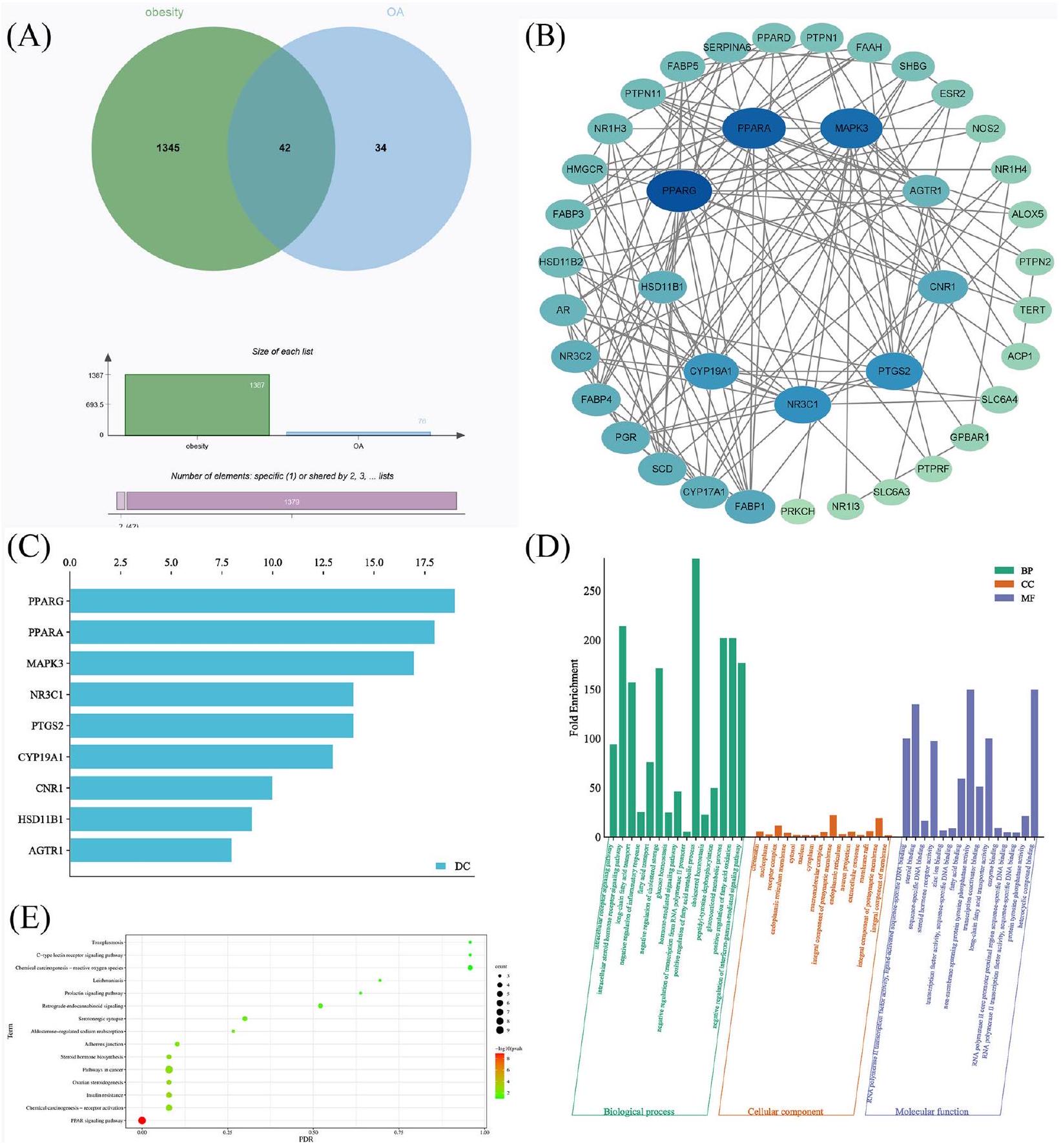
قد تكون دراسات قاعدة البيانات المرتبطة بقاعدة البيانات مرتبطة بتحديث قاعدة البيانات. ركزنا على مسار إشارة PPAR استنادًا إلى نتائج إثراء الأهداف الأساسية والمسارات، ويظهر الشكل التخطيطي لـ OA في علاج السمنة من خلال استهداف مسار إشارة PPAR في الشكل 2. تشير هذه النتائج إلى أن OA يمكن أن تظهر فوائد مضادة للسمنة عبر مجموعة متنوعة من الطرق، حيث يلعب مسار إشارة PPAR دورًا رئيسيًا في عملية OA المضادة للسمنة.
الربط الجزيئي
| معرف KEGG | مسار الإشارة | عد |
| Hsa01100 | مسارات الأيض – الإنسان العاقل | 10 |
| Hsa03320 | مسار إشارة PPAR – الإنسان (هومو سابينس) | ٩ |
| hsa05200 | مسارات السرطان – الإنسان العاقل | 9 |
| hsa05207 | السرطنة الكيميائية – تنشيط المستقبلات – الإنسان هومو سابينس | ٦ |
| hsa04931 | مقاومة الأنسولين – إنسان هومو سابينس | ٥ |
| hsa04520 | وصلة الالتصاق – الإنسان العاقل | ٤ |
| hsa04723 | إشارات الكانابينويد العكسية – الإنسان العاقل | ٤ |
| hsa00140 | تخليق هرمونات الستيرويد – الإنسان العاقل | ٤ |
| hsa05022 | مسارات التنكس العصبي – أمراض متعددة – إنسان هومو سابينس | ٤ |
| hsa05208 | السرطان الكيميائي – أنواع الأكسجين التفاعلية – الإنسان هومو سابينس | ٤ |
| أهداف | RMSD |
| PPARG | 0.018 |
| PPARA | 0.038 |
| MAPK3 | 0.031 |
| NR3C1 | 0.006 |
| PTGS2 | 0.037 |
| CYP19A1 | 0.532 |
| CNR1 | 0.027 |
| HSD11B1 | 0.019 |
| AGTR1 | 0.028 |
محاكاة الديناميكا الجزيئية
| أهداف | طاقة الربط (كج/مول) | التفاعلات الكارهة للماء | روابط الهيدروجين | جسور الملح |
| PPARG | -24.85 | أرج 262، ألا 263، ثر 266 | – | ليسين 185، أرجينين 262 |
| PPARA | -٢٦.١٥ | ليس 116، ليس 116 | – CYS 128 | – |
| MAPK3 | -24.18 | فال 63، أرج 64، أرج 64، ليس 65، برو 193 | ASN 161، PHE 346، THR 347 | – |
| NR3C1 | -24.64 | GLU 540، GLU 540، ARG 611، TYR 660 | TRP 610، TYR 660 | – |
| PTGS2 | -26.15 | PHE 205، VAL 344، TYR 348 | تي آر 385، جلايسين 526، جلايسين 533، ليوسين 534 | – |
| CYP19A1 | -29.20 | GLN 225، LYS 243، TYR 244، TYR 244، ASP 476 | تي آر 244، آسب 476 | ليس 243 |
| CNR1 | -22.55 | PHE 208، LEU 209، ILE 212، ALA 236، MET 240، LEU 286 | – | – |
| HSD11B1 | -٣٤.١٤ | إيل 121، ليو 126، تير 177، فال 180، ليو 217، ثر 222، ألا 223، ألا 226، إيل 230 | – | – |
| AGTR1 | -31.59 | TRP 84، PHE 182، LEU 195، LEU 195، LYS 199، THR 260 | THR 260 | لي 199، هيس 256 |
تجربة حيوانية
OA يمنع زيادة الوزن في الفئران البدينة
تنظم OA اضطرابات استقلاب الجليكوليبيد
OA يحسن التغيرات النسيجية المرضية في الفئران التي تتغذى على نظام غذائي عالي الدهون
OA يخفف من استجابة الالتهاب الناتجة عن النظام الغذائي عالي الدهون
OA يزيد من إفراز GLP-1 عن طريق تنظيم TGR5
خفض النظام الغذائي تعبير TGR5، مع بيانات أقل بكثير في مجموعة النموذج (
يؤثر OA على تخليق الدهون عبر PPAR
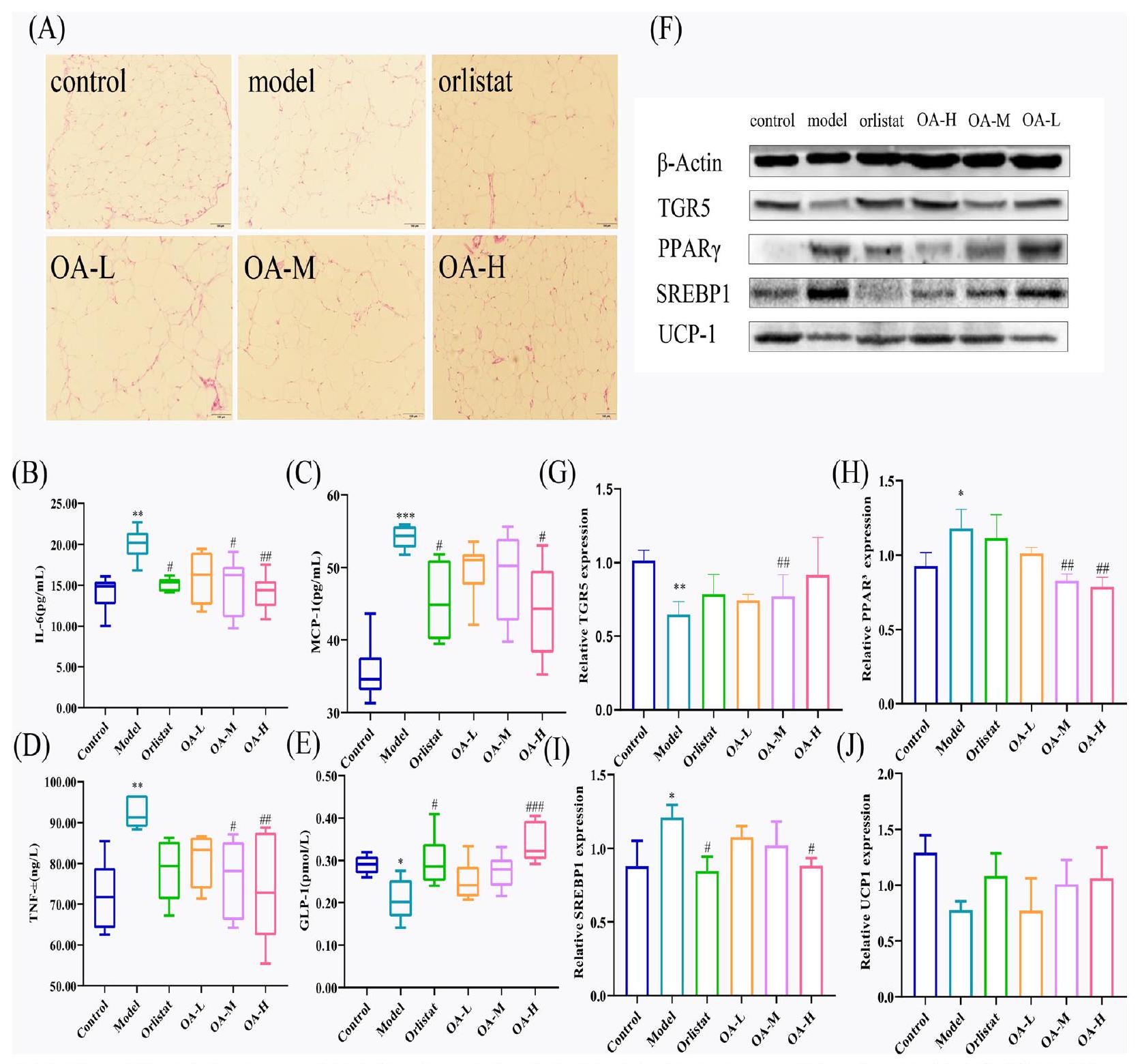
OA تؤثر على استهلاك الطاقة مع TGR5/UCP-1
تعزيز تعبير بروتين TGR5 / UCP-1، مما يسرع من استهلاك الطاقة. ومع ذلك، لا يزال يتعين التحقق من صحة هذه الفرضية من خلال أبحاث أخرى.
طرق
تحليل الصيدلة الشبكية
الحصول على الأهداف المرتبطة بالتهاب المفاصل والسمنة
للحصول على الأهداف المقابلة لـ OA (احتمالية > 0). بعد ذلك، قاعدة بيانات GeneCards (https://www. genecards.org/) [31] تم البحث عن الأهداف المتعلقة بالمرض باستخدام الكلمة الرئيسية “السمنة”، تلا ذلك تطبيق الفلاتر (درجة الصلة > 2) لاستخراج المجموعة النهائية من أهداف المرض. ثم تم تحليل القائمتين من الأهداف التي تم الحصول عليها باستخدام منصة المعلوماتية الحيوية (http://www. bioinformatics.com.cn/) لتحديد الأهداف التي تتداخل بين OA والسمنة.
بناء شبكة تفاعل البروتينات (PPI)
فحص الأهداف الأساسية لـ OA المضادة للسمنة
تحليل إثراء علم الجينات (GO)
تحليل إثراء مسار KEGG
تحليل التداخل الجزيئي
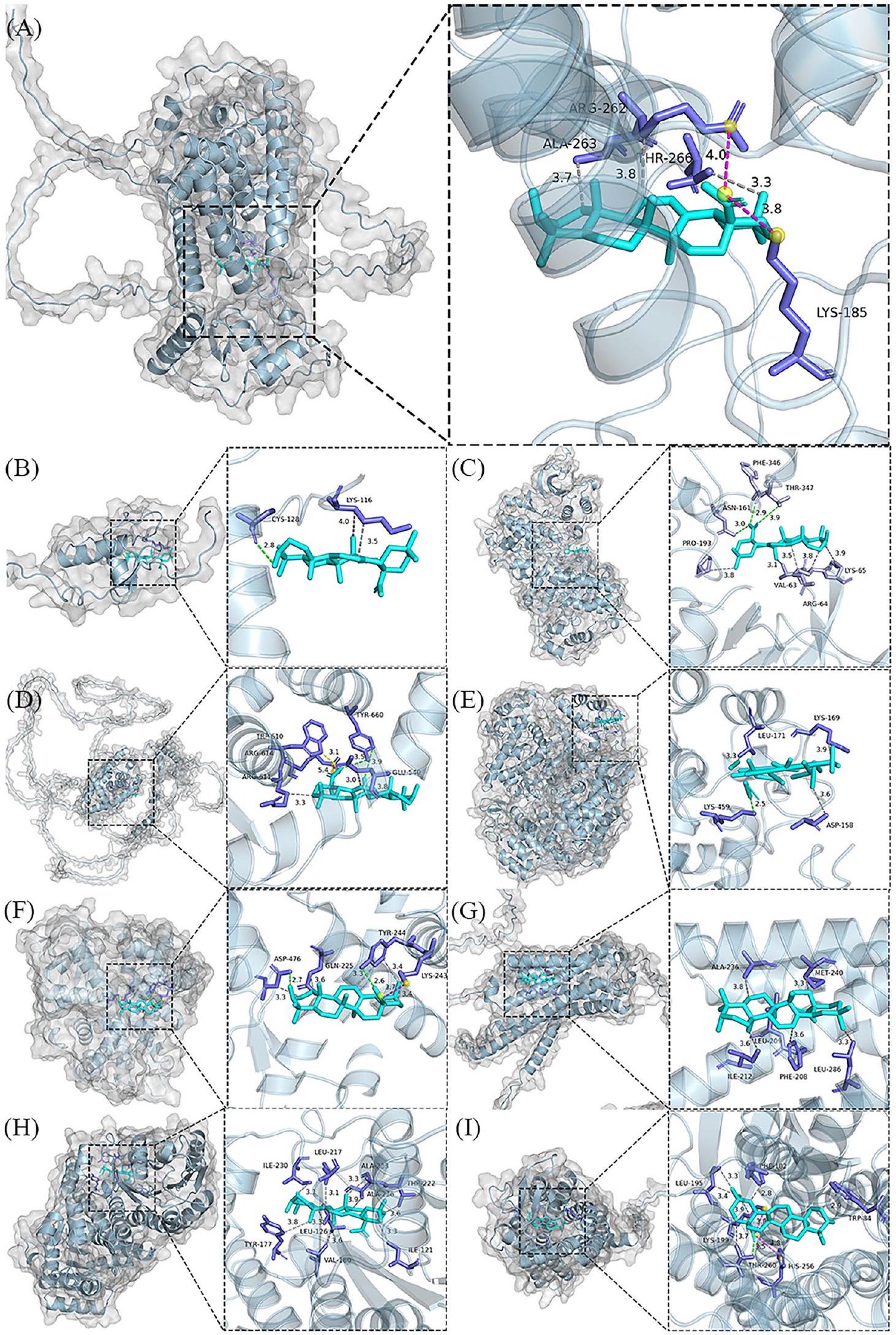
واستخدمت OA كروابط للتداخل الجزيئي عبر AutoDock. في البداية، تم الحصول على الهيكل ثلاثي الأبعاد لـ OA بتنسيق “mol2” من قاعدة بيانات نظام الطب التقليدي الصيني ومنصة التحليل (TCMSP، https://old.tcmsp-e.com/tcmsp.php) [37]، ثم تم حفظه بتنسيق “PDB” باستخدام Pymol 2.5.7 (Portland، OR، الولايات المتحدة). تم تنزيل هيكل البروتين للأهداف الأساسية بتنسيق “PDB” من قواعد بيانات البروتين PDB (https:// www.rcsb.org) [38] وقواعد بيانات alphafold (https:// alphafold.com) [39]. بعد ذلك، تمت إزالة الروابط لجزيئات الهدف باستخدام برنامج Pymol، وتم معالجة الجزيئات مسبقًا باستخدام أداة AutoDock 1.5.7، بما في ذلك الهيدروجين، وإضافة الإلكترونات، وإضافة الجذر. في الخطوة النهائية، تم استخدام برنامج AutoDock 4.2.6 للتداخل الجزيئي شبه المرن، وتم تقييم إمكانات الربط بواسطة درجة الارتباط. ثم تم اختيار نموذج التداخل الأمثل، وتم تحليل التفاعلات غير التساهمية لمجمع البروتين-الرابط على المستوى الذري بواسطة أداة الويب Protein-ligand Interaction Profiler (PLIP) (https://plip-tool. biotec.tu-dresden.de/plip-web/plip/index) [40]. أخيرًا، تم إجراء إعادة التداخل لتقييم أنماط الربط قبل التداخل الجزيئي الرسمي، تلاها تحليل بصري باستخدام برنامج Pymol.
محاكاة الديناميكا الجزيئية
اختبار في الجسم الحي
المواد الكيميائية والأجسام المضادة
الحيوانات التجريبية وظروف التغذية
التجميع وإقامة النموذج
الإدارة والتعامل
حساب زيادة الوزن وقياس وزن الكبد والدهون الإبيديديمية
قياسات مستويات الجلوكوز في الدم والدهون
الدراسات النسيجية
اختبار الامتصاص المناعي المرتبط بالإنزيم (ELISA)
البلوت الغربي
تم استخدام تقنية الفصل الكهربائي للهلام بولي أكريلاميد كبريتات الصوديوم (SDS-PAGE) لفصل البروتينات، والتي تم نقلها بعد ذلك إلى أغشية فلوريد البولي فينيليدين (PVDF). تم تحضين الأغشية طوال الليل مع الأجسام المضادة الأولية والثانوية المخففة في TBST. تم إجراء تصوير كيميائي ضوئي عن طريق إضافة قطرات المطور إلى غشاء PVDF. مستويات تعبير البروتين لـ PPAR
التحليل الإحصائي
- الموافقة: تم الموافقة على تجارب الحيوانات في هذه الدراسة من قبل لجنة أخلاقيات الحيوانات في جامعة قوانغدونغ للصيدلة.
- وفقًا: تم تنفيذ الطرق وفقًا للإرشادات واللوائح المعنية.
نقاش
تأثير OA على الأهداف الأساسية باستخدام طرق الربط الجزيئي، وتم التحقق من استقرار الربط وسعة الربط باستخدام محاكاة الديناميكا الجزيئية. في الوقت نفسه، تم استخدام التجارب الحيوانية للتحقق من الأهداف الأساسية والمسارات.
التمثيل الغذائي، هجرة الخلايا، وتمايز الخلايا [62]. يلعب دورًا في العديد من الأمراض مثل تمثيل الدهون الكبدي، تمثيل الدهون في الكبد، واضطرابات التمثيل الغذائي القلبي، إلخ [63، 64]. أظهرت الأبحاث السابقة أن اللبتين، وحمض البروتوكاتيكويك، وجزيئات أخرى يمكن أن تحارب السمنة وتصلب الشرايين من خلال MAPK3/ ERK1 [65، 66]. NR3C1 (عضو مجموعة 3 من عائلة المستقبلات النووية 1)، الذي يشفر مستقبل الجلوكوكورتيكويد، يشارك في الاستجابة الالتهابية، تكاثر الخلايا، وتمايزها في الأنسجة المستهدفة [67]. وقد تم الإبلاغ عن أنه يمارس تأثيرات مفيدة على السمنة، واضطراب تمثيل الجلوكوز، والخلل الدهني [68-70]. وبالمثل، يُتوقع أن يكون PTGS2 (سينثاز البروستاجلاندين-إنيدروكسي 2) وCYP19A1 (عضو عائلة السيتوكروم P450 19 فرع A 1) أهدافًا محورية لتقليل أو عكس فرط شحميات الدم والسمنة [68، 71، 72]. بالإضافة إلى ذلك، تم تحديد CNR1 (مستقبل الكانابينويد 1) كهدف دوائي واعد لعلاج السمنة، حيث أدى حذف CNR1 في الفئران إلى تحسين مقاومة الأنسولين، وإجهاد ER، وتراكم الدهون [73]. على العكس، وُجد أن الإفراط في التعبير عن HSD11B1 (إيزوزيم 11-بيتا-هيدروكسيستيرود ديهيدروجيناز) الذي يحفز تحويل الشكل النشط من الكورتيزول [74]، يؤدي إلى السمنة الحشوية، ومرض السكري المقاوم للأنسولين، والخلل الدهني [75]. AGTR1 (مستقبل الأنجيوتنسين II من النوع 1) هو لاعب رئيسي في نظام الرينين-أنجيوتنسين (RAS) الذي يمكن أن يزيد من ضغط الدم ومقاومة الأنسولين بينما يمنع أيضًا تحلل الدهون، ويحافظ على توازن الطاقة، ويقلل الالتهاب [76]. تم استخدام الربط الجزيئي والمحاكاة الديناميكية الجزيئية للتحقق من الآلية الجزيئية لتدخل OA في السمنة. كشفت دراسات الربط الجزيئي أن OA لديه affinity عالية للأهداف الرئيسية التي حددتها الصيدلة الشبكية. أظهرت نتائج المحاكاة الديناميكية الجزيئية أن ارتباط OA بالأهداف الأساسية كان أكثر استقرارًا، مما يشير إلى أن البروتينات الأساسية التي تم افتراضها سابقًا كانت بالفعل الروابط الحاسمة في عمل OA المضاد للسمنة.
يمكن أن يحسن كل من الاضطرابات الالتهابية وعمليات التمثيل الغذائي للغلوكوز والدهون. تشير هذه النتيجة إلى أن قدرة OA على مكافحة السمنة قد تتضمن هذه الآلية.
في الختام، تجمع هذه الدراسة بين علم الأدوية الشبكي، والربط الجزيئي، والمحاكاة الديناميكية الجزيئية، والتحقق التجريبي على الحيوانات لأول مرة لدراسة فعالية وآلية عمل OA في علاج السمنة. تكشف النتائج عن إمكانيات OA في علاج السمنة وتوفر اتجاهًا جديدًا لتطوير أدوية وأطعمة مشتقة من OA، مما يساهم في اكتشاف وتطوير الموارد الطبيعية المتعلقة بالسمنة. علاوة على ذلك، استكشفت هذه الدراسة تأثيرات الأدوية على السمنة من خلال تراكم الدهون، مما يوفر للباحثين رؤى جديدة لتطوير أدوية السمنة. لا يمكن إنكار أن هناك بعض القيود في دراستنا. يعتمد علم الأدوية الشبكي، والربط الجزيئي، والمحاكاة الديناميكية الجزيئية على البيانات والخوارزميات، وقد تختلف نتائجها عن النتائج الفعلية بسبب قيود قاعدة البيانات والبرمجيات. بالإضافة إلى ذلك، بسبب قيود الوقت والموارد، لم نتمكن من تأكيد جميع الأهداف المتوقعة تجريبيًا، أو دمج الأبحاث على الحيوانات والخلايا مع التحقق السريري وطرق أخرى، مما يمنعنا من الكشف الكامل عن آلية عمل OA المضادة للسمنة. سنقوم بإجراء اختبارات إضافية في المستقبل لفحص المسارات الجزيئية المحتملة وراء تأثيرات OA المضادة للسمنة بمزيد من التفصيل.
الخاتمة
معلومات إضافية
الشكر والتقدير
مساهمات المؤلفين
التمويل
توفر البيانات والمواد
الإعلانات
موافقة الأخلاقيات والموافقة على المشاركة
الموافقة على النشر
المصالح المتنافسة
تفاصيل المؤلف
تم النشر عبر الإنترنت: 21 يناير 2024
References
- Upadhyay J, Farr O, Perakakis N, Ghaly W, Mantzoros C. Obesity as a disease. Med Clin N Am. 2018;102:13-33.
- Fontaine KR, Redden DT, Wang C, Westfall AO, Allison DB. Years of life lost due to obesity. JAMA. 2003;289:187-93.
- Afshin A, Forouzanfar MH, Reitsma MB, Sur P, Estep K, Lee A, Marczak L, Mokdad AH, Moradi-Lakeh M, Naghavi M, Salama JS, Vos T, Abate KH, Abbafati C, Ahmed MB, Al-Aly Z, Alkerwi A, Al-Raddadi R, Amare AT, Amberbir A, Amegah AK, Amini E, Amrock SM, Anjana RM, Arnlov J, Asayesh H, Banerjee A, Barac A, Baye E, Bennett DA, Beyene AS, Biadgilign S, Biryukov S, Bjertness E, Boneya DJ, Campos-Nonato I, Carrero JJ, Cecilio P, Cercy K, Ciobanu LG, Cornaby L, Damtew SA, Dandona L, Dandona R, Dharmaratne SD, Duncan BB, Eshrati B, Esteghamati A, Feigin VL, Fernandes JC, Furst T, Gebrehiwot TT, Gold A, Gona PN, Goto A, Habtewold TD, Hadush KT, Hafezi-Nejad N, Hay SI, Horino M, Islami F, Kamal R, Kasaeian A, Katikireddi SV, Kengne AP, Kesavachandran CN, Khader YS, Khang YH, Khubchandani J, Kim D, Kim YJ, Kinfu Y, Kosen S, Ku T, Defo BK, Kumar GA, Larson HJ, Leinsalu M, Liang
4. Boutari C, Mantzoros CS. A 2022 update on the epidemiology of obesity and a call to action: as its twin COVID-19 pandemic appears to be receding, the obesity and dysmetabolism pandemic continues to rage on. Metabolism. 2022;133: 155217.
5. Chooi YC, Ding C, Magkos F. The epidemiology of obesity. Metabolism. 2019;92:6-10.
6. Kivimaki M, Strandberg T, Pentti J, Nyberg ST, Frank P, Jokela M, Ervasti J, Suominen SB, Vahtera J, Sipila PN, Lindbohm JV, Ferrie JE. Body-mass index and risk of obesity-related complex multimorbidity: an observational multicohort study. Lancet Diabetes Endocrinol. 2022;10:253-63.
7. Reid TJ, Korner J. Medical and surgical treatment of obesity. Med Clin North Am. 2022;106:837-52.
8. Walter CP, Bleske BE, Dorsch MP. Pharmacotherapy for weight loss: the cardiovascular effects of the old and new agents. J Clin Pharm Ther. 2014;39:475-84.
9. Lan JP, Tong RC, Sun XM, Zhang HY, Sun S, Xiong AZ, Wang ZT, Yang L. Comparison of the main chemical composition of Plantago asiatica L . and P. depressa Willd. seed extracts and their anti-obesity effects in highfat diet-induced obese mice. Phytomedicine. 2021;81: 153362.
10. Martel J, Ojcius DM, Chang CJ, Lin CS, Lu CC, Ko YF, Tseng SF, Lai HC, Young JD. Anti-obesogenic and antidiabetic effects of plants and mushrooms. Nat Rev Endocrinol. 2017;13:149-60.
11. Wan Y, Xia J, Xu JF, Chen L, Yang Y, Wu JJ, Tang F, Ao H, Peng C. Nuciferine, an active ingredient derived from lotus leaf, lights up the way for the potential treatment of obesity and obesity-related diseases. Pharmacol Res. 2022;175: 106002.
12. Castellano JM, Espinosa JM, Perona JS. Modulation of lipid transport and adipose tissue deposition by small lipophilic compounds. Front Cell Dev Biol. 2020;8: 555359.
13. Yang YH, Dai SY, Deng FH, Peng LH, Li C, Pei YH. Recent advances in medicinal chemistry of oleanolic acid derivatives. Phytochemistry. 2022;203: 113397.
14. Luo HQ, Shen J, Chen CP, Ma X, Lin C, Ouyang Q, Xuan CX, Liu J, Sun HB, Liu J. Lipid-lowering effects of oleanolic acid in hyperlipidemic patients. Chin J Nat Med. 2018;16:339-46.
15. Liu D, Jin X, Yu G, Wang M, Liu L, Zhang W, Wu J, Wang F, Yang J, Luo Q, Cai L, Yang X, Ke X, Qu Y, Xu Z, Jia L, Chen WL. Oleanolic acid blocks the purine salvage pathway for cancer therapy by inactivating SOD1 and stimulating lysosomal proteolysis. Mol Ther Oncolytics. 2021;23:107-23.
16. Santos-Lozano JM, Rada M, Lapetra J, Guinda A, Jimenez-Rodriguez MC, Cayuela JA, Angel-Lugo A, Vilches-Arenas A, Gomez-Martin AM, OrtegaCalvo M, Castellano JM. Prevention of type 2 diabetes in prediabetic patients by using functional olive oil enriched in oleanolic acid: The PREDIABOLE study, a randomized controlled trial. Diabetes Obes Metab. 2019;21:2526-34.
17. de Melo CL, Queiroz MGR, Fonseca SGC, Bizerra AMC, Lemos TLG, Melo TS, Santos FA, Rao VS. Oleanolic acid, a natural triterpenoid that improves blood glucose tolerance in normal mice and ameliorates visceral obesity in mice fed a high-fat diet. Chem-Biol Interact. 2010;185:59-65.
18. Dzubak P, Hajduch M, Vydra D, Hustova A, Kvasnica M, Biedermann D, Markova L, Urban M, Sarek J. Pharmacological activities of natural triterpenoids and their therapeutic implications. Nat Prod Rep. 2006;23:394-411.
19. Kaushik S, Dar L, Kaushik S, Yadav JP. Anti-dengue activity of supercritical extract and isolated oleanolic acid of Leucas cephalotes using in vitro and silico approach. Bmc Complement Med. 2021;21:227.
20. de Moura EDM, Dos Reis SA, da Conceicao LL, Sediyama C, Pereira SS, de Oliveira LL, Gouveia Peluzio MDC, Martinez JA, Milagro Fl. Diet-induced
obesity in animal models: points to consider and influence on metabolic markers. Diabetol Metab Syndr. 2021;13:32.
21. Djeziri FZ, Belarbi M, Murtaza B, Hichami A, Benammar C, Khan NA. Oleanolic acid improves diet-induced obesity by modulating fat preference and inflammation in mice. Biochimie. 2018;152:110-20.
22. Wang S, Du LB, Jin L, Wang Z, Peng J, Liao N, Zhao YY, Zhang JL, Pauluhn J, Hai CX, Wang X, Li WL. Nano-oleanolic acid alleviates metabolic dysfunctions in rats with high fat and fructose diet. Biomed Pharmacother. 2018;108:1181-7.
23. Liu J. Pharmacology of oleanolic acid and ursolic acid. J Ethnopharmacol. 1995;49:57-68.
24. Claro-Cala CM, Jimenez-Altayo F, Zagmutt S, Rodriguez-Rodriguez R. Molecular mechanisms underlying the effects of olive oil triterpenic acids in obesity and related diseases. Nutrients. 2022;14:1606.
25. Jiao W, Mi S, Sang Y, Jin Q, Chitrakar B, Wang X, Wang S. Integrated network pharmacology and cellular assay for the investigation of an anti-obesity effect of 6-shogaol. Food Chem. 2022;374: 131755.
26. Xia QD, Xun Y, Lu JL, Lu YC, Yang YY, Zhou P, Hu J, Li C, Wang SG. Network pharmacology and molecular docking analyses on Lianhua Qingwen capsule indicate Akt1 is a potential target to treat and prevent COVID-19. Cell Proliferat. 2020;53: e12949.
27. Yang B, Yang N, Chen Y, Zhu M, Lian Y, Xiong Z, Wang B, Feng L, Jia X. An integrated strategy for effective-component discovery of astragali radix in the treatment of lung cancer. Front Pharmacol. 2020;11: 580978.
28. Singh A, Vanga SK, Orsat V, Raghavan V. Application of molecular dynamic simulation to study food proteins: A review. Crit Rev Food Sci Nutr. 2018;58:2779-89.
29. Kim S, Chen J, Cheng T, Gindulyte A, He J, He S, Li Q, Shoemaker BA, Thiessen PA, Yu B, Zaslavsky L, Zhang J, Bolton EE. PubChem in 2021: new data content and improved web interfaces. Nucleic Acids Res. 2021;49:D1388-95.
30. Daina A, Michielin O, Zoete V. SwissTargetPrediction: updated data and new features for efficient prediction of protein targets of small molecules. Nucleic Acids Res. 2019;47:W357-64.
31. Stelzer G, Rosen N, Plaschkes I, Zimmerman S, Twik M, Fishilevich S, Stein TI, Nudel R, Lieder I, Mazor Y, Kaplan S, Dahary D, Warshawsky D, Guan-Golan Y, Kohn A, Rappaport N, Safran M, Lancet D. The GeneCards Suite: From Gene Data Mining to Disease Genome Sequence Analyses, Curr Protoc Bioinformatics, 2016;54, 130 31-31 3033.
32. Szklarczyk D, Gable AL, Nastou KC, Lyon D, Kirsch R, Pyysalo S, Doncheva NT, Legeay M, Fang T, Bork P, Jensen LJ, von Mering C. The STRING database in 2021: customizable protein-protein networks, and functional characterization of user-uploaded gene/measurement sets. Nucleic Acids Res. 2021;49:D605-12.
33. Shannon P, Markiel A, Ozier O, Baliga NS, Wang JT, Ramage D, Amin N, Schwikowski B, Ideker T. Cytoscape: a software environment for integrated models of biomolecular interaction networks. Genome Res. 2003;13:2498-504.
34. Tang Y, Li M, Wang J, Pan Y, Wu FX. CytoNCA: a cytoscape plugin for centrality analysis and evaluation of protein interaction networks. Biosystems. 2015;127:67-72.
35. Sherman BT, Hao M, Qiu J, Jiao X, Baseler MW, Lane HC, Imamichi T, Chang W, DAVID: a web server for functional enrichment analysis and functional annotation of gene lists,. update). Nucleic Acids Res. 2021;2022. https://doi.org/10.1093/nar/gkac194.
36. UniProt C. UniProt: the universal protein knowledgebase in 2021. Nucleic Acids Res. 2021;49:D480-9.
37. Ru J, Li P, Wang J, Zhou W, Li B, Huang C, Li P, Guo Z, Tao W, Yang Y, Xu X, Li Y, Wang Y, Yang L. TCMSP: a database of systems pharmacology for drug discovery from herbal medicines. J Cheminform. 2014;6:13.
38. Burley SK, Bhikadiya C, Bi C, Bittrich S, Chen L, Crichlow GV, Christie CH, Dalenberg K, Di Costanzo L, Duarte JM, Dutta S, Feng Z, Ganesan S, Goodsell DS, Ghosh S, Green RK, Guranovic V, Guzenko D, Hudson BP, Lawson CL, Liang Y, Lowe R, Namkoong H, Peisach E, Persikova I, Randle C, Rose A, Rose Y, Sali A, Segura J, Sekharan M, Shao C, Tao YP, Voigt M, Westbrook JD, Young JY, Zardecki C, Zhuravleva M. RCSB Protein Data Bank: powerful new tools for exploring 3D structures of biological macromolecules for basic and applied research and education in fundamental biology, biomedicine, biotechnology, bioengineering and energy sciences. Nucleic Acids Res. 2021;49:D437-51.
39. Jumper J, Evans R, Pritzel A, Green T, Figurnov M, Ronneberger O, Tunyasuvunakool K, Bates R, Zidek A, Potapenko A, Bridgland A, Meyer C, Kohl SAA, Ballard AJ, Cowie A, Romera-Paredes B, Nikolov S, Jain R, Adler J, Back T, Petersen S, Reiman D, Clancy E, Zielinski M, Steinegger M, Pacholska M, Berghammer T, Bodenstein S, Silver D, Vinyals O, Senior AW, Kavukcuoglu K, Kohli P, Hassabis D. Highly accurate protein structure prediction with AlphaFold. Nature. 2021;596:583-9.
40. Adasme MF, Linnemann KL, Bolz SN, Kaiser F, Salentin S, Haupt VJ, Schroeder M. PLIP 2021: expanding the scope of the proteinligand interaction profiler to DNA and RNA. Nucleic Acids Res. 2021;49:W530-4.
41. Song
42. Dong Y, Zheng Y, Zhu L, Li T, Guan Y, Zhao S, Wang Q, Wang J, Li L. Hua-Tan-Sheng-Jing decoction treats obesity with oligoasthenozoospermia by up-regulating the PI3K-AKT and down-regulating the JNK MAPK signaling pathways: at the crossroad of obesity and oligoasthenozoospermia. Front Pharmacol. 2022;13: 896434.
43. Su S, Wu G, Cheng X, Fan J, Peng J, Su H, Xu Z, Cao M, Long Z, Hao Y, Li G, Li S, Hai C, Wang X. Oleanolic acid attenuates PCBs-induced adiposity and insulin resistance via HNF1b-mediated regulation of redox and PPARgamma signaling. Free Radic Biol Med. 2018;124:122-34.
44. Li W, Zeng H, Xu M, Huang C, Tao L, Li J, Zhang T, Chen H, Xia J, Li C, Li X. Oleanolic acid improves obesity-related inflammation and insulin resistance by regulating macrophages activation. Front Pharmacol. 2021;12: 697483.
45. Pan Y, Zhou F, Song Z, Huang H, Chen Y, Shen Y, Jia Y, Chen J. Oleanolic acid protects against the pathogenesis of atherosclerosis, possibly via FXR-mediated angiotensin (Ang)-(1-7) upregulation. Biomed Pharmacother. 2018;97:1694-700.
46. Fernandez-Aparicio A, Schmidt-RioValle J, Perona JS, Correa-Rodriguez M, Castellano JM, Gonzalez-Jimenez E. Potential protective effect of oleanolic acid on the components of metabolic syndrome: a systematic review. J Clin Med. 2019;8:1294.
47. Li M, Han Z, Bei W, Rong X, Guo J, Hu X. Oleanolic acid attenuates insulin resistance via NF-kappaB to regulate the IRS1-GLUT4 pathway in HepG2 cells. Evid Based Complement Alternat Med. 2015;2015: 643102.
48. Nyakudya TT, Isaiah S, Ayeleso A, Ndhlala AR, Mukwevho E, Erlwanger KH. Short-Term neonatal oral administration of oleanolic acid protects against fructose-induced oxidative stress in the skeletal muscles of suckling rats. Molecules. 2019;24:661.
49. Teixeira LL, Pilon G, Coutinho CP, Dudonne S, Dube P, Houde V, Desjardins Y, Lajolo FM, Marette A, Hassimotto NMA. Purple grumixama anthocyanins (Eugenia brasiliensis Lam.) attenuate obesity and insulin resistance in high-fat diet mice. Food Funct. 2021;12:3680-91.
50. Shi L, Karrar E, Wang X. Sesamol ameliorates hepatic lipid accumulation and oxidative stress in steatosis HepG2 cells via the PPAR signaling pathway. J Food Biochem. 2021;45: e13976.
51. Wu L, Guo C, Wu J. Therapeutic potential of PPARgamma natural agonists in liver diseases. J Cell Mol Med. 2020;24:2736-48.
52. Robinson-Rechavi M, Carpentier AS, Duffraisse M, Laudet V. How many nuclear hormone receptors are there in the human genome? Trends Genet. 2001;17:554-6.
53. Montaigne D, Butruille L, Staels B. PPAR control of metabolism and cardiovascular functions. Nat Rev Cardiol. 2021;18:809-23.
54. Derosa G, Sahebkar A, Maffioli P. The role of various peroxisome prolifera-tor-activated receptors and their ligands in clinical practice. J Cell Physiol. 2018;233:153-61.
55. Botta M, Audano M, Sahebkar A, Sirtori CR, Mitro N, Ruscica M. PPAR agonists and metabolic syndrome: an established role? Int J Mol Sci. 2018;19:1197.
56. Pawlak M, Lefebvre P, Staels B. Molecular mechanism of PPAR alpha action and its impact on lipid metabolism, inflammation, and fibrosis in nonalcoholic fatty liver disease. J Hepatol. 2015;62:720-33.
57. Hu N, Chen CY, Wang JH, Huang J, Yao DH, Li CL. Atorvastatin ester regulates lipid metabolism in hyperlipidemia rats via the PPAR-signaling pathway and HMGCR expression in the liver. Int J Mol Sci. 2021;22:11107.
58. Magadum A, Engel FB. PPARbeta/delta: linking metabolism to regeneration. Int J Mol Sci. 2018;19:2013.
59. Braissant O, Foufelle F, Scotto C, Dauca M, Wahli W. Differential expression of peroxisome proliferator-activated receptors (PPARs): tissue distribution of PPAR-alpha, -beta, and -gamma in the adult rat. Endocrinology. 1996;137:354-66.
60. Berger J, Moller DE. The mechanisms of action of PPARs. Annu Rev Med. 2002;53:409-35.
61. Corrales P, Vidal-Puig A, Medina-Gomez G. PPARs and Metabolic Disorders Associated with Challenged Adipose Tissue Plasticity. Int J Mol Sci. 2018;19:2124.
62. Lavoie H, Gagnon J, Therrien M. ERK signaling: a master regulator of cell behavior, life, and fate. Nat Rev Mol Cell Biol. 2020;21:607-32.
63. Kwong E, Li Y, Hylemon PB, Zhou H. Bile acids and sphingosine-1-phosphate receptor 2 in hepatic lipid metabolism. Acta Pharm Sin B. 2015;5:151-7.
64. Hara T, Sata M, Fukuda D. Emerging roles of protease-activated receptors in cardiometabolic disorders. J Cardiol. 2022. https://doi.org/10.1016/j. jjcc.2022.09.013.
65. Zheng J, Li Q, He L, Weng H, Su D, Liu X, Ling W, Wang D. Protocatechuic acid inhibits vulnerable atherosclerotic lesion progression in older apoe-/- mice. J Nutr. 2020;150:1167-77.
66. Khan AS, Subramaniam S, Dramane G, Khelifi D, Khan NA. ERK1 and ERK2 activation modulates diet-induced obesity in mice. Biochimie. 2017;137:78-87.
67. Vitellius G, Trabado S, Bouligand J, Delemer B, Lombes M. Pathophysiology of glucocorticoid signaling. Ann Endocrinol. 2018;79:98-106.
68. Zhang QH, Cui XY, Wang D, Jin Y, Guan YX. Anti-obesity effect of escin: a study on high-fat diet-induced obese mice. Eur Rev Med Pharmacol Sci. 2022;26:7797-812.
69. Li R, Jia Y, Pan S, Li X, Song H, Zhao R. Glucocorticoid receptor mediates the effect of high-fat diet on mitochondrial oxidative phosphorylation in mouse liver. DNA Cell Biol. 2016;35:51-8.
70. Giordano R, Marzotti S, Berardelli R, Karamouzis I, Brozzetti A, D’Angelo V, Mengozzi G, Mandrile G, Giachino D, Migliaretti G, Bini V, Falorni A, Ghigo E, Arvat E. BCII polymorphism of the glucocorticoid receptor gene is associated with increased obesity, impaired glucose metabolism, and dyslipidemia in patients with Addison’s disease. Clin Endocrinol. 2012;77:863-70.
71. Suriagandhi V, Nachiappan V. Therapeutic target analysis and molecular mechanism of melatonin – leptin resistance induced obesity: a systematic study of network pharmacology. Front Endocrinol. 2022;13: 927576.
72. Huang J, Zhao L, Sun J, Wang L, Gu J, Liu X, Yang M, Wang Y, Zhang N, Zhu J, Xu S, Ren X, Su Y. Clinical evidence and potential mechanisms of complementary treatment of ling gui zhu gan formula for the management of serum lipids and obesity. Evid Based Complement Alternat Med. 2022;2022:7714034.
73. Pei SJ, Zhu HY, Guo JH, Zhang X, Deng ZJ. Knockout of CNR1 prevents metabolic stress-induced cardiac injury through improving insulin resistance (IR) injury and endoplasmic reticulum (ER) stress by promoting AMPK-alpha activation. Biochem Biophys Res Commun. 2018;503:744-51.
74. Tomlinson JW, Walker EA, Bujalska IJ, Draper N, Lavery GG, Cooper MS, Hewison M, Stewart PM. 11beta-hydroxysteroid dehydrogenase type 1: a tissue-specific regulator of glucocorticoid response. Endocr Rev. 2004;25:831-66.
75. Masuzaki H, Paterson J, Shinyama H, Morton NM, Mullins JJ, Seckl JR, Flier JS. A transgenic model of visceral obesity and the metabolic syndrome. Science. 2001;294:2166-70.
76. Pahlavani M, Kalupahana NS, Ramalingam L, Moustaid-Moussa N. Regulation, and functions of the renin-angiotensin system in white and brown adipose tissue. Compr Physiol. 2017;7:1137-50.
77. Bertholet AM, Kazak L, Chouchani ET, Bogaczynska MG, Paranjpe I, Wainwright GL, Betourne A, Kajimura S, Spiegelman BM, Kirichok Y. Mitochondrial Patch Clamp of Beige Adipocytes Reveals UCP1-Positive and UCP1-Negative Cells Both Exhibiting Futile Creatine Cycling, Cell Metab, 2017, 25, 811-+.
78. Gulyaeva O, Dempersmier J, Sul HS. Genetic and epigenetic control of adipose development. Biochim Biophys Acta Mol Cell Biol Lipids. 2019;1864:3-12.
79. Jia P, Wu X, Pan T, Xu S, Hu J, Ding X. Uncoupling protein 1 inhibits mitochondrial reactive oxygen species generation and alleviates acute kidney injury. EBio Med. 2019;49:331-40.
80. Clarke KJ, Porter RK. Uncoupling protein 1 dependent reactive oxygen species production by thymus mitochondria. Int J Biochem Cell Biol. 2013;45:81-9.
81. Shimano H, Sato R. SREBP-regulated lipid metabolism: convergent physiology – divergent pathophysiology. Nat Rev Endocrinol. 2017;13:710-30.
82. DeBose-Boyd RA, Ye J. SREBPs in Lipid Metabolism, Insulin Signaling, and Beyond, Trends Biochem Sci,
83. Thomas C, Gioiello A, Noriega L, Strehle A, Oury J, Rizzo G, Macchiarulo A, Yamamoto H, Mataki C, Pruzanski M, Pellicciari R, Auwerx J, Schoonjans K. TGR5-mediated bile acid sensing controls glucose homeostasis. Cell Metab. 2009;10:167-77.
84. Watanabe M, Houten SM, Mataki C, Christoffolete MA, Kim BW, Sato H, Messaddeq N, Harney JW, Ezaki O, Kodama T, Schoonjans K, Bianco AC, Auwerx J. Bile acids induce energy expenditure by promoting intracellular
85. Castellanos-Jankiewicz A, Guzman-Quevedo O, Fenelon VS, Zizzari P, Quarta C, Bellocchio L, Tailleux A, Charton J, Fernando D, Henricsson M, Piveteau C, Simon V, Allard C, Quemener S, Guinot V, Hennuyer N, Perino A, Duveau A, Maitre M, Leste-Lasserre T, Clark S, Dupuy N, Cannich A, Gonzales D, Deprez B, Mithieux G, Dombrowicz D, Backhed F, Prevot V, Marsicano G, Staels B, Schoonjans K, Cota D. Hypothalamic bile acid-TGR5 signaling protects from obesity. Cell Metab. 2021;33(1483-1492): e1410.
86. Hira T, Pinyo J, Hara H. What Is GLP-1 Doing in Obesity? Trends Endocrinol Metab. 2020;31:71-80.
87. Drucker DJ. GLP-1 physiology informs the pharmacotherapy of obesity. Mol Metab. 2022;57: 101351.
ملاحظة الناشر
- (انظر الشكل في الصفحة التالية.)
الشكل 3 نماذج ربط جزيئي لـ OA مع أهداف محتملة مضادة للسمنة. A PPARG، B PPARA، C MAPK3، D NR3C1، E PTGS2، F CYP19A1، G CNR1، H HSD11B1 و I AGTR1
DOI: https://doi.org/10.1186/s12967-023-04840-x
PMID: https://pubmed.ncbi.nlm.nih.gov/38246999
Publication Date: 2024-01-21
Integrating network pharmacology and animal experimental validation to investigate the action mechanism of oleanolic acid in obesity
Abstract
Background Obesity, a condition associated with the development of widespread cardiovascular disease, metabolic disorders, and other health complications, has emerged as a significant global health issue. Oleanolic acid (OA), a pentacyclic triterpenoid compound that is widely distributed in various natural plants, has demonstrated potential antiinflammatory and anti-atherosclerotic properties. However, the mechanism by which OA fights obesity has not been well studied.
Method Network pharmacology was utilized to search for potential targets and pathways of OA against obesity. Molecular docking and molecular dynamics simulations were utilized to validate the interaction of OA with core targets, and an animal model of obesity induced by high-fat eating was then employed to confirm the most central of these targets. Results The network pharmacology study thoroughly examined 42 important OA targets for the treatment of obesity. The key biological processes (BP), cellular components (CC), and molecular functions (MF) of OA for anti-obesity were identified using GO enrichment analysis, including intracellular receptor signaling, intracellular steroid hormone receptor signaling, chromatin, nucleoplasm, receptor complex, endoplasmic reticulum membrane, and RNA polymerase II transcription Factor Activity. The KEGG/DAVID database enrichment study found that metabolic pathways, PPAR signaling pathways, cancer pathways/PPAR signaling pathways, insulin resistance, and ovarian steroidogenesis all play essential roles in the treatment of obesity and OA . The protein-protein interaction (PPI) network was used to screen nine main targets: PPARG, PPARA, MAPK3, NR3C1, PTGS2, CYP19A1, CNR1, HSD11B1, and AGTR1. Using molecular docking technology, the possible binding mechanism and degree of binding between OA and each important target were validated, demonstrating that OA has a good binding potential with each target. The molecular dynamics simulation’s Root Mean Square Deviation (RMSD), and Radius of Gyration (Rg) further demonstrated that OA has strong binding stability with each target. Additional animal studies confirmed the significance of the core target PPARG and the core pathway PPAR signaling pathway in OA anti-obesity. Conclusion Overall, our study utilized a multifaceted approach to investigate the value and mechanisms of OA in treating obesity, thereby providing a novel foundation for the identification and development of natural drug treatments.
Introduction
Oleanolic acid (OA), a pentacyclic triterpenoid, is an active ingredient in herbs such as Fructus Ligustri Lucidi and Folium Camellia Sinensis, as well as in numerous common foods including olive leaves, Apple, Grape, Ginger, and Mango [12]. In China, OA has been utilized as a hepatoprotective medication [13], and there is clinical proof of its effectiveness in treating hyperlipidemia [14]. Furthermore, multiple clinical studies have demonstrated the potential of various OA derivatives to prevent or treat a range of diseases, including cancer, diabetes, and viral infections [13, 15, 16]. Previous research has identified OA as possessing diverse pharmacological properties, such as antiviral, antibacterial, anticancer, anti-inflammatory, antioxidant, hepatoprotective, and gastroprotective effects [17-19]. OA has been shown to improve aberrant alterations in lipid parameters, reduce hepatic microvesicular steatosis, increase leptin content, significantly reduce visceral fat, improve glucose tolerance, and elevate insulin levels [20,21]. It also reduced systemic inflammation, promoted hepatic lipogenesis, and enhanced the taste perception of dietary fat in mice fed the HFD [21]. Meanwhile, OA derivatives, such as Nano-OA, exhibit similar lipid-lowering effects [22]. Although the hypolipidemic effect of OA has been confirmed [23, 24], further research is required to elucidate
the mechanisms underlying its potential role in combating obesity.
Results
Network pharmacology analysis

of the two database studies may be connected to the database’s update. We focused on the PPAR signaling pathway based on the results of the enrichment of core targets and pathways, and the schematic diagram of OA in the treatment of obesity by targeting the PPAR signaling pathway is shown in Fig. 2. These findings imply that OA can exhibit anti-obesity benefits via a variety of routes, among which the PPAR signaling pathway plays a key role in OA’s anti-obesity process.
Molecular docking
| KEGG ID | Signaling pathway | Count |
| hsa01100 | Metabolic pathways – Homo sapiens human | 10 |
| hsa03320 | PPAR signaling pathway – Homo sapiens human | 9 |
| hsa05200 | Pathways in cancer – Homo sapiens human | 9 |
| hsa05207 | Chemical carcinogenesis – receptor activation – Homo sapiens human | 6 |
| hsa04931 | Insulin resistance – Homo sapiens human | 5 |
| hsa04520 | Adherens junction – Homo sapiens humann | 4 |
| hsa04723 | Retrograde endocannabinoid signaling – Homo sapiens human | 4 |
| hsa00140 | Steroid hormone biosynthesis – Homo sapiens human | 4 |
| hsa05022 | Pathways of neurodegeneration – multiple diseases – Homo sapiens human | 4 |
| hsa05208 | Chemical carcinogenesis – reactive oxygen species – Homo sapiens human | 4 |

| Targets | RMSD |
| PPARG | 0.018 |
| PPARA | 0.038 |
| MAPK3 | 0.031 |
| NR3C1 | 0.006 |
| PTGS2 | 0.037 |
| CYP19A1 | 0.532 |
| CNR1 | 0.027 |
| HSD11B1 | 0.019 |
| AGTR1 | 0.028 |
Molecular dynamics simulation
| Targets | Binding Energy (kJ/mol) | Hydrophobic Interactions | Hydrogen Bonds | Salt Bridges |
| PPARG | -24.85 | ARG 262, ALA 263, THR 266 | – | LYS 185, ARG 262 |
| PPARA | -26.15 | LYS 116, LYS 116 | – CYS 128 | – |
| MAPK3 | -24.18 | VAL 63, ARG 64, ARG 64, LYS 65, PRO 193 | ASN 161, PHE 346, THR 347 | – |
| NR3C1 | -24.64 | GLU 540, GLU 540, ARG 611, TYR 660 | TRP 610, TYR 660 | – |
| PTGS2 | -26.15 | PHE 205, VAL 344, TYR 348 | TYR 385, GLY 526, GLY 533, LEU 534 | – |
| CYP19A1 | -29.20 | GLN 225, LYS 243, TYR 244, TYR 244, ASP 476 | TYR 244, ASP 476 | LYS 243 |
| CNR1 | -22.55 | PHE 208, LEU 209, ILE 212, ALA 236, MET 240, LEU 286 | – | – |
| HSD11B1 | -34.14 | ILE 121, LEU 126, TYR 177, VAL 180, LEU 217, THR 222, ALA 223, ALA 226, ILE 230 | – | – |
| AGTR1 | -31.59 | TRP 84, PHE 182, LEU 195, LEU 195, LYS 199, THR 260 | THR 260 | LYS 199, HIS 256 |
Animal experiment
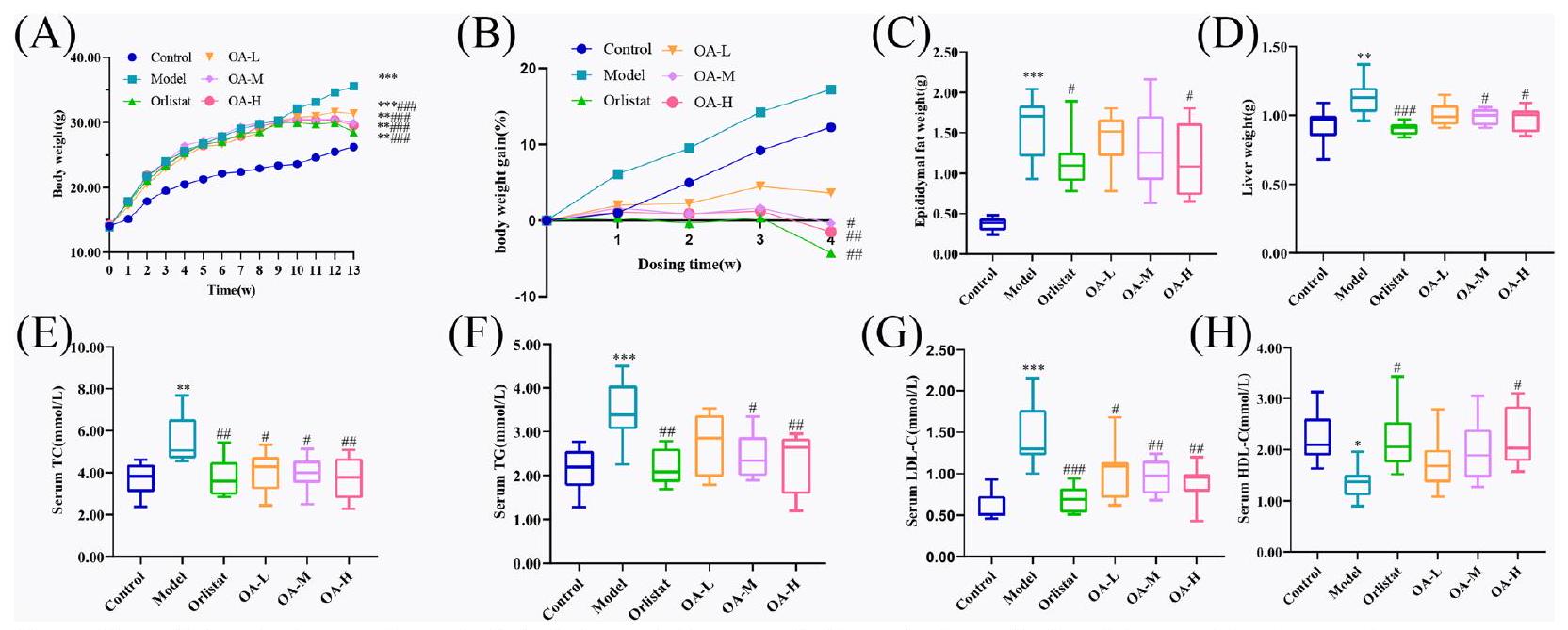
OA prevents weight gain in obese mice
OA regulates disorders of glycolipid metabolism


OA improves tissue pathological changes in HFD-fed mice
OA alleviates HFD-induced inflammation response
OA increases GLP-1 secretion by regulating TGR5
diet reduced TGR5 expression, with considerably lower data in the model group (
OA affects lipid synthesis via PPAR

OA affects energy consumption with TGR5/UCP-1
enhancement of TGR5 / UCP-1 protein expression, hence accelerating energy expenditure. However, this hypothesis still needs to be validated by other research.
Methods
Network pharmacology analysis
Obtaining targets associated with OA and obesity
ch/) [30] to obtain OA’s corresponding targets (probability> 0). Next, the GeneCards database (https://www. genecards.org/) [31] was searched for disease-related targets using the keyword “obesity”, followed by applying filters (Relevance score> 2) to extract the final set of disease targets. The two lists of targets obtained were then analyzed using the bioinformatics platform (http://www. bioinformatics.com.cn/) to identify the targets that overlap between OA and obesity.
Constructing protein-protein interaction (PPI) network
Screening core targets of OA anti-obesity
Gene ontology (GO) enrichment analysis
KEGG pathway enrichment analysis
Molecular docking analysis
and OA was utilized as ligands for molecular docking via AutoDock. Initially, the 3D structure of OA was obtained in “mol2” format from the China Traditional Medicine System Drug Database and Analysis Platform (TCMSP, https://old.tcmsp-e.com/tcmsp.php) [37], and then saved in “PDB” format using Pymol 2.5.7 (Portland, OR, US). The protein structure of the core targets in “PDB” format was downloaded from protein databases PDB (https:// www.rcsb.org) [38] and the alphafold databases (https:// alphafold.com) [39]. Next, the ligands of the target macromolecules were removed using Pymol software, and the molecules were pretreated using AutoDock tool 1.5.7 software, including hydrogenation, electron addition, and root addition. In the final step, AutoDock 4.2.6 software was employed for molecular semi-flexible docking, and the binding potential was assessed by an affinity score. The optimal docking model was then selected, and the noncovalent interactions of the protein-ligand complex at the atomic level were analyzed by the Protein-ligand Interaction Profiler (PLIP) web tool (https://plip-tool. biotec.tu-dresden.de/plip-web/plip/index) [40]. Finally, redocking was conducted to evaluate binding patterns before the formal molecular docking, followed by visual analysis using Pymol software.
Molecular dynamics simulation
In vivo assay
Chemicals and antibodies
Experimental animals and feeding conditions
Grouping and model establishment
Administration and handling
Calculation of weight gain and weight measurement of liver and epididymal fat
Measurements of blood glucose and lipid levels
Histologic studies
Enzyme-linked immunosorbent assay (ELISA)
Western blot
sulfate-polyacrylamide gel electrophoresis (SDS-PAGE) was used to separate the proteins, which were then transferred to polyvinylidene fluoride (PVDF) membranes. The membranes were incubated overnight with primary and secondary antibodies diluted in TBST. Chemiluminescence imaging was performed by adding developer drops to the PVDF membrane. Protein expression levels of PPAR
Statistical analysis
- Approval: The animal experiments in this study were approved by the Animal Ethics Committee of Guangdong Pharmaceutical University.
- Accordance: The methods were carried out by the relevant guidelines and regulations.
Discussion
effect of OA to the core targets using molecular docking methods, and further verified the binding stability and binding capacity using molecular dynamics simulations. At the same time, animal experiments were used to verify the core targets and pathways.
metabolism, cell migration, and cell differentiation [62]. It plays a role in various diseases such as hepatic lipid metabolism, liver lipid metabolism, cardiac metabolic disorders, etc [63, 64]. Prior research has demonstrated that Leptin, Protocatechuic Acid, and other molecules can combat obesity and atherosclerosis through MAPK3/ ERK1 [65, 66]. NR3C1 (Nuclear Receptor Subfamily 3 Group C Member 1), which encodes the glucocorticoid receptor, is involved in the inflammatory response, cell proliferation, and differentiation in target tissues [67]. It has been reported to exert beneficial effects on obesity, impaired glucose metabolism, and dyslipidemia [68-70]. Similarly, PTGS2(prostaglandin-endoperoxide synthase 2) and CYP19A1(cytochrome P450 family 19 subfamily A member 1) are also predicted to be pivotal target for reducing or reversing hyperlipidemia and obesity [68, 71, 72]. Additionally, the CNR1 (cannabinoid receptor 1) has been identified as a promising drug target for the treatment of obesity, with CNR1 knockout in mice resulting in improvements in insulin resistance, ER stress, and lipid accumulation [73]. Conversely, overexpression of HSD11B1 (corticosteroid 11-beta-dehydrogenase isozyme 1), which catalyzes the conversion of the active form of cortisol [74], has been found to result in visceral obesity, insulin-resistant diabetes, and dyslipidemia [75]. AGTR1 (angiotensin II receptor type 1) is a key player in the renin-angiotensin system (RAS) that can increase blood pressure and insulin resistance while also inhibiting lipolysis, maintaining energy homeostasis, and reducing inflammation [76]. Molecular docking and molecular dynamic simulation were used to validate the molecular mechanism of OA intervention in obesity. The molecular docking studies revealed that OA has a high affinity for the key targets identified by network pharmacology. The results of molecular dynamics simulation revealed that OA binding to the core targets was all more stable, indicating that the previously hypothesized core proteins were indeed the critical linkages in OA’s anti-obesity action.
can improve both inflammatory and glucolipid metabolism disorders. This finding suggests that OA’s ability to combat obesity may involve this mechanism.
In conclusion, this study combines network pharmacology, molecular docking, molecular dynamic simulation, and animal experimental verification for the first time to study the effectiveness and mechanism of action of OA in the treatment of obesity. The findings reveal the potential of OA in the treatment of obesity and provide a novel direction for the development of OA-derived drugs and food, thereby contributing to the discovery and development of natural resources related to obesity. Furthermore, this study explored the effects of medications on obesity through lipid accumulation, providing researchers with new insights for developing obesity drugs. Undeniably, there are some limitations in our study. Network pharmacology, molecular docking, and molecular dynamic simulation are reliant on data and algorithms, and their outcomes may differ from actual results due to database and software limitations. Additionally, due to time and resource limitations, we were unable to experimentally confirm all of the expected targets, or to combine animal and cell research with clinical validation and other methods, preventing us from fully revealing the anti-obesity mechanism of OA. We will conduct additional tests in the future to examine the potential molecular pathways behind the anti-obesity effects of OA in greater detail.
Conclusion
Supplementary Information
Acknowledgements
Author contributions
Funding
Availability of data and materials
Declarations
Ethics approval and consent to participate
Consent for publication
Competing interests
Author details
Published online: 21 January 2024
References
- Upadhyay J, Farr O, Perakakis N, Ghaly W, Mantzoros C. Obesity as a disease. Med Clin N Am. 2018;102:13-33.
- Fontaine KR, Redden DT, Wang C, Westfall AO, Allison DB. Years of life lost due to obesity. JAMA. 2003;289:187-93.
- Afshin A, Forouzanfar MH, Reitsma MB, Sur P, Estep K, Lee A, Marczak L, Mokdad AH, Moradi-Lakeh M, Naghavi M, Salama JS, Vos T, Abate KH, Abbafati C, Ahmed MB, Al-Aly Z, Alkerwi A, Al-Raddadi R, Amare AT, Amberbir A, Amegah AK, Amini E, Amrock SM, Anjana RM, Arnlov J, Asayesh H, Banerjee A, Barac A, Baye E, Bennett DA, Beyene AS, Biadgilign S, Biryukov S, Bjertness E, Boneya DJ, Campos-Nonato I, Carrero JJ, Cecilio P, Cercy K, Ciobanu LG, Cornaby L, Damtew SA, Dandona L, Dandona R, Dharmaratne SD, Duncan BB, Eshrati B, Esteghamati A, Feigin VL, Fernandes JC, Furst T, Gebrehiwot TT, Gold A, Gona PN, Goto A, Habtewold TD, Hadush KT, Hafezi-Nejad N, Hay SI, Horino M, Islami F, Kamal R, Kasaeian A, Katikireddi SV, Kengne AP, Kesavachandran CN, Khader YS, Khang YH, Khubchandani J, Kim D, Kim YJ, Kinfu Y, Kosen S, Ku T, Defo BK, Kumar GA, Larson HJ, Leinsalu M, Liang
4. Boutari C, Mantzoros CS. A 2022 update on the epidemiology of obesity and a call to action: as its twin COVID-19 pandemic appears to be receding, the obesity and dysmetabolism pandemic continues to rage on. Metabolism. 2022;133: 155217.
5. Chooi YC, Ding C, Magkos F. The epidemiology of obesity. Metabolism. 2019;92:6-10.
6. Kivimaki M, Strandberg T, Pentti J, Nyberg ST, Frank P, Jokela M, Ervasti J, Suominen SB, Vahtera J, Sipila PN, Lindbohm JV, Ferrie JE. Body-mass index and risk of obesity-related complex multimorbidity: an observational multicohort study. Lancet Diabetes Endocrinol. 2022;10:253-63.
7. Reid TJ, Korner J. Medical and surgical treatment of obesity. Med Clin North Am. 2022;106:837-52.
8. Walter CP, Bleske BE, Dorsch MP. Pharmacotherapy for weight loss: the cardiovascular effects of the old and new agents. J Clin Pharm Ther. 2014;39:475-84.
9. Lan JP, Tong RC, Sun XM, Zhang HY, Sun S, Xiong AZ, Wang ZT, Yang L. Comparison of the main chemical composition of Plantago asiatica L . and P. depressa Willd. seed extracts and their anti-obesity effects in highfat diet-induced obese mice. Phytomedicine. 2021;81: 153362.
10. Martel J, Ojcius DM, Chang CJ, Lin CS, Lu CC, Ko YF, Tseng SF, Lai HC, Young JD. Anti-obesogenic and antidiabetic effects of plants and mushrooms. Nat Rev Endocrinol. 2017;13:149-60.
11. Wan Y, Xia J, Xu JF, Chen L, Yang Y, Wu JJ, Tang F, Ao H, Peng C. Nuciferine, an active ingredient derived from lotus leaf, lights up the way for the potential treatment of obesity and obesity-related diseases. Pharmacol Res. 2022;175: 106002.
12. Castellano JM, Espinosa JM, Perona JS. Modulation of lipid transport and adipose tissue deposition by small lipophilic compounds. Front Cell Dev Biol. 2020;8: 555359.
13. Yang YH, Dai SY, Deng FH, Peng LH, Li C, Pei YH. Recent advances in medicinal chemistry of oleanolic acid derivatives. Phytochemistry. 2022;203: 113397.
14. Luo HQ, Shen J, Chen CP, Ma X, Lin C, Ouyang Q, Xuan CX, Liu J, Sun HB, Liu J. Lipid-lowering effects of oleanolic acid in hyperlipidemic patients. Chin J Nat Med. 2018;16:339-46.
15. Liu D, Jin X, Yu G, Wang M, Liu L, Zhang W, Wu J, Wang F, Yang J, Luo Q, Cai L, Yang X, Ke X, Qu Y, Xu Z, Jia L, Chen WL. Oleanolic acid blocks the purine salvage pathway for cancer therapy by inactivating SOD1 and stimulating lysosomal proteolysis. Mol Ther Oncolytics. 2021;23:107-23.
16. Santos-Lozano JM, Rada M, Lapetra J, Guinda A, Jimenez-Rodriguez MC, Cayuela JA, Angel-Lugo A, Vilches-Arenas A, Gomez-Martin AM, OrtegaCalvo M, Castellano JM. Prevention of type 2 diabetes in prediabetic patients by using functional olive oil enriched in oleanolic acid: The PREDIABOLE study, a randomized controlled trial. Diabetes Obes Metab. 2019;21:2526-34.
17. de Melo CL, Queiroz MGR, Fonseca SGC, Bizerra AMC, Lemos TLG, Melo TS, Santos FA, Rao VS. Oleanolic acid, a natural triterpenoid that improves blood glucose tolerance in normal mice and ameliorates visceral obesity in mice fed a high-fat diet. Chem-Biol Interact. 2010;185:59-65.
18. Dzubak P, Hajduch M, Vydra D, Hustova A, Kvasnica M, Biedermann D, Markova L, Urban M, Sarek J. Pharmacological activities of natural triterpenoids and their therapeutic implications. Nat Prod Rep. 2006;23:394-411.
19. Kaushik S, Dar L, Kaushik S, Yadav JP. Anti-dengue activity of supercritical extract and isolated oleanolic acid of Leucas cephalotes using in vitro and silico approach. Bmc Complement Med. 2021;21:227.
20. de Moura EDM, Dos Reis SA, da Conceicao LL, Sediyama C, Pereira SS, de Oliveira LL, Gouveia Peluzio MDC, Martinez JA, Milagro Fl. Diet-induced
obesity in animal models: points to consider and influence on metabolic markers. Diabetol Metab Syndr. 2021;13:32.
21. Djeziri FZ, Belarbi M, Murtaza B, Hichami A, Benammar C, Khan NA. Oleanolic acid improves diet-induced obesity by modulating fat preference and inflammation in mice. Biochimie. 2018;152:110-20.
22. Wang S, Du LB, Jin L, Wang Z, Peng J, Liao N, Zhao YY, Zhang JL, Pauluhn J, Hai CX, Wang X, Li WL. Nano-oleanolic acid alleviates metabolic dysfunctions in rats with high fat and fructose diet. Biomed Pharmacother. 2018;108:1181-7.
23. Liu J. Pharmacology of oleanolic acid and ursolic acid. J Ethnopharmacol. 1995;49:57-68.
24. Claro-Cala CM, Jimenez-Altayo F, Zagmutt S, Rodriguez-Rodriguez R. Molecular mechanisms underlying the effects of olive oil triterpenic acids in obesity and related diseases. Nutrients. 2022;14:1606.
25. Jiao W, Mi S, Sang Y, Jin Q, Chitrakar B, Wang X, Wang S. Integrated network pharmacology and cellular assay for the investigation of an anti-obesity effect of 6-shogaol. Food Chem. 2022;374: 131755.
26. Xia QD, Xun Y, Lu JL, Lu YC, Yang YY, Zhou P, Hu J, Li C, Wang SG. Network pharmacology and molecular docking analyses on Lianhua Qingwen capsule indicate Akt1 is a potential target to treat and prevent COVID-19. Cell Proliferat. 2020;53: e12949.
27. Yang B, Yang N, Chen Y, Zhu M, Lian Y, Xiong Z, Wang B, Feng L, Jia X. An integrated strategy for effective-component discovery of astragali radix in the treatment of lung cancer. Front Pharmacol. 2020;11: 580978.
28. Singh A, Vanga SK, Orsat V, Raghavan V. Application of molecular dynamic simulation to study food proteins: A review. Crit Rev Food Sci Nutr. 2018;58:2779-89.
29. Kim S, Chen J, Cheng T, Gindulyte A, He J, He S, Li Q, Shoemaker BA, Thiessen PA, Yu B, Zaslavsky L, Zhang J, Bolton EE. PubChem in 2021: new data content and improved web interfaces. Nucleic Acids Res. 2021;49:D1388-95.
30. Daina A, Michielin O, Zoete V. SwissTargetPrediction: updated data and new features for efficient prediction of protein targets of small molecules. Nucleic Acids Res. 2019;47:W357-64.
31. Stelzer G, Rosen N, Plaschkes I, Zimmerman S, Twik M, Fishilevich S, Stein TI, Nudel R, Lieder I, Mazor Y, Kaplan S, Dahary D, Warshawsky D, Guan-Golan Y, Kohn A, Rappaport N, Safran M, Lancet D. The GeneCards Suite: From Gene Data Mining to Disease Genome Sequence Analyses, Curr Protoc Bioinformatics, 2016;54, 130 31-31 3033.
32. Szklarczyk D, Gable AL, Nastou KC, Lyon D, Kirsch R, Pyysalo S, Doncheva NT, Legeay M, Fang T, Bork P, Jensen LJ, von Mering C. The STRING database in 2021: customizable protein-protein networks, and functional characterization of user-uploaded gene/measurement sets. Nucleic Acids Res. 2021;49:D605-12.
33. Shannon P, Markiel A, Ozier O, Baliga NS, Wang JT, Ramage D, Amin N, Schwikowski B, Ideker T. Cytoscape: a software environment for integrated models of biomolecular interaction networks. Genome Res. 2003;13:2498-504.
34. Tang Y, Li M, Wang J, Pan Y, Wu FX. CytoNCA: a cytoscape plugin for centrality analysis and evaluation of protein interaction networks. Biosystems. 2015;127:67-72.
35. Sherman BT, Hao M, Qiu J, Jiao X, Baseler MW, Lane HC, Imamichi T, Chang W, DAVID: a web server for functional enrichment analysis and functional annotation of gene lists,. update). Nucleic Acids Res. 2021;2022. https://doi.org/10.1093/nar/gkac194.
36. UniProt C. UniProt: the universal protein knowledgebase in 2021. Nucleic Acids Res. 2021;49:D480-9.
37. Ru J, Li P, Wang J, Zhou W, Li B, Huang C, Li P, Guo Z, Tao W, Yang Y, Xu X, Li Y, Wang Y, Yang L. TCMSP: a database of systems pharmacology for drug discovery from herbal medicines. J Cheminform. 2014;6:13.
38. Burley SK, Bhikadiya C, Bi C, Bittrich S, Chen L, Crichlow GV, Christie CH, Dalenberg K, Di Costanzo L, Duarte JM, Dutta S, Feng Z, Ganesan S, Goodsell DS, Ghosh S, Green RK, Guranovic V, Guzenko D, Hudson BP, Lawson CL, Liang Y, Lowe R, Namkoong H, Peisach E, Persikova I, Randle C, Rose A, Rose Y, Sali A, Segura J, Sekharan M, Shao C, Tao YP, Voigt M, Westbrook JD, Young JY, Zardecki C, Zhuravleva M. RCSB Protein Data Bank: powerful new tools for exploring 3D structures of biological macromolecules for basic and applied research and education in fundamental biology, biomedicine, biotechnology, bioengineering and energy sciences. Nucleic Acids Res. 2021;49:D437-51.
39. Jumper J, Evans R, Pritzel A, Green T, Figurnov M, Ronneberger O, Tunyasuvunakool K, Bates R, Zidek A, Potapenko A, Bridgland A, Meyer C, Kohl SAA, Ballard AJ, Cowie A, Romera-Paredes B, Nikolov S, Jain R, Adler J, Back T, Petersen S, Reiman D, Clancy E, Zielinski M, Steinegger M, Pacholska M, Berghammer T, Bodenstein S, Silver D, Vinyals O, Senior AW, Kavukcuoglu K, Kohli P, Hassabis D. Highly accurate protein structure prediction with AlphaFold. Nature. 2021;596:583-9.
40. Adasme MF, Linnemann KL, Bolz SN, Kaiser F, Salentin S, Haupt VJ, Schroeder M. PLIP 2021: expanding the scope of the proteinligand interaction profiler to DNA and RNA. Nucleic Acids Res. 2021;49:W530-4.
41. Song
42. Dong Y, Zheng Y, Zhu L, Li T, Guan Y, Zhao S, Wang Q, Wang J, Li L. Hua-Tan-Sheng-Jing decoction treats obesity with oligoasthenozoospermia by up-regulating the PI3K-AKT and down-regulating the JNK MAPK signaling pathways: at the crossroad of obesity and oligoasthenozoospermia. Front Pharmacol. 2022;13: 896434.
43. Su S, Wu G, Cheng X, Fan J, Peng J, Su H, Xu Z, Cao M, Long Z, Hao Y, Li G, Li S, Hai C, Wang X. Oleanolic acid attenuates PCBs-induced adiposity and insulin resistance via HNF1b-mediated regulation of redox and PPARgamma signaling. Free Radic Biol Med. 2018;124:122-34.
44. Li W, Zeng H, Xu M, Huang C, Tao L, Li J, Zhang T, Chen H, Xia J, Li C, Li X. Oleanolic acid improves obesity-related inflammation and insulin resistance by regulating macrophages activation. Front Pharmacol. 2021;12: 697483.
45. Pan Y, Zhou F, Song Z, Huang H, Chen Y, Shen Y, Jia Y, Chen J. Oleanolic acid protects against the pathogenesis of atherosclerosis, possibly via FXR-mediated angiotensin (Ang)-(1-7) upregulation. Biomed Pharmacother. 2018;97:1694-700.
46. Fernandez-Aparicio A, Schmidt-RioValle J, Perona JS, Correa-Rodriguez M, Castellano JM, Gonzalez-Jimenez E. Potential protective effect of oleanolic acid on the components of metabolic syndrome: a systematic review. J Clin Med. 2019;8:1294.
47. Li M, Han Z, Bei W, Rong X, Guo J, Hu X. Oleanolic acid attenuates insulin resistance via NF-kappaB to regulate the IRS1-GLUT4 pathway in HepG2 cells. Evid Based Complement Alternat Med. 2015;2015: 643102.
48. Nyakudya TT, Isaiah S, Ayeleso A, Ndhlala AR, Mukwevho E, Erlwanger KH. Short-Term neonatal oral administration of oleanolic acid protects against fructose-induced oxidative stress in the skeletal muscles of suckling rats. Molecules. 2019;24:661.
49. Teixeira LL, Pilon G, Coutinho CP, Dudonne S, Dube P, Houde V, Desjardins Y, Lajolo FM, Marette A, Hassimotto NMA. Purple grumixama anthocyanins (Eugenia brasiliensis Lam.) attenuate obesity and insulin resistance in high-fat diet mice. Food Funct. 2021;12:3680-91.
50. Shi L, Karrar E, Wang X. Sesamol ameliorates hepatic lipid accumulation and oxidative stress in steatosis HepG2 cells via the PPAR signaling pathway. J Food Biochem. 2021;45: e13976.
51. Wu L, Guo C, Wu J. Therapeutic potential of PPARgamma natural agonists in liver diseases. J Cell Mol Med. 2020;24:2736-48.
52. Robinson-Rechavi M, Carpentier AS, Duffraisse M, Laudet V. How many nuclear hormone receptors are there in the human genome? Trends Genet. 2001;17:554-6.
53. Montaigne D, Butruille L, Staels B. PPAR control of metabolism and cardiovascular functions. Nat Rev Cardiol. 2021;18:809-23.
54. Derosa G, Sahebkar A, Maffioli P. The role of various peroxisome prolifera-tor-activated receptors and their ligands in clinical practice. J Cell Physiol. 2018;233:153-61.
55. Botta M, Audano M, Sahebkar A, Sirtori CR, Mitro N, Ruscica M. PPAR agonists and metabolic syndrome: an established role? Int J Mol Sci. 2018;19:1197.
56. Pawlak M, Lefebvre P, Staels B. Molecular mechanism of PPAR alpha action and its impact on lipid metabolism, inflammation, and fibrosis in nonalcoholic fatty liver disease. J Hepatol. 2015;62:720-33.
57. Hu N, Chen CY, Wang JH, Huang J, Yao DH, Li CL. Atorvastatin ester regulates lipid metabolism in hyperlipidemia rats via the PPAR-signaling pathway and HMGCR expression in the liver. Int J Mol Sci. 2021;22:11107.
58. Magadum A, Engel FB. PPARbeta/delta: linking metabolism to regeneration. Int J Mol Sci. 2018;19:2013.
59. Braissant O, Foufelle F, Scotto C, Dauca M, Wahli W. Differential expression of peroxisome proliferator-activated receptors (PPARs): tissue distribution of PPAR-alpha, -beta, and -gamma in the adult rat. Endocrinology. 1996;137:354-66.
60. Berger J, Moller DE. The mechanisms of action of PPARs. Annu Rev Med. 2002;53:409-35.
61. Corrales P, Vidal-Puig A, Medina-Gomez G. PPARs and Metabolic Disorders Associated with Challenged Adipose Tissue Plasticity. Int J Mol Sci. 2018;19:2124.
62. Lavoie H, Gagnon J, Therrien M. ERK signaling: a master regulator of cell behavior, life, and fate. Nat Rev Mol Cell Biol. 2020;21:607-32.
63. Kwong E, Li Y, Hylemon PB, Zhou H. Bile acids and sphingosine-1-phosphate receptor 2 in hepatic lipid metabolism. Acta Pharm Sin B. 2015;5:151-7.
64. Hara T, Sata M, Fukuda D. Emerging roles of protease-activated receptors in cardiometabolic disorders. J Cardiol. 2022. https://doi.org/10.1016/j. jjcc.2022.09.013.
65. Zheng J, Li Q, He L, Weng H, Su D, Liu X, Ling W, Wang D. Protocatechuic acid inhibits vulnerable atherosclerotic lesion progression in older apoe-/- mice. J Nutr. 2020;150:1167-77.
66. Khan AS, Subramaniam S, Dramane G, Khelifi D, Khan NA. ERK1 and ERK2 activation modulates diet-induced obesity in mice. Biochimie. 2017;137:78-87.
67. Vitellius G, Trabado S, Bouligand J, Delemer B, Lombes M. Pathophysiology of glucocorticoid signaling. Ann Endocrinol. 2018;79:98-106.
68. Zhang QH, Cui XY, Wang D, Jin Y, Guan YX. Anti-obesity effect of escin: a study on high-fat diet-induced obese mice. Eur Rev Med Pharmacol Sci. 2022;26:7797-812.
69. Li R, Jia Y, Pan S, Li X, Song H, Zhao R. Glucocorticoid receptor mediates the effect of high-fat diet on mitochondrial oxidative phosphorylation in mouse liver. DNA Cell Biol. 2016;35:51-8.
70. Giordano R, Marzotti S, Berardelli R, Karamouzis I, Brozzetti A, D’Angelo V, Mengozzi G, Mandrile G, Giachino D, Migliaretti G, Bini V, Falorni A, Ghigo E, Arvat E. BCII polymorphism of the glucocorticoid receptor gene is associated with increased obesity, impaired glucose metabolism, and dyslipidemia in patients with Addison’s disease. Clin Endocrinol. 2012;77:863-70.
71. Suriagandhi V, Nachiappan V. Therapeutic target analysis and molecular mechanism of melatonin – leptin resistance induced obesity: a systematic study of network pharmacology. Front Endocrinol. 2022;13: 927576.
72. Huang J, Zhao L, Sun J, Wang L, Gu J, Liu X, Yang M, Wang Y, Zhang N, Zhu J, Xu S, Ren X, Su Y. Clinical evidence and potential mechanisms of complementary treatment of ling gui zhu gan formula for the management of serum lipids and obesity. Evid Based Complement Alternat Med. 2022;2022:7714034.
73. Pei SJ, Zhu HY, Guo JH, Zhang X, Deng ZJ. Knockout of CNR1 prevents metabolic stress-induced cardiac injury through improving insulin resistance (IR) injury and endoplasmic reticulum (ER) stress by promoting AMPK-alpha activation. Biochem Biophys Res Commun. 2018;503:744-51.
74. Tomlinson JW, Walker EA, Bujalska IJ, Draper N, Lavery GG, Cooper MS, Hewison M, Stewart PM. 11beta-hydroxysteroid dehydrogenase type 1: a tissue-specific regulator of glucocorticoid response. Endocr Rev. 2004;25:831-66.
75. Masuzaki H, Paterson J, Shinyama H, Morton NM, Mullins JJ, Seckl JR, Flier JS. A transgenic model of visceral obesity and the metabolic syndrome. Science. 2001;294:2166-70.
76. Pahlavani M, Kalupahana NS, Ramalingam L, Moustaid-Moussa N. Regulation, and functions of the renin-angiotensin system in white and brown adipose tissue. Compr Physiol. 2017;7:1137-50.
77. Bertholet AM, Kazak L, Chouchani ET, Bogaczynska MG, Paranjpe I, Wainwright GL, Betourne A, Kajimura S, Spiegelman BM, Kirichok Y. Mitochondrial Patch Clamp of Beige Adipocytes Reveals UCP1-Positive and UCP1-Negative Cells Both Exhibiting Futile Creatine Cycling, Cell Metab, 2017, 25, 811-+.
78. Gulyaeva O, Dempersmier J, Sul HS. Genetic and epigenetic control of adipose development. Biochim Biophys Acta Mol Cell Biol Lipids. 2019;1864:3-12.
79. Jia P, Wu X, Pan T, Xu S, Hu J, Ding X. Uncoupling protein 1 inhibits mitochondrial reactive oxygen species generation and alleviates acute kidney injury. EBio Med. 2019;49:331-40.
80. Clarke KJ, Porter RK. Uncoupling protein 1 dependent reactive oxygen species production by thymus mitochondria. Int J Biochem Cell Biol. 2013;45:81-9.
81. Shimano H, Sato R. SREBP-regulated lipid metabolism: convergent physiology – divergent pathophysiology. Nat Rev Endocrinol. 2017;13:710-30.
82. DeBose-Boyd RA, Ye J. SREBPs in Lipid Metabolism, Insulin Signaling, and Beyond, Trends Biochem Sci,
83. Thomas C, Gioiello A, Noriega L, Strehle A, Oury J, Rizzo G, Macchiarulo A, Yamamoto H, Mataki C, Pruzanski M, Pellicciari R, Auwerx J, Schoonjans K. TGR5-mediated bile acid sensing controls glucose homeostasis. Cell Metab. 2009;10:167-77.
84. Watanabe M, Houten SM, Mataki C, Christoffolete MA, Kim BW, Sato H, Messaddeq N, Harney JW, Ezaki O, Kodama T, Schoonjans K, Bianco AC, Auwerx J. Bile acids induce energy expenditure by promoting intracellular
85. Castellanos-Jankiewicz A, Guzman-Quevedo O, Fenelon VS, Zizzari P, Quarta C, Bellocchio L, Tailleux A, Charton J, Fernando D, Henricsson M, Piveteau C, Simon V, Allard C, Quemener S, Guinot V, Hennuyer N, Perino A, Duveau A, Maitre M, Leste-Lasserre T, Clark S, Dupuy N, Cannich A, Gonzales D, Deprez B, Mithieux G, Dombrowicz D, Backhed F, Prevot V, Marsicano G, Staels B, Schoonjans K, Cota D. Hypothalamic bile acid-TGR5 signaling protects from obesity. Cell Metab. 2021;33(1483-1492): e1410.
86. Hira T, Pinyo J, Hara H. What Is GLP-1 Doing in Obesity? Trends Endocrinol Metab. 2020;31:71-80.
87. Drucker DJ. GLP-1 physiology informs the pharmacotherapy of obesity. Mol Metab. 2022;57: 101351.
Publisher’s Note
- *Correspondence:
Xuguang Hu
hxguang21@163.com
Full list of author information is available at the end of the article - (See figure on next page.)
Fig. 3 Molecular docking models of OA with possible core anti-obesity targets. A PPARG, B PPARA, C MAPK3, D NR3C1, E PTGS2, F CYP19A1, G CNR1, H HSD11B1 and I AGTR1
