DOI: https://doi.org/10.1186/s12964-023-01302-1
PMID: https://pubmed.ncbi.nlm.nih.gov/38347575
تاريخ النشر: 2024-02-12
كشف آليات وتحديات مقاومة الأدوية لعلاج السرطان
الملخص
يواجه علاج السرطان العديد من العقبات، والمقاومة هي واحدة منها. تتطور استراتيجيات علاج السرطان بسبب القدرة على المقاومة الفطرية والمكتسبة، التي تحكمها إشارات وراثية، وإيبيجينيتية، وبروتينية، واستقلابية، أو بيئية دقيقة، مما يمكّن في النهاية خلايا السرطان المختارة من البقاء والتقدم في ظل ظروف غير مواتية. على الرغم من أن آلية مقاومة الأدوية تُدرس على نطاق واسع لتوليد أدوية جديدة تستهدف بشكل أفضل من الأدوية الحالية. ومع ذلك، بسبب المرونة الأوسع في مقاومة الأدوية المكتسبة، يجب استكشاف خيارات علاجية متقدمة ذات فعالية أفضل. تعتبر العلاج المركب بديلاً له معدل نجاح أفضل على الرغم من أن خطر الآثار الجانبية المتزايدة هو أمر شائع. علاوة على ذلك، فإن العلاج المناعي الدقيق الرائد حديثًا هو أحد الطرق للتغلب على مقاومة الأدوية وقد أحدث ثورة في علاج السرطان إلى حد كبير مع القيد الوحيد المتمثل في كونه محددًا فرديًا ويحتاج إلى مزيد من الاهتمام. ستركز هذه المراجعة على التحديات والاستراتيجيات التي اختارتها خلايا السرطان لتحمل العلاجات الحالية على المستوى الجزيئي، كما تسلط الضوء على الخيارات العلاجية الناشئة – مثل الخيارات المناعية، والخيارات القائمة على الخلايا الجذعية التي قد تثبت أن لديها إمكانات أفضل لمواجهة مشكلة مقاومة العلاج الحالية.
مقدمة
الحصول على) وتطوير العلاجات المستهدفة من الجيل التالي أمر حاسم لتلبية الاحتياجات الطبية [13، 14].
تساهم التفاعلات المعقدة بين العوامل الداخلية (الفطرية) والعوامل الخارجية (المكتسبة) لخلايا السرطان في مقاومة السرطان تجاه العلاجات المختلفة. تشمل العوامل الداخلية الطفرات الجينية الموجودة مسبقًا، وتنوع الأورام، وتنشيط مسارات الدفاع داخل الخلايا، التي تمنح المقاومة من خلال تنشيط مسارات ورمية مختلفة، وتغيير أهداف الأدوية، وتقليل الحساسية تجاه العلاجات، وتعزيز آليات إصلاح الحمض النووي، بالإضافة إلى تنشيط مسارات البقاء، مما يمكن خلايا السرطان من التهرب من التأثيرات السامة للعلاجات. بينما تشمل العوامل الخارجية بشكل رئيسي مكونات بيئة الورم الدقيقة (TME) التي تشارك بنشاط في قدرة خلايا السرطان على التهرب من التأثيرات السامة لمختلف العلاجات المضادة للسرطان. تشمل مكونات TME المختلفة مصفوفة خارج الخلية المعدلة (ECM)، وخلايا السدى المرتبطة بالورم، وعوامل النمو، والحويصلات خارج الخلوية (EVs)، وخلايا المناعة، وغيرها. تؤثر مصفوفة ECM الصلبة والمكثفة المرتبطة بالورم على استجابة الأدوية من خلال تقليل نقل الأدوية واحتجاز الأدوية من خلال الارتباط المباشر بها، وبالتالي تمثل آلية مهمة لمقاومة الأدوية في العديد من الأورام الصلبة. تعتبر الخلايا الليفية المرتبطة بالسرطان (CAFs) مكونًا آخر من مكونات TME التي تلعب أدوارًا مهمة في نمو الورم، والانتقال، ومقاومة العلاج السرطاني من خلال إفراز عوامل نمو مختلفة مثل عامل نمو الكبد (HGF) وعامل نمو البشرة (EGF)؛ والسيتوكينات مثل عامل الخلايا السدوية المستمدة من الخلايا (SDF-1) والإنترلوكين-6 (IL-6). يمكن أن تحتجز الحويصلات خارج الخلوية (EVs) لخلايا السرطان المقاومة للأدوية وتنقل الأدوية إلى ECM. علاوة على ذلك، قد يتم نقل EVs من خلايا السرطان المقاومة للأدوية إلى نظيراتها الحساسة للأدوية، وبالتالي تلعب دورًا في النقل الأفقي لمقاومة الأدوية في خلايا السرطان من خلال توصيل حمولات متخصصة تشمل بروتينات مرتبطة بمقاومة الأدوية (P-gp، ABCG2، ABCA3، إلخ)، والأحماض النووية (mt DNA، mRNAs، miRNAs)، والمواد الأيضية الورمية، وبروتينات مضادة للاستماتة. قد يكون استهداف مكونات خلايا السرطان المقاومة للأدوية الداخلية وبيئة الورم الدقيقة، بمفردها أو بالاشتراك مع العلاجات المضادة للسرطان، نهجًا أفضل في تعزيز فعالية علاجات السرطان وتحسين نتائج المرضى. يتم مناقشة الآليات التفصيلية للعوامل الخارجية والداخلية المتورطة في مقاومة الأدوية والاستراتيجيات لإيقافها في الأقسام أدناه.
الاعتراف وتدمير خلايا السرطان مع مزيد من التحمل وتحسن في الشفاء. تُظهر العلاجات المضادة لـ CTLA و PD-1/PD-L1 المستخدمة على نطاق واسع نشاطًا مضادًا للورم بشكل ملحوظ من خلال تعطيل المنظمين السلبيين لجهاز المناعة التكيفي المضاد للسرطان، على الرغم من أن الحد الأدنى من فرصة المقاومة وقيودها على مجموعة معينة من السرطان لا تزال مصدر قلق. تتبع خلايا السرطان قاعدة ضغط الاختيار الدارويني لتحقيق سمات مقاومة الأدوية على المستويات الجينومية، والإبيجينومية، والبروتيومية من أجل بقاء الأجدر. مع ظهور الفحوصات عالية الإنتاجية، أصبح الرابط بين تباين الورم ومقاومة الأدوية موجودًا، مما يشير إلى أنه تحت ضغط دوائي انتقائي، تنقسم بعض خلايا الورم وتشكل مجموعة فرعية من الخلايا التي قد تحقق ميزات تمكنها من أن تصبح غير مستجيبة لدواء معين مع مرور الوقت. يتم تمثيل الميزات المتناقضة لخلايا السرطان وخلايا مقاومة الأدوية في الشكل 1.
تباين الورم هو عامل حاسم لمقاومة الأدوية
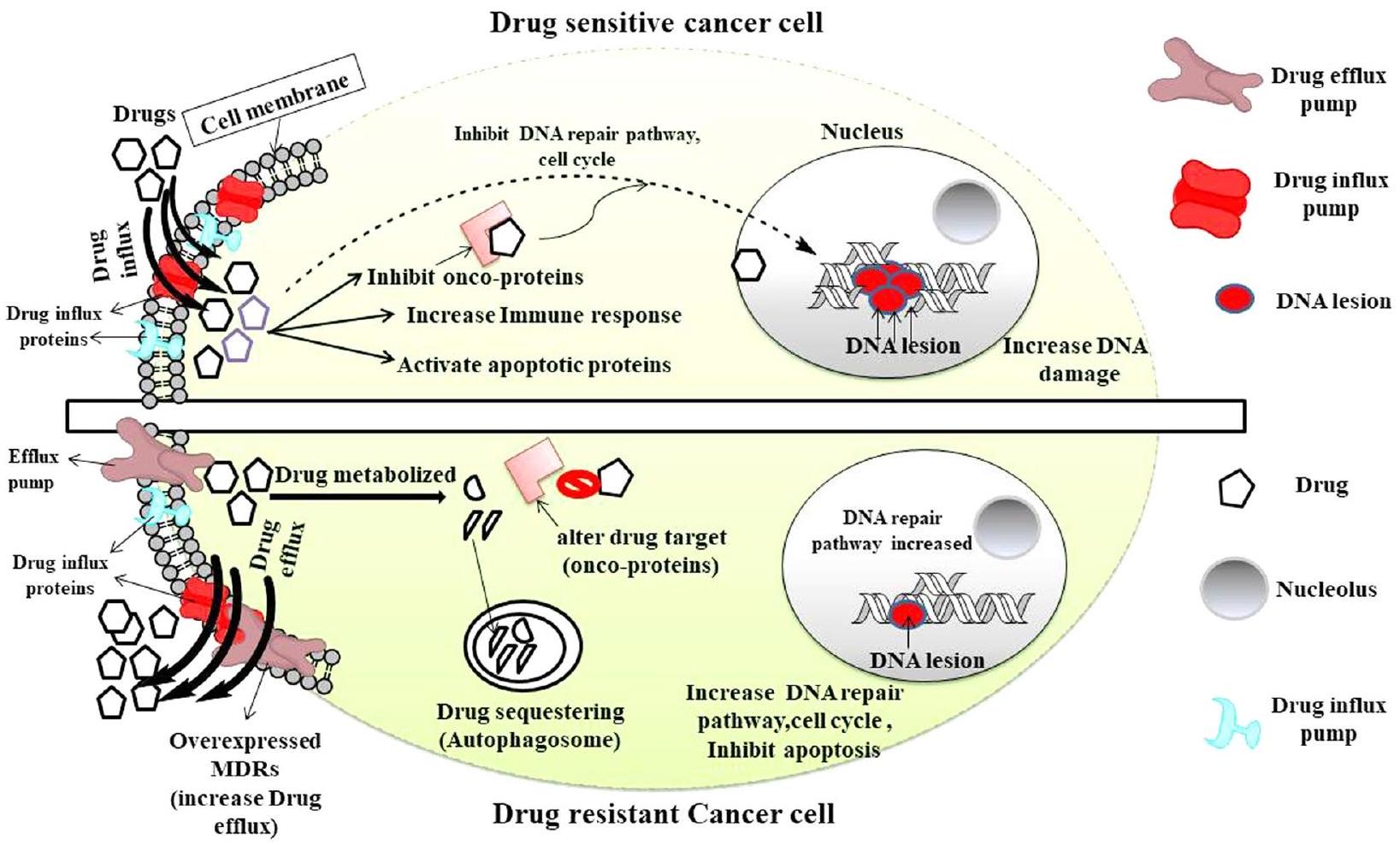
تقود الانقسام وعدم استقرار الكروموسومات إلى فقدان عشوائي للكروموسومات في مناطق مختلفة من الورم، مما يؤدي إلى تباين طفرات مع نتيجة ظهور سلالات فرعية تنافسية متطورة وخلايا جذعية سرطانية (CSCs) والعكس صحيح. هذه السلالات التي تشكلت حتى الآن تتوسع إما بشكل تسلسلي أو من خلال نهج متفرع وتولد مزيدًا من التنوع الجيني بأي من الطريقتين، والذي يتم اختياره تحت ضغط تطوري مع نمو أفضل، ومقاومة، وميزة بقاء. تمكّننا الأساليب الحديثة ذات الإنتاجية العالية مثل تسلسل RNA على مستوى الخلية الواحدة وتوصيف الطفرات من التحقيق في الديناميات التطورية التي تحدث في مجموعة خلايا الورم المحددة في نفس المرضى أو مرضى مختلفين، ولها دور بارز في العلاج الفردي. تساهم هذه التغيرات الجينومية المتنوعة في اكتساب خصائص مفيدة مثل مقاومة الأدوية وتكرار الورم في هذه الخلايا السرطانية المختارة. تسهم تطورات الورم في ظهور مجموعة فرعية من الخلايا مقاومة لعدة أدوية (MDR) مع استجابات علاجية متباينة للأدوية مقارنة بخلايا الورم الأولية. من ناحية أخرى، يلعب الضغط الكيميائي دورًا مهمًا في تشكيل سلالات فرعية مقاومة أكثر تطورًا مع نتائج أسوأ. أظهرت الدراسات الحديثة أن التباين الورمي المتواجد مع المشهد المناعي.
تحليل في مجموعة من المرضى الذين يعانون من سرطان الكبد الخلوي البشري المرتبط بفيروس التهاب الكبد B (HCC) يعيق تسلل خلايا T وبالتالي ينظم البيئة المناعية المثبطة داخل الورم. هذه النتيجة حاسمة لتصميم علاجات مناعية فعالة يمكن إعطاؤها بشكل فردي أو بالاشتراك مع العلاجات الكيميائية الحالية لجعل خلايا السرطان المقاومة أكثر حساسية مع نتائج أفضل. وبالتالي، فإن تباين الورم والمشهد المناعي المتنوع يمثلان عقبات كبيرة لفهم المقاومة، ويمكن أن يساهم المزيد من التركيز على هذه القضية الصعبة في توجيه أفضل لمستقبل علاج السرطان.
التغيرات الجينية والوراثية كاستجابة تكيفية للعلاج الكيميائي التي تؤثر بشكل كبير على مقاومة الأدوية
منطقة GSTp و MDR1 و uPA و O(6)-methylguanine DNA methyltransferase (MGMT) ميثلة بشكل كبير، ولكن في خلايا MCF-7 المقاومة للعقاقير، كانت هذه المحفزات ميثلة بشكل منخفض وكان لها دور كبير في المقاومة. تم ربط انخفاض ميثلة جين MDR1 بزيادة تعبير بروتين تدفق الدواء (P-glycoprotein؛ P-gp) الذي بدوره مسؤول عن المقاومة للدوكسوروبيسين (DOX) [80-83].
| اسم الدواء | دراسة تجريبية | نوع السرطان | المرحلة السريرية | مرجع |
| سونيديجيب (LDE225) | لدى LDE225 القدرة على تعطيل جيوب خلايا السرطان الجذعية والتغلب على مقاومة الدوكيتاكسل | سرطان الثدي الثلاثي السلبي | 1ب | [112] |
| RO4929097 | تم عكس مقاومة مضادات الأندروجين التي تتوسطها خلايا السدى، مقاومة التاموكسيفين، ومقاومة الإشعاع | ورم دبقي خبيث متكرر | 1 | [113] |
| PF-03084014 | مواجهة مقاومة الدوكيتاكسيل في خلايا السرطان الجذعية | الورم الليفي الدسمي | 1 | [114] |
| PRI-724 | يمكن أن يتغلب على مقاومة السيسبلاتين في خلايا السرطان الجذعية ويقلل من تعبير SOX2 و CD44 | تليف الكبد المرتبط بفيروس التهاب الكبد الوبائي C | 1 | [115] |
| فيسموديجيب (GDC-0449) | لديها القدرة على التغلب على الإشعاع، ومقاومة الكاربوبلاتين/الإرولوتينيب بالإضافة إلى خصائص الخلايا الجذعية. | سرطانات الخلايا القاعدية المتعددة (MIKIE) | 2 | [116] |
تلعب خلايا الساق السرطانية دورًا رئيسيًا في تطوير مقاومة الأدوية وانتكاسة الورم
الإشارات التنموية غير المنظمة التي تنظم مساهمة خلايا السرطان الجذعية في مقاومة العلاج الكيميائي
تعتمد بقاء الخلايا والوظائف الخلوية تحت نقص المغذيات، نقص الأكسجين، أو في مقاومة الأدوية، على عملية فسيولوجية محفوظة تطورياً تعرف باسم الالتهام الذاتي. من المثير للاهتمام أن السرطان والخلايا الجذعية السرطانية تستغل هذه العملية الاستقلابية لدعم تكوين الأورام، والحفاظ على القدرة على التعددية، وتقدم الورم، والانتكاس. على سبيل المثال، CD44
نظير الخلايا غير الجذعية، مما يثبط البلعمة الذاتية من خلال حذف ATG5 باستخدام CRISPR/Cas9 مما يجعل هذه الخلايا الجذعية السرطانية حساسة للعلاج الكيميائي. من المعروف أن البلعمة الذاتية تقلل من الإجهاد التأكسدي الناتج عن العلاج الكيميائي في الخلايا الطبيعية والسرطانية وخلايا CSC، مما يحميها من موت الخلايا. بالمثل، في الخلايا الجذعية السرطانية والخلايا الجذعية الطبيعية، يقوم إنزيم العلامة ديهيدروجيناز الألدهيد (ALDH) بأكسدة الألدهيدات داخل الخلايا ويحميها من العواقب الضارة للجذور الحرة للأكسجين (ROS). أظهرت دراسة مفاجئة أن الشكل المتغير من ALDH (ALDH1A3) مسؤول عن مقاومة جرعات أقل من تيموزولوميد في الورم الدبقي. أظهرت الجرعات الأعلى من تيموزولوميد أنها تحفز التفاعل الفيزيائي المباشر بين ALDH1A3 وبروتين موائم البلعمة الذاتية p62، مما يؤدي إلى تحللها وتقليل المقاومة. أظهر ييو وآخرون (2016) الاعتماد الورمي وخصائص الخلايا الجذعية.
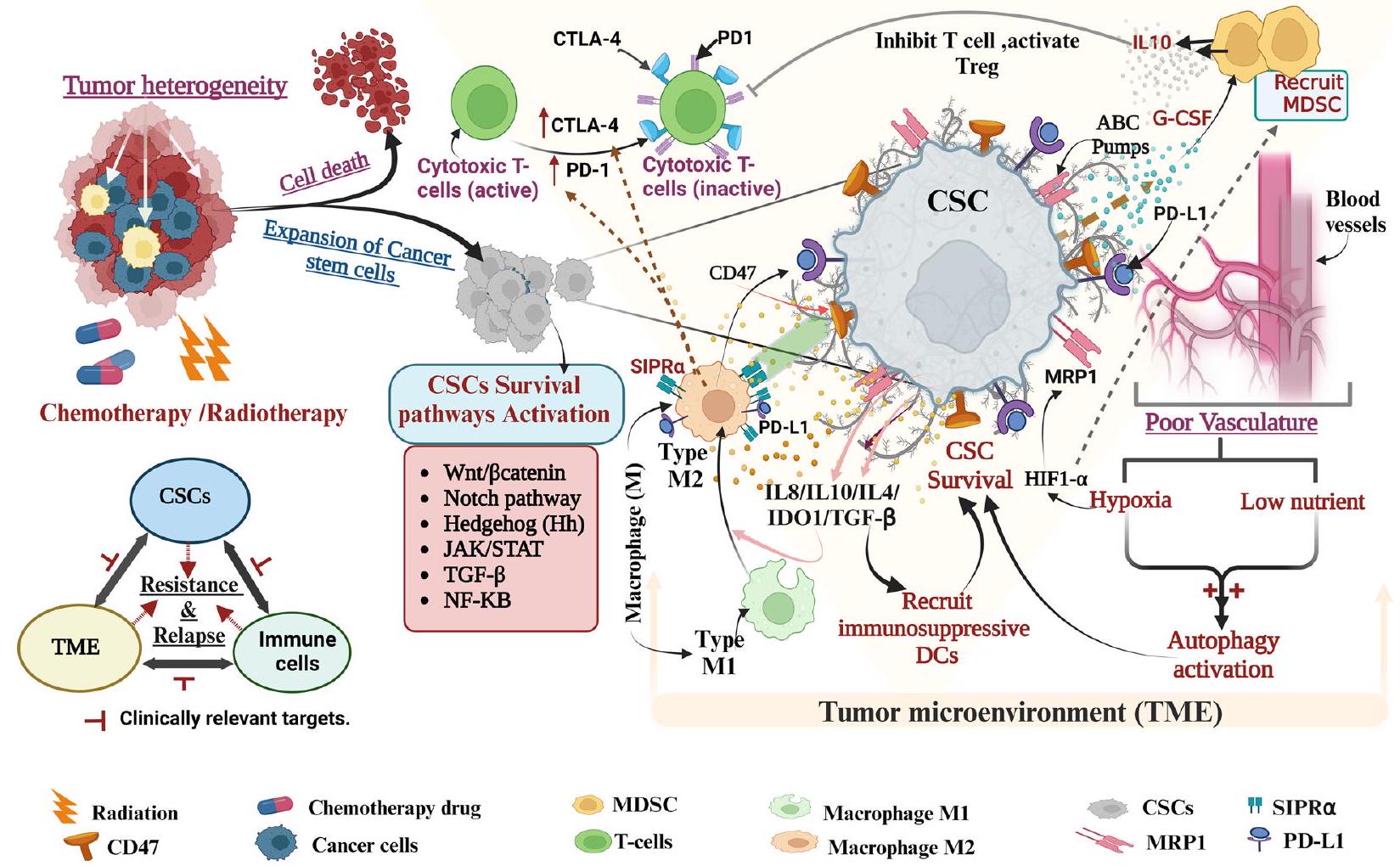
تتحكم خلايا السلف في جهاز المناعة لدى المضيف وتساعد في مقاومة الأدوية
تقوم TAMs المفعلة بواسطة CSCs أيضًا بتثبيط السمية الخلوية للخلايا التائية من خلال الإفراط في التعبير عن مستقبلات نقاط التفتيش المناعية السرطانية مثل بروتين ligand 1 المبرمج للموت (PD-L1) و(CD80/CD86) التي تتفاعل مع بروتين الموت الخلوي المبرمج-1 (PD-1) وبروتين-4 المرتبط بالخلايا التائية السامة (CTLA-4) على السطح
السرطان، الخلايا الجذعية السرطانية المستندة إلى نقص الأكسجين تجذب الخلايا المناعية المثبطة للورم إلى موقع الورم من خلال إشارات ENTPD2/CD39 L1 وتوقف علاج PD1، وتقليل الخلايا المناعية المثبطة يجعل هذه الخلايا أكثر حساسية لـ 5-FU. تم تقديم خلايا CAR T (خلايا T المستقبلة لمستضدات هجينة) مؤخرًا كعلاج معدّل لخلايا الدم البيضاء المضيفة، وقد أحدثت ثورة في العلاج المناعي ضد السرطان لتجاوز حاجز التخصص وعدم الاستجابة للعلاج القياسي، وتم تجربتها لأول مرة على مرضى اللوكيميا اللمفاوية الحادة (ALL) مع تحسن في الشفاء. ومع ذلك، فإن التهديد المستمر لتطوير متلازمة إطلاق السيتوكينات (CRS) والانتكاسة المبكرة لسرطان الدم الإيجابي للمستضد (فقدان المراقبة النشطة لخلايا CAR T) أو الانتكاسة اللاحقة (فقدان المستضد) للورم، قد حد من نطاق خلايا CAR T المحددة. ومع ذلك، فإن العلاج المشترك الأخير لخلايا CAR T مع المناعية المعدلة (مثبطات نقاط التفتيش المناعية مثل حجب PDL1-PD1) قد أظهر نتائج واعدة من حيث عمق ودوام العلاج سريريًا. الوصف أعلاه يسلط الضوء على أهمية التفاعل الثلاثي ‘CSC-TME-المناعة’ في توسيع الورم ومقاومة العلاج وأهميته السريرية. تم عرض تمثيل شامل لارتباط المناعة والخلايا الجذعية السرطانية في مقاومة الأدوية في الشكل 2.
برامج EMT الديناميكية ومقاومة الأدوية مرتبطة ببعضها البعض بشكل متبادل
علاوة على ذلك، كشفت مجموعة من عينات سرطان الثدي الكبيرة أن العينات التي تعبر عن ZEB1 بشكل مفرط كانت أقل استجابة لعلاج الإبيروبيسين الجيني السام. عند تحليل الآلية، وُجد أن ZEB ينشط بشكل فعال كيناز أتاكسي-تيلانجيكتاز (ATM) على المستوى النسخي من خلال تعزيز تشكيل مركب ZEB1/p300/PCAF، مما أدى إلى تنشيط مسار إصلاح الحمض النووي من خلال إعادة التركيب المتماثل. يرتبط EMT وبيئة خلايا الورم (TME) من خلال محور FBXW7-ZEB2 لتعزيز تكوين خلايا السرطان الجذعية القولونية ومقاومة العلاج الكيميائي. علاوة على ذلك، من خلال FZD7/Wnt/
دور في تنظيم تكوين الأورام ومقاومة العلاج الكيميائي [195].
دور تدرج الرقم الهيدروجيني عبر العضيات في مقاومة الأدوية
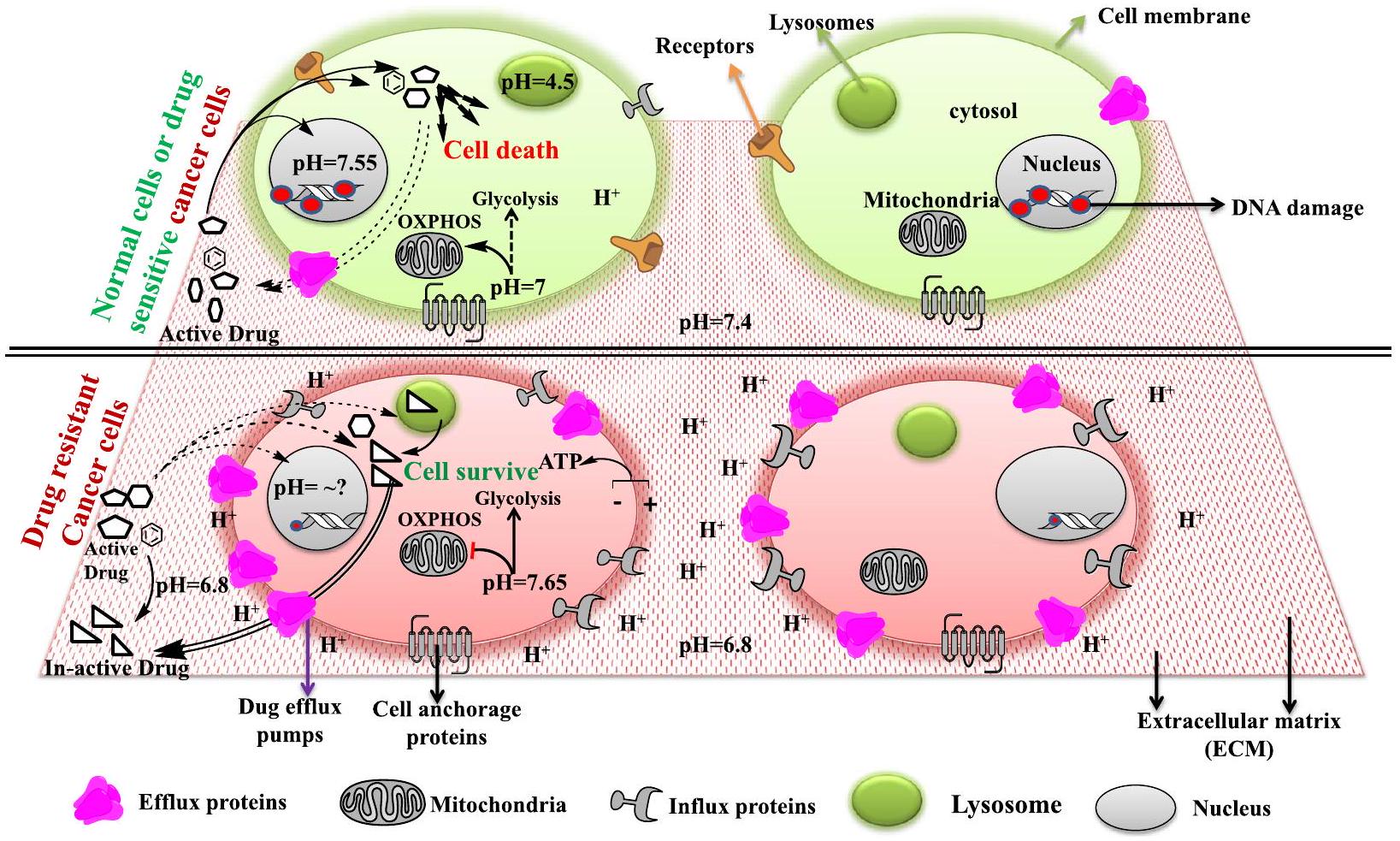
الموقع تحت الخلوي مع تأثير أقل على تغيير الرقم الهيدروجيني. تعتبر الإندوليسوزومات مكونًا مهمًا من العملية التحليلية داخل الخلايا المعروفة باسم الالتهام الذاتي، والتي تزيل الجزيئات الحيوية الزائدة وغير المعروفة والمشوهة وغير المستخدمة من الخلايا عن طريق تكسيرها إلى وحدات بناء أصغر أو وحدات طاقة للخلايا. تشير تقارير متنوعة إلى أن الرقم الهيدروجيني للإندوليسوزوم يلعب دورًا محوريًا في مقاومة الأدوية من خلال تراكم وتخزين أدوية العلاج الكيميائي المختلفة (وهي في الغالب قاعدة ضعيفة) التي تدخل إليها إما عن طريق الانتشار السلبي أو من خلال مضخات P-gp المدمجة في الغشاء، ثم يتم طردها لاحقًا عن طريق الإخراج الخلوي، مما يؤدي إلى توليد خلايا سرطانية مقاومة للعلاج الكيميائي. لذا فإن سلامة نفاذية غشاء الليزوزوم ضرورية لتكون خلايا السرطان مقاومة، ويمكن اعتبارها موضوعًا مهمًا من الناحية العلاجية. أظهرت دراسات متنوعة أن المحفزات لنفاذية غشاء الليزوزوم مثل الكلوروكين يمكن أن تؤدي إلى موت خلايا السرطان المقاومة عن طريق إطلاق الأدوية المخزنة والبروتيازومات مثل الكاتيبسين للعمل على النواة وتحفيز موت الخلايا المبرمج. علاوة على ذلك، يعزز الكلوروكين إطلاق أكسيد النيتريك الذي يثبط بشكل فعال نشاط P-gp ويؤدي إلى تراكم أدوية العلاج الكيميائي، مما يؤدي إلى الموت في سرطان الكبد المقاوم.
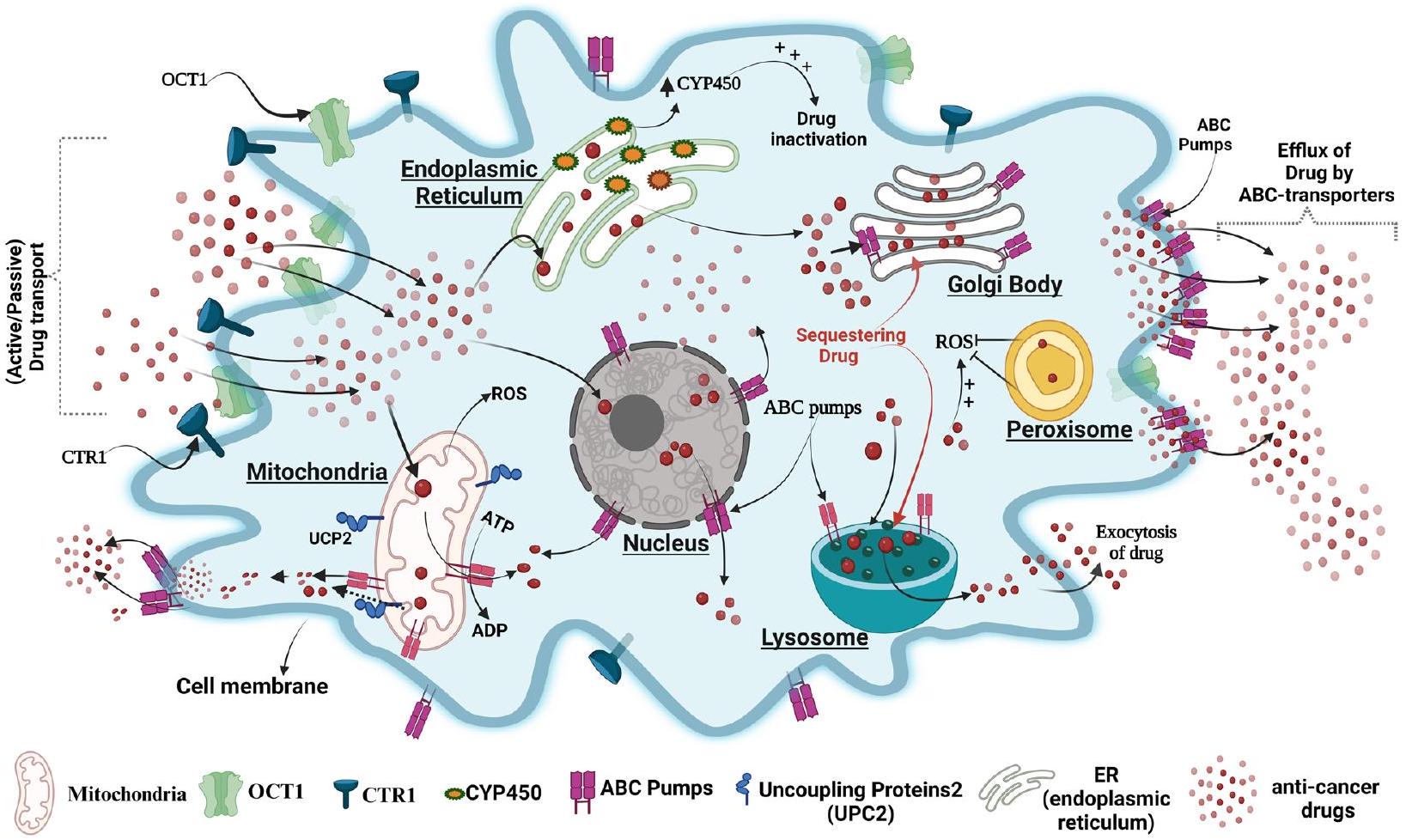
تظهر الدراسات أن الشبكة الإندوبلازمية (ER) تلعب دورًا حيويًا في استقلاب الأدوية بسبب وجود إنزيم “السيتوكروم P450” (CYPs) الذي يستقلب الأدوية. أظهر لين وآخرون أن تعبير إنزيم السيتوكروم P450 CYP1B1 كان أعلى في خلايا A549 المقاومة للتاكسول مقارنة بخلايا A549 الأصلية، وأن تثبيط CYP1B1 بواسطة 4 هيدروكسي-إيمودين زاد من حساسيتها للتاكسول. تعمل الجسم جولجي (GB) كوسيلة لنقل ما بعد الترجمة.
يلعب المحور دورًا في مقاومة الأدوية أيضًا. في خلايا مقاومة الورم الدبقي، يتم التعبير عن GB بشكل مفرط لمختلف MDRs ويحتجز الأدوية من خلال نظامها السري الذي يقوم لاحقًا بتصدير الأدوية خارج الخلايا، وتُعكس هذه العملية بواسطة مثبطات P-gp مثل S9788 و PSC833، التي تعكس مقاومة الأدوية. تلعب البيروكسيسوم، وهو مريح للإجهاد التأكسدي للخلايا، أيضًا دورًا في المقاومة كما في اللمفوما تجاه الفورينستات من خلال تقليل توليد ROS، وقد أظهر تثبيط نشاط البيروكسيسوم زيادة حساسية هذه الخلايا للأدوية. النواة هي مركز التحكم في الخلايا حقيقية النواة وقد أفادت الدراسات السابقة بتعبير أنواع مختلفة من ناقلات ABC مثل P-gp على غشاء النواة ودورها في مقاومة أنواع مختلفة من الأدوية المضادة للسرطان مثل دوكسوروبيسين في أنواع مختلفة من السرطانات مثل الورم الدبقي متعدد الأشكال (LN299). يتم عرض تمثيل صورة لدور العضية في مقاومة الأدوية في الشكل 4.
تحمي خلايا المناعة وبيئة الورم خلايا السرطان من الأدوية المضادة للسرطان
الخلايا الليفية النشطة (CAFs): هي خلايا ليفية نشطة، مرتبطة بكثرة بالخلايا السرطانية في البيئة المجاورة للورم (TME)، حيث تساهم مجموعة متنوعة من الإشارات المسرطنة مثل عوامل النمو، الكيموكينات، الإكسوزومات، وغيرها، التي تفرزها CAFs، في تمكين الخلايا السرطانية من الخضوع لعملية التحول الظهاري (EMT)، وتفادي العلاجات، وزيادة احتمالية عودة الورم [241-244]. تستجيب CAF للعقار الكيميائي سيسبلاتين المستخدم ضد سرطان الخلايا الحرشفية في المريء (ESCC)، من خلال إفراز جزيء الإشارة الباراكراين مثبط منشط البلازمينوجين 1 (PAI-1). PAI-1
في المقابل، يحفز بقاء مسارات AKT و MAPK لحماية ESCC من تلف الحمض النووي الناتج عن ROS وموت الخلايا تحت علاج الأدوية الكيميائية، مما يدعم المقاومة. علاوة على ذلك، فإن عامل نمو الكبد (HGF) أو TGF الذي تفرزه خلايا CAF
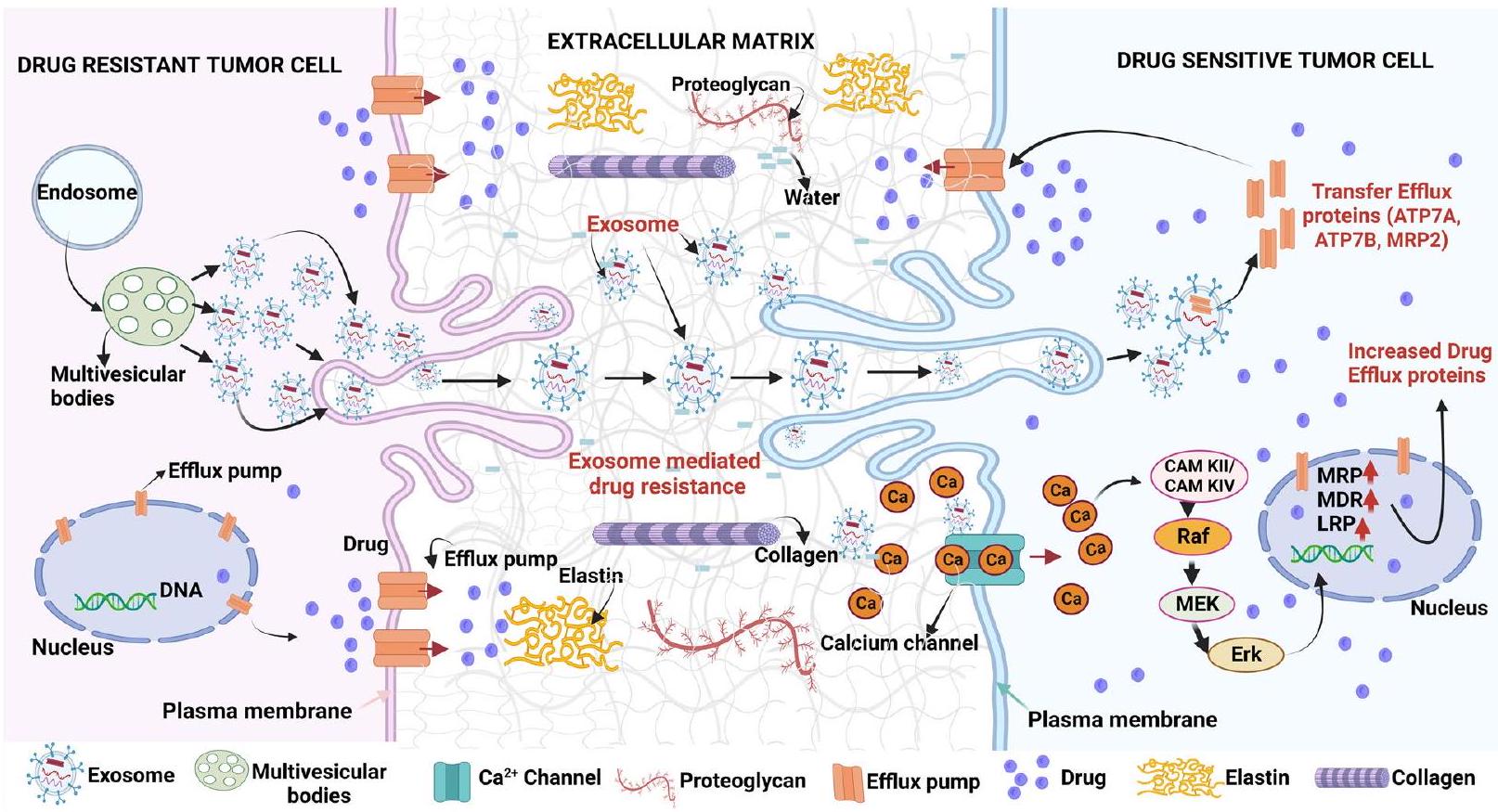
ii) ECM: يعتبر المصفوفة خارج الخلوية (ECM) عاملاً آخر يتكون من بروتينات ليفية مثل الكولاجين، والإيلاستين، والبروتيوغليكانات، والعناصر الدقيقة، والماء، وما إلى ذلك، وتلعب دورًا نشطًا في مقاومة العلاج (الشكل 5) [250، 251]. تختلف مصفوفة الورم بشكل كبير في التركيب والتكوين وتظهر تسللًا ليفيًا/ليفيا عضليًا، يتبعه تراكم كبير لمصفوفة كولاجينية أو نسيج ديسموبلاستي، مما يعوق توصيل الأدوية المضادة للسرطان إلى خلايا السرطان [251، 252]. وُجد أن سرطان المبيض المقاوم للسيكلوفسفاميد يعبر عن COL11A1 (الكولاجين من النوع الحادي عشر
مقاومة العلاج [255،256]. لقد أظهرت الدراسات أن خلايا السرطان في البيئة المجهرية للورم/المصفوفة خارج الخلوية التي تفتقر إلى الكولاجين أو الفيبرو نكتين أو كليهما كانت أكثر حساسية تجاه السيسبلاتين مقارنة بنظيراتها [257]. واستمرارًا في هذا السياق، زادت فعالية الأوكساليبلاتين ضد خلايا سرطان القولون من خلال قمع
iii) الإكسوزومات: الإكسوزومات، المعروفة أيضًا باسم الحويصلات خارج الخلوية (EVs)، التي تنتجها الأجسام متعددة الحويصلات الكبيرة (MVBs)، تسهل التفاعل بين الخلايا من خلال نقل الحمولات الحيوية عبر الخلايا وتدفق الجزيئات غير المرغوب فيها في الخلايا السليمة [259]. إنها وسطاء إشارات مهمون، تلعب أدوارًا في نمو الورم، وإعادة تشكيل بيئة الورم، والنقائل، وتكوين الأوعية، فضلاً عن مقاومة العلاج [260، 261]. تقوم خلايا سرطانية مختلفة باختطاف EVs لتدفق الأدوية المضادة للسرطان مما يؤدي بدوره إلى مقاومة الأدوية كما هو موضح في الشكل 5 [262، 263]. تندمج الإكسوزومات المستمدة من خلايا الجذع الميزانشيمي (MSC) في خلايا الورم المعدي، مما يعزز تنشيط مسارات إشارات CaM-Ks وRaf/MEK/ERK downstream، مما يزيد من تعبير البروتينات المرتبطة بمقاومة الأدوية مما يؤدي إلى مقاومة كيميائية للسرطان المعدي [264]. أظهرت خلايا سرطان المبيض المقاومة للأدوية زيادة في تصدير الإكسوزومات من سيسبلاتين مع تجنيد الناقلات النازحة ATP7A وATP7B وMRP2 (ABCB2) في الخلايا المجاورة لحماية موت الخلايا الناتج عن الأدوية [265].
iv) نقص الأكسجين: تؤدي الأوعية الدموية الشاذة في الورم والطلب العالي على الأكسجين إلى نقص الأكسجين، وانخفاض توفر العناصر الغذائية مثل الجلوكوز والأحماض الأمينية الحيوية [266، 267]. يحفز حرمان الأكسجين عامل نقص الأكسجين (HIF)-
v) خلايا المناعة: أكثر أنواع خلايا المناعة انتشارًا في بيئة الورم هي البلعميات المرتبطة بالورم (TAMs) [273]. تم ربط غزو TAMs إلى بيئة الورم بتوقعات سيئة واستجابة غير كافية للعوامل الكيميائية في مرضى السرطان [274، 275]. تستمد TAMs من وحيدات Ly6C الدائرية
[276]. يلعب نمط M2 دورًا في مقاومة العلاج ويحفز استجابات Th2. في الميكروبيئة السرطانية، يكون استقطاب البلعميات من M1 إلى M2 شائعًا [277]. يحفز العلاج الدوائي TAMs لتتطور إلى بلعميات M2 المستقطبة المثبطة للمناعة مما يمنح مقاومة كيميائية في خلايا سرطانية مختلفة. لقد أظهرت الدراسات أن ROS تتراكم في خلايا السرطان المعدي بعد التعرض لـ 5-FU تنشط إشارات HIF
تغيرات/تنظيمات الأيض تحكم مقاومة الأدوية في خلايا السرطان
علاوة على ذلك، يلعب أيض ATP أيضًا دورًا عميقًا في النظام المناعي أو المناعي المعدل الذي يحافظ في الظروف الطبيعية على توازن الجسم من خلال الحفاظ على التوازن بين التحفيز المناعي، وقمع المناعة، والدفاع ضد الأمراض المناعية الذاتية. تقوم خلايا السرطان بتمثيل ATP خارج الخلية إلى أدينوزين خارج الخلية المثبط للمناعة (eADO) بواسطة إنزيمات CD39 (إكستونيوكليوتيداز ثلاثي الفوسفات ثنائي الفوسفات-هيدراز 1) وCD73 (5′-نيوكليوتيداز) المثبتة على الغشاء [296]. على الرغم من أن مسارًا غير تقليدي آخر يشارك أيضًا في توليد الأدينوزين والذي يشمل نشاط CD38 (NAD
مقاومة خلايا الورم للعلاج المناعي. يؤثر تنشيط مسار الأدينوزين على تنشيط اللمفاويات بما في ذلك الخلايا المناعية المتسللة إلى الورم، والخلايا النخاعية، وخلايا السدى المرتبطة بالورم، وبالتالي تساعد خلايا الورم على التهرب من استجابة الخلايا المناعية المضادة للورم مما يؤدي إلى مقاومة العلاج [297]. لذا فإن استهداف CD39 وCD73 ومستقبلات الأدينوزين في وجود علاج نقاط التفتيش المناعية (مضاد PDL1/PD1؛ مضاد CTLA-4) يمكن أن يثبت أنه استراتيجية مناعية جديدة ضد خلايا السرطان المقاومة للمناعة. على سبيل المثال، أظهر العلاج المشترك لمثبط CD39 (POM1) والأجسام المضادة المضادة لـ PD1 وCTLA-4 في نموذج الفئران المزروعة بخلايا الميلانوما B16-F10 انخفاضًا ملحوظًا في عبء الورم وزيادة في بقاء الفئران الحاملة للورم [298]. على الرغم من أن بيانات القوارض واعدة جدًا، إلا أن القضية الرئيسية في هذه النتيجة هي ما إذا كان يمكن ترجمتها إلى البشر.
تمثيل الأدوية ومقاومة خلايا السرطان
عملها [305]. ومع ذلك، تظهر خلايا السرطان مقاومة للأدوية عن طريق تغيير موقع تحفيزي أو هيكل إنزيم مما يؤثر على تفاعلها مع الأدوية وبالتالي يؤدي إلى تغيير في طريقة عملها. على سبيل المثال، تعبر خلايا AML المقاومة للسيترابين (نظير السيتيدين) عن كيناز الديوكسي سيتيدين المتحور (منشط السيترابين)، مما يقلل من نشاط الدواء وبالتالي تطور المقاومة [306، 307]. وبالمثل، فإن مقاومة دوكسوروبيسين (نشط) في سرطان البروستاتا والثدي ترجع أساسًا إلى التحول الإنزيمي إلى دوكسوروبيسينول (غير نشط) بواسطة الإنزيم المفرط التعبير عنه ألدو-كيتو ريدوكتاز، وقد أظهرت العلاجات المركبة أنها فعالة جدًا في زيادة النشاط العلاجي لـ DOX [308، 309].
تغيير هدف الدواء
تظهر خلايا السرطان مقاومة للأدوية إما عن طريق تقليل الامتصاص أو تعزيز تدفق الأدوية أو كليهما من خلال مستقبلات وناقلات متحورة [315]. يمكن أن تنقل الناقلات المرتبطة بالغشاء المعروفة باسم بولي ببتيدات نقل الأنيونات العضوية (مثل OATP1B1 وOATP1B3 وOATP1A2) باكليتاكسيل، ميثوتريكسات، فلافوبيريدول، مثبطات كيناز التيروزين، إيرينوتيكان، سيسبلاتين وتلعب دورًا حاسمًا في المقاومة في الزوائد في الأمعاء الغليظة وسرطان القولون [316-319]. علاوة على ذلك، في سرطان الكبد الخلوي (HCC) وسرطان القناة الصفراوية (CGC) واللوكيميا النخاعية المزمنة (CML) يبدو أن تراكم أدوية السرطان أقل
(مثل إيماتينيب) يبدو أنه يتم بوساطة انخفاض في تعبير OATP1B1 وOATP1B3 وOATP1A2 أو وظيفتها [317، 320، 321]. أظهرت دراسات مختلفة أن ناقل الكاتيون العضوي-1 (OCT1) يشارك في امتصاص أدوية مضادة للسرطان الكاتيونية القوية، مثل سيسبلاتين، الأنثراسيكلينات، وسورافينيب وأن نشاطه غير منظم في العديد من السرطانات مثل سرطان القولون والكبد [322-325]. بالإضافة إلى ذلك، تم إثبات أن امتصاص إيماتينيب في CML يعتمد على تعبير OCT1 ويعتبر درجة تعبير OCT1 علامة حيوية مفيدة للتنبؤ بفعالية العلاج القائم على إيماتينيب في مرضى اللوكيميا [326]. تم مؤخرًا إظهار أن الناقل النحاسي عالي الألفة (CTR1) ينقل أدوية البلاتين، مما يبرز الوظيفة الحاسمة لـ CTR1 في حساسية أدوية البلاتين في العلاج الكيميائي للسرطان [327، 328]. أظهر تجربة سريرية واعدة من المرحلة الأولى باستخدام العلاج المشترك للترينتين (مخلب نحاسي) وكاربوبلاتين أن CTR1 يحقق امتصاصًا أعلى لسيسبلاتين ونتائج أفضل [329، 330].
تستخدم الخلايا السرطانية البلعمة الذاتية وإجهاد الشبكة الإندوبلازمية (UPR) للحصول على الدعم لمقاومة الأدوية
الموت الخلوي المبرمج من النوع 2 [133، 341]. في أنواع مختلفة من السرطان، يمكن أن تلعب الأوتوفاجي دورًا متناقضًا إما في دعم أو تحفيز الموت وتعتمد على السياق [342-347]. ومع ذلك، فإن دور الأوتوفاجي في مقاومة الأدوية هو موضوع ناشئ وفهم عميق لهذه العلاقة يمكن أن يكون حاسمًا من الناحية العلاجية للحد من أنواع السرطان المختلفة. أظهرت دراسات متعددة أن الأوتوفاجي يلعب دورًا مركزيًا في مقاومة الأدوية من خلال إعادة تدوير الجزيئات الحيوية، وتفكيك البروتينات والأعضاء المشوهة، وبالتالي منع تلف الحمض النووي [348-350]. وقد اقترحت بعض التقارير أن استجابة تلف الحمض النووي يمكن أن تنشط أيضًا الأوتوفاجي عبر مسار إصلاح ATM (الذي يسبب عدم التنسيق) وإعادة التركيب المتماثل (HR) [351، 352]. وقد وُجد أن دواء الأنثراسيكلين إبيروبيسين الذي يحفز الأوتوفاجي يزيد من تنظيم بروتينات P-gp ويقلل من تنظيم مسار إشارة NF-кB، مما يعيق تفعيل الموت الخلوي ويعزز مقاومة الأدوية [353]. تم تعزيز حساسية التاموكسيفين في خلايا سرطان الثدي الإيجابية لمستقبلات الإستروجين المقاومة من خلال تثبيط الأوتوفاجي، مما يؤدي إلى تحفيز موت الخلايا [354، 355]. في الأورام السليفة المعوية، يتسبب العلاج المشترك مع إيماتينيب ومثبطات الأوتوفاجي (مثل الكلوروكين) في تحفيز الموت الخلوي [356، 357]. الشبكة الإندوبلازمية (ER) هي هيكل فرعي خلوي أساسي يحافظ على توازن الخلايا ويمكن أن تتعطل بسبب مجموعة متنوعة من الحالات المرضية مثل السرطان، مما يؤدي إلى تحفيز إجهاد الشبكة الإندوبلازمية، والذي إذا استمر يمكن أن يقتل خلايا السرطان من خلال تحفيز الموت الخلوي والموت الحديدي أو يساعدها على النمو والبقاء وتحفيز مقاومة الأدوية إذا تم تنشيطه بشكل معتدل [358]. يؤدي إجهاد الشبكة الإندوبلازمية إلى UPR، الذي يتم تنظيمه بواسطة إنزيم يتطلب الإينوزيتول-1 (IRE1).
من الموت الخلوي المبرمج. تم إظهار أن الالتهام الذاتي يؤثر على بعض الإنزيمات المقاومة للأدوية الحاسمة (إنزيمات إزالة السموم) مثل إنزيم الألدهايد ديهيدروجيناز (ALD1A3) وبالتالي يتوسط في مقاومة الأدوية المكتسبة في خلايا الورم الدبقي البشري المعالجة بالتيموزولوميد. في خلايا المبيض البشري (متلازمة المبيض المتعدد الكيسات – PCOS) يلعب تحفيز الالتهام الذاتي الشاذ عند إطلاق مجموعة الصندوق عالي الحركة 1 (HMGB1) دورًا في تحقيق مقاومة الأنسولين من خلال تقليل تنظيم IRS-1 وAKT وانتقال GLUT4. بينما في خلايا سرطان الكبد الخلوي، تم إظهار أن HMGB1 يعزز مقاومة الدوكسوروبيسين من خلال تحفيز الالتهام الذاتي AMPK وحمايتها من الموت الخلوي المبرمج من النوع 1. وُجد أن الالتهام الذاتي يلعب دورًا وقائيًا خلويًا في خلايا مقاومة الموت TNF-TRAIL من خلال احتجاز وتفكيك وتعطيل الكاسبيز 8، مما يحمي خلايا السرطان من الموت. ومع ذلك، هناك تقارير عديدة تفيد بأن تنشيط الالتهام الذاتي لفترة طويلة أو مستدامة يمكن أن يؤدي إلى الموت الخلوي المبرمج من النوع 2. على سبيل المثال، يؤدي علاج الريسفيراترول (مركب نباتي مشتق) إلى تحفيز إشارة قوية للالتهام الذاتي (تراكم p62) من خلال تنشيط JNK مما يؤدي إلى الموت في خلايا اللوكيميا المزمنة الميول النخاعية المقاومة للإيماتينيب. في بعض الحالات، يؤدي تنشيط الالتهام الذاتي إلى قمع مقاومة الأدوية ويحفز الموت المعتمد على العلاج أو غير المعتمد عليه. على سبيل المثال، أظهر العلاج المشترك لـ ABT-88 (مثبط بروتين البوليميراز) والتيموزولوميد أنه ي Sensitize خلايا الورم الدبقي المقاومة للتيموزولوميد من خلال تحفيز انكسارات مزدوجة في الحمض النووي وتفعيل الالتهام الذاتي القاتل والموت الخلوي في الوقت نفسه. لذا، فإن الالتهام الذاتي في معظم الحالات يعارض تأثير الأدوية المضادة للسرطان على خلايا الموت من خلال الموت الخلوي المبرمج، وبالتالي يعمل كمسار خلوي دفاعي ويدعم مقاومة الأدوية.
تغيير البروتيوم استجابةً للأدوية الكيميائية المعروفة لتحقيق مقاومة الأدوية (الشكل 6)
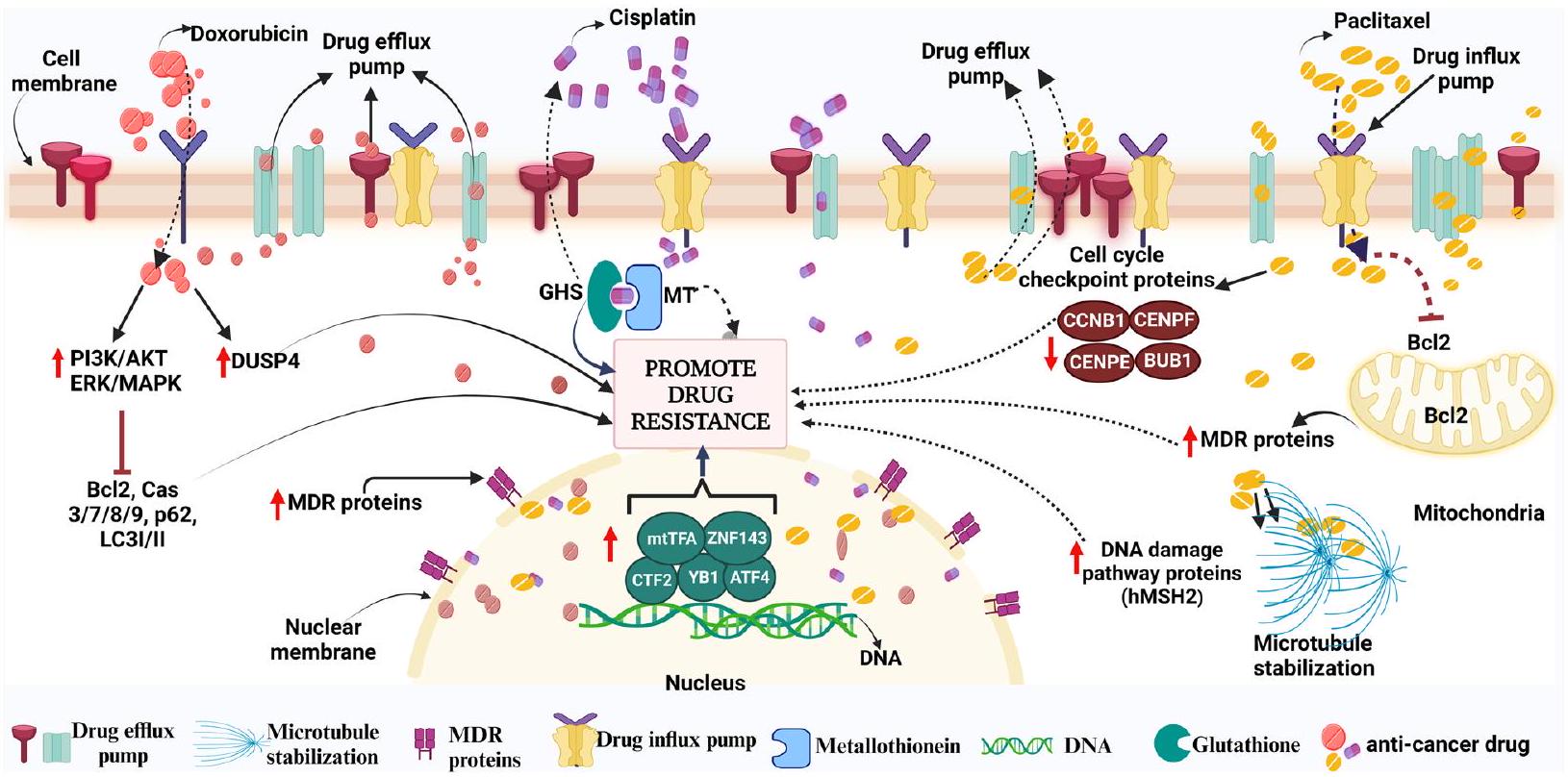
وبالمثل، فإن مقاومة السيسبلاتين تظل عائقًا في علاج أنواع مختلفة من السرطان. عند دخولها إلى الخلية، تتفاعل مع مجموعة من الجزيئات بخلاف الحمض النووي، مثل الجلوتاثيون المحتوي على الكبريت (GSH)/المعدن الثيونيين (MT) التي تحبس السيسبلاتين ثم تقضي عليه من الخلية. علاوة على ذلك، فإن عامل الزنك الأصبع 143 (ZNF143)، وبروتين ربط Y-box-1 (YB-1)، وعامل النسخ المنشط 4 (ATF4)، وعامل النسخ المرتبط بـ CCAAT 2 (CTF2)، وبروتينات إصلاح الحمض النووي (مثل منتج جين XRCC1؛ YB1؛ إلخ) وعامل النسخ الميتوكوندري A (mtTFA) هي بعض عوامل النسخ التي تم ربطها بمقاومة CDDP. دواء معروف آخر هو باكليتاكسيل (PTX)، الذي ينتمي إلى فئة التاكسين من العوامل المضادة للسرطان التي تؤثر على الاستقرار الطبيعي للأنابيب الدقيقة أثناء انقسام الخلايا.
فعّال ضد أنواع مختلفة من السرطان مثل سرطان الثدي والمبيض، إلخ. على الرغم من أن التوبولين هو الهدف الرئيسي لـ PTX، فقد تم اكتشاف أنه يهاجم أيضًا الميتوكوندريا ويعيق نشاط بروتين مثبط الموت الخلوي Bcl-2 (بروتين سرطان الدم B للخلايا). مثل الأدوية المضادة للسرطان الأخرى، يمكن أن يؤدي علاج PTX إلى مقاومة عن طريق تحفيز التعبير المفرط لبروتين المحرك MCAK (الذي يؤدي إلى تفكيك التوبولين)، ويؤثر على الدهون الغشائية (مثل سينثاز الأحماض الدهنية، ليبين، إلخ)، وبروتينات نقاط تفتيش دورة الخلية المعدلة (BUB1، CCNB1، CENPE، CENPF)، وزيادة بروتينات مسارات إصلاح تلف الحمض النووي (مثل hMSH2)، وارتفاع بروتينات الطرد (MDR1، MDR3، إلخ).
نقاش
تحويل البيئة المضادة للورم إلى بيئة داعمة ومؤيدة للورم. علاوة على ذلك، يمكن لبعض خلايا الورم أن تحفز إعادة برمجة خلايا السدى وخلايا المناعة، مما يؤدي إلى إفراز عوامل متنوعة مثل السيتوكينات، التي تعزز تقدم الورم وتثبط موت الخلايا. بالإضافة إلى ذلك، ظهرت خلايا السرطان الجذعية كلاعبين رئيسيين في المشهد المعقد لمقاومة أدوية السرطان. تكتسب هذه الخلايا قدرات ملحوظة لمقاومة العلاجات التقليدية، مما يؤدي إلى تكرار المرض وانتشاره كما تم شرحه بالتفصيل في القسم الرابع. تلعب خلايا السرطان الجذعية دورًا رئيسيًا في تطوير مقاومة الأدوية وانتكاسة الورم. تساهم الخصائص الفريدة لخلايا السرطان الجذعية، مثل التجديد الذاتي وإمكانية التمايز، وزيادة التعبير عن لجنات مثبطة للمناعة على سطح الغشاء وإفراز مجموعة متنوعة من العوامل الكيميائية/السيتوكينات، بشكل جماعي في مقاومة العلاج وتطرح تحديات كبيرة في تحقيق الشفاء على المدى الطويل. لذلك، فإن استهداف خلايا السرطان الجذعية يفتح آفاقًا لاستراتيجيات علاجية مبتكرة يمكن استخدامها بالاشتراك مع أدوية كيميائية أخرى قد تحمل وعدًا في التغلب على مقاومة الأدوية وتحسين نتائج المرضى. واحدة من الآليات السائدة لمقاومة الأدوية التي تحتاج إلى التركيز عليها هي المشاركة في طرد الأدوية الكارهة للماء، التي تسهلها ناقلات ABC المعتمدة على ATP. أحد الأعضاء المدروسة جيدًا من ناقلات ABC، P-gp، وهو بروتين غشائي متكامل، يتم تنظيمه بشكل متكرر في أنواع مختلفة من الأورام. على وجه التحديد، من المحتمل أن يلعب فهم شامل للآليات المعقدة التي تكمن وراء مقاومة الأدوية المتعددة في خلايا السرطان دورًا محوريًا في تطوير أساليب مبتكرة لعلاج السرطان في السنوات القادمة. ومع ذلك، هناك حاجة إلى مزيد من العمل من المستوى الجذري باستخدام اختبارات عالية الإنتاجية (على مستوى الخلية الواحدة) لمختلف أقسام نفس عينات الورم للعثور على هدف محدد للعلاج أو العلاج المركب كما تم مناقشته أعلاه. قد يساعد تحقيق هذا الهدف في معالجة خلايا السرطان العدوانية والمقاومة المتطورة مما يؤدي إلى تقليل الشدة وتحسين بقاء المرضى.
| اختصارات | |
| مراكز خدمة العملاء | خلايا السرطان الجذعية |
| CTLA-4 | مستضد مرتبط بالخلايا التائية السامة للخلايا 4 |
| PD-1 | بروتين موت الخلايا المبرمج 1 |
| PD-L1 | الليغاند المبرمج للموت 1 |
| سرطان الرئة غير صغير الخلايا | سرطان الرئة غير صغير الخلايا |
| MDR | مقاومة متعددة الأدوية |
| HCC | سرطان الخلايا الكبدية |
| DTP | المقاوم للعقاقير |
| DNMTs | ميثيل ترانسفيراز الحمض النووي |
| دي إن إيه م | ميثيلation الحمض النووي |
| 5-FU | 5-فلورويوراسيل |
| P-gp | بروتين جليكوبروتين P |
| بي سي إل-2 | ليمفومة خلايا ب 2 |
| بي سي إل-إكس إل | ليمفوما الخلايا البائية – كبيرة جداً |
| البرامج المعتمدة على الإنترنت | مثبطات الموت الخلوي |
| عرض | مُعَذِّب الموت المرتبط بنطاق BH3 |
| باكس | بروتين X المرتبط بـ Bcl-2 |
| بم | وسيط Bcl-2 المتفاعل مع موت الخلايا |
| بومه | منظم موت الخلايا المبرمج المعزز بواسطة P53 |
| CRC | سرطان القولون والمستقيم |
| دوكس | دوكسوروبيسين |
| MAPK | كيناز البروتين المنشط بواسطة الميتوجين |
| KMTs | ميثيل ترانسفيراز ليسين الهيستون |
| PRMTs | نقل الميثيل للأرجينين بروتين |
| جي إس إتش | جلوتاثيون |
| دي إن إيه-بي كيه | كيناز البروتين المعتمد على الحمض النووي |
| SASP | الظاهرة الإفرازية المرتبطة بالشيخوخة |
| مكافحة غسل الأموال | سرطان الدم النخاعي الحاد |
| نوتش 1 | بروتين 1 المتماثل لموقع نوتش العصبي |
| Wnt | موقع تكامل مرتبط بدون أجنحة |
| TGF-
|
عامل النمو المحول-
|
| الخلايا الجذعية الجنينية | خلايا جذعية جنينية |
| SOX2 | منطقة تحديد الجنس Y-box 2 |
| أكتوبر 4 | عامل النسخ المرتبط بالثماني 4 |
| KLF4 | عامل كروبل الشبيه 4 |
| SALL4 | بروتين شبيه سال 4 |
| فوكس إم 1 | بروتين فوكهيد صندوق M1 |
| هـهـ | القنفذ |
| شيك | كيناز نقطة التفتيش |
| يو بي سي | سرطان المثانة urothelial |
| فني الطوارئ الطبية | الانتقال من الظهارة إلى الميزانشيم |
| ميت | الانتقال من الخلايا الميزانشيمية إلى الخلايا الظهارية |
| سرطان الثدي الثلاثي السلبي | سرطان الثدي الثلاثي السلبي |
| ABCG | عائلة ناقلات ATP الفرعية G |
| FBXO21 | بروتين F-Box 21 |
| GSTp | جلوتاثيون S-ترانسفيراز ب |
| uPA | منشط بلازمينوجين يوروكيناز |
| HIF-1α | عامل تحفيز نقص الأكسجين-1 ألفا |
| كريسبر/كاس9 | تكرارات متباعدة بانتظام قصيرة متطابقة/ كاسبيز 9 |
| ATG | مرتبط بالالتهام الذاتي |
| روس | أنواع الأكسجين التفاعلية |
| ALDH | ديهيدروجيناز الألدهيد |
| TNBCSC | خلايا جذعية لسرطان الثدي الثلاثي السلبية |
| جاك/ستات | كيناز جانوس / ناقلات الإشارات ومفعّلات النسخ |
| إل | إنترلوكين |
| CCL2 | الليغاند الكيميائي CC-2 |
| CSF1 | عامل تحفيز المستعمرات 1 |
| CSF2 | عامل تحفيز المستعمرات 2 |
| HGF | عامل نمو الكبد |
| ميف | عامل تثبيط هجرة البلعميات |
| CX3CL1 | الليغاند الكيميائي C-X3-C |
| PGE2 | بروستاجلاندين E2 |
| SDF-1 | عامل مشتق من الخلايا الداعمة 1 |
| لوكس | ليزيل أوكسيداز |
| CCL3 | الليغاند الكيميائي CC-3 |
| CCL5 | الليغاند الكيميائي CC 5 |
| VEGF-A | عامل نمو بطانة الأوعية الدموية A |
| PTN | بليوتروفين |
| HMGB1 | مجموعة الصناديق عالية الحركة 1 |
| تام | البلاعم المرتبطة بالورم |
| EGFR | مستقبل عامل نمو البشرة |
| IDO1 | إنزيم إندولامين 2,3-ديوكسيجيناز 1 |
| PGE | بروستاجلاندين |
| ENTPD2 | دي فوسفات هيدروكسيلاز 2 |
| قرص مضغوط | عناقيد التمايز |
| MDSC | خلايا مثبطة مشتقة من النخاع الشوكي |
| خلايا CART | خلايا T مستقبلات المستضدات الكيميرية |
| CRS | متلازمة إطلاق السيتوكينات |
| زEB1 | البروتين المرتبط بصندوق الزنك E-box 1 |
| HDAC | هيستون ديأسيتيلز |
| الصراف الآلي | المتغير في التأتكسيا-تيلانجكتازيا |
| PCAF | عامل مرتبط بـ p300/CBP |
| FBXW7 | بروتين يحتوي على نطاق F-box وتكرار WD 7 |
| TWIST1 | بروتين مرتبط بالتواء 1 |
| LMP | نفاذية غشاء الليزوزوم |
| UCP2 | بروتين مفكك الارتباط 2 |
| mTOR | هدف الراباميسين في الثدييات |
| NF-kB | عامل النسخ النووي – كابا بي |
| STAT3 | موصل الإشارة ومفعل النسخ 3 |
| سعر البيع المقترح | بروتين مقاومة متعدد الأدوية |
| مسار | الليغاند المحفز للموت الخلوي المرتبط بالورم |
| جي بي إم | ورم دبقي متعدد الأشكال |
| فيم | فيمنتين |
| VDAC | قناة الأنيونات المعتمدة على الجهد |
| OHCP | 4-هيدروكسي-سيكلوفوسفاميد |
| OATPs | بروتينات نقل الأنيونات العضوية |
| LRP | بروتين مرتبط بمقاومة الرئة |
| UPR | استجابة البروتين غير المطوي |
| LC3 | سلسلة خفيفة 3 |
| سي إم إل | خلايا اللوكيميا النقوية المزمنة (CML) |
| JNK | كيناز الطرف N لجون |
| DUSP4 | فوسفاتاز البروتين ذو الخصوصية المزدوجة 4 |
| إي آر كيه | كيناز الإشارة المنظمة خارج الخلية |
| ATF4 | تفعيل عامل النسخ 4 |
| ZNF143 | عامل إصبع الزنك 143 |
| YB-1 | بروتين ربط Y-box-1 |
| بوبا1 | تبرعم غير مقيد بواسطة البنزيدازولات 1 |
| CCNB1 | سايكلين ب1 |
| سينبي | بروتين مركز الكروموسوم E |
| CENPF | بروتين مركزية الكروموسوم F |
شكر وتقدير
مساهمات المؤلفين
تمويل
توفر البيانات والمواد
الإعلانات
موافقة الأخلاقيات والموافقة على المشاركة
موافقة على النشر
المصالح المتنافسة
نُشر على الإنترنت: 12 فبراير 2024
References
- Nikolaou
, et al. The challenge of drug resistance in cancer treatment: a current overview. Clin Exp Metastasis. 2018;35(4):309-18. - Saha M, Sarkar A. Review on Multiple Facets of Drug Resistance: A Rising Challenge in the 21st Century. J Xenobiot. 2021;11(4):197-214.
- Boumahdi S, de Sauvage FJ. The great escape: tumour cell plasticity in resistance to targeted therapy. Nat Rev Drug Discov. 2020;19(1):39-56.
- Michaelis M, Wass MN, Cinatl J. Drug-adapted cancer cell lines as preclinical models of acquired resistance. Cancer Drug Resist. 2019;2(3):447-56.
- Oun R, Moussa YE, Wheate NJ. The side effects of platinumbased chemotherapy drugs: a review for chemists. Dalton Trans. 2018;47(19):6645-53.
- Tao JJ, Visvanathan K, Wolff AC. Long term side effects of adjuvant chemotherapy in patients with early breast cancer. Breast. 2015;24:S149-53.
- Aramini
, et al. Dissecting tumor growth: The role of cancer stem cells in drug resistance and recurrence. Cancers (Basel). 2022;14(4):976. - Masoudi M, Gopalan V. Roles of Cancer Stem Cells in Therapy Resistance and Disease Recurrence. In: Cancer Stem Cells: Basic Concept and Therapeutic Implications. Springer; 2023. p. 149-65.
- Zhang Z, et al. Overcoming cancer therapeutic bottleneck by drug repurposing. Signal Transduct Target Ther. 2020;5(1):113.
- DeRidder L, et al. The past, present, and future of chemotherapy with a focus on individualization of drug dosing. J Control Release. 2022;352:840-60.
- XuH , et al. Targeting strategies for bone diseases: signaling pathways and clinical studies. Signal Transduct Target Ther. 2023;8(1):202.
- Miglietta F, et al. Endocrine sequelae of hematopoietic stem cell transplantation: Effects on mineral homeostasis and bone metabolism. Front Endocrinol (Lausanne). 2023;13:1085315.
- Mansoori
, et al. The different mechanisms of cancer drug resistance: brief review. Adv Pharm Bull. 2017;7(3):339. - Wang
, Zhang , Chen . Drug resistance and combating drug resistance in cancer. Cancer Drug Resist. 2019;2(2):141. - Dzobo K, et al. Not Everyone Fits the Mold: Intratumor and Intertumor Heterogeneity and Innovative Cancer Drug Design and Development. OMICS. 2018;22(1):17-34.
- Labrie M, et al. Therapy resistance: opportunities created by adaptive responses to targeted therapies in cancer. Nat Rev Cancer. 2022;22(6):323-39.
- Nussinov R, Tsai CJ, Jang H. Anticancer drug resistance: An update and perspective. Drug Resist Updat. 2021;59:100796.
- Dzobo K, Senthebane DA, Dandara C. The Tumor Microenvironment in Tumorigenesis and Therapy Resistance Revisited. Cancers (Basel). 2023;15(2):376.
- Dzobo K, et al. Advances in Therapeutic Targeting of Cancer Stem Cells within the Tumor Microenvironment: An Updated Review. Cells. 2020;9(8):1896.
- Senthebane DA, et al. The role of tumor microenvironment in chemoresistance: to survive, keep your enemies closer. Int J Mol Sci. 2017;18(7):1586.
- Khan SU, Fatima K, Malik F, Kalkavan H, Wani A. Cancer metastasis: Molecular mechanisms and clinical perspectives. Pharmacol Ther. 2023;250:108522. https://doi.org/10.1016/j.pharmthera.2023.108522.
- Yang Q, et al. Extracellular Vesicles in Cancer Drug Resistance: Roles, Mechanisms, and Implications. Adv Sci (Weinh). 2022;9(34):e2201609.
- Namee NM, O’Driscoll L. Extracellular vesicles and anti-cancer drug resistance. Biochim Biophys Acta Rev Cancer. 2018;1870(2):123-36.
- Wu S. Fu L, Tyrosine kinase inhibitors enhanced the efficacy of conventional chemotherapeutic agent in multidrug resistant cancer cells. Mol Cancer. 2018;17(1):1-13.
- Roviello G , et al. Apatinib: a novel receptor tyrosine kinase inhibitor for the treatment of gastric cancer. Cancer Lett. 2016;372(2):187-91.
- Martin SK, et al. N-terminal targeting of androgen receptor variant enhances response of castration resistant prostate cancer to taxane chemotherapy. Mol Oncol. 2015;9(3):628-39.
- Fujita K, Nonomura N. Role of androgen receptor in prostate cancer: a review. World J Mens Health. 2019;37(3):288-95.
- Asano
, et al. Clinical verification of sensitivity to preoperative chemotherapy in cases of androgen receptor-expressing positive breast cancer. Br J Cancer. 2016;114(1):14-20. - Barton VN, et al. Androgen receptor supports an anchorage-independent, cancer stem cell-like population in triple-negative breast cancer. Cancer Res. 2017;77(13):3455-66.
- Niikura N, et al. Changes in tumor expression of HER2 and hormone receptors status after neoadjuvant chemotherapy in 21755 patients from the Japanese breast cancer registry. Ann Oncol. 2016;27(3):480-7.
- Tolaney SM, et al. Abemaciclib plus trastuzumab with or without fulvestrant versus trastuzumab plus standard-of-care chemotherapy in women with hormone receptor-positive, HER2-positive advanced breast cancer (monarcHER): a randomised, open-label, phase 2 trial. Lancet Oncol. 2020;21(6):763-75.
- Calabrò L, et al. Efficacy and safety of an intensified schedule of tremelimumab for chemotherapy-resistant malignant mesothelioma: an openlabel, single-arm, phase 2 study. Lancet Respir Med. 2015;3(4):301-9.
- Calabrò L, et al. Tremelimumab for patients with chemotherapy-resistant advanced malignant mesothelioma: an open-label, single-arm, phase 2 trial. Lancet Oncol. 2013;14(11):1104-11.
- Noguchi T, et al. Temporally Distinct PD-L1 Expression by Tumor and Host Cells Contributes to Immune EscapeThe Role of Tumor versus Host PD-L1 in Tumor Immune Escape. Cancer Immunol Res. 2017;5(2):106-17.
- Juneja VR, et al. PD-L1 on tumor cells is sufficient for immune evasion in immunogenic tumors and inhibits CD8 T cell cytotoxicity. J Exp Med. 2017;214(4):895-904.
- Ribas A, Wolchok JD. Cancer immunotherapy using checkpoint blockade. Science. 2018;359(6382):1350-5.
- Shi Z-D, et al. Tumor cell plasticity in targeted therapy-induced resistance: mechanisms and new strategies. Signal Transduct Target Ther. 2023;8(1):113.
- Prasad B, et al. Exploration of space to achieve scientific breakthroughs. Biotechnol Adv. 2020;43:107572.
- Jin H, Wang L, Bernards R. Rational combinations of targeted cancer therapies: background, advances and challenges. Nat Rev Drug Discovery. 2023;22(3):213-34.
- Lim Z-F, Ma PC. Emerging insights of tumor heterogeneity and drug resistance mechanisms in lung cancer targeted therapy. J Hematol Oncol. 2019;12(1):1-18.
- Krook MA, et al. Tumor heterogeneity and acquired drug resistance in FGFR2-fusion-positive cholangiocarcinoma through rapid research autopsy. Cold Spring Harb Mol Case Stud. 2019;5(4):a004002.
- Guillon J, et al. Chemotherapy-induced senescence, an adaptive mechanism driving resistance and tumor heterogeneity. Cell Cycle. 2019;18(19):2385-97.
- Hass R, von der Ohe J, Ungefroren HJC. Impact of the tumor microenvironment on tumor heterogeneity and consequences for cancer cell plasticity and stemness. Cancers (Basel). 2020;12(12):3716.
- Punter KB, Chu CE, Chan YW. Mitochondrial dynamics and oxidative phosphorylation as critical targets in cancer. Endocr Relat Cancer. 2023;30(1):e220229.
- Hanselmann RG, Welter C. Origin of Cancer: Cell work is the Key to Understanding Cancer Initiation and Progression. Front Cell Dev Biol. 2022;10:787995. https://doi.org/10.3389/fcell.2022.787995.
- Jiang W, et al. Personalized medicine of non-gene-specific chemotherapies for non-small cell lung cancer. Acta Pharm Sin B. 2021;11(11):3406-16.
- Capdevila J, et al. Molecular diagnosis and targeted treatment of advanced follicular cell-derived thyroid cancer in the precision medicine era. Cancer Treat Rev. 2022;106:102380.
- Bhang H-e C, et al. Studying clonal dynamics in response to cancer therapy using high-complexity barcoding. Nat Med. 2015;21(5):440-8.
- Jamal-Hanjani M, et al. Tracking the evolution of non-small-cell lung cancer. N Engl J Med. 2017;376(22):2109-21.
- Singh AK, et al. Tumor heterogeneity and cancer stem cell paradigm: updates in concept, controversies and clinical relevance. Int J Cancer. 2015;136(9):1991-2000.
- Huang, R., P.-K.J.S.T. Zhou, and T. Therapy. DNA damage repair: Historical perspectives, mechanistic pathways and clinical translation for targeted cancer therapy. Signal Transduct Target Ther. 2021;6(1):254.
- Roberts SA, Gordenin DA. Hypermutation in human cancer genomes: footprints and mechanisms. Nat Rev Cancer. 2014;14(12):786-800.
- Khan SU, et al. Redox balance and autophagy regulation in cancer progression and their therapeutic perspective. Med Oncol. 2023;40(1):1-21.
- Sui Q , et al. Genetic and microenvironmental differences in nonsmoking lung adenocarcinoma patients compared with smoking patients. Transl Lung Cancer Res. 2020;9(4):1407.
- Anagnostou V, et al. Multimodal genomic features predict outcome of immune checkpoint blockade in non-small-cell lung cancer. Nat Cancer. 2020;1(1):99-111.
- Connor AA. Gallinger S, Pancreatic cancer evolution and heterogeneity: integrating omics and clinical data. Nat Rev Cancer. 2022;22(3):131-42.
- Killcoyne S, Fitzgerald RC. Evolution and progression of Barrett’s oesophagus to oesophageal cancer. Nat Rev Cancer. 2021;21(11):731-41.
- Sjödahl G, et al. Molecular changes during progression from nonmuscle invasive to advanced urothelial carcinoma. Int J Cancer. 2020;146(9):2636-47.
- van Niekerk G, et al. C ancer stem cells: A product of clonal evolution? Int J Cancer. 2017;140(5):993-9.
- Losic B, et al. Intratumoral heterogeneity and clonal evolution in liver cancer. Nat Commun. 2020;11(1):1-15.
- Zardavas D, et al. Clinical management of breast cancer heterogeneity. Nat Rev Clin Oncol. 2015;12(7):381-94.
- AI-Rawi DH, Bakhoum SF. Chromosomal instability as a source of genomic plasticity. Curr Opin Genet Dev. 2022;74:101913.
- van den Bosch T, Derks S, Miedema DMJC. Chromosomal Instability Selection and Competition: Factors That Shape the Level of Karyotype Intra-Tumor Heterogeneity. Cancers (Basel). 2022;14(20):4986.
- Comaills V, Castellano-Pozo MJB. Chromosomal Instability in Genome Evolution: From Cancer to Macroevolution. Biology (Basel). 2023;12(5):671.
- Majc B, et al. Epithelial-to-mesenchymal transition as the driver of changing carcinoma and glioblastoma microenvironment. Biochim Biophys Acta Mol Cell Res. 2020;1867(10):118782.
- Levitin HM, Yuan J, Sims PA. Single-cell transcriptomic analysis of tumor heterogeneity. Trends Cancer. 2018;4(4):264-8.
- Fan J, et al. Linking transcriptional and genetic tumor heterogeneity through allele analysis of single-cell RNA-seq data. Genome Res. 2018;28(8):1217-27.
- Peng J, et al. Single-cell RNA-seq highlights intra-tumoral heterogeneity and malignant progression in pancreatic ductal adenocarcinoma. Cell Res. 2019;29(9):725-38.
- Ding S , Chen X , Shen KJCC. Single-cell RNA sequencing in breast cancer: Understanding tumor heterogeneity and paving roads to individualized therapy. Cancer Commun (Lond). 2020;40(8):329-44.
- Hata AN, et al. Tumor cells can follow distinct evolutionary paths to become resistant to epidermal growth factor receptor inhibition. Nat Med. 2016;22(3):262-9.
- Ho DW-H, et al. Single-cell RNA sequencing shows the immunosuppressive landscape and tumor heterogeneity of HBV-associated hepatocellular carcinoma. Nat Commun. 2021;12(1):1-14.
- Cheng Y, He C, Wang M, Ma X, Mo F, Yang S, Han J, Wei X. Targeting epigenetic regulators for cancer therapy: mechanisms and advances in clinical trials. Signal Transduct Target Ther. 2019;4:62. https://doi.org/10. 1038/s41392-019-0095-0.
- Mancarella D, Plass C. Epigenetic signatures in cancer: proper controls, current challenges and the potential for clinical translation. Genome Med. 2021;13:1-12.
- Mikubo M , et al. Mechanism of drug tolerant persister cancer cells: the landscape and clinical implication for therapy. J Thorac Oncol. 2021;16(11):1798-809.
- De Conti G, Dias MH , Bernards RJC. Fighting drug resistance through the targeting of drug-tolerant persister cells. Cancers (Basel). 2021;13(5):1118.
- Toh TB, Lim JJ, Chow EK-H. Epigenetics in cancer stem cells. Mol Cancer. 2017;16(1):1-20.
- Fouad MA, et al. Impact of global DNA methylation in treatment outcome of colorectal cancer patients. Front Pharmacol. 2018;9:1173.
- Lee DD, et al. DNA methylation of the TERT promoter and its impact on human cancer. Curr Opin Genet Dev. 2020;60:17-24.
- Wu Q, et al. Methylation of miR-129-5p CpG island modulates multidrug resistance in gastric cancer by targeting ABC transporters. Oncotarget. 2014;5(22):11552-63.
- Ni Y, et al. The role of tumor-stroma interactions in drug resistance within tumor microenvironment. Front Cell Dev Biol. 2021;9:637675.
- Shi Y, et al. Genome-wide DNA methylation analysis of breast cancer MCF-7/Taxol cells with MeDIP-Seq. PLoS One. 2020;15(12):e0241515.
- Aguiari G, et al. Dysregulation of Transglutaminase type 2 through GATA3 defines aggressiveness and Doxorubicin sensitivity in breast cancer. Int J Biol Sci. 2022;18(1):1.
- Mosca L, et al. Therapeutic potential of the natural compound S-adenosylmethionine as a chemoprotective synergistic agent in breast, and head and neck cancer treatment: Current status of research. Int J Mol Sci. 2020;21(22):8547.
- Williams MM, Cook RS. Bcl-2 family proteins in breast development and cancer: could Mcl-1 targeting overcome therapeutic resistance? Oncotarget. 2015;6(6):3519.
- Baharudin R, et al. Identification of predictive DNA methylation biomarkers for chemotherapy response in colorectal cancer. Front Pharmacol. 2017;8:47.
- Yang C, et al. Histone methyltransferase and drug resistance in cancers. J Exp Clin Cancer Res. 2020;39(1):173.
- Liu C-W, et al. Histone Methyltransferase G9a Drives Chemotherapy Resistance by Regulating the Glutamate-Cysteine Ligase Catalytic Subunit in Head and Neck Squamous Cell CarcinomaG9a Modulates GCLC Expression and Chemoresistance. Mol Cancer Ther. 2017;16(7):1421-34.
- Musiani D, et al. PRMT1 Is Recruited via DNA-PK to Chromatin Where It Sustains the Senescence-Associated Secretory Phenotype in Response to Cisplatin. Cell Rep. 2020;30(4):1208-1222.e9.
- Walcher L, et al. Cancer stem cells—origins and biomarkers: perspectives for targeted personalized therapies. Front Immunol. 2020;11:1280.
- Trumpp A, Haas SJC. Cancer stem cells: the adventurous journey from hematopoietic to leukemic stem cells. Cells. 2022;185(8):1266-70.
- Kapoor-Narula U, Lenka NJC. Cancer stem cells and tumor heterogeneity: Deciphering the role in tumor progression and metastasis. Cytokine. 2022;157:155968.
- De Angelis ML, Francescangeli F, Zeuner AJC. Breast cancer stem cells as drivers of tumor chemoresistance, dormancy and relapse: new challenges and therapeutic opportunities. Cancers (Basel). 2019;11(10):1569.
- Paul R, et al. Cell plasticity, senescence, and quiescence in cancer stem cells: Biological and therapeutic implications. Pharmacol Ther. 2022;231:107985.
- Tang H, et al. miR-200c suppresses stemness and increases cellular sensitivity to trastuzumab in HER2+ breast cancer. J Cell Mol Med. 2019;23(12):8114-27.
- Dittmer A, Dittmer JJC. Carcinoma-associated fibroblasts promote growth of Sox2-expressing breast cancer cells. Cancers (Basel). 2020;12(11):3435.
- Giuli MV, et al. Notch signaling in female cancers: A multifaceted node to overcome drug resistance. Cancer Drug Resist. 2021;4(4):805.
- Mourkioti I, et al. Interplay of Developmental Hippo-Notch Signaling Pathways with the DNA Damage Response in Prostate Cancer. Cells. 2022;11(15):2449.
- Aggarwal V, et al. NOTCH signaling: Journey of an evolutionarily conserved pathway in driving tumor progression and its modulation as a therapeutic target. Crit Rev Oncol Hematol. 2021;164:103403.
- Yan Q, et al. Oncofetal proteins and cancer stem cells. 2022.
- Sun D , et al. The IVF-generated human embryonic microenvironment reverses progestin resistance in endometrial cancer cells by inducing cancer stem cell differentiation. Cancer Lett. 2022;526:311-21.
- Yang Y, et al. Emerging agents that target signaling pathways in cancer stem cells. J Hematol Oncol. 2020;13:1-18.
- Cheng S-W, et al. Lin28B is an oncofetal circulating cancer stem cell-like marker associated with recurrence of hepatocellular carcinoma. PLoS ONE. 2013;8(11):e80053.
- Karsten U, Goletz S. What makes cancer stem cell markers different? Springerplus. 2013;2(1):1-8.
- Liu L, et al. Chemotherapy Induces Breast Cancer Stemness in Association with Dysregulated MonocytosisMCPs Mediate Chemother-apy-Induced Cancer Stemness. Clin Cancer Res. 2018;24(10):2370-82.
- Alhaddad L, Osipov AN, Leonov S. The Molecular and Cellular Strategies of Glioblastoma and Non-Small-Cell Lung Cancer Cells Conferring Radioresistance. Int J Mol Sci. 2022;23(21):13577.
- Bazan, N.G., et al., Multiprong control of glioblastoma multiforme invasiveness: blockade of pro-inflammatory signaling, anti-angiogenesis, and homeostasis restoration. 2021: p. 1-5.
- Angom RS, Mondal SK, Wang F, Madamsetty VS, Wang E, Dutta SK, Gulani Y, Sarabia-Estrada R, Sarkaria JN, Quiñones-Hinojosa A, Mukhopadhyay D. Ablation of neuropilin-1 improves the therapeutic response in conventional drug-resistant glioblastoma multiforme. Oncogene. 2020;39(48):7114-26. https://doi.org/10.1038/ s41388-020-01462-1.
- Bazan NG, Reid MM, Flores VAC, Gallo JE, Lewis W, Belayev L. Multiprong control of glioblastoma multiforme invasiveness: blockade of pro-inflammatory signaling, anti-angiogenesis, and homeostasis restoration. Cancer Metastasis Rev. 2021;40(3):643-7. https://doi.org/ 10.1007/s10555-021-09987-X.
- Douyère M, Chastagner P, Douyère M. Chastagner P, Boura, Neuropi-lin-1: a key protein to consider in the progression of pediatric brain tumors. Front Oncol. 2021;11:665634.
- Zhang Q, et al. miR34a/GOLPH3 axis abrogates urothelial bladder cancer chemoresistance via reduced cancer stemness. Theranostics. 2017;7(19):4777.
- Allegra A, et al. The cancer stem cell hypothesis: a guide to potential molecular targets. Cancer Invest. 2014;32(9):470-95.
- Castelli V, et al. The great escape: the power of cancer stem cells to evade programmed cell death. Cancers (Basel). 2021;13(2):328.
- Pan E, et al. Phase I study of RO4929097 with bevacizumab in patients with recurrent malignant glioma. J Neurooncol. 2016;130:571-9.
- Villalobos VM, et al. Long-term follow-up of desmoid fibromatosis treated with PF-03084014, an oral gamma secretase inhibitor. Ann Surg Oncol. 2018;25:768-75.
- Kimura K, et al. Safety, tolerability, and preliminary efficacy of the antifibrotic small molecule PRI-724, a CBP/
-catenin inhibitor, in patients with hepatitis C virus-related cirrhosis: a single-center, open-label, dose escalation phase 1 trial. EBioMedicine. 2017;23:79-87. - Dréno
, et al. Two intermittent vismodegib dosing regimens in patients with multiple basal-cell carcinomas (MIKIE): a randomised, regimen-controlled, double-blind, phase 2 trial. Lancet Oncol. 2017;18(3):404-12. - Shibue T, Weinberg RA. EMT, CSCs, and drug resistance: the mechanistic link and clinical implications. Nat Rev Clin Oncol. 2017;14(10):611-29.
- Khan SU, Fatima K, Malik F. Understanding the cell survival mechanism of anoikis-resistant cancer cells during different steps of metastasis. Clin Exp Metastasis. 2022;39(5):715-26. https://doi.org/10.1007/ s10585-022-10172-9.
- Shibata M, Hoque MOJC. Targeting cancer stem cells: a strategy for effective eradication of cancer. Cancers (Basel). 2019;11(5):732.
- Zinzi L, et al. ABC transporters in CSCs membranes as a novel target for treating tumor relapse. Front Pharmacol. 2014;5:163.
- Zhao
, et al. Eradication of cancer stem cells in triple negative breast cancer using doxorubicin/pluronic polymeric micelles. Nanomedicine. 2020;24:102124. - Kim EJ, et al. NRF2 knockdown resensitizes 5-fluorouracil-resistant pancreatic cancer cells by suppressing HO-1 and ABCG2 expression. Int J Mol Sci. 2020;21(13):4646.
- Eldaly, S.M., S.A. Gouhar, and M.T.J.E.J.o.C. Abo-elfadl, The Influence of 5-Fluorouracil on Drug Transporters is a Dose-Dependent Effect Mediated by Altered Expression of miRNAs. 2022. 65(8): p. 737-748.
- Kukal S, Guin D, Rawat C, Bora S, Mishra MK, Sharma P, Paul PR, Kanojia N, Grewal GK, Kukreti S, Saso L, Kukreti R. Multidrug efflux transporter ABCG2: expression and regulation. Cell Mol Life Sci. 2021;78(21-22):6887-939. https://doi.org/10.1007/s00018-021-03901-y.
- Hsu HH, Chen MC, Baskaran R, Lin YM, Day CH, Lin YJ, Tu CC, Vijaya Padma V, Kuo WW, Huang CY. Oxaliplatin resistance in colorectal cancer cells is mediated via activation of ABCG2 to alleviate ER stress induced apoptosis. J Cell Physiol. 2018;233(7):5458-67. https://doi.org/10.1002/jcp.26406.
- Ravindranath AK , et al. CD44 promotes multi-drug resistance by protecting P-glycoprotein from FBXO21-mediated ubiquitination. Oncotarget. 2015;6(28):26308.
- Zhang H, et al. Cancer stem cells, epithelial-mesenchymal transition, ATP and their roles in drug resistance in cancer. Cancer Drug Resist. 2021;4(3):684.
- Lv Y, et al. Hypoxia-inducible factor-1 a induces multidrug resistance protein in colon cancer. Onco Targets Ther. 2015;8:1941.
- Nedeljković M, Damjanović A. Mechanisms of chemotherapy resistance in triple-negative breast cancer-how we can rise to the challenge. Cells. 2019;8(9):957.
- Kim H, et al. The hypoxic tumor microenvironment in vivo selects the cancer stem cell fate of breast cancer cells. Breast Cancer Res. 2018;20(1):1-15.
- Jing
, et al. Role of hypoxia in cancer therapy by regulating the tumor microenvironment. Mol Cancer. 2019;18:1-15. - Khan, S.U., K. Fatima, and F. Malik, Understanding the cell survival mechanism of anoikis-resistant cancer cells during different steps of metastasis. Clin Exp Metastasis, 2022.
- Khan SU, et al. Activation of lysosomal mediated cell death in the course of autophagy by mTORC1 inhibitor. Sci Rep. 2022;12(1):1-13.
- Wani A, et al. Crocetin promotes clearance of amyloid-
by inducing autophagy via the STK11/LKB1-mediated AMPK pathway. Autophagy. 2021;17(11):3813-32. - Han Y, et al. Role of autophagy in breast cancer and breast cancer stem cells. Int J Oncol. 2018;52(4):1057-70.
- Gong C, et al. Beclin 1 and autophagy are required for the tumorigenicity of breast cancer stem-like/progenitor cells. Oncogene. 2013;32(18):2261-72.
- Pagotto A, et al. Autophagy inhibition reduces chemoresistance and tumorigenic potential of human ovarian cancer stem cells. Cell Death Dis. 2017;8(7):e2943-e2943.
- Liu W, et al. PRDX1 activates autophagy via the PTEN-AKT signaling pathway to protect against cisplatin-induced spiral ganglion neuron damage. Autophagy. 2021;17(12):4159-81.
- Khan SU, Rayees S, Sharma P, Malik F. Targeting redox regulation and autophagy systems in cancer stem cells. Clin Exp Med. 2023;23(5):140523. https://doi.org/10.1007/s10238-022-00955-5.
- Raha D, et al. The Cancer Stem Cell Marker Aldehyde Dehydrogenase Is Required to Maintain a Drug-Tolerant Tumor Cell SubpopulationAldehyde Dehydrogenase Contributes to Cancer Drug Resistance. Cancer Res. 2014;74(13):3579-90.
- Wu W, et al. Aldehyde dehydrogenase 1A3 (ALDH1A3) is regulated by autophagy in human glioblastoma cells. Cancer Lett. 2018;417:112-23.
- Yeo SK, et al. Autophagy Differentially Regulates Distinct Breast Cancer Stem-like Cells in Murine Models via EGFR/Stat3 and Tgfß/Smad SignalingRegulation of Distinct Breast Cancer Stem Cells by Autophagy. Cancer Res. 2016;76(11):3397-410.
- Shih P-C, Mei K. Role of STAT3 signaling transduction pathways in cancer stem cell-associated chemoresistance. Drug Discov Today. 2021;26(6):1450-8.
- Jin, M L, Jeong K W, Histone modifications in drug-resistant cancers: From a cancer stem cell and immune evasion perspective. 2023: 1-15.
- Zeng F, et al. Comprehensive profiling identifies a novel signature with robust predictive value and reveals the potential drug resistance mechanism in glioma. Cell Commun Signal. 2020;18:1-13.
- Chung AW, et al. Tocilizumab overcomes chemotherapy resistance in mesenchymal stem-like breast cancer by negating autocrine IL-1A induction of IL-6. NPJ Breast Cancer. 2022;8(1):1-10.
- Dianat-Moghadam H, et al. Cancer stem cells-emanated therapy resistance: implications for liposomal drug delivery systems. J Control Release. 2018;288:62-83.
- Codony-Servat J, Rosell R. Cancer stem cells and immunoresistance: clinical implications and solutions. Transl Lung Cancer Res. 2015;4(6):689-703. https://doi.org/10.3978/j.issn.2218-6751.2015.12.11.
- Najafi M. Mortezaee K, Majidpoor J, Cancer stem cell (CSC) resistance drivers. Life Sci. 2019;234:116781.
- Walker ND, et al. Exosomes from differentially activated macrophages influence dormancy or resurgence of breast cancer cells within bone marrow stroma. Cell Death Dis. 2019;10(2):59.
- Müller L, et al. Bidirectional crosstalk between cancer stem cells and immune cell subsets. Front Immunol. 2020;11:140.
- The role of stem cells in small-cell lung cancer: Evidence from chemoresistance to immunotherapy Seminars in Cancer Biology. 2022; 87160-169. https://doi.org/10.1016/j.semcancer.2022.11.006.
- Balaji S, et al. Emerging role of tumor microenvironment derived exosomes in therapeutic resistance and metastasis through epithelial-to-mesenchymal transition. Life Sci. 2021;280:119750.
- Luo S, et al. Macrophages Are a Double-Edged Sword: Molecular Crosstalk between Tumor-Associated Macrophages and Cancer Stem Cells. Biomolecules. 2022;12(6):850.
- Park SY, et al. Interleukin-8 is related to poor chemotherapeutic response and tumourigenicity in hepatocellular carcinoma. Eur J Cancer. 2014;50(2):341-50.
- Lei, M.M.L. and T.K.W. Lee, Cancer stem cells: Emerging key players in immune evasion of cancers. Frontiers in Cell and Developmental Biology, 2021: p. 1643.
- Lei Q, et al. Resistance mechanisms of anti-PD1/PDL1 therapy in solid tumors. Front Cell Dev Biol. 2020;8:672.
- Dianat-Moghadam H, et al. Immune evader cancer stem cells direct the perspective approaches to cancer immunotherapy. Stem Cell Res Ther. 2022;13(1):1-12.
- Lei MML, Lee TKW. Cancer Stem Cells: Emerging Key Players in Immune Evasion of Cancers. Front Cell Dev Biol. 2021;9:692940. https://doi.org/ 10.3389/fcell.2021.692940.
- Lee TKW, et al. Blockade of CD47-mediated cathepsin S/protease-activated receptor 2 signaling provides a therapeutic target for hepatocellular carcinoma. Hepatology. 2014;60(1):179-91.
- Nigro A, et al. Enhanced expression of CD47 is associated with offtarget resistance to tyrosine kinase inhibitor gefitinib in NSCLC. Front Immunol. 2020;10:3135.
- Zhong M , et al. Induction of tolerogenic dendritic cells by activated TGF-
Akt/Smad2 signaling in RIG-I-deficient stemness-high human liver cancer cells. BMC Cancer. 2019;19(1):1-15. - Ma Y, et al. Dendritic cells in the cancer microenvironment. J Cancer. 2013;4(1):36.
- Ravindran S, Rasool S, Maccalli CJCM. The cross talk between cancer stem cells/cancer initiating cells and tumor microenvironment: the missing piece of the puzzle for the efficient targeting of these cells with immunotherapy. Cancer Microenviron. 2019;12(2):133-48.
- Dianat-Moghadam H, Sharifi M, Salehi R, Keshavarz M, Shahgolzari M, Amoozgar Z. Engaging stemness improves cancer immunotherapy. Cancer Lett. 2023;554:216007. https://doi.org/10.1016/j.canlet.2022. 216007.
- Solis-Castillo LA, et al. Tumor-infiltrating regulatory T cells, CD8/Treg ratio, and cancer stem cells are correlated with lymph node metastasis in patients with early breast cancer. Breast Cancer. 2020;27(5):837-49.
- Komura N, et al. The role of myeloid-derived suppressor cells in increasing cancer stem-like cells and promoting PD-L1 expression in epithelial ovarian cancer. Cancer Immunol Immunother. 2020;69(12):2477-99.
- Tomić S, et al. Prostaglanin-E2 potentiates the suppressive functions of human mononuclear myeloid-derived suppressor cells and increases their capacity to expand IL-10-producing regulatory T cell subsets. Front Immunol. 2019;10:475.
- Wu B, et al. Cross-talk between cancer stem cells and immune cells: potential therapeutic targets in the tumor immune microenvironment. Mol Cancer. 2023;22(1):1-22.
- Chiu DK-C, et al. Hypoxia inducible factor HIF-1 promotes myeloidderived suppressor cells accumulation through ENTPD2/CD39L1 in hepatocellular carcinoma. Nat Commun. 2017;8(1):1-12.
- Xu M, et al. Interactions between interleukin-6 and myeloid-derived suppressor cells drive the chemoresistant phenotype of hepatocellular cancer. Exp Cell Res. 2017;351(2):142-9.
- Gardner RA, et al. Intent-to-treat leukemia remission by CD19 CART cells of defined formulation and dose in children and young adults. Exp Cell Res. 2017;129(25):3322-31.
- Maude SL, et al. Tisagenlecleucel in children and young adults with B-cell lymphoblastic leukemia. N Engl J Med. 2018;378(5):439-48.
- Fry TJ, et al. CD22-targeted CAR T cells induce remission in B-ALL that is naive or resistant to CD19-targeted CAR immunotherapy. Nat Med. 2018;24(1):20-8.
- Lee DW, et al. T cells expressing CD19 chimeric antigen receptors for acute lymphoblastic leukaemia in children and young adults: a phase 1 dose-escalation trial. Lancet. 2015;385(9967):517-28.
- Al-Haideri M, et al. CAR-T cell combination therapy: the next revolution in cancer treatment. Cancer Cell Int. 2022;22(1):1-26.
- Bozorgi A, Bozorgi M, Khazaei M. Immunotherapy and immunoengineering for breast cancer; a comprehensive insight into CAR-T cell therapy advancements, challenges and prospects. Cell Oncol (Dordr). 2022;45(5):755-77.
- Sterner RC, Sterner RM. CAR-T cell therapy: current limitations and potential strategies. Blood Cancer J. 2021;11(4):69. https://doi.org/10. 1038/s41408-021-00459-7.
- Scarfò I, Maus MV. Current approaches to increase CAR T cell potency in solid tumors: targeting the tumor microenvironment. J Immunother Cancer. 2017;5(1):28. https://doi.org/10.1186/s40425-017-0230-9.
- Du B, Shim JS. Targeting epithelial-mesenchymal transition (EMT) to overcome drug resistance in cancer. Molecules. 2016;21(7):965.
- Ribatti D, Tamma R, Annese T. Epithelial-mesenchymal transition in cancer: a historical overview. Transl Oncol. 2020;13(6):100773.
- Yang J, et al. Guidelines and definitions for research on epithelial-mesenchymal transition. Nat Rev Mol Cell Biol. 2020;21(6):341-52.
- Cevatemre B, et al. Pyruvate dehydrogenase contributes to drug resistance of lung cancer cells through epithelial mesenchymal transition. Front Cell Dev Biol. 2022;9:738916.
- Das V, et al. The basics of epithelial-mesenchymal transition (EMT): A study from a structure, dynamics, and functional perspective. J Cell Physiol. 2019;234(9):14535-55.
- Lu J, et al. ZEB1: catalyst of immune escape during tumor metastasis. Biomed Pharmacother. 2022;153:113490.
- Funato N, Yanagisawa H. TBX1 targets the miR-200-ZEB2 axis to induce epithelial differentiation and inhibit stem cell properties. Sci Rep. 2022;12(1):20188.
- Bencivenga M, Decimo I, Malpeli G. A therapeutic perspective for proliferative vitreoretinopathy based on the inhibition of epithelialmesenchymal transition by miR-194. Ann Transl Med. 2020;8(8):525. https://doi.org/10.21037/atm.2020.03.181.
- Title AC, et al. The miR-200-Zeb1 axis regulates key aspects of
-cell function and survival in vivo. Mol Metab. 2021;53:101267. - Dai Y, et al. Copy number gain of ZEB1 mediates a double-negative feedback loop with miR-33a-5p that regulates EMT and bone metastasis of prostate cancer dependent on TGF-
signaling. Theranostics. 2019;9(21):6063. - Li, D., et al., Heterogeneity and plasticity of epithelial-mesenchymal transition (EMT) in cancer metastasis: Focusing on partial EMT and regulatory mechanisms. 2023: p. e13423.
- Meidhof
, et al. ZEB 1 -associated drug resistance in cancer cells is reversed by the class I HDAC inhibitor mocetinostat. EMBO Mol Med. 2015;7(6):831-47. - Zhang
, et al. ZEB1 confers chemotherapeutic resistance to breast cancer by activating ATM. Cell Death Dis. 2018;9(2):1-15. - Xie SL, et al. SOX8 regulates cancer stem-like properties and cisplatininduced EMT in tongue squamous cell carcinoma by acting on the Wnt/
-catenin pathway. Int J Cancer. 2018;142(6):1252-65. - Liang Y, et al. Epigenetic activation of TWIST1 by MTDH promotes cancer stem-like cell traits in breast cancer. Cancer Res. 2015;75(17):3672-80.
- Mukherjee P, et al. Modulation of SOX2 expression delineates an endpoint for paclitaxel-effectiveness in breast cancer stem cells. Sci Rep. 2017;7(1):1-16.
- Li Q, et al. Subcellular drug distribution: mechanisms and roles in drug efficacy, toxicity, resistance, and targeted delivery. Drug Metab Rev. 2018;50(4):430-47.
- Sakhrani NM, Padh H. Organelle targeting: third level of drug targeting. Drug Des Devel Ther. 2013;7:585.
- Boedtkjer E, Pedersen SF. The acidic tumor microenvironment as a driver of cancer. Annu Rev Physiol. 2020;82:103-26.
- Audero MM, Prevarskaya N, Pla A. F, Ca2+ Signalling and Hypoxia/Acidic Tumour Microenvironment Interplay in Tumour Progression. Int J Mol Sci. 2022;23(13):7377.
- Goswami KK, et al. Lactic acid in alternative polarization and function of macrophages in tumor microenvironment. Hum Immunol. 2022;83(5):409-17.
- Kato Y, et al. Acidic extracellular microenvironment and cancer. Cancer Cell Int. 2013;13(1):1-8.
- Pilon-Thomas S, et al. Neutralization of tumor acidity improves antitumor responses to immunotherapy. Cancer Res. 2016;76(6):1381-90.
- Guo Y, et al. Mechanisms of chemotherapeutic resistance and the application of targeted nanoparticles for enhanced chemotherapy in colorectal cancer. J Nanobiotechnology. 2022;20(1):1-24.
- Dhar G, Sen S, Chaudhuri G. Acid Gradient Across Plasma Membrane can Drive Phosphate-Bond Synthesis in Cancer Cells: Acidic Tumor Milieu can Act as a Potential Energy Source. PLoS ONE. 2015;29:725.28.
- Dhar G, Sen S, Chaudhuri GJPO. Acid gradient across plasma membrane can drive phosphate bond synthesis in cancer cells: acidic tumor milieu as a potential energy source. PLoS ONE. 2015;10(4):e0124070.
- Halcrow PW, et al. Overcoming chemoresistance: Altering pH of cellular compartments by chloroquine and hydroxychloroquine. Front Cell Dev Biol. 2021;9:627639.
- Pérez-Tomás R, Pérez-Guillén IJC. Lactate in the tumor microenvironment: An essential molecule in cancer progression and treatment. Cancers (Basel). 2020;12(11):3244.
- Xiang Y-L, et al. Zwitterionic meso-silica/polypeptide hybrid nanoparticles for efficient azithromycin delivery and photodynamic therapy for synergistic treatment of drug-resistant bacterial infection. Int J Biol Macromol. 2022;219:597-610.
- Hulikova A. Swietach P, Nuclear proton dynamics and interactions with calcium signaling. J Mol Cell Cardiol. 2016;96:26-37.
- Ji X, et al. Chemoresistance mechanisms of breast cancer and their countermeasures. Biomed Pharmacother. 2019;114:108800.
- Yuan D, et al. Interruption of endolysosomal trafficking leads to stroke brain injury. Exp Neurol. 2021;345:113827.
- Polanco JC, et al. Exosomes induce endolysosomal permeabilization as a gateway by which exosomal tau seeds escape into the cytosol. Acta Neuropathol. 2021;141:235-56.
- McGuire C, et al. Regulation of V-ATPase activity. Front Biosci (Landmark Ed). 2017;22(4):609-22.
- Webb BA, et al. pHLARE: a new biosensor reveals decreased lysosome pH in cancer cells. Mol Biol Cell. 2021;32(2):131-42.
- Tang T, Yang ZY, Wang D, Yang XY, Wang J, Li L, Wen Q, Gao L, Bian XW, Yu SC. The role of lysosomes in cancer development and progression. Cell Biosci. 2020;10(1):131. https://doi.org/10.1186/s13578-020-00489-x.
- Zhitomirsky B, Assaraf RK. Lysosomes as mediators of drug resistance in cancer. Drug Resist Updat. 2016;24:23-33.
- Geisslinger F, et al. Targeting lysosomes in cancer as promising strategy to overcome chemoresistance-a mini review. Front Oncol. 2020;10:1156.
- Hraběta J, et al. Drug sequestration in lysosomes as one of the mechanisms of chemoresistance of cancer cells and the possibilities of its inhibition. Int J Mol Sci. 2020;21(12):4392.
- Al-Akra L, et al. Tumor stressors induce two mechanisms of intracellular P-glycoprotein-mediated resistance that are overcome by lysosomaltargeted thiosemicarbazones. J Biol Chem. 2018;293(10):3562-87.
- Fu Z, et al. CQ sensitizes human pancreatic cancer cells to gemcitabine through the lysosomal apoptotic pathway via reactive oxygen species. Mol Oncol. 2018;12(4):529-44.
- Sharapova TN, et al. FasL on the surface of Tag7 (PGRP-S)-activated lymphocytes induces necroptosis in HLA-negative tumor cells with the involvement of lysosomes and mitochondria. Biochimie. 2018;152:174-80.
- Seebacher NA, et al. A mechanism for overcoming P-glycoproteinmediated drug resistance: novel combination therapy that releases stored doxorubicin from lysosomes via lysosomal permeabilization using Dp44mT or DpC. Cell Death Dis. 2016;7(12):e2510-e2510.
- Salaroglio IC, et al. Increasing intratumor C/EBP-
LIP and nitric oxide levels overcome resistance to doxorubicin in triple negative breast cancer. J Exp Clin Cancer Res. 2018;37(1):1-20. - Champagne DP, et al. Fine-tuning of CD8+ T cell mitochondrial metabolism by the respiratory chain repressor MCJ dictates protection to influenza virus. Immunity. 2016;44(6):1299-311.
- Barbier-Torres
, et al. The mitochondrial negative regulator MCJ is a therapeutic target for acetaminophen-induced liver injury. Nat Commun. 2017;8(1):1-11. - Hatle KM, et al. MCJ/DnaJC15, an endogenous mitochondrial repressor of the respiratory chain that controls metabolic alterations. Mol Cell Biol. 2013;33(11):2302-14.
- Giddings EL, et al. Mitochondrial ATP fuels ABC transporter-mediated drug efflux in cancer chemoresistance. Nat Commun. 2021;12(1):2804.
- Miglietta S. Deciphering the role of the mitochondrial chaperonine MCJ in ovarian cancer. 2021.
- Shubin AV, et al. Cytoplasmic vacuolization in cell death and survival. Oncotarget. 2016;7(34):55863.
- Lin H, et al. Overcoming Taxol-resistance in A549 cells: A comprehensive strategy of targeting P-gp transporter, AKT/ERK pathways, and cytochrome P450 enzyme CYP1B1 by 4-hydroxyemodin. Biochem Pharmacol. 2020;171:113733.
- Belisario DC, et al. Hypoxia dictates metabolic rewiring of tumors: implications for chemoresistance. Cells. 2020;9(12):2598.
- Liang W, et al. Role of reactive oxygen species in tumors based on the ‘seed and soil’theory: A complex interaction. Oncol Rep. 2021;46(3):1-15.
- Pascual-Ahuir A, Manzanares-Estreder S, Proft M. Pro- and Antioxidant Functions of the Peroxisome-Mitochondria Connection and Its Impact on Aging and Disease. Oxid Med Cell Longev. 2017;2017:9860841. https://doi.org/10.1155/2017/9860841.
- Jacqueroux E , et al. Value of quantifying ABC transporters by mass spectrometry and impact on in vitro-to-in vivo prediction of transportermediated drug-drug interactions of rivaroxaban. Eur J Pharm Biopharm. 2020;148:27-37.
- Dang Y, et al. P-Gp and TOPO II Expression and Their Clinical Significance in Colon Cancer. Ann Clin Lab Sci. 2020;50(5):584-90.
- Heming CP, et al. P-glycoprotein and cancer: what do we currently know? 2022.
- Mirzaei S, Gholami MH, Hashemi F, Zabolian A, Farahani MV, Hushmandi K, Zarrabi A, Goldman A, Ashrafizadeh M, Orive G. Advances in understanding the role of P-gp in doxorubicin resistance: Molecular pathways, therapeutic strategies, and prospects. Drug Discov Today. 2022;27(2):436-55. https://doi.org/10.1016/j.drudis.2021.09.020.
- Khalaf K, et al. Aspects of the tumor microenvironment involved in immune resistance and drug resistance. Front Immunol. 2021;12:656364.
- Xu M, et al. Targeting the tumor stroma for cancer therapy. Mol Cancer. 2022;21(1):208.
- Winkler J, Abisoye-Ogunniyan A, Metcalf KJ, Werb Z. Concepts of extracellular matrix remodelling in tumour progression and metastasis. Nat Commun. 2020;11(1):5120. https://doi.org/10.1038/ s41467-020-18794-X.
- Hu J, et al. CAFs secreted exosomes promote metastasis and chemotherapy resistance by enhancing cell stemness and epithelial-mesenchymal transition in colorectal cancer. Mol Cance. 2019;18(1):1-15.
- Jena BC, et al. Cancer associated fibroblast mediated chemoresistance: A paradigm shift in understanding the mechanism of tumor progression. Biochim Biophys Acta Rev Cancer. 2020;1874(2):188416.
- Haider T, et al. Drug resistance in cancer: mechanisms and tackling strategies. Pharmacol Rep. 2020;72(5):1125-51.
- Guo Z, Ashrafizadeh M, Zhang W, Zou R, Sethi G, Zhang X. Molecular profile of metastasis, cell plasticity and EMT in pancreatic cancer: a pre-clinical connection to aggressiveness and drug resistance. Cancer Metastasis Rev. 2023. https://doi.org/10.1007/s10555-023-10125-y.
- Che Y, et al. Cisplatin-activated PAI-1 secretion in the cancer-associated fibroblasts with paracrine effects promoting esophageal squamous cell carcinoma progression and causing chemoresistance. Cell Death Dis. 2018;9(7):1-13.
- Luraghi P, et al. MET Signaling in Colon Cancer Stem-like Cells Blunts the Therapeutic Response to EGFR InhibitorsMET in Colon Cancer-Initiating Cells. Can Res. 2014;74(6):1857-69.
- Fiori ME, et al. Cancer-associated fibroblasts as abettors of tumor progression at the crossroads of EMT and therapy resistance. Mol Cancer. 2019;18(1):1-16.
- Hamidi H, Ivaska J. Every step of the way: integrins in cancer progression and metastasis. Nat Rev Cancer. 2018;18(9):533-48.
- Fang Z, Meng Q, Xu J, Wang W, Zhang B, Liu J, Liang C, Hua J, Zhao Y, Yu X, Shi S. Signaling pathways in cancer-associated fibroblasts: recent advances and future perspectives. Cancer Commun (Lond). 2023;43(1):3-41. https://doi.org/10.1002/cac2.12392.
- Martino MM, et al. Growth factors engineered for super-affinity to the extracellular matrix enhance tissue healing. Science. 2014;343(6173):885-8.
- Walker C, Mojares E, del Río Hernández A. Role of extracellular matrix in development and cancer progression. Int J Mol Sci. 2018;19(10):3028.
- Kaemmerer E, Loessner D, Avery VM. Addressing the tumour microenvironment in early drug discovery: a strategy to overcome drug resistance and identify novel targets for cancer therapy. Drug Discov Today. 2021;26(3):663-76.
- Teng PN, et al. Identification of candidate circulating cisplatin-resistant biomarkers from epithelial ovarian carcinoma cell secretomes. Br J Cancer. 2014;110(1):123-32.
- Wu Y-H, et al. COL11A1 confers chemoresistance on ovarian cancer cells through the activation of Akt/c/EBP
pathway and PDK1 stabilization. Oncotarget. 2015;6(27):23748. - Cooper J, Giancotti FG. Integrin Signaling in Cancer: Mechanotransduction, Stemness, Epithelial Plasticity, and Therapeutic Resistance. Cancer Cell. 2019;35(3):347-67. https://doi.org/10.1016/j.ccell.2019.01.007.
- Murphy JM, Rodriguez YAR, Jeong K, Ahn EE, Lim SS. Targeting focal adhesion kinase in cancer cells and the tumor microenvironment. Exp Mol Med. 2020;52(6):877-86. https://doi.org/10.1038/ s12276-020-0447-4.
- Senthebane DA, et al. The role of tumor microenvironment in chemoresistance: 3D extracellular matrices as accomplices. Int J Mol Sci. 2018;19(10):2861.
- Hong Y, Rao Y. Current status of nanoscale drug delivery systems for colorectal cancer liver metastasis. Biomed Pharmacother. 2019;114:108764. https://doi.org/10.1016/j.biopha.2019.108764.
- Mashouri L, Yousefi H, Aref AR, Ahadi AM, Molaei F, Alahari SK. Exosomes: Composition, Biogenesis, and Mechanisms in Cancer Metastasis and Drug Resistance. Mol Cancer. 2019;18(1):75.
- Yang E, et al. Exosome-mediated metabolic reprogramming: The emerging role in tumor microenvironment remodeling and its influence on cancer progression. Signal Transduct Target Ther. 2020;5(1):1-13.
- Dianat-Moghadam H , et al. The role of circulating tumor cells in the metastatic cascade: biology, technical challenges, and clinical relevance. Cancers. 2020;12(4):867.
- Li S, Yi M, Dong B, Jiao Y, Luo S, Wu K. The roles of exosomes in cancer drug resistance and its therapeutic application. Clin Transl Med. 2020;10(8):e257. https://doi.org/10.1002/ctm2.257.
- Steinbichler TB, Dudás J, Skvortsov S, Ganswindt U, Riechelmann H, Skvortsova II. Therapy resistance mediated by exosomes. Mol Cancer. 2019;18(1):58. https://doi.org/10.1186/s12943-019-0970-X.
- Ji R, et al. Exosomes derived from human mesenchymal stem cells confer drug resistance in gastric cancer. Cell Cycle. 2015;14(15):2473-83.
- Lukanović D, et al. The contribution of copper efflux transporters ATP7A and ATP7B to chemoresistance and personalized medicine in ovarian cancer. Biomed Pharmacother. 2020;129:110401.
- Zaidi
, et al. Quantitative visualization of hypoxia and proliferation gradients within histological tissue sections. Front Bioeng Biotechnol. 2019;7:397. - Singh D, et al. Overexpression of hypoxia-inducible factor and metabolic pathways: possible targets of cancer. Cell Biosci. 2017;7(1):1-9.
- Warfel NA, El-Deiry WS. HIF-1 signaling in drug resistance to chemotherapy. Curr Med Chem. 2014;21(26):3021-8.
- Xia Y, Jiang L, Zhong T. The role of HIF-1 a in chemo-/radioresistant tumors. Onco Targets Ther. 2018;11:3003.
- Nishimoto A, et al. HIF-1a activation under glucose deprivation plays a central role in the acquisition of anti-apoptosis in human colon cancer cells. Int J Oncol. 2014;44(6):2077-84.
- Zhao Q, et al. Enhancement of drug sensitivity by knockdown of HIF-1a in gastric carcinoma cells. Oncol Res. 2016;23(3):129.
- Chen W-L, et al. Cycling hypoxia induces chemoresistance through the activation of reactive oxygen species-mediated B-cell lymphoma extra-long pathway in glioblastoma multiforme. J Transl Med. 2015;13(1):1-13.
- Gao J, Liang Y, Wang L. Shaping polarization of tumor-associated macrophages in cancer immunotherapy. Front Immunol. 2022;13:888713.
- Lei X, et al. Immune cells within the tumor microenvironment: Biological functions and roles in cancer immunotherapy. Cancer Lett. 2020:470:126-33.
- Larionova I, et al. Interaction of tumor-associated macrophages and cancer chemotherapy. Oncoimmunology. 2019;8(7):e1596004.
- Liu Y, Cao X. The origin and function of tumor-associated macrophages. Cell Mol Immunol. 2015;12(1):1-4.
- Moradi-Chaleshtori M , et al. In vitro and in vivo evaluation of antitumoral effect of M1 phenotype induction in macrophages by miR-130 and miR-33 containing exosomes. Cancer Immunol Immunother. 2021;70(5):1323-39.
- Yu S, et al. Activated HIF1a of tumor cells promotes chemoresistance development via recruiting GDF15-producing tumor-associated macrophages in gastric cancer. Cancer Immunol Immunother. 2020;69(10):1973-87.
- Incio J, et al. Obesity-Induced Inflammation and Desmoplasia Promote Pancreatic Cancer Progression and Resistance to Chemotherapy. Cancer Discov. 2016;6(8):852-69.
- Zhang Y, et al. Targeting and exploitation of tumor-associated neutrophils to enhance immunotherapy and drug delivery for cancer treatment. Cancer Biol Med. 2020;17(1):32.
- Pylaeva E, Lang S, Jablonska J. The essential role of type I interferons in differentiation and activation of tumor-associated neutrophils. Front Immunol. 2016;7:629.
- Liu X-D, et al. Resistance to Antiangiogenic Therapy Is Associated with an Immunosuppressive Tumor Microenvironment in Metastatic Renal Cell CarcinomaAntiangiogenic Therapy Increases PD-L1 Expression. Cancer Immunol Res. 2015;3(9):1017-29.
- Wang D, et al. Colorectal cancer cell-derived CCL20 recruits regulatory T cells to promote chemoresistance via FOXO1/CEBPB/NF-kB signaling. J Immunother Cancer. 2019;7(1):215.
- Long Y, et al. Dysregulation of Glutamate Transport Enhances Treg Function That Promotes VEGF Blockade Resistance in Glioblastoma. Cancer Res. 2020;80(3):499-509.
- Desbats MA, et al. Metabolic plasticity in chemotherapy resistance. Front Oncol. 2020;10:281.
- Ragni M, et al. Amino acids in cancer and cachexia: An integrated view. Cancers (Basel). 2022;14(22):5691.
- Phan LM, et al. Cancer metabolic reprogramming: importance, main features, and potentials for precise targeted anti-cancer therapies. Cancer Biol Med. 2014;11(1):1.
- Cao J, et al. Effects of altered glycolysis levels on CD8+ T cell activation and function. Cell Death Dis. 2023;14(7):407.
- Khodaei T, et al. Drug delivery for metabolism targeted cancer immunotherapy. Adv Drug Deliv Rev. 2022;184:114242.
- Baek G, et al. MCT4 defines a glycolytic subtype of pancreatic cancer with poor prognosis and unique metabolic dependencies. Cell Rep. 2014;9(6):2233-49.
- Ruprecht B, et al. Lapatinib resistance in breast cancer cells is accompanied by phosphorylation-mediated reprogramming of glycolysis. Cancer Res. 2017;77(8):1842-53.
- Zhang Y, et al. Targeting Glucose Metabolism Enzymes in Cancer Treatment: Current and Emerging Strategies. Cancers (Basel). 2022;14(19):4568.
- Zhao H, et al. Up-regulation of glycolysis promotes the stemness and EMT phenotypes in gemcitabine-resistant pancreatic cancer cells. J Cell Mol Med. 2017;21(9):2055-67.
- Varghese E, et al. Targeting glucose metabolism to overcome resistance to anticancer chemotherapy in breast cancer. Cancers (Basel). 2020;12(8):2252.
- Krasnov GS, et al. Targeting VDAC-bound hexokinase II: a promising approach for concomitant anti-cancer therapy. Expert Opin Ther Targets. 2013;17(10):1221-33.
- Boison D. Yegutkin G G, Adenosine metabolism: emerging concepts for cancer therapy. Cancer Cell. 2019;36(6):582-96.
- Allard B, et al. The adenosine pathway in immuno-oncology. Nat Rev Clin Oncol. 2020;17(10):611-29.
- Sade-Feldman M, et al. Defining T cell states associated with response to checkpoint immunotherapy in melanoma. Cell. 2019;176(1):404.
- Song Y, et al. Drug-metabolizing cytochrome P450 enzymes have multifarious influences on treatment outcomes. Clin Pharmacokinet. 2021;60:585-601.
- Parashar A, Manoj K. Murburn precepts for cytochrome P450 mediated drug/xenobiotic metabolism and homeostasis. Curr Drug Metab. 2021;22(4):315-26.
- Vredenburg G, den Braver-Sewradj S, van Vugt-Lussenburg BM, Vermeulen NP, Commandeur JN, Vos JC. Activation of the anticancer drugs cyclophosphamide and ifosfamide by cytochrome P450 BM3 mutants. Toxicol Lett. 2015;232(1):182-92. https://doi.org/10.1016/j.toxlet.2014. 11.005.
- Voelcker, G. Enzyme catalyzed decomposition of 4-hydroxycyclophosphamide. in The Open Conference Proceedings Journal. 2017.
- Yang W, et al. Cyclophosphamide Exposure Causes Long-Term Detrimental Effect of Oocytes Developmental Competence Through Affecting the Epigenetic Modification and Maternal Factors’Transcription During Oocyte Growth. Front Cell Dev Biol. 2021;9:682060.
- Bagdasaryan AA, et al. Pharmacogenetics of drug metabolism: the role of gene polymorphism in the regulation of doxorubicin safety and efficacy. Cancers (Basel). 2022;14(21):5436.
- Zahreddine H, Borden KLB. Mechanisms and insights into drug resistance in cancer. Front Pharmacol. 2013,4:28.
- Arwanih, E.Y., et al., Resistance mechanism of acute myeloid leukemia cells against daunorubicin and cytarabine: a literature review. 2022. 14(12).
- Levin M, et al. Surmounting Cytarabine-resistance in acute myeloblastic leukemia cells and specimens with a synergistic combination of hydroxyurea and azidothymidine. Cell Death Dis. 2019;10(6):390.
- Costea T, et al. Alleviation of multidrug resistance by flavonoid and nonflavonoid compounds in breast, lung, colorectal and prostate cancer. Int J Mol Sci. 2020;21(2):401.
- Varela-López A, et al. An update on the mechanisms related to cell death and toxicity of doxorubicin and the protective role of nutrients. Food Chem Toxicol. 2019;134:110834.
- Guo J, et al. Reprogramming of glutamine metabolism via glutamine synthetase silencing induces cisplatin resistance in A2780 ovarian cancer cells. BMC Cancer. 2021;21(1):1-11.
- Wang SF, Chang YL, Fang WL, Li AF, Chen CF, Yeh TS, Hung GY, Huang KH, Lee HC. Growth differentiation factor 15 induces cisplatin resistance through upregulation of xCT expression and glutathione synthesis in gastric cancer. Cancer Sci. 2023;114(8):3301-17. https://doi.org/10. 1111/cas. 15869.
- Housman G, et al. Drug resistance in cancer: an overview. Cancers (Basel). 2014;6(3):1769-92.
- To C, et al. An allosteric inhibitor against the therapy-resistant mutant forms of EGFR in non-small cell lung cancer. Nat Cancer. 2022;3(4):402-17.
- Wu PS, Lin MH, Hsiao JC, Lin PY, Pan SH, Chen YJ. EGFR-T790M MutationDerived Interactome Rerouted EGFR Translocation Contributing to Gefitinib Resistance in Non-Small Cell Lung Cancer. Mol Cell Proteomics. 2023;22(9):100624. https://doi.org/10.1016/j.mcpro.2023.100624.
- Chong CR, Jänne PA. The quest to overcome resistance to EGFR-targeted therapies in cancer. Nat Med. 2013;19(11):1389-400.
- Schulte RR, Ho RH. Organic anion transporting polypeptides: emerging roles in cancer pharmacology. Mol Pharmacol. 2019;95(5):490-506.
- Brecht K, Schäfer AM, Meyer zu Schwabedissen HE. Uptake Transporters of the SLC21, SLC22A, and SLC15A Families in Anticancer TherapyModulators of Cellular Entry or Pharmacokinetics? Cancers (Basel). 2020;12(8):2263.
- Klein I, et al. Glycyrrhizic Acid Prevents Paclitaxel-Induced Neuropathy via Inhibition of OATP-Mediated Neuronal Uptake. Cells. 2023;12(9):1249.
- Zhao D, Long X, Wang J. Transporter-mediated drug-drug interactions involving poly (ADP-ribose) polymerase inhibitors. Oncol Lett. 2023;25(4):1-8.
- Zhou S, Zeng S, Shu Y. Drug-drug interactions at organic cation transporter 1. Front Pharmacol. 2021;12:628705.
- Lin L, Yee SW, Kim RB, Giacomini KM. SLC transporters as therapeutic targets: emerging opportunities. Nat Rev Drug Discov. 2015;14(8):54360. https://doi.org/10.1038/nrd4626.
- Siemer
, et al. Profiling cisplatin resistance in head and neck cancer: A critical role of the VRAC ion channel for chemoresistance. Cancers (Basel). 2021;13(19):4831. - Cabral LKD, Tiribelli C, Sukowati CHJC. Sorafenib resistance in hepatocellular carcinoma: the relevance of genetic heterogeneity. Cancers (Basel). 2020;12(6):1576.
- Otter M , et al. Expression and functional contribution of different organic cation transporters to the cellular uptake of doxorubicin into human breast cancer and cardiac tissue. Int J Mol Sci. 2021;23(1):255.
- Edemir B. Identification of Prognostic Organic Cation and Anion Transporters in Different Cancer Entities by In Silico Analysis. Int J Mol Sci. 2020;21(12):4491. https://doi.org/10.3390/ijms21124491.
- Marin JJ, et al. The role of reduced intracellular concentrations of active drugs in the lack of response to anticancer chemotherapy. Acta Pharmacol Sin. 2014;35(1):1-10.
- Ortiz M, et al. Mechanisms of chemotherapy resistance in ovarian cancer. Cancer Drug Resist. 2022;5(2):304.
- Arnesano F, Natile G. Interference between copper transport systems and platinum drugs. Semin Cancer Biol. 2021;76:173-88. https://doi. org/10.1016/j.semcancer.2021.05.023.
- De Luca A, et al. Copper homeostasis as target of both consolidated and innovative strategies of anti-tumor therapy. J Trace Elem Med Biol. 2019;55:204-13.
- Shiqun, Shao Jingxing, Si Youqing, Shen. Copper as the Target for Anticancer Nanomedicine. Abstr Adv Ther. 2019;2(5):10.1002/adtp.v2.5. https://doi.org/10.1002/adtp. 201800147.
- Mohammad IS, et al. Understanding of human ATP binding cassette superfamily and novel multidrug resistance modulators to overcome MDR. Biomed Pharmacother. 2018;100:335-48.
- Wu Z-X, et al. Elevated ABCB1 expression confers acquired resistance to aurora kinase inhibitor GSK-1070916 in cancer cells. Front Pharmacol. 2021;11:615824.
- Martins-Gomes C, Silva AM. Natural Products as a Tool to Modulate the Activity and Expression of Multidrug Resistance Proteins of Intestinal Barrier. J Xenobiot. 2023;13(2):172-92.
- Barnett-Griness O, Rennert G, Lejbkowicz F, Pinchev M, Saliba W, Gronich N. Association Between ABCG2, ABCB1, ABCC2 Efflux Transporter Single-Nucleotide Variants and Irinotecan Adverse Effects in Patients With Colorectal Cancer: A Real-Life Study. Clin Pharmacol Ther. 2023;113(3):704-11. https://doi.org/10.1002/cpt.2833.
- Petruzzelli R, Polishchuk RSJC. Activity and trafficking of copper-transporting ATPases in tumor development and defense against platinumbased drugs. Cells. 2019;8(9):1080.
- Zhang B, Burke R. Copper homeostasis and the ubiquitin proteasome system. Metallomics. 2023;15(3):mfad010.
- Petruzzelli R, Polishchuk RS. Activity and Trafficking of Copper-Transporting ATPases in Tumor Development and Defense against PlatinumBased Drugs. Cells. 2019;8(9):1080. https://doi.org/10.3390/cells80910 80.
- Zhang Z, et al. Reversing drug resistance of cisplatin by hsp90 inhibitors in human ovarian cancer cells. Int J Clin Exp Med. 2015;8(5):6687.
- Przystupski, D., et al., The modulatory effect of green tea catechin on drug resistance in human ovarian cancer cells. 2019. 28(5): 657-667.
- Hejmady S, Pradhan R, Alexander A, Agrawal M, Singhvi G, Gorain B, Tiwari S, Kesharwani P, Dubey SK. Recent advances in targeted nanomedicine as promising antitumor therapeutics. Drug Discov Today. 2020;25(12):2227-44. https://doi.org/10.1016/j.drudis.2020.09.031.
- Li X, He S, Ma B. Autophagy and autophagy-related proteins in cancer. Mol Cancer. 2020;19(1):12. https://doi.org/10.1186/s12943-020-1138-4.
- Man SM, Jenkins BJ. Context-dependent functions of pattern recognition receptors in cancer. Nat Rev Cancer. 2022;22(7):397-413.
- Manzoor S, et al. Autophagy: A versatile player in the progression of colorectal cancer and drug resistance. Nat Rev Cancer. 2022;12:924290.
- Marsh T, Debnath JJA. Autophagy suppresses breast cancer metastasis by degrading NBR1. Autophagy. 2020;16(6):1164-5.
- Khan SU, Fatima K, Singh U, Singh PP, Malik F. Small molecule “4ab” induced autophagy and endoplasmic reticulum stress-mediated death of aggressive cancer cells grown under adherent and floating conditions. Med Oncol. 2023;40(4):121. https://doi.org/10.1007/ s12032-023-01963-5.
- Assi M, Kimmelman AC. Impact of context-dependent autophagy states on tumor progression. Nat Cancer. 2023;4(5):596-607. https://doi. org/10.1038/s43018-023-00546-7.
- Dower CM, Wills CA, Frisch SM, Wang HG. Mechanisms and context underlying the role of autophagy in cancer metastasis. Autophagy. 2018;14(7):1110-28. https://doi.org/10.1080/15548627.2018.1450020.
- White E. Autophagy and p53. Cold Spring Harb Perspect Med. 2016;6(4):a026120.
- Levy JMM, Towers CG, Thorburn A. Targeting autophagy in cancer. Nat Rev Cancer. 2017;17(9):528-42.
- Rahman MA, et al. Recent Update and Drug Target in Molecular and Pharmacological Insights into Autophagy Modulation in Cancer Treatment and Future Progress. Cells. 2023;12(3):458.
- Anand SK, et al. Entrenching role of cell cycle checkpoints and autophagy for maintenance of genomic integrity. DNA Repair (Amst). 2020;86:102748.
- Ueno S, Sudo T. Hirasawa A, ATM: Functions of ATM kinase and its relevance to hereditary tumors. Int J Mol Sci. 2022;23(1):523.
- Zhang L-H, et al. Enhanced autophagy reveals vulnerability of P-gp mediated epirubicin resistance in triple negative breast cancer cells. Apoptosis. 2016;21(4):473-88.
- Liu ZR, Song Y, Wan LH, Zhang YY, Zhou LM. Over-expression of miR451a can enhance the sensitivity of breast cancer cells to tamoxifen by regulating
, estrogen receptor a, and autophagy. Life Sci. 2016;149:104-13. https://doi.org/10.1016/j.lfs.2016.02.059. - Lee MH, Koh D, Na H, Ka NL, Kim S, Kim HJ, Hong S, Shin YK, Seong JK, Lee MO. MTA1 is a novel regulator of autophagy that induces tamoxifen resistance in breast cancer cells. Autophagy. 2018;14(5):812-24. https:// doi.org/10.1080/15548627.2017.1388476.
- Liu W, et al. Targeting the WEE1 kinase strengthens the antitumor activity of imatinib via promoting KIT autophagic degradation in gastrointestinal stromal tumors. Gastric Cancer. 2020;23:39-51.
- Liu T, et al. Combination of an autophagy inducer and an autophagy inhibitor: a smarter strategy emerging in cancer therapy. Front Pharmacol. 2020;11:408.
- Avril T, Vauléon E, Chevet E. Endoplasmic reticulum stress signaling and chemotherapy resistance in solid cancers. Oncogenesis. 2017;6(8):e373. https://doi.org/10.1038/oncsis.2017.72.
- Pavlović N, Calitz C, Thanapirom K, Mazza G, Rombouts K, Gerwins P, Heindryckx F. Inhibiting IRE1a-endonuclease activity decreases tumor burden in a mouse model for hepatocellular carcinoma. Elife. 2020;9:e55865. https://doi.org/10.7554/eLife.55865.
- Khaled J, Kopsida M, Lennernäs H, Heindryckx F. Drug Resistance and Endoplasmic Reticulum Stress in Hepatocellular Carcinoma. Cells. 2022;11(4):632. https://doi.org/10.3390/cells11040632.
- Hetz C, Zhang K, Kaufman RJ. Mechanisms, regulation and functions of the unfolded protein response. Nat Rev Mol Cell Biol. 2020;21:421-38. https://doi.org/10.1038/s41580-020-0250-z.
- Khaled J, et al. Drug resistance and endoplasmic reticulum stress in hepatocellular carcinoma. Cells. 2022;11(4):632.
- Wang H, Mi K. Emerging roles of endoplasmic reticulum stress in the cellular plasticity of cancer cells. Front Oncol. 2023;13:1110881.
- Zhang C, et al. HMGB1-induced aberrant autophagy contributes to insulin resistance in granulosa cells in PCOS. FASEB J. 2020;34(7):9563-74.
- Li J, Zhou W, Mao Q, Gao D, Xiong L, Hu X, Zheng Y, Xu X. HMGB1 Promotes Resistance to Doxorubicin in Human Hepatocellular Carcinoma Cells by Inducing Autophagy via the AMPK/mTOR Signaling Pathway. Front Oncol. 2021;11:739145. https://doi.org/10.3389/fonc.2021.739145.
- Yu G, Klionsky DJ. Life and Death Decisions-The Many Faces of Autophagy in Cell Survival and Cell Death. Biomolecules. 2022;12(7):866.
- Gupta R, et al. Autophagy and apoptosis cascade: which is more prominent in neuronal death? Cell Mol Life Sci. 2021;78(24):8001-47.
- Booth LA, Roberts JL, Dent P. The role of cell signaling in the crosstalk between autophagy and apoptosis in the regulation of tumor cell survival in response to sorafenib and neratinib. Semin Cancer Biol. 2020;66:129-39. https://doi.org/10.1016/j.semcancer.2019.10.013.
- Anjum J, Mitra S, Das R, Alam R, Mojumder A, Emran TB, Islam F, Rauf A, Hossain MJ, Aljohani ASM, Abdulmonem WA, Alsharif KF, Alzahrani KJ, Khan H. A renewed concept on the MAPK signaling pathway in
cancers: Polyphenols as a choice of therapeutics. Pharmacol Res. 2022;184:106398. https://doi.org/10.1016/j.phrs.2022.106398. - Zhu S, Cao L, Yu Y, Yang L, Yang M, Liu K, Huang J, Kang R, Livesey KM, Tang D. Inhibiting autophagy potentiates the anticancer activity of IFN1@/IFNa in chronic myeloid leukemia cells. Autophagy. 2013;9(3):317-27. https://doi.org/10.4161/auto.22923.
- Balvers RK, et al. ABT-888 enhances cytotoxic effects of temozolomide independent of MGMT status in serum free cultured glioma cells. J Transl Med. 2015;13(1):1-10.
- Christowitz C, et al. Mechanisms of doxorubicin-induced drug resistance and drug resistant tumour growth in a murine breast tumour model. BMC Cancer. 2019;19(1):757.
- Mei Y, et al. Overexpression of RSK4 reverses doxorubicin resistance in human breast cancer cells via PI3K/AKT signalling pathway. J Biochem. 2020;167(6):603-11.
- AI-Malky HS, AI Harthi SE, Osman A-MM. Major obstacles to doxorubicin therapy: Cardiotoxicity and drug resistance. J Oncol Pharm Pract. 2020;26(2):434-44.
- Ramani S, Park S. HSP27 role in cardioprotection by modulating chemotherapeutic doxorubicin-induced cell death. J Mol Med (Berl). 2021;99(6):771-84.
- Panneerpandian P, Rao DB, Ganesan K. Calcium channel blockers lercanidipine and amlodipine inhibit YY1/ERK/TGF-
mediated transcription and sensitize the gastric cancer cells to doxorubicin. Toxicol In Vitro. 2021;74:105152. - Kang
, et al. DUSP4 promotes doxorubicin resistance in gastric cancer through epithelial-mesenchymal transition. Oncotarget. 2017;8(55):94028. - Lee H-J, Choi C-H. Characterization of SN38-resistant T47D breast cancer cell sublines overexpressing BCRP, MRP1, MRP2, MRP3, and MRP4. BMC Cancer. 2022;22(1):446.
- Cho CJ, et al. The modulation study of multiple drug resistance in bladder cancer by curcumin and resveratrol. Oncol Lett. 2019;18(6):6869-76.
- Gupta SK, Singh P, Ali V, Verma M. Role of membrane-embedded drug efflux
transporters in the cancer chemotherapy. Oncol Rev. 2020;14(2):448. https://doi.org/10.4081/oncol.2020. - Monteran L, et al. Chemotherapy-induced complement signaling modulates immunosuppression and metastatic relapse in breast cancer. Nat Commun. 2022;13(1):1-19.
- Makovec T. Cisplatin and beyond: molecular mechanisms of action and drug resistance development in cancer chemotherapy. Radiol Oncol. 2019;53(2):148-58.
- Bulathge AW, et al. Comparative cisplatin reactivity towards human Zn7-metallothionein-2 and MTF-1 zinc fingers: potential implications in anticancer drug resistance. Metallomics. 2022;14(9):mfac061.
- Lee IT-L, et al. Cancer-Derived Extracellular Vesicles as Biomarkers for Cutaneous Squamous Cell Carcinoma: A Systematic Review. Cancers (Basel). 2022;14(20):5098.
- Horibe S, et al. Mitochondrial DNA mutations are involved in the acquisition of cisplatin resistance in human lung cancer A549 cells. Oncol Rep. 2022;47(2):1-12.
- Lilienthal I, Herold N. Targeting Molecular Mechanisms Underlying Treatment Efficacy and Resistance in Osteosarcoma: A Review of Current and Future Strategies. Int J Mol Sci. 2020;21(18):6885. https://doi. org/10.3390/ijms21186885.
- Barbuti AM, Chen Z-S. Paclitaxel through the ages of anticancer therapy: exploring its role in chemoresistance and radiation therapy. Cancers. 2015;7(4):2360-71.
- Germain N, et al. Lipid metabolism and resistance to anticancer treatment. Biology (Basel). 2020;9(12):474.
- Zhang J, Yin D, Li H. hMSH2 expression is associated with paclitaxel resistance in ovarian carcinoma, and inhibition of hMSH2 expression in vitro restores paclitaxel sensitivity. Oncol Rep. 2014;32(5):2199-206.
- Mir MA, Khan SU, Aisha S. Cell Cycle Dysregulation in Breast Cancer. In: Therapeutic potential of Cell Cycle Kinases in Breast Cancer. Springer; 2023. p. 103-31.
- Lopes D, et al. a-Tubulin detyrosination links the suppression of MCAK activity with taxol cytotoxicity. J Cell Biol. 2022;222(2):e202205092.
- Comparetti, E.J., et al., Cancer cell membrane-derived nanoparticles improve the activity of gemcitabine and paclitaxel on pancreatic
cancer cells and coordinate immunoregulatory properties on professional antigen-presenting cells. 2020. 1(6): p. 1775-1787. - Mohiuddin M, Kasahara K. Paclitaxel impedes EGFR-mutated PC9 cell growth via reactive oxygen species-mediated DNA damage and EGFR/ PI3K/AKT/mTOR signaling pathway suppression. Cancer Genomics Proteomics. 2021;18(5):645-59.
- Indini A, et al. NAD/NAMPT and mTOR pathways in melanoma: Drivers of drug resistance and prospective therapeutic targets. Int J Mol Sci. 2022;23(17):9985.
- Dogan E, et al. Cancer stem cells in tumor modeling: Challenges and future directions. Advanced Nanobiomed Res. 2021;1(11):2100017.
- Vasan N, Baselga J, Hyman DM. A view on drug resistance in cancer. Nature. 2019;575(7782):299-309.
- Oshimori N, Guo Y, Taniguchi S. An emerging role for cellular crosstalk in the cancer stem cell niche. J Pathol. 2021;254(4):384-94.
- Li Y, et al. Drug resistance and Cancer stem cells. Cell Commun Signal. 2021;19(1):19.
- Batlle E, Clevers H. Cancer stem cells revisited. Nat Med. 2017;23(10):1124-34.
- Mirzaei S, Gholami MH, Hashemi F, Zabolian A, Farahani MV, Hushmandi K, Zarrabi A, Goldman A, Ashrafizadeh M, Orive G. Advances in understanding the role of P-gp in doxorubicin resistance: Molecular pathways, therapeutic strategies, and prospects. Drug Discov Today. 2022;27(2):436-55. https://doi.org/10.1016/j.drudis.2021.09.020.
ملاحظة الناشر
ساهم سمير الله خان وكانيز فاطمة بالتساوي.
*المراسلة:
سمير الله خان
muminsameer@gmail.com
فايز مالك
fmalik@iiim.res.in
¹ قسم الأورام الطبية المسالك البولية، جامعة تكساس MD أندرسون لمركز السرطان، هولكومب بوليفارد، هيوستن، تكساس 77030، الولايات المتحدة الأمريكية
قسم علم الأدوية السرطانية، CSIR-المعهد الهندي للطب التكاملي، سريناغار-190005، جامو وكشمير، الهند
أكاديمية البحث العلمي والابتكار (AcSIR)، غازي آباد-201002، الهند
DOI: https://doi.org/10.1186/s12964-023-01302-1
PMID: https://pubmed.ncbi.nlm.nih.gov/38347575
Publication Date: 2024-02-12
Unveiling the mechanisms and challenges of cancer drug resistance
Abstract
Cancer treatment faces many hurdles and resistance is one among them. Anti-cancer treatment strategies are evolving due to innate and acquired resistance capacity, governed by genetic, epigenetic, proteomic, metabolic, or microenvironmental cues that ultimately enable selected cancer cells to survive and progress under unfavorable conditions. Although the mechanism of drug resistance is being widely studied to generate new target-based drugs with better potency than existing ones. However, due to the broader flexibility in acquired drug resistance, advanced therapeutic options with better efficacy need to be explored. Combination therapy is an alternative with a better success rate though the risk of amplified side effects is commonplace. Moreover, recent groundbreaking precision immune therapy is one of the ways to overcome drug resistance and has revolutionized anticancer therapy to a greater extent with the only limitation of being individual-specific and needs further attention. This review will focus on the challenges and strategies opted by cancer cells to withstand the current therapies at the molecular level and also highlights the emerging therapeutic options -like immunological, and stem cell-based options that may prove to have better potential to challenge the existing problem of therapy resistance.
Introduction
acquired) and developing next-generation targeted therapies is crucial for medical need [13, 14].
A complex interplay between intrinsic (innate) and extrinsic (acquired) factors of the cancer cell contributes to cancer resistance towards various therapies. Intrinsic factors include pre-existing genetic mutations, tumor heterogeneity, and activation of intracellular defense pathways, that confer resistance by activating various oncogenic pathways, altering drug targets, desensitization towards therapies, enhancing DNA repair mechanisms, as well as activation of survival pathways, thereby potentiating cancer cells to evade the cytotoxic effects of treatments [15]. While extrinsic factors mainly include tumor microenvironment (TME) components that actively participate in cancer cell ability to evade the cytotoxic effects of various anticancer therapeutics[16, 17]. The various TME components include the altered extracellular matrix (ECM), tumor-associated stromal cells, growth factors, extracellular vesicles (EVs), immune cells, etc. Tumor-associated rigid and condensed ECM affects the drug response by reducing drug transport and sequestering drugs through direct binding with it thus represents a significant mechanism of drug resistance in many solid tumors [18, 19]. Cancer-associated fibroblasts (CAFs) are another TME component that plays significant roles in tumor growth, metastasis and cancer therapy resistance by secreting various growth factors like Hepatocyte growth factor (HGF) and Epidermal growth factor (EGF); cytokines such as stromal cell-derived factor 1 (SDF-1) and interleukin-6 (IL-6) [20, 21]. Extracellular vesicles (EVs) of drug-resistant cancer cells can sequester and transport drugs to ECM. Moreover, EVs may be transferred from drug-resistant cancer cell to drug-sensitive counterparts and thus plays a role in the horizontal transfer of drug resistance in cancer cells by delivering specialized cargo which includes drug resistance-related proteins (P-gp, ABCG2, ABCA3, etc.), nucleic acids (mt DNA, mRNAs, miRNAs), onco-metabolites, antiapoptotic proteins [22, 23]. Targeting drug-resistant cancer cell-intrinsic and tumor microenvironment components, alone or in combination with anticancer therapies may prove to be a better approach in enhancing the efficacy of cancer treatments and improving patient outcomes. The details mechanism of the extrinsic and intrinsic factors implicated in drug resistance and the strategies to inhibit them are discussed in the below sections.
recognition and destruction of cancer cells with more tolerability and better remission. Widely used AntiCTLA and anti-PD-1/PD-L1 therapy remarkably show antitumor activity by dysfunctioning the negative regulators of the anticancer adaptive immune system, though the minimum chance of resistance and its limitations to a certain subset of cancer remains the concern [32-36]. Cancer cells follow the Darwinian selection pressure rule to achieve drug-resistant traits at genomic, epigenomic and proteomic levels for the survival of their fittest [3739]. With the advent of high throughput assays, the link between tumor heterogeneity and drug resistance came into existence which suggests that under selective drug pressure, few tumor cells divide and form a subpopulation of cells that may achieve features that enable them to become non-responsive to a particular drug over time [40-43]. Contrasting features of cancer cells and drugresistant cells are represented in Fig. 1.
Tumor heterogeneity is a decisive factor for drug resistance

division and chromosomal instability lead to a random chromosomal loss in various tumor regions that result in mutational heterogeneity with the outcome of the rise of evolving competitive sub-clones and Cancer stem cells (CSCs) and vice versa [62-64]. These clones formed so far expand either sequentially or by a branched approach and generate more genetic diversity in either way which is further selected under evolutionary pressure with better growth, resistance, and survival advantage [65]. Recent high throughput assays exercises like single-cell RNA sequencing and mutation characterization enable us to investigate and identify the evolutionary dynamics that occur in the particular tumor cell population in the same or different patients and have a flagging role in individualized therapy [66-69]. These diverse genomic changes contribute to acquiring beneficial properties like drug resistance and tumor recurrence in these selected cancer cells [68]. Tumor evolution contributes to the emergence of a multi-drug resistant (MDR) subpopulation of cells with varying treatment responses to the drugs than that of the primary tumor cells [70]. Chemotherapeutic pressure on the other hand plays an important role in the formation of more evolved resistant sub-clones with poorer outcomes [49, 70]. Recent studies have shown that co-existing tumor heterogeneity and immune landscape
analysis in a cohort of patients with HBV-associated human hepatocellular carcinoma (HCC) inhibits the T-cell infiltration and thus regulates the intratumoral immune suppressive microenvironment [71]. The finding is crucial to design effectual immune therapies that can be given individually or in combination with existing chemotherapies to sensitize the resistant cancer cells with better outcomes. Thus tumor heterogeneity and varied immune landscape are big hurdles to understanding the resistance and more emphasis on this difficult issue can give a better future direction to the cancer therapy.
Genetic and epigenetic alteration as an adaptive response to chemotherapy that majorly influences the drug resistance
region of GSTp, MDR1,uPA , and O(6)-methylguanine DNA methyltransferase (MGMT) are significantly methylated but in drug-resistant MCF-7 cells, these promoters were hypo-methylated and had a significant role in resistance. MDR1 gene’s hypo-methylation was linked to drug efflux protein (P-glycoprotein; P-gp) overexpression which in turn is responsible for resistance to doxorubicin (DOX) [80-83].
| Drug Name | Experimental study | Type of Cancer | Clinical Phase | Reference |
| Sonidegib (LDE225) | LDE225 has the potential to disrupt CSC niches and overcome docetaxel resistance | TNBC | 1b | [112] |
| RO4929097 | CSC-mediated antiandrogen resistance, tamoxifen resistance, and radiation resistance are reversed | Recurrent Malignant Glioma | 1 | [113] |
| PF-03084014 | Counteracting docetaxel resistance in CSC | Desmoid Fibromatosis | 1 | [114] |
| PRI-724 | It could overcome cisplatin resistance in CSCs and reduce the expression of SOX2 and CD44 | Hepatitis C Virus-related Cirrhosis | 1 | [115] |
| Vismodegib (GDC-0449) | It has the potential to overcome radiation, carboplatin/erlotinib resistance as well as stemness | Multiple basal-cell carcinomas (MIKIE) | 2 | [116] |
CSCs play a key role in developing drug resistance and tumor relapse
Dysregulated developmental Cues that regulate CSCs contribution to chemoresistance
Cell survival and cellular functions under nutrient deprivation, hypoxia, or in drug resistance, depend on an evolutionary conserved physiological process known as autophagy [53, 132-134]. Interestingly cancer and CSCs exploit this catabolic process to support tumorigenesis, maintain pluripotency, tumor progression, and relapse [135, 136]. For example, CD44
non-stem cell counterpart, thus inhibiting autophagy by CRISPR/Cas9 ATG5 knockout making these CSCs chemosensitive [137]. Autophagy is known to reduce the chemotherapy-mediated oxidative stress in normal, cancer and CSCs thus protecting them from cell death [53, 138, 139]. Similarly, in cancer stem cells and normal stem cells, the marker enzyme aldehyde dehydrogenase (ALDH) oxidizes intracellular aldehydes and shields them from harmful consequences of reactive oxygen species (ROS) [140]. A surprising study has shown that the ALDH isoform (ALDH1A3) is responsible for lower doses of Temozolomide resistance in glioblastoma. Higher doses of Temozolomide were shown to induce direct physical interaction of ALDH1A3 with autophagy adaptor protein p62 thus leading to their degradation and reducing the resistance [141]. Yeo et al. (2016) demonstrated the tumorigenic dependence and stemness of
CSCs control the host immune system and mediate drug resistance
CSCs-activated TAMs also inhibit T-cell cytotoxicity by overexpressing cancer immune checkpoint receptors such as programmed death ligand protein1 (PD-L1), and (CD80/CD86) that interact with programmed cell death protein-1 (PD-1) and cytotoxic T-lymphocyteassociated protein-4 (CTLA-4) on the surface of
carcinoma, the hypoxia-mediated CSCs attract MDSCs to the tumor site through ENTPD2/CD39 L1 signaling and halt the PD1 treatment, and the reduction of MDSC sensitizes these cells to 5-FU [170, 171]. Recently introduced CAR T cell (chimeric antigen receptor T cell) is an altered host white blood cell therapy, that has revolutionized anti-cancer immune therapy to cross the barrier of specificity and non-responders to standard therapy and was first tried on acute lymphoblastic leukemia (ALL) patients with better remission [172-174]. However, due to the constant threat of developing cytokine release syndrome (CRS) and early relapse of antigen-positive leukemia (loss of active CAR T cell-mediated surveillance) or later relapse (loss of antigen) of the tumor, limited the range of specific CAR T cells [175]. However recent cotreatment of CAR T cells with immunomodulators (immune check point inhibitors like PDL1-PD1 blockage) has shown to be promising in terms of the depth and durability of the treatment clinically [176-179]. The above description insights us into the importance of the ‘CSC-TME-immune’ triangular (Fig. 2) signaling interaction in tumor expansion and therapy resistance and its clinical significance. A comprehensive representation of immune and CSC in drug resistance linkage has been shown in Fig. 2.
Dynamic EMT programs and drug resistance are mutually linked to each other

Moreover, a cohort of large breast cancer samples has revealed that ZEB1overexpressing samples were less responsive to genotoxic epirubicin treatment. Upon dissecting the mechanism it was found that ZEB is actively activating ataxia-telangiectasia mutated (ATM) kinase at the transcriptional level by promoting the ZEB1/ p300/PCAF complex formation which resulted in the activation of homologous recombination DNA repair pathway [192]. EMT and the tumor cells microenvironment (TME) are linked through the FBXW7-ZEB2 axis to promote colorectal CSC formation and chemoresistance [40]. Moreover, through the FZD7/Wnt/
role in regulating tumorigenicity and chemoresistance [195].
Role of pH gradient across organelles in drug resistance

subcellular location with less effect on pH change [214, 215] Fig. 3. Endolysosomes are an important component of the intracellular catabolic process called autophagy which clears the extra, unknown, deformed, and unused biomolecules from the cells by breaking them into smaller building blocks or energy units for the cell [53, 132-134]. Various reports suggest that endolysosome’s pH plays a pivotal role in drug resistance by accumulating and sequestering various chemotherapeutic drugs (a mostly weak base) that enter into them either by passive diffusion or through membrane-embedded P-gp pumps and are later expelled by exocytosis and thus generate chemoresistant cancer cells [216-219]. So the integrity of lysosomal membrane permeability (LMP) is necessary for cancer cells to be resistant and can be considered a therapeutically important subject. Various studies have shown that LMP inducers like chloroquine (CQ) can result in resistant cancer cell death by releasing sequestered drugs and proteasomes like cathepsin to act on the nucleus and induce apoptosis [220-222]. Moreover, CQ promotes the release of NO that efficiently inhibits P-gp activity and leads to the accumulation of chemotherapeutic drugs thus leading to death in the resistant hepatic carcinoma [223].

Studies show thatEndoplasmic reticulum (ER) plays a vital role in drug metabolism due to the presence of the drug-metabolizing enzyme “cytochrome P450″(CYPs). Lin. et al. demonstrated that the expression of cytochrome P450 enzyme CYP1B1 was higher in taxolresistant A549 cells compared to its parental A549 cells and inhibition of CYP1B1 by 4 hydroxy-emodin increased their sensitivity to Taxol [230]. The Golgi body (GB) functions as a post-translational trafficking
hub and has a role in drug resistance as well. In glioblastoma resistance cells, GB overexpresses various MDRs and sequesters the drugs by their secretary system which later exports drugs out of the cells, the process is reversed by P-gp inhibitors such as S9788 and PSC833, which reverses drug resistance [196]. The peroxisome an oxidative stress reliever of cells also plays a role in resistance as in lymphoma towards vorinostat by reducing ROS generation and inhibition of peroxisome activity was shown to increase the drug sensitivity of these cells [231-233]. The nucleus is the control center of eukaryotic cells and previous studies reported the expression of various kinds of ABC transporters such as P-gp on the membrane of the nucleus and its role in resistance to various anti-cancer drugs like doxorubicin in various cancers such as glioblastoma multiform (LN299) [234-237]. An image representation of the organelle’s role in drug resistance is shown in Fig. 4.

Immune cells and tumor microenvironment protect cancer cells from anti-cancer drugs
i) CAFs (stromal cells): are activated fibroblasts, abundantly associated with cancerous cells in the TME, a variety of oncogenic cues like growth factors, chemokines, exosomes, etc. contributed by CAFs allow cancer cells to undergo EMT, evade therapies and potentiate them for tumor relapse [241-244]. CAF responds to the chemotherapeutic drug cisplatin used against esophageal squamous cell carcinoma (ESCC), by secreting paracrine signaling molecule plasminogen activator inhibitor 1 (PAI-1). PAI-1
in turn stimulates the survival of AKT and MAPK pathways to protect ESCC from ROS-mediated DNA damage and cell death under chemotherapeutic drug treatment thus supporting resistance [245]. Moreover, CAF-secreted hepatocyte growth factor (HGF) or TGF-
ii) ECM: The ECM is another factor that is made up of fibrous proteins like collagen, elastin, proteoglycans, microelements, water, etc., and plays an active role in therapeutic resistance (Fig. 5) [250, 251]. Tumor ECM is substantially different in structure and composition and exhibits fibroblastic/myofibroblastic infiltration, followed by the considerable buildup of collagenous matrix or desmoplastic stroma, obstructing anti-cancer drug delivery to cancer cells [251, 252]. It was found that cisplatin-resistant ovarian malignancy overexpresses COL11A1 (collagen type XI
therapeutic resistance [255,256]. It has been shown that cancer cells TME/ECM lacking collagen or fibronectin or both were more sensitive towards cisplatin than their counterparts [257]. In continuing with this, Oxaliplatin’s effectiveness against colon cancer cells was increased by suppressing the
iii) Exosomes: Exosomes, also known as extracellular vesicles (EVs), which are produced by big multivesicular bodies (MVBs), facilitate cell-to-cell interaction by transporting bioactive cargos, across cells and efflux of undesirable molecules in healthy cells [259]. They are important signaling intercessors, playing roles in tumor growth, TME remodeling, metastasis, angiogenesis, as well as treatment resistance [260, 261]. Various cancerous cells hijack EVs for anti-cancer drug efflux which in turn results in drug resistance as shown in Fig. 5 [262, 263]. Mesenchymal stem cell (MSC) derived exosomes integrate into gastric tumor cells, enhancing CaM-Ks and downstream Raf/MEK/ERK signaling cascades pathway activation, which increases the expression of MDRrelated proteins resulting in gastric carcinoma chemoresistance [264]. Ovarian cancer drug-resistant cells showed increased cisplatin exosomal export along with the recruitment of efflux transporters ATP7A, ATP7B, and MRP2 (ABCB2) in neighboring cells to protect drug-mediated cell death [265].
iv) Hypoxia: The tumor’s aberrant vasculature and high oxygen demand result in hypoxia, and reduced availability of nutrients like glucose and vital amino acids [266, 267]. Oxygen deprivation stimulates hypoxia-inducible factor (HIF)-
v) Immune cells: The most prevalent immune cell types in TME are tumor-associated macrophages (TAMs) [273]. TAMs invasion into TME has been linked to poor prognosis and inadequate response to chemotherapeutic agents in cancer patients [274, 275]. TAMs are derived from circulatory Ly6C
ties [276]. The M2 phenotype plays a role in treatment resistance and induces Th2 responses. In the cancer microenvironment, the polarization of macrophages from M1 to M2 is prevalent [277]. Drug treatment stimulates TAMs to develop into immunosuppressive M2-polarized macrophages which confer chemoresistance in various cancerous cells. It has been shown that ROS builds up in the gastric carcinoma cells after exposure to 5-FU activates HIF
Metabolic changes/regulations govern drug resistance in cancer cells
Moreover, ATP metabolism also has a profound role in the onco-immune or immunomodulatory system which in normal conditions maintains the body’s homeostasis by maintaining the balance between immune-stimulatory, immune suppression, and autoimmune disease defense. Cancer cells metabolize extracellular ATP into immunosuppressive extracellular adenosine (eADO) by transmembrane ectopeptidases CD39 (ectonucleotidase triphosphate diphosphate-hydrolase 1) and mem-brane-anchored CD73 (5′-nucleotidase) [296]. Although another noncanonical pathway also participates in the generation of adenosine which includes the activity of CD38 (NAD
or tumor cells resistance to immune therapy. Adenosine pathway activation affects lymphocyte activation including tumor-infiltrating immune cells, myeloid cells, and tumor-associated stromal cells, and tumor cells consequently helping cancer cells to evade from anti-tumor immune cell response which results in therapy resistance [297]. So targeting CD39, CD73, and adenosine receptors in the presence of immune checkpoint therapy (anti-PDL1/PD1; ANTI CTLA-4) can prove to be a novel immunotherapeutic strategy against immune-resistant cancer cells. For example, combined treatment of Poly-oxotungstate-1 (POM1; CD39 inhibitor) and anti-PD1 and CTLA-4 antibodies in B16-F10 (melanoma cells) transplanted mice model showed a remarkable reduction in tumor burden and increase in the survival of tumorbearing mice [298]. Although rodent data is very promising but the key issue of this finding is wheather this can be translated in humans.
Cancer cell drug Metabolism and resistance
their action [305]. However, cancer cells exhibit resistance to drugs by altering a catalytic site or structure of an enzyme which affects their interaction with drugs and thus culminates in their mode of action. For example, cytarabine (cytidine analog) resistant AML cells express mutated deoxycytidine kinase (cytarabine activator), reduce the activity of the drug and thus develop resistance [306, 307]. Similarly, doxorubicin (active) resistance in prostate and breast cancer is mainly due to enzymatic transformation into doxorubicinol (inactive) by the overexpressed enzyme Aldo-keto reductase, combination therapy is shown to be very effective in increasing therapeutic activity of DOX [308, 309].
Drug target alteration
Cancer cells show drug resistance either by reducing uptake or enhancing the efflux of drugs or both through mutated receptors and transporters [315]. Membrane-bound transporters called organic aniontransporting polypeptides (like OATP1B1, OATP1B3, and OATP1A2) can transport paclitaxel, methotrexate, flavopiridol, tyrosine kinase inhibitors, irinotecan, cisplatin and plays a crucial role in the resistance in polyps of the large intestine and colon cancer [316-319]. Moreover in hepatocellular carcinoma (HCC), cholangiocarcinoma (CGC), and Chronic myeloid leukemia (CML) a lesser accumulation of anticancer drugs
(like imatinib) seems to be mediated by a decrease in OATP1B1, OATP1B3, and OATP1A2 expression or their function [317, 320, 321]. Various studies have shown that organic cation transporter-1 (OCT1) is involved in the uptake of potent cationic anti-tumor drugs, such as Cisplatin, anthracyclines, and sorafenib and its activity is dysregulated in many cancers such as colon and liver cancer [322-325]. In addition, it has been demonstrated that imatinib uptake in CML is dependent on OCT1 expression and the degree of OCT1 expression is considered a useful biomarker for predicting the efficacy of imatinib-based therapy in leukemia patients [326]. The high-affinity copper transporter (CTR1) has been recently shown to transport platinum drugs, emphasizing the crucial function of CTR1 in platinum-drug sensitivity in cancer chemotherapy [327, 328]. A promising phase I clinical trial using cotreatment of trientine (a copper chelator) and carboplatin has shown CTR1-mediated higher cisplatin uptake and better outcomes [329, 330].
Autophagy and ER stress (UPR) are utilized by cancer cells to gain support for drug resistance
programmed cell death type 2 [133, 341]. In various types of cancer, autophagy can play a contrasting role in either supporting or inducing death and is context-dependent [342-347]. However, the role of autophagy in drug resistance is an emerging topic and a deep understanding of this relationship can prove to be therapeutically very crucial to curb various cancer types. Various studies have shown that autophagy plays a central role in drug resistance by recycling biomolecules, degrading deformed proteins and organelles, and thus preventing DNA damage [348-350]. Some reports have suggested that DNA damage response can also activate autophagy via ataxia telangiectasia mutated (ATM) and homologous recombination (HR) repair pathway [351, 352]. It has been found that an anthracycline drug epirubicin-mediated autophagy upregulates P-gp proteins and downregulates the NF-кB signaling pathway thus hindering activation of apoptosis and promoting drug resistance [353]. Tamoxifen sensitivity was enhanced in resistant estrogen recep-tor-positive breast cancer cells by autophagy inhibition, thus inducing cell death [354, 355]. In gastrointestinal stromal tumors, cotreatment of imatinib and autophagy inhibitors (like chloroquine) cause apoptosis [356, 357]. The endoplasmic reticulum (ER) is an essential subcellular structure that maintains cellular homeostasis and can be disrupted by a variety of pathological conditions like cancer, resulting in induction of ER stress which if remains sustained can either kill cancer cells by induction of apoptosis and ferroptosis or help them to grow, survive and induce drug resistance if moderately activated [358]. ER stress induces UPR ,which is regulated by ino-sitol-requiring enzyme-1 (IRE1
from apoptosis [362, 363]. Autophagy is shown to influence some crucial drug-resistant enzymes (detoxifying enzymes) like aldehyde dehydrogenase (ALD1A3) and thus arbitrate in acquired drug resistance in temozolo-mide-treated human glioblastoma cells [141]. In human ovarian cells (Polycystic ovarian syndrome- PCOS) aberrant autophagy induction upon release of the high mobility group box 1(HMGB1) plays a role in achieving insulin resistance by downregulating IRS-1, AKT, and GLUT4 translocation [364]. While in hepatocellular carcinoma cells, HMGB1 is shown to promote doxorubicin resistance by inducing AMPK-autophagy and protecting them from programmed cell death type 1 [365]. Autophagy is found to play a cytoprotective role in TNF-TRAIL deathresistant cells by sequestering, degrading, and dysfunctioning of caspase 8 protecting cancer cells to undergo death [366-368]. However, there are ample reports that prolonged or sustained autophagy activation can result in programmed cell death type 2. For example, resveratrol (plant-derived phytoalexin) treatment triggers a strong signal for autophagy (p62 accumulation) through JNK activation leading to death in imatinib-resistant chronic myelogenic leukemia cells (CML) [369, 370]. In some cases, autophagy activation supresses drug resistance and induces therapeutic dependent or independent death. For example, co-treatment of ABT-88 (polymerase protein inhibitor) and temozolomide has been shown to sensitize temozolomide-resistant glioma cells by inducing double DNA strand breaks and coincidently coactivating lethal autophagy and apoptosis [371]. So autophagy in most cases opposes the anti-cancer drug action of culminating cells by apoptosis, thus acting as a defensive cellular pathway and supporting drug resistance.
Proteomic alteration in response to known chemotherapeutic drugs to achieve drug resistance (Fig. 6)

Likewise, resistance to cisplatin remains a barrier in the treatment of various cancers [382]. Upon entering the cell, it reacts with a range of molecules apart from DNA, such as sulfur-containing glutathione (GSH)/metallothionein (MT) which trap cisplatin and then eliminate it from the cell[383]. Moreover, Zinc-finger factor 143 (ZNF143), Y-box binding protein-1 (YB-1), activating transcription factor 4 (ATF4), CCAAT-binding transcription factor 2 (CTF2), DNA repair proteins (e.g., the product of the XRCC1 gene; YB1; etc.) and mitochondrial transcription factor A (mtTFA) are a few transcription factors that have been linked to CDDP resistance [384-386]. Another well-known drug is paclitaxel (PTX), which belongs to the taxane class of anticancer agents that affect the normal stability of microtubules during cell division and is
effective on various cancers like breast, ovarian, etc. Although tubulin is the primary target for PTX, it has additionally been discovered to attack the mitochondria and block the activity of the apoptotic inhibitor protein Bcl-2 (B-cell Leukemia 2) [387]. Like other antineoplastic drugs, PTX treatment can induce resistance by inducing overexpression of the motor protein MCAK (leads to tubulin depolymerization), affecting membrane lipids (fatty acid synthase, Lipin, etc.), modified cell cycle checkpoint proteins (BUB1, CCNB1, CENPE, CENPF), enhanced DNA damage repair pathway proteins (like hMSH2), elevated efflux proteins (MDR1, MDR3, etc. [388-393].
Discussion
anti-tumorigenic environment into a supportive, protumorigenic environment [15, 16]. Moreover, some of the tumor cells can induce the reprogramming of stromal cells and immune cells, inducing the secretion of diverse factors like cytokines, that enhance tumor progression and suppress cell death [396]. Besides, CSCs have emerged as key players in the intricate landscape of cancer drug resistance [397]. They acquire remarkable abilities to resist conventional treatments, driving disease recurrence and metastasis as has been detailed explained in Section-4. “CSCs play a key role in developing drug resistance and tumor relapse”. CSCs unique properties, such as self-renewal and differentiation potential, upregulated surface membrane immune inhibitory ligands and release of various chemo/cytokines collectively contribute to therapy resistance and pose significant challenges in achieving long-term remission [398, 399]. Therefore targeting CSCs opens possibilities for innovative therapeutic strategies that can be used in combination with another chemotherapeutic drug that can hold promise in overcoming drug resistance and potentially improve patient outcomes. One of the prevailing mechanisms of drug resistance that needs to be focused on, is the involvement in the expulsion of hydrophobic drugs, facilitated by ATP-dependent ABC transporters [400]. A well-studied member of the ABC transporter, P-gp, an integral membrane protein, is frequently upregulated in diverse malignancies [400]. Specifically, gaining a comprehensive understanding of intricate mechanisms underlying multidrug resistance (MDR) in cancer cells is likely to play a pivotal role in the development of innovative approaches to cancer therapy in the coming years. However, more work is needed to be done from root level using high throughput assays (single cell level) of different sections of the same tumor samples to find a specific target/s for therapy or combination therapy as discussed above. Achieving this goal might address the evolving aggressive and resistant cancer cells leading to reduced severity and improving the survival of patients.
| Abbreviations | |
| CSCs | Cancer stem cells |
| CTLA-4 | Cytotoxic T lymphocyte-associated antigen 4 |
| PD-1 | Programmed Cell Death Protein 1 |
| PD-L1 | Programmed Death Ligand 1 |
| NSCLC | Non-small-cell lung cancer |
| MDR | Multi-drug resistant |
| HCC | Hepatocellular carcinoma |
| DTP | Drug-tolerant persister |
| DNMTs | DNA Methyltransferases |
| DNAm | DNA methylation |
| 5-FU | 5-Fluorouracil |
| P-gp | P-glycoprotein |
| Bcl-2 | B-cell lymphoma 2 |
| Bcl-xl | B-cell lymphoma-extra large |
| IAPs | Inhibitors of apoptosis |
| Bid | BH3 interacting-domain death agonist |
| Bax | Bcl-2 Associated X-protein |
| Bim | Bcl-2 Interacting Mediator of cell death |
| PUMA | P53 upregulated modulator of apoptosis |
| CRC | Colorectal carcinoma |
| DOX | Doxorubicin |
| MAPK | Mitogen-activated protein kinase |
| KMTs | Histone lysine methyltransferase |
| PRMTs | Protein arginine methyl transferase |
| GSH | Glutathione |
| DNA-PK | DNA-dependent protein kinase |
| SASP | Senescence-associated secretory phenotype |
| AML | Acute myeloid leukemia |
| Notch 1 | Neurogenic locus notch homolog protein 1 |
| Wnt | Wingless-related integration site |
| TGF-
|
Transforming growth factor-
|
| ESCs | Embryonic stem cells |
| SOX2 | Sex determining region Y-box 2 |
| OCT4 | Octamer-binding transcription factor 4 |
| KLF4 | Kruppel-like factor 4 |
| SALL4 | Sal-like protein 4 |
| FOXM1 | Forkhead box protein M1 |
| Hh | Hedgehog |
| Chk | Checkpoint kinase |
| UBC | Urothelial bladder cancer |
| EMT | Epithelial-to-mesenchymal transition |
| MET | Mesenchymal-to-epithelial transition |
| TNBC | Triple-negative breast cancer |
| ABCG | ATP binding cassette subfamily G |
| FBXO21 | F-Box Protein 21 |
| GSTp | Glutathione S-transferase p |
| uPA | Urokinase Plasminogen Activator |
| HIF-1a | Hypoxia Inducible factor-1 a |
| CRISPR/Cas9 | Clustered regularly interspaced short palindromic repeats/ Caspase 9 |
| ATG | Autophagy-related |
| ROS | Reactive oxygen species |
| ALDH | Aldehyde dehydrogenase |
| TNBCSC | Triple-negative breast cancer stem cells |
| JAK/STAT | Janus Kinase/signal transducers and activators of transcription |
| IL | Interleukin |
| CCL2 | CC-Chemokine ligand 2 |
| CSF1 | Colony stimulating factor 1 |
| CSF2 | Colony stimulating factor 2 |
| HGF | Hepatocyte growth factor |
| MIF | Macrophage migration inhibitory factor |
| CX3CL1 | C-X3-C motif Chemokine ligand 1 |
| PGE2 | Prostaglandin E2 |
| SDF-1 | Stromal cell-derived factor 1 |
| LOX | Lysyl oxidase |
| CCL3 | CC-Chemokine ligand 3 |
| CCL5 | CC Chemokine ligand 5 |
| VEGF-A | Vascular endothelial growth factor A |
| PTN | Pleiotrophin |
| HMGB1 | High-mobility group box 1 |
| TAMs | Tumor-associated macrophages |
| EGFR | Epidermal growth factor receptor |
| IDO1 | Indoleamine 2,3-dioxygenase 1 |
| PGE | Prostaglandin |
| ENTPD2 | Ectonucleoside triphosphate diphosphohydrolase 2 |
| CD | Clusters of differentiation |
| MDSC | Myeloid-derived suppressor cells |
| CART cell | Chimeric antigen receptor T cell |
| CRS | Cytokine release syndrome |
| ZEB1 | Zinc finger E-box binding homeobox 1 |
| HDAC | Histone deacetylase |
| ATM | Ataxia-telangiectasia mutated |
| PCAF | p300/CBP-associated factor |
| FBXW7 | F-box and WD repeat domain-containing 7 |
| TWIST1 | Twist-related protein 1 |
| LMP | Lysosomal membrane permeability |
| UCP2 | Uncoupler protein 2 |
| mTOR | Mammalian target of rapamycin |
| NF-kB | Nuclear factor- kB |
| STAT3 | Signal transducer and activator of transcription3 |
| MRP | Multidrug resistance protein |
| TRAIL | Tumor related apoptosis-inducing ligand |
| GBM | Glioblastoma multiforme |
| VIM | Vimentin |
| VDAC | Voltage-dependent anion channel |
| OHCP | 4-Hydroxy-cyclophosphamide |
| OATPs | Organic anion-transporting polypeptides |
| LRP | Lung resistance-related protein |
| UPR | Unfolded protein response |
| LC3 | Light chain 3 |
| CML | Chronic myelogenic leukemia cells (CML) |
| JNK | Jun N-terminal kinase |
| DUSP4 | Dual specificity protein phosphatase 4 |
| ERK | Extracellular signal-regulated kinase |
| ATF4 | Activating transcription factor 4 |
| ZNF143 | Zinc-finger factor 143 |
| YB-1 | Y-box binding protein-1 |
| BUB1 | Budding uninhibited by benzimidazoles 1 |
| CCNB1 | Cyclin B1 |
| CENPE | Centromere protein E |
| CENPF | Centromere protein F |
Acknowledgements
Authors’ contributions
Funding
Availability of data and materials
Declarations
Ethics approval and consent to participate
Consent for publication
Competing interests
Published online: 12 February 2024
References
- Nikolaou
, et al. The challenge of drug resistance in cancer treatment: a current overview. Clin Exp Metastasis. 2018;35(4):309-18. - Saha M, Sarkar A. Review on Multiple Facets of Drug Resistance: A Rising Challenge in the 21st Century. J Xenobiot. 2021;11(4):197-214.
- Boumahdi S, de Sauvage FJ. The great escape: tumour cell plasticity in resistance to targeted therapy. Nat Rev Drug Discov. 2020;19(1):39-56.
- Michaelis M, Wass MN, Cinatl J. Drug-adapted cancer cell lines as preclinical models of acquired resistance. Cancer Drug Resist. 2019;2(3):447-56.
- Oun R, Moussa YE, Wheate NJ. The side effects of platinumbased chemotherapy drugs: a review for chemists. Dalton Trans. 2018;47(19):6645-53.
- Tao JJ, Visvanathan K, Wolff AC. Long term side effects of adjuvant chemotherapy in patients with early breast cancer. Breast. 2015;24:S149-53.
- Aramini
, et al. Dissecting tumor growth: The role of cancer stem cells in drug resistance and recurrence. Cancers (Basel). 2022;14(4):976. - Masoudi M, Gopalan V. Roles of Cancer Stem Cells in Therapy Resistance and Disease Recurrence. In: Cancer Stem Cells: Basic Concept and Therapeutic Implications. Springer; 2023. p. 149-65.
- Zhang Z, et al. Overcoming cancer therapeutic bottleneck by drug repurposing. Signal Transduct Target Ther. 2020;5(1):113.
- DeRidder L, et al. The past, present, and future of chemotherapy with a focus on individualization of drug dosing. J Control Release. 2022;352:840-60.
- XuH , et al. Targeting strategies for bone diseases: signaling pathways and clinical studies. Signal Transduct Target Ther. 2023;8(1):202.
- Miglietta F, et al. Endocrine sequelae of hematopoietic stem cell transplantation: Effects on mineral homeostasis and bone metabolism. Front Endocrinol (Lausanne). 2023;13:1085315.
- Mansoori
, et al. The different mechanisms of cancer drug resistance: brief review. Adv Pharm Bull. 2017;7(3):339. - Wang
, Zhang , Chen . Drug resistance and combating drug resistance in cancer. Cancer Drug Resist. 2019;2(2):141. - Dzobo K, et al. Not Everyone Fits the Mold: Intratumor and Intertumor Heterogeneity and Innovative Cancer Drug Design and Development. OMICS. 2018;22(1):17-34.
- Labrie M, et al. Therapy resistance: opportunities created by adaptive responses to targeted therapies in cancer. Nat Rev Cancer. 2022;22(6):323-39.
- Nussinov R, Tsai CJ, Jang H. Anticancer drug resistance: An update and perspective. Drug Resist Updat. 2021;59:100796.
- Dzobo K, Senthebane DA, Dandara C. The Tumor Microenvironment in Tumorigenesis and Therapy Resistance Revisited. Cancers (Basel). 2023;15(2):376.
- Dzobo K, et al. Advances in Therapeutic Targeting of Cancer Stem Cells within the Tumor Microenvironment: An Updated Review. Cells. 2020;9(8):1896.
- Senthebane DA, et al. The role of tumor microenvironment in chemoresistance: to survive, keep your enemies closer. Int J Mol Sci. 2017;18(7):1586.
- Khan SU, Fatima K, Malik F, Kalkavan H, Wani A. Cancer metastasis: Molecular mechanisms and clinical perspectives. Pharmacol Ther. 2023;250:108522. https://doi.org/10.1016/j.pharmthera.2023.108522.
- Yang Q, et al. Extracellular Vesicles in Cancer Drug Resistance: Roles, Mechanisms, and Implications. Adv Sci (Weinh). 2022;9(34):e2201609.
- Namee NM, O’Driscoll L. Extracellular vesicles and anti-cancer drug resistance. Biochim Biophys Acta Rev Cancer. 2018;1870(2):123-36.
- Wu S. Fu L, Tyrosine kinase inhibitors enhanced the efficacy of conventional chemotherapeutic agent in multidrug resistant cancer cells. Mol Cancer. 2018;17(1):1-13.
- Roviello G , et al. Apatinib: a novel receptor tyrosine kinase inhibitor for the treatment of gastric cancer. Cancer Lett. 2016;372(2):187-91.
- Martin SK, et al. N-terminal targeting of androgen receptor variant enhances response of castration resistant prostate cancer to taxane chemotherapy. Mol Oncol. 2015;9(3):628-39.
- Fujita K, Nonomura N. Role of androgen receptor in prostate cancer: a review. World J Mens Health. 2019;37(3):288-95.
- Asano
, et al. Clinical verification of sensitivity to preoperative chemotherapy in cases of androgen receptor-expressing positive breast cancer. Br J Cancer. 2016;114(1):14-20. - Barton VN, et al. Androgen receptor supports an anchorage-independent, cancer stem cell-like population in triple-negative breast cancer. Cancer Res. 2017;77(13):3455-66.
- Niikura N, et al. Changes in tumor expression of HER2 and hormone receptors status after neoadjuvant chemotherapy in 21755 patients from the Japanese breast cancer registry. Ann Oncol. 2016;27(3):480-7.
- Tolaney SM, et al. Abemaciclib plus trastuzumab with or without fulvestrant versus trastuzumab plus standard-of-care chemotherapy in women with hormone receptor-positive, HER2-positive advanced breast cancer (monarcHER): a randomised, open-label, phase 2 trial. Lancet Oncol. 2020;21(6):763-75.
- Calabrò L, et al. Efficacy and safety of an intensified schedule of tremelimumab for chemotherapy-resistant malignant mesothelioma: an openlabel, single-arm, phase 2 study. Lancet Respir Med. 2015;3(4):301-9.
- Calabrò L, et al. Tremelimumab for patients with chemotherapy-resistant advanced malignant mesothelioma: an open-label, single-arm, phase 2 trial. Lancet Oncol. 2013;14(11):1104-11.
- Noguchi T, et al. Temporally Distinct PD-L1 Expression by Tumor and Host Cells Contributes to Immune EscapeThe Role of Tumor versus Host PD-L1 in Tumor Immune Escape. Cancer Immunol Res. 2017;5(2):106-17.
- Juneja VR, et al. PD-L1 on tumor cells is sufficient for immune evasion in immunogenic tumors and inhibits CD8 T cell cytotoxicity. J Exp Med. 2017;214(4):895-904.
- Ribas A, Wolchok JD. Cancer immunotherapy using checkpoint blockade. Science. 2018;359(6382):1350-5.
- Shi Z-D, et al. Tumor cell plasticity in targeted therapy-induced resistance: mechanisms and new strategies. Signal Transduct Target Ther. 2023;8(1):113.
- Prasad B, et al. Exploration of space to achieve scientific breakthroughs. Biotechnol Adv. 2020;43:107572.
- Jin H, Wang L, Bernards R. Rational combinations of targeted cancer therapies: background, advances and challenges. Nat Rev Drug Discovery. 2023;22(3):213-34.
- Lim Z-F, Ma PC. Emerging insights of tumor heterogeneity and drug resistance mechanisms in lung cancer targeted therapy. J Hematol Oncol. 2019;12(1):1-18.
- Krook MA, et al. Tumor heterogeneity and acquired drug resistance in FGFR2-fusion-positive cholangiocarcinoma through rapid research autopsy. Cold Spring Harb Mol Case Stud. 2019;5(4):a004002.
- Guillon J, et al. Chemotherapy-induced senescence, an adaptive mechanism driving resistance and tumor heterogeneity. Cell Cycle. 2019;18(19):2385-97.
- Hass R, von der Ohe J, Ungefroren HJC. Impact of the tumor microenvironment on tumor heterogeneity and consequences for cancer cell plasticity and stemness. Cancers (Basel). 2020;12(12):3716.
- Punter KB, Chu CE, Chan YW. Mitochondrial dynamics and oxidative phosphorylation as critical targets in cancer. Endocr Relat Cancer. 2023;30(1):e220229.
- Hanselmann RG, Welter C. Origin of Cancer: Cell work is the Key to Understanding Cancer Initiation and Progression. Front Cell Dev Biol. 2022;10:787995. https://doi.org/10.3389/fcell.2022.787995.
- Jiang W, et al. Personalized medicine of non-gene-specific chemotherapies for non-small cell lung cancer. Acta Pharm Sin B. 2021;11(11):3406-16.
- Capdevila J, et al. Molecular diagnosis and targeted treatment of advanced follicular cell-derived thyroid cancer in the precision medicine era. Cancer Treat Rev. 2022;106:102380.
- Bhang H-e C, et al. Studying clonal dynamics in response to cancer therapy using high-complexity barcoding. Nat Med. 2015;21(5):440-8.
- Jamal-Hanjani M, et al. Tracking the evolution of non-small-cell lung cancer. N Engl J Med. 2017;376(22):2109-21.
- Singh AK, et al. Tumor heterogeneity and cancer stem cell paradigm: updates in concept, controversies and clinical relevance. Int J Cancer. 2015;136(9):1991-2000.
- Huang, R., P.-K.J.S.T. Zhou, and T. Therapy. DNA damage repair: Historical perspectives, mechanistic pathways and clinical translation for targeted cancer therapy. Signal Transduct Target Ther. 2021;6(1):254.
- Roberts SA, Gordenin DA. Hypermutation in human cancer genomes: footprints and mechanisms. Nat Rev Cancer. 2014;14(12):786-800.
- Khan SU, et al. Redox balance and autophagy regulation in cancer progression and their therapeutic perspective. Med Oncol. 2023;40(1):1-21.
- Sui Q , et al. Genetic and microenvironmental differences in nonsmoking lung adenocarcinoma patients compared with smoking patients. Transl Lung Cancer Res. 2020;9(4):1407.
- Anagnostou V, et al. Multimodal genomic features predict outcome of immune checkpoint blockade in non-small-cell lung cancer. Nat Cancer. 2020;1(1):99-111.
- Connor AA. Gallinger S, Pancreatic cancer evolution and heterogeneity: integrating omics and clinical data. Nat Rev Cancer. 2022;22(3):131-42.
- Killcoyne S, Fitzgerald RC. Evolution and progression of Barrett’s oesophagus to oesophageal cancer. Nat Rev Cancer. 2021;21(11):731-41.
- Sjödahl G, et al. Molecular changes during progression from nonmuscle invasive to advanced urothelial carcinoma. Int J Cancer. 2020;146(9):2636-47.
- van Niekerk G, et al. C ancer stem cells: A product of clonal evolution? Int J Cancer. 2017;140(5):993-9.
- Losic B, et al. Intratumoral heterogeneity and clonal evolution in liver cancer. Nat Commun. 2020;11(1):1-15.
- Zardavas D, et al. Clinical management of breast cancer heterogeneity. Nat Rev Clin Oncol. 2015;12(7):381-94.
- AI-Rawi DH, Bakhoum SF. Chromosomal instability as a source of genomic plasticity. Curr Opin Genet Dev. 2022;74:101913.
- van den Bosch T, Derks S, Miedema DMJC. Chromosomal Instability Selection and Competition: Factors That Shape the Level of Karyotype Intra-Tumor Heterogeneity. Cancers (Basel). 2022;14(20):4986.
- Comaills V, Castellano-Pozo MJB. Chromosomal Instability in Genome Evolution: From Cancer to Macroevolution. Biology (Basel). 2023;12(5):671.
- Majc B, et al. Epithelial-to-mesenchymal transition as the driver of changing carcinoma and glioblastoma microenvironment. Biochim Biophys Acta Mol Cell Res. 2020;1867(10):118782.
- Levitin HM, Yuan J, Sims PA. Single-cell transcriptomic analysis of tumor heterogeneity. Trends Cancer. 2018;4(4):264-8.
- Fan J, et al. Linking transcriptional and genetic tumor heterogeneity through allele analysis of single-cell RNA-seq data. Genome Res. 2018;28(8):1217-27.
- Peng J, et al. Single-cell RNA-seq highlights intra-tumoral heterogeneity and malignant progression in pancreatic ductal adenocarcinoma. Cell Res. 2019;29(9):725-38.
- Ding S , Chen X , Shen KJCC. Single-cell RNA sequencing in breast cancer: Understanding tumor heterogeneity and paving roads to individualized therapy. Cancer Commun (Lond). 2020;40(8):329-44.
- Hata AN, et al. Tumor cells can follow distinct evolutionary paths to become resistant to epidermal growth factor receptor inhibition. Nat Med. 2016;22(3):262-9.
- Ho DW-H, et al. Single-cell RNA sequencing shows the immunosuppressive landscape and tumor heterogeneity of HBV-associated hepatocellular carcinoma. Nat Commun. 2021;12(1):1-14.
- Cheng Y, He C, Wang M, Ma X, Mo F, Yang S, Han J, Wei X. Targeting epigenetic regulators for cancer therapy: mechanisms and advances in clinical trials. Signal Transduct Target Ther. 2019;4:62. https://doi.org/10. 1038/s41392-019-0095-0.
- Mancarella D, Plass C. Epigenetic signatures in cancer: proper controls, current challenges and the potential for clinical translation. Genome Med. 2021;13:1-12.
- Mikubo M , et al. Mechanism of drug tolerant persister cancer cells: the landscape and clinical implication for therapy. J Thorac Oncol. 2021;16(11):1798-809.
- De Conti G, Dias MH , Bernards RJC. Fighting drug resistance through the targeting of drug-tolerant persister cells. Cancers (Basel). 2021;13(5):1118.
- Toh TB, Lim JJ, Chow EK-H. Epigenetics in cancer stem cells. Mol Cancer. 2017;16(1):1-20.
- Fouad MA, et al. Impact of global DNA methylation in treatment outcome of colorectal cancer patients. Front Pharmacol. 2018;9:1173.
- Lee DD, et al. DNA methylation of the TERT promoter and its impact on human cancer. Curr Opin Genet Dev. 2020;60:17-24.
- Wu Q, et al. Methylation of miR-129-5p CpG island modulates multidrug resistance in gastric cancer by targeting ABC transporters. Oncotarget. 2014;5(22):11552-63.
- Ni Y, et al. The role of tumor-stroma interactions in drug resistance within tumor microenvironment. Front Cell Dev Biol. 2021;9:637675.
- Shi Y, et al. Genome-wide DNA methylation analysis of breast cancer MCF-7/Taxol cells with MeDIP-Seq. PLoS One. 2020;15(12):e0241515.
- Aguiari G, et al. Dysregulation of Transglutaminase type 2 through GATA3 defines aggressiveness and Doxorubicin sensitivity in breast cancer. Int J Biol Sci. 2022;18(1):1.
- Mosca L, et al. Therapeutic potential of the natural compound S-adenosylmethionine as a chemoprotective synergistic agent in breast, and head and neck cancer treatment: Current status of research. Int J Mol Sci. 2020;21(22):8547.
- Williams MM, Cook RS. Bcl-2 family proteins in breast development and cancer: could Mcl-1 targeting overcome therapeutic resistance? Oncotarget. 2015;6(6):3519.
- Baharudin R, et al. Identification of predictive DNA methylation biomarkers for chemotherapy response in colorectal cancer. Front Pharmacol. 2017;8:47.
- Yang C, et al. Histone methyltransferase and drug resistance in cancers. J Exp Clin Cancer Res. 2020;39(1):173.
- Liu C-W, et al. Histone Methyltransferase G9a Drives Chemotherapy Resistance by Regulating the Glutamate-Cysteine Ligase Catalytic Subunit in Head and Neck Squamous Cell CarcinomaG9a Modulates GCLC Expression and Chemoresistance. Mol Cancer Ther. 2017;16(7):1421-34.
- Musiani D, et al. PRMT1 Is Recruited via DNA-PK to Chromatin Where It Sustains the Senescence-Associated Secretory Phenotype in Response to Cisplatin. Cell Rep. 2020;30(4):1208-1222.e9.
- Walcher L, et al. Cancer stem cells—origins and biomarkers: perspectives for targeted personalized therapies. Front Immunol. 2020;11:1280.
- Trumpp A, Haas SJC. Cancer stem cells: the adventurous journey from hematopoietic to leukemic stem cells. Cells. 2022;185(8):1266-70.
- Kapoor-Narula U, Lenka NJC. Cancer stem cells and tumor heterogeneity: Deciphering the role in tumor progression and metastasis. Cytokine. 2022;157:155968.
- De Angelis ML, Francescangeli F, Zeuner AJC. Breast cancer stem cells as drivers of tumor chemoresistance, dormancy and relapse: new challenges and therapeutic opportunities. Cancers (Basel). 2019;11(10):1569.
- Paul R, et al. Cell plasticity, senescence, and quiescence in cancer stem cells: Biological and therapeutic implications. Pharmacol Ther. 2022;231:107985.
- Tang H, et al. miR-200c suppresses stemness and increases cellular sensitivity to trastuzumab in HER2+ breast cancer. J Cell Mol Med. 2019;23(12):8114-27.
- Dittmer A, Dittmer JJC. Carcinoma-associated fibroblasts promote growth of Sox2-expressing breast cancer cells. Cancers (Basel). 2020;12(11):3435.
- Giuli MV, et al. Notch signaling in female cancers: A multifaceted node to overcome drug resistance. Cancer Drug Resist. 2021;4(4):805.
- Mourkioti I, et al. Interplay of Developmental Hippo-Notch Signaling Pathways with the DNA Damage Response in Prostate Cancer. Cells. 2022;11(15):2449.
- Aggarwal V, et al. NOTCH signaling: Journey of an evolutionarily conserved pathway in driving tumor progression and its modulation as a therapeutic target. Crit Rev Oncol Hematol. 2021;164:103403.
- Yan Q, et al. Oncofetal proteins and cancer stem cells. 2022.
- Sun D , et al. The IVF-generated human embryonic microenvironment reverses progestin resistance in endometrial cancer cells by inducing cancer stem cell differentiation. Cancer Lett. 2022;526:311-21.
- Yang Y, et al. Emerging agents that target signaling pathways in cancer stem cells. J Hematol Oncol. 2020;13:1-18.
- Cheng S-W, et al. Lin28B is an oncofetal circulating cancer stem cell-like marker associated with recurrence of hepatocellular carcinoma. PLoS ONE. 2013;8(11):e80053.
- Karsten U, Goletz S. What makes cancer stem cell markers different? Springerplus. 2013;2(1):1-8.
- Liu L, et al. Chemotherapy Induces Breast Cancer Stemness in Association with Dysregulated MonocytosisMCPs Mediate Chemother-apy-Induced Cancer Stemness. Clin Cancer Res. 2018;24(10):2370-82.
- Alhaddad L, Osipov AN, Leonov S. The Molecular and Cellular Strategies of Glioblastoma and Non-Small-Cell Lung Cancer Cells Conferring Radioresistance. Int J Mol Sci. 2022;23(21):13577.
- Bazan, N.G., et al., Multiprong control of glioblastoma multiforme invasiveness: blockade of pro-inflammatory signaling, anti-angiogenesis, and homeostasis restoration. 2021: p. 1-5.
- Angom RS, Mondal SK, Wang F, Madamsetty VS, Wang E, Dutta SK, Gulani Y, Sarabia-Estrada R, Sarkaria JN, Quiñones-Hinojosa A, Mukhopadhyay D. Ablation of neuropilin-1 improves the therapeutic response in conventional drug-resistant glioblastoma multiforme. Oncogene. 2020;39(48):7114-26. https://doi.org/10.1038/ s41388-020-01462-1.
- Bazan NG, Reid MM, Flores VAC, Gallo JE, Lewis W, Belayev L. Multiprong control of glioblastoma multiforme invasiveness: blockade of pro-inflammatory signaling, anti-angiogenesis, and homeostasis restoration. Cancer Metastasis Rev. 2021;40(3):643-7. https://doi.org/ 10.1007/s10555-021-09987-X.
- Douyère M, Chastagner P, Douyère M. Chastagner P, Boura, Neuropi-lin-1: a key protein to consider in the progression of pediatric brain tumors. Front Oncol. 2021;11:665634.
- Zhang Q, et al. miR34a/GOLPH3 axis abrogates urothelial bladder cancer chemoresistance via reduced cancer stemness. Theranostics. 2017;7(19):4777.
- Allegra A, et al. The cancer stem cell hypothesis: a guide to potential molecular targets. Cancer Invest. 2014;32(9):470-95.
- Castelli V, et al. The great escape: the power of cancer stem cells to evade programmed cell death. Cancers (Basel). 2021;13(2):328.
- Pan E, et al. Phase I study of RO4929097 with bevacizumab in patients with recurrent malignant glioma. J Neurooncol. 2016;130:571-9.
- Villalobos VM, et al. Long-term follow-up of desmoid fibromatosis treated with PF-03084014, an oral gamma secretase inhibitor. Ann Surg Oncol. 2018;25:768-75.
- Kimura K, et al. Safety, tolerability, and preliminary efficacy of the antifibrotic small molecule PRI-724, a CBP/
-catenin inhibitor, in patients with hepatitis C virus-related cirrhosis: a single-center, open-label, dose escalation phase 1 trial. EBioMedicine. 2017;23:79-87. - Dréno
, et al. Two intermittent vismodegib dosing regimens in patients with multiple basal-cell carcinomas (MIKIE): a randomised, regimen-controlled, double-blind, phase 2 trial. Lancet Oncol. 2017;18(3):404-12. - Shibue T, Weinberg RA. EMT, CSCs, and drug resistance: the mechanistic link and clinical implications. Nat Rev Clin Oncol. 2017;14(10):611-29.
- Khan SU, Fatima K, Malik F. Understanding the cell survival mechanism of anoikis-resistant cancer cells during different steps of metastasis. Clin Exp Metastasis. 2022;39(5):715-26. https://doi.org/10.1007/ s10585-022-10172-9.
- Shibata M, Hoque MOJC. Targeting cancer stem cells: a strategy for effective eradication of cancer. Cancers (Basel). 2019;11(5):732.
- Zinzi L, et al. ABC transporters in CSCs membranes as a novel target for treating tumor relapse. Front Pharmacol. 2014;5:163.
- Zhao
, et al. Eradication of cancer stem cells in triple negative breast cancer using doxorubicin/pluronic polymeric micelles. Nanomedicine. 2020;24:102124. - Kim EJ, et al. NRF2 knockdown resensitizes 5-fluorouracil-resistant pancreatic cancer cells by suppressing HO-1 and ABCG2 expression. Int J Mol Sci. 2020;21(13):4646.
- Eldaly, S.M., S.A. Gouhar, and M.T.J.E.J.o.C. Abo-elfadl, The Influence of 5-Fluorouracil on Drug Transporters is a Dose-Dependent Effect Mediated by Altered Expression of miRNAs. 2022. 65(8): p. 737-748.
- Kukal S, Guin D, Rawat C, Bora S, Mishra MK, Sharma P, Paul PR, Kanojia N, Grewal GK, Kukreti S, Saso L, Kukreti R. Multidrug efflux transporter ABCG2: expression and regulation. Cell Mol Life Sci. 2021;78(21-22):6887-939. https://doi.org/10.1007/s00018-021-03901-y.
- Hsu HH, Chen MC, Baskaran R, Lin YM, Day CH, Lin YJ, Tu CC, Vijaya Padma V, Kuo WW, Huang CY. Oxaliplatin resistance in colorectal cancer cells is mediated via activation of ABCG2 to alleviate ER stress induced apoptosis. J Cell Physiol. 2018;233(7):5458-67. https://doi.org/10.1002/jcp.26406.
- Ravindranath AK , et al. CD44 promotes multi-drug resistance by protecting P-glycoprotein from FBXO21-mediated ubiquitination. Oncotarget. 2015;6(28):26308.
- Zhang H, et al. Cancer stem cells, epithelial-mesenchymal transition, ATP and their roles in drug resistance in cancer. Cancer Drug Resist. 2021;4(3):684.
- Lv Y, et al. Hypoxia-inducible factor-1 a induces multidrug resistance protein in colon cancer. Onco Targets Ther. 2015;8:1941.
- Nedeljković M, Damjanović A. Mechanisms of chemotherapy resistance in triple-negative breast cancer-how we can rise to the challenge. Cells. 2019;8(9):957.
- Kim H, et al. The hypoxic tumor microenvironment in vivo selects the cancer stem cell fate of breast cancer cells. Breast Cancer Res. 2018;20(1):1-15.
- Jing
, et al. Role of hypoxia in cancer therapy by regulating the tumor microenvironment. Mol Cancer. 2019;18:1-15. - Khan, S.U., K. Fatima, and F. Malik, Understanding the cell survival mechanism of anoikis-resistant cancer cells during different steps of metastasis. Clin Exp Metastasis, 2022.
- Khan SU, et al. Activation of lysosomal mediated cell death in the course of autophagy by mTORC1 inhibitor. Sci Rep. 2022;12(1):1-13.
- Wani A, et al. Crocetin promotes clearance of amyloid-
by inducing autophagy via the STK11/LKB1-mediated AMPK pathway. Autophagy. 2021;17(11):3813-32. - Han Y, et al. Role of autophagy in breast cancer and breast cancer stem cells. Int J Oncol. 2018;52(4):1057-70.
- Gong C, et al. Beclin 1 and autophagy are required for the tumorigenicity of breast cancer stem-like/progenitor cells. Oncogene. 2013;32(18):2261-72.
- Pagotto A, et al. Autophagy inhibition reduces chemoresistance and tumorigenic potential of human ovarian cancer stem cells. Cell Death Dis. 2017;8(7):e2943-e2943.
- Liu W, et al. PRDX1 activates autophagy via the PTEN-AKT signaling pathway to protect against cisplatin-induced spiral ganglion neuron damage. Autophagy. 2021;17(12):4159-81.
- Khan SU, Rayees S, Sharma P, Malik F. Targeting redox regulation and autophagy systems in cancer stem cells. Clin Exp Med. 2023;23(5):140523. https://doi.org/10.1007/s10238-022-00955-5.
- Raha D, et al. The Cancer Stem Cell Marker Aldehyde Dehydrogenase Is Required to Maintain a Drug-Tolerant Tumor Cell SubpopulationAldehyde Dehydrogenase Contributes to Cancer Drug Resistance. Cancer Res. 2014;74(13):3579-90.
- Wu W, et al. Aldehyde dehydrogenase 1A3 (ALDH1A3) is regulated by autophagy in human glioblastoma cells. Cancer Lett. 2018;417:112-23.
- Yeo SK, et al. Autophagy Differentially Regulates Distinct Breast Cancer Stem-like Cells in Murine Models via EGFR/Stat3 and Tgfß/Smad SignalingRegulation of Distinct Breast Cancer Stem Cells by Autophagy. Cancer Res. 2016;76(11):3397-410.
- Shih P-C, Mei K. Role of STAT3 signaling transduction pathways in cancer stem cell-associated chemoresistance. Drug Discov Today. 2021;26(6):1450-8.
- Jin, M L, Jeong K W, Histone modifications in drug-resistant cancers: From a cancer stem cell and immune evasion perspective. 2023: 1-15.
- Zeng F, et al. Comprehensive profiling identifies a novel signature with robust predictive value and reveals the potential drug resistance mechanism in glioma. Cell Commun Signal. 2020;18:1-13.
- Chung AW, et al. Tocilizumab overcomes chemotherapy resistance in mesenchymal stem-like breast cancer by negating autocrine IL-1A induction of IL-6. NPJ Breast Cancer. 2022;8(1):1-10.
- Dianat-Moghadam H, et al. Cancer stem cells-emanated therapy resistance: implications for liposomal drug delivery systems. J Control Release. 2018;288:62-83.
- Codony-Servat J, Rosell R. Cancer stem cells and immunoresistance: clinical implications and solutions. Transl Lung Cancer Res. 2015;4(6):689-703. https://doi.org/10.3978/j.issn.2218-6751.2015.12.11.
- Najafi M. Mortezaee K, Majidpoor J, Cancer stem cell (CSC) resistance drivers. Life Sci. 2019;234:116781.
- Walker ND, et al. Exosomes from differentially activated macrophages influence dormancy or resurgence of breast cancer cells within bone marrow stroma. Cell Death Dis. 2019;10(2):59.
- Müller L, et al. Bidirectional crosstalk between cancer stem cells and immune cell subsets. Front Immunol. 2020;11:140.
- The role of stem cells in small-cell lung cancer: Evidence from chemoresistance to immunotherapy Seminars in Cancer Biology. 2022; 87160-169. https://doi.org/10.1016/j.semcancer.2022.11.006.
- Balaji S, et al. Emerging role of tumor microenvironment derived exosomes in therapeutic resistance and metastasis through epithelial-to-mesenchymal transition. Life Sci. 2021;280:119750.
- Luo S, et al. Macrophages Are a Double-Edged Sword: Molecular Crosstalk between Tumor-Associated Macrophages and Cancer Stem Cells. Biomolecules. 2022;12(6):850.
- Park SY, et al. Interleukin-8 is related to poor chemotherapeutic response and tumourigenicity in hepatocellular carcinoma. Eur J Cancer. 2014;50(2):341-50.
- Lei, M.M.L. and T.K.W. Lee, Cancer stem cells: Emerging key players in immune evasion of cancers. Frontiers in Cell and Developmental Biology, 2021: p. 1643.
- Lei Q, et al. Resistance mechanisms of anti-PD1/PDL1 therapy in solid tumors. Front Cell Dev Biol. 2020;8:672.
- Dianat-Moghadam H, et al. Immune evader cancer stem cells direct the perspective approaches to cancer immunotherapy. Stem Cell Res Ther. 2022;13(1):1-12.
- Lei MML, Lee TKW. Cancer Stem Cells: Emerging Key Players in Immune Evasion of Cancers. Front Cell Dev Biol. 2021;9:692940. https://doi.org/ 10.3389/fcell.2021.692940.
- Lee TKW, et al. Blockade of CD47-mediated cathepsin S/protease-activated receptor 2 signaling provides a therapeutic target for hepatocellular carcinoma. Hepatology. 2014;60(1):179-91.
- Nigro A, et al. Enhanced expression of CD47 is associated with offtarget resistance to tyrosine kinase inhibitor gefitinib in NSCLC. Front Immunol. 2020;10:3135.
- Zhong M , et al. Induction of tolerogenic dendritic cells by activated TGF-
Akt/Smad2 signaling in RIG-I-deficient stemness-high human liver cancer cells. BMC Cancer. 2019;19(1):1-15. - Ma Y, et al. Dendritic cells in the cancer microenvironment. J Cancer. 2013;4(1):36.
- Ravindran S, Rasool S, Maccalli CJCM. The cross talk between cancer stem cells/cancer initiating cells and tumor microenvironment: the missing piece of the puzzle for the efficient targeting of these cells with immunotherapy. Cancer Microenviron. 2019;12(2):133-48.
- Dianat-Moghadam H, Sharifi M, Salehi R, Keshavarz M, Shahgolzari M, Amoozgar Z. Engaging stemness improves cancer immunotherapy. Cancer Lett. 2023;554:216007. https://doi.org/10.1016/j.canlet.2022. 216007.
- Solis-Castillo LA, et al. Tumor-infiltrating regulatory T cells, CD8/Treg ratio, and cancer stem cells are correlated with lymph node metastasis in patients with early breast cancer. Breast Cancer. 2020;27(5):837-49.
- Komura N, et al. The role of myeloid-derived suppressor cells in increasing cancer stem-like cells and promoting PD-L1 expression in epithelial ovarian cancer. Cancer Immunol Immunother. 2020;69(12):2477-99.
- Tomić S, et al. Prostaglanin-E2 potentiates the suppressive functions of human mononuclear myeloid-derived suppressor cells and increases their capacity to expand IL-10-producing regulatory T cell subsets. Front Immunol. 2019;10:475.
- Wu B, et al. Cross-talk between cancer stem cells and immune cells: potential therapeutic targets in the tumor immune microenvironment. Mol Cancer. 2023;22(1):1-22.
- Chiu DK-C, et al. Hypoxia inducible factor HIF-1 promotes myeloidderived suppressor cells accumulation through ENTPD2/CD39L1 in hepatocellular carcinoma. Nat Commun. 2017;8(1):1-12.
- Xu M, et al. Interactions between interleukin-6 and myeloid-derived suppressor cells drive the chemoresistant phenotype of hepatocellular cancer. Exp Cell Res. 2017;351(2):142-9.
- Gardner RA, et al. Intent-to-treat leukemia remission by CD19 CART cells of defined formulation and dose in children and young adults. Exp Cell Res. 2017;129(25):3322-31.
- Maude SL, et al. Tisagenlecleucel in children and young adults with B-cell lymphoblastic leukemia. N Engl J Med. 2018;378(5):439-48.
- Fry TJ, et al. CD22-targeted CAR T cells induce remission in B-ALL that is naive or resistant to CD19-targeted CAR immunotherapy. Nat Med. 2018;24(1):20-8.
- Lee DW, et al. T cells expressing CD19 chimeric antigen receptors for acute lymphoblastic leukaemia in children and young adults: a phase 1 dose-escalation trial. Lancet. 2015;385(9967):517-28.
- Al-Haideri M, et al. CAR-T cell combination therapy: the next revolution in cancer treatment. Cancer Cell Int. 2022;22(1):1-26.
- Bozorgi A, Bozorgi M, Khazaei M. Immunotherapy and immunoengineering for breast cancer; a comprehensive insight into CAR-T cell therapy advancements, challenges and prospects. Cell Oncol (Dordr). 2022;45(5):755-77.
- Sterner RC, Sterner RM. CAR-T cell therapy: current limitations and potential strategies. Blood Cancer J. 2021;11(4):69. https://doi.org/10. 1038/s41408-021-00459-7.
- Scarfò I, Maus MV. Current approaches to increase CAR T cell potency in solid tumors: targeting the tumor microenvironment. J Immunother Cancer. 2017;5(1):28. https://doi.org/10.1186/s40425-017-0230-9.
- Du B, Shim JS. Targeting epithelial-mesenchymal transition (EMT) to overcome drug resistance in cancer. Molecules. 2016;21(7):965.
- Ribatti D, Tamma R, Annese T. Epithelial-mesenchymal transition in cancer: a historical overview. Transl Oncol. 2020;13(6):100773.
- Yang J, et al. Guidelines and definitions for research on epithelial-mesenchymal transition. Nat Rev Mol Cell Biol. 2020;21(6):341-52.
- Cevatemre B, et al. Pyruvate dehydrogenase contributes to drug resistance of lung cancer cells through epithelial mesenchymal transition. Front Cell Dev Biol. 2022;9:738916.
- Das V, et al. The basics of epithelial-mesenchymal transition (EMT): A study from a structure, dynamics, and functional perspective. J Cell Physiol. 2019;234(9):14535-55.
- Lu J, et al. ZEB1: catalyst of immune escape during tumor metastasis. Biomed Pharmacother. 2022;153:113490.
- Funato N, Yanagisawa H. TBX1 targets the miR-200-ZEB2 axis to induce epithelial differentiation and inhibit stem cell properties. Sci Rep. 2022;12(1):20188.
- Bencivenga M, Decimo I, Malpeli G. A therapeutic perspective for proliferative vitreoretinopathy based on the inhibition of epithelialmesenchymal transition by miR-194. Ann Transl Med. 2020;8(8):525. https://doi.org/10.21037/atm.2020.03.181.
- Title AC, et al. The miR-200-Zeb1 axis regulates key aspects of
-cell function and survival in vivo. Mol Metab. 2021;53:101267. - Dai Y, et al. Copy number gain of ZEB1 mediates a double-negative feedback loop with miR-33a-5p that regulates EMT and bone metastasis of prostate cancer dependent on TGF-
signaling. Theranostics. 2019;9(21):6063. - Li, D., et al., Heterogeneity and plasticity of epithelial-mesenchymal transition (EMT) in cancer metastasis: Focusing on partial EMT and regulatory mechanisms. 2023: p. e13423.
- Meidhof
, et al. ZEB 1 -associated drug resistance in cancer cells is reversed by the class I HDAC inhibitor mocetinostat. EMBO Mol Med. 2015;7(6):831-47. - Zhang
, et al. ZEB1 confers chemotherapeutic resistance to breast cancer by activating ATM. Cell Death Dis. 2018;9(2):1-15. - Xie SL, et al. SOX8 regulates cancer stem-like properties and cisplatininduced EMT in tongue squamous cell carcinoma by acting on the Wnt/
-catenin pathway. Int J Cancer. 2018;142(6):1252-65. - Liang Y, et al. Epigenetic activation of TWIST1 by MTDH promotes cancer stem-like cell traits in breast cancer. Cancer Res. 2015;75(17):3672-80.
- Mukherjee P, et al. Modulation of SOX2 expression delineates an endpoint for paclitaxel-effectiveness in breast cancer stem cells. Sci Rep. 2017;7(1):1-16.
- Li Q, et al. Subcellular drug distribution: mechanisms and roles in drug efficacy, toxicity, resistance, and targeted delivery. Drug Metab Rev. 2018;50(4):430-47.
- Sakhrani NM, Padh H. Organelle targeting: third level of drug targeting. Drug Des Devel Ther. 2013;7:585.
- Boedtkjer E, Pedersen SF. The acidic tumor microenvironment as a driver of cancer. Annu Rev Physiol. 2020;82:103-26.
- Audero MM, Prevarskaya N, Pla A. F, Ca2+ Signalling and Hypoxia/Acidic Tumour Microenvironment Interplay in Tumour Progression. Int J Mol Sci. 2022;23(13):7377.
- Goswami KK, et al. Lactic acid in alternative polarization and function of macrophages in tumor microenvironment. Hum Immunol. 2022;83(5):409-17.
- Kato Y, et al. Acidic extracellular microenvironment and cancer. Cancer Cell Int. 2013;13(1):1-8.
- Pilon-Thomas S, et al. Neutralization of tumor acidity improves antitumor responses to immunotherapy. Cancer Res. 2016;76(6):1381-90.
- Guo Y, et al. Mechanisms of chemotherapeutic resistance and the application of targeted nanoparticles for enhanced chemotherapy in colorectal cancer. J Nanobiotechnology. 2022;20(1):1-24.
- Dhar G, Sen S, Chaudhuri G. Acid Gradient Across Plasma Membrane can Drive Phosphate-Bond Synthesis in Cancer Cells: Acidic Tumor Milieu can Act as a Potential Energy Source. PLoS ONE. 2015;29:725.28.
- Dhar G, Sen S, Chaudhuri GJPO. Acid gradient across plasma membrane can drive phosphate bond synthesis in cancer cells: acidic tumor milieu as a potential energy source. PLoS ONE. 2015;10(4):e0124070.
- Halcrow PW, et al. Overcoming chemoresistance: Altering pH of cellular compartments by chloroquine and hydroxychloroquine. Front Cell Dev Biol. 2021;9:627639.
- Pérez-Tomás R, Pérez-Guillén IJC. Lactate in the tumor microenvironment: An essential molecule in cancer progression and treatment. Cancers (Basel). 2020;12(11):3244.
- Xiang Y-L, et al. Zwitterionic meso-silica/polypeptide hybrid nanoparticles for efficient azithromycin delivery and photodynamic therapy for synergistic treatment of drug-resistant bacterial infection. Int J Biol Macromol. 2022;219:597-610.
- Hulikova A. Swietach P, Nuclear proton dynamics and interactions with calcium signaling. J Mol Cell Cardiol. 2016;96:26-37.
- Ji X, et al. Chemoresistance mechanisms of breast cancer and their countermeasures. Biomed Pharmacother. 2019;114:108800.
- Yuan D, et al. Interruption of endolysosomal trafficking leads to stroke brain injury. Exp Neurol. 2021;345:113827.
- Polanco JC, et al. Exosomes induce endolysosomal permeabilization as a gateway by which exosomal tau seeds escape into the cytosol. Acta Neuropathol. 2021;141:235-56.
- McGuire C, et al. Regulation of V-ATPase activity. Front Biosci (Landmark Ed). 2017;22(4):609-22.
- Webb BA, et al. pHLARE: a new biosensor reveals decreased lysosome pH in cancer cells. Mol Biol Cell. 2021;32(2):131-42.
- Tang T, Yang ZY, Wang D, Yang XY, Wang J, Li L, Wen Q, Gao L, Bian XW, Yu SC. The role of lysosomes in cancer development and progression. Cell Biosci. 2020;10(1):131. https://doi.org/10.1186/s13578-020-00489-x.
- Zhitomirsky B, Assaraf RK. Lysosomes as mediators of drug resistance in cancer. Drug Resist Updat. 2016;24:23-33.
- Geisslinger F, et al. Targeting lysosomes in cancer as promising strategy to overcome chemoresistance-a mini review. Front Oncol. 2020;10:1156.
- Hraběta J, et al. Drug sequestration in lysosomes as one of the mechanisms of chemoresistance of cancer cells and the possibilities of its inhibition. Int J Mol Sci. 2020;21(12):4392.
- Al-Akra L, et al. Tumor stressors induce two mechanisms of intracellular P-glycoprotein-mediated resistance that are overcome by lysosomaltargeted thiosemicarbazones. J Biol Chem. 2018;293(10):3562-87.
- Fu Z, et al. CQ sensitizes human pancreatic cancer cells to gemcitabine through the lysosomal apoptotic pathway via reactive oxygen species. Mol Oncol. 2018;12(4):529-44.
- Sharapova TN, et al. FasL on the surface of Tag7 (PGRP-S)-activated lymphocytes induces necroptosis in HLA-negative tumor cells with the involvement of lysosomes and mitochondria. Biochimie. 2018;152:174-80.
- Seebacher NA, et al. A mechanism for overcoming P-glycoproteinmediated drug resistance: novel combination therapy that releases stored doxorubicin from lysosomes via lysosomal permeabilization using Dp44mT or DpC. Cell Death Dis. 2016;7(12):e2510-e2510.
- Salaroglio IC, et al. Increasing intratumor C/EBP-
LIP and nitric oxide levels overcome resistance to doxorubicin in triple negative breast cancer. J Exp Clin Cancer Res. 2018;37(1):1-20. - Champagne DP, et al. Fine-tuning of CD8+ T cell mitochondrial metabolism by the respiratory chain repressor MCJ dictates protection to influenza virus. Immunity. 2016;44(6):1299-311.
- Barbier-Torres
, et al. The mitochondrial negative regulator MCJ is a therapeutic target for acetaminophen-induced liver injury. Nat Commun. 2017;8(1):1-11. - Hatle KM, et al. MCJ/DnaJC15, an endogenous mitochondrial repressor of the respiratory chain that controls metabolic alterations. Mol Cell Biol. 2013;33(11):2302-14.
- Giddings EL, et al. Mitochondrial ATP fuels ABC transporter-mediated drug efflux in cancer chemoresistance. Nat Commun. 2021;12(1):2804.
- Miglietta S. Deciphering the role of the mitochondrial chaperonine MCJ in ovarian cancer. 2021.
- Shubin AV, et al. Cytoplasmic vacuolization in cell death and survival. Oncotarget. 2016;7(34):55863.
- Lin H, et al. Overcoming Taxol-resistance in A549 cells: A comprehensive strategy of targeting P-gp transporter, AKT/ERK pathways, and cytochrome P450 enzyme CYP1B1 by 4-hydroxyemodin. Biochem Pharmacol. 2020;171:113733.
- Belisario DC, et al. Hypoxia dictates metabolic rewiring of tumors: implications for chemoresistance. Cells. 2020;9(12):2598.
- Liang W, et al. Role of reactive oxygen species in tumors based on the ‘seed and soil’theory: A complex interaction. Oncol Rep. 2021;46(3):1-15.
- Pascual-Ahuir A, Manzanares-Estreder S, Proft M. Pro- and Antioxidant Functions of the Peroxisome-Mitochondria Connection and Its Impact on Aging and Disease. Oxid Med Cell Longev. 2017;2017:9860841. https://doi.org/10.1155/2017/9860841.
- Jacqueroux E , et al. Value of quantifying ABC transporters by mass spectrometry and impact on in vitro-to-in vivo prediction of transportermediated drug-drug interactions of rivaroxaban. Eur J Pharm Biopharm. 2020;148:27-37.
- Dang Y, et al. P-Gp and TOPO II Expression and Their Clinical Significance in Colon Cancer. Ann Clin Lab Sci. 2020;50(5):584-90.
- Heming CP, et al. P-glycoprotein and cancer: what do we currently know? 2022.
- Mirzaei S, Gholami MH, Hashemi F, Zabolian A, Farahani MV, Hushmandi K, Zarrabi A, Goldman A, Ashrafizadeh M, Orive G. Advances in understanding the role of P-gp in doxorubicin resistance: Molecular pathways, therapeutic strategies, and prospects. Drug Discov Today. 2022;27(2):436-55. https://doi.org/10.1016/j.drudis.2021.09.020.
- Khalaf K, et al. Aspects of the tumor microenvironment involved in immune resistance and drug resistance. Front Immunol. 2021;12:656364.
- Xu M, et al. Targeting the tumor stroma for cancer therapy. Mol Cancer. 2022;21(1):208.
- Winkler J, Abisoye-Ogunniyan A, Metcalf KJ, Werb Z. Concepts of extracellular matrix remodelling in tumour progression and metastasis. Nat Commun. 2020;11(1):5120. https://doi.org/10.1038/ s41467-020-18794-X.
- Hu J, et al. CAFs secreted exosomes promote metastasis and chemotherapy resistance by enhancing cell stemness and epithelial-mesenchymal transition in colorectal cancer. Mol Cance. 2019;18(1):1-15.
- Jena BC, et al. Cancer associated fibroblast mediated chemoresistance: A paradigm shift in understanding the mechanism of tumor progression. Biochim Biophys Acta Rev Cancer. 2020;1874(2):188416.
- Haider T, et al. Drug resistance in cancer: mechanisms and tackling strategies. Pharmacol Rep. 2020;72(5):1125-51.
- Guo Z, Ashrafizadeh M, Zhang W, Zou R, Sethi G, Zhang X. Molecular profile of metastasis, cell plasticity and EMT in pancreatic cancer: a pre-clinical connection to aggressiveness and drug resistance. Cancer Metastasis Rev. 2023. https://doi.org/10.1007/s10555-023-10125-y.
- Che Y, et al. Cisplatin-activated PAI-1 secretion in the cancer-associated fibroblasts with paracrine effects promoting esophageal squamous cell carcinoma progression and causing chemoresistance. Cell Death Dis. 2018;9(7):1-13.
- Luraghi P, et al. MET Signaling in Colon Cancer Stem-like Cells Blunts the Therapeutic Response to EGFR InhibitorsMET in Colon Cancer-Initiating Cells. Can Res. 2014;74(6):1857-69.
- Fiori ME, et al. Cancer-associated fibroblasts as abettors of tumor progression at the crossroads of EMT and therapy resistance. Mol Cancer. 2019;18(1):1-16.
- Hamidi H, Ivaska J. Every step of the way: integrins in cancer progression and metastasis. Nat Rev Cancer. 2018;18(9):533-48.
- Fang Z, Meng Q, Xu J, Wang W, Zhang B, Liu J, Liang C, Hua J, Zhao Y, Yu X, Shi S. Signaling pathways in cancer-associated fibroblasts: recent advances and future perspectives. Cancer Commun (Lond). 2023;43(1):3-41. https://doi.org/10.1002/cac2.12392.
- Martino MM, et al. Growth factors engineered for super-affinity to the extracellular matrix enhance tissue healing. Science. 2014;343(6173):885-8.
- Walker C, Mojares E, del Río Hernández A. Role of extracellular matrix in development and cancer progression. Int J Mol Sci. 2018;19(10):3028.
- Kaemmerer E, Loessner D, Avery VM. Addressing the tumour microenvironment in early drug discovery: a strategy to overcome drug resistance and identify novel targets for cancer therapy. Drug Discov Today. 2021;26(3):663-76.
- Teng PN, et al. Identification of candidate circulating cisplatin-resistant biomarkers from epithelial ovarian carcinoma cell secretomes. Br J Cancer. 2014;110(1):123-32.
- Wu Y-H, et al. COL11A1 confers chemoresistance on ovarian cancer cells through the activation of Akt/c/EBP
pathway and PDK1 stabilization. Oncotarget. 2015;6(27):23748. - Cooper J, Giancotti FG. Integrin Signaling in Cancer: Mechanotransduction, Stemness, Epithelial Plasticity, and Therapeutic Resistance. Cancer Cell. 2019;35(3):347-67. https://doi.org/10.1016/j.ccell.2019.01.007.
- Murphy JM, Rodriguez YAR, Jeong K, Ahn EE, Lim SS. Targeting focal adhesion kinase in cancer cells and the tumor microenvironment. Exp Mol Med. 2020;52(6):877-86. https://doi.org/10.1038/ s12276-020-0447-4.
- Senthebane DA, et al. The role of tumor microenvironment in chemoresistance: 3D extracellular matrices as accomplices. Int J Mol Sci. 2018;19(10):2861.
- Hong Y, Rao Y. Current status of nanoscale drug delivery systems for colorectal cancer liver metastasis. Biomed Pharmacother. 2019;114:108764. https://doi.org/10.1016/j.biopha.2019.108764.
- Mashouri L, Yousefi H, Aref AR, Ahadi AM, Molaei F, Alahari SK. Exosomes: Composition, Biogenesis, and Mechanisms in Cancer Metastasis and Drug Resistance. Mol Cancer. 2019;18(1):75.
- Yang E, et al. Exosome-mediated metabolic reprogramming: The emerging role in tumor microenvironment remodeling and its influence on cancer progression. Signal Transduct Target Ther. 2020;5(1):1-13.
- Dianat-Moghadam H , et al. The role of circulating tumor cells in the metastatic cascade: biology, technical challenges, and clinical relevance. Cancers. 2020;12(4):867.
- Li S, Yi M, Dong B, Jiao Y, Luo S, Wu K. The roles of exosomes in cancer drug resistance and its therapeutic application. Clin Transl Med. 2020;10(8):e257. https://doi.org/10.1002/ctm2.257.
- Steinbichler TB, Dudás J, Skvortsov S, Ganswindt U, Riechelmann H, Skvortsova II. Therapy resistance mediated by exosomes. Mol Cancer. 2019;18(1):58. https://doi.org/10.1186/s12943-019-0970-X.
- Ji R, et al. Exosomes derived from human mesenchymal stem cells confer drug resistance in gastric cancer. Cell Cycle. 2015;14(15):2473-83.
- Lukanović D, et al. The contribution of copper efflux transporters ATP7A and ATP7B to chemoresistance and personalized medicine in ovarian cancer. Biomed Pharmacother. 2020;129:110401.
- Zaidi
, et al. Quantitative visualization of hypoxia and proliferation gradients within histological tissue sections. Front Bioeng Biotechnol. 2019;7:397. - Singh D, et al. Overexpression of hypoxia-inducible factor and metabolic pathways: possible targets of cancer. Cell Biosci. 2017;7(1):1-9.
- Warfel NA, El-Deiry WS. HIF-1 signaling in drug resistance to chemotherapy. Curr Med Chem. 2014;21(26):3021-8.
- Xia Y, Jiang L, Zhong T. The role of HIF-1 a in chemo-/radioresistant tumors. Onco Targets Ther. 2018;11:3003.
- Nishimoto A, et al. HIF-1a activation under glucose deprivation plays a central role in the acquisition of anti-apoptosis in human colon cancer cells. Int J Oncol. 2014;44(6):2077-84.
- Zhao Q, et al. Enhancement of drug sensitivity by knockdown of HIF-1a in gastric carcinoma cells. Oncol Res. 2016;23(3):129.
- Chen W-L, et al. Cycling hypoxia induces chemoresistance through the activation of reactive oxygen species-mediated B-cell lymphoma extra-long pathway in glioblastoma multiforme. J Transl Med. 2015;13(1):1-13.
- Gao J, Liang Y, Wang L. Shaping polarization of tumor-associated macrophages in cancer immunotherapy. Front Immunol. 2022;13:888713.
- Lei X, et al. Immune cells within the tumor microenvironment: Biological functions and roles in cancer immunotherapy. Cancer Lett. 2020:470:126-33.
- Larionova I, et al. Interaction of tumor-associated macrophages and cancer chemotherapy. Oncoimmunology. 2019;8(7):e1596004.
- Liu Y, Cao X. The origin and function of tumor-associated macrophages. Cell Mol Immunol. 2015;12(1):1-4.
- Moradi-Chaleshtori M , et al. In vitro and in vivo evaluation of antitumoral effect of M1 phenotype induction in macrophages by miR-130 and miR-33 containing exosomes. Cancer Immunol Immunother. 2021;70(5):1323-39.
- Yu S, et al. Activated HIF1a of tumor cells promotes chemoresistance development via recruiting GDF15-producing tumor-associated macrophages in gastric cancer. Cancer Immunol Immunother. 2020;69(10):1973-87.
- Incio J, et al. Obesity-Induced Inflammation and Desmoplasia Promote Pancreatic Cancer Progression and Resistance to Chemotherapy. Cancer Discov. 2016;6(8):852-69.
- Zhang Y, et al. Targeting and exploitation of tumor-associated neutrophils to enhance immunotherapy and drug delivery for cancer treatment. Cancer Biol Med. 2020;17(1):32.
- Pylaeva E, Lang S, Jablonska J. The essential role of type I interferons in differentiation and activation of tumor-associated neutrophils. Front Immunol. 2016;7:629.
- Liu X-D, et al. Resistance to Antiangiogenic Therapy Is Associated with an Immunosuppressive Tumor Microenvironment in Metastatic Renal Cell CarcinomaAntiangiogenic Therapy Increases PD-L1 Expression. Cancer Immunol Res. 2015;3(9):1017-29.
- Wang D, et al. Colorectal cancer cell-derived CCL20 recruits regulatory T cells to promote chemoresistance via FOXO1/CEBPB/NF-kB signaling. J Immunother Cancer. 2019;7(1):215.
- Long Y, et al. Dysregulation of Glutamate Transport Enhances Treg Function That Promotes VEGF Blockade Resistance in Glioblastoma. Cancer Res. 2020;80(3):499-509.
- Desbats MA, et al. Metabolic plasticity in chemotherapy resistance. Front Oncol. 2020;10:281.
- Ragni M, et al. Amino acids in cancer and cachexia: An integrated view. Cancers (Basel). 2022;14(22):5691.
- Phan LM, et al. Cancer metabolic reprogramming: importance, main features, and potentials for precise targeted anti-cancer therapies. Cancer Biol Med. 2014;11(1):1.
- Cao J, et al. Effects of altered glycolysis levels on CD8+ T cell activation and function. Cell Death Dis. 2023;14(7):407.
- Khodaei T, et al. Drug delivery for metabolism targeted cancer immunotherapy. Adv Drug Deliv Rev. 2022;184:114242.
- Baek G, et al. MCT4 defines a glycolytic subtype of pancreatic cancer with poor prognosis and unique metabolic dependencies. Cell Rep. 2014;9(6):2233-49.
- Ruprecht B, et al. Lapatinib resistance in breast cancer cells is accompanied by phosphorylation-mediated reprogramming of glycolysis. Cancer Res. 2017;77(8):1842-53.
- Zhang Y, et al. Targeting Glucose Metabolism Enzymes in Cancer Treatment: Current and Emerging Strategies. Cancers (Basel). 2022;14(19):4568.
- Zhao H, et al. Up-regulation of glycolysis promotes the stemness and EMT phenotypes in gemcitabine-resistant pancreatic cancer cells. J Cell Mol Med. 2017;21(9):2055-67.
- Varghese E, et al. Targeting glucose metabolism to overcome resistance to anticancer chemotherapy in breast cancer. Cancers (Basel). 2020;12(8):2252.
- Krasnov GS, et al. Targeting VDAC-bound hexokinase II: a promising approach for concomitant anti-cancer therapy. Expert Opin Ther Targets. 2013;17(10):1221-33.
- Boison D. Yegutkin G G, Adenosine metabolism: emerging concepts for cancer therapy. Cancer Cell. 2019;36(6):582-96.
- Allard B, et al. The adenosine pathway in immuno-oncology. Nat Rev Clin Oncol. 2020;17(10):611-29.
- Sade-Feldman M, et al. Defining T cell states associated with response to checkpoint immunotherapy in melanoma. Cell. 2019;176(1):404.
- Song Y, et al. Drug-metabolizing cytochrome P450 enzymes have multifarious influences on treatment outcomes. Clin Pharmacokinet. 2021;60:585-601.
- Parashar A, Manoj K. Murburn precepts for cytochrome P450 mediated drug/xenobiotic metabolism and homeostasis. Curr Drug Metab. 2021;22(4):315-26.
- Vredenburg G, den Braver-Sewradj S, van Vugt-Lussenburg BM, Vermeulen NP, Commandeur JN, Vos JC. Activation of the anticancer drugs cyclophosphamide and ifosfamide by cytochrome P450 BM3 mutants. Toxicol Lett. 2015;232(1):182-92. https://doi.org/10.1016/j.toxlet.2014. 11.005.
- Voelcker, G. Enzyme catalyzed decomposition of 4-hydroxycyclophosphamide. in The Open Conference Proceedings Journal. 2017.
- Yang W, et al. Cyclophosphamide Exposure Causes Long-Term Detrimental Effect of Oocytes Developmental Competence Through Affecting the Epigenetic Modification and Maternal Factors’Transcription During Oocyte Growth. Front Cell Dev Biol. 2021;9:682060.
- Bagdasaryan AA, et al. Pharmacogenetics of drug metabolism: the role of gene polymorphism in the regulation of doxorubicin safety and efficacy. Cancers (Basel). 2022;14(21):5436.
- Zahreddine H, Borden KLB. Mechanisms and insights into drug resistance in cancer. Front Pharmacol. 2013,4:28.
- Arwanih, E.Y., et al., Resistance mechanism of acute myeloid leukemia cells against daunorubicin and cytarabine: a literature review. 2022. 14(12).
- Levin M, et al. Surmounting Cytarabine-resistance in acute myeloblastic leukemia cells and specimens with a synergistic combination of hydroxyurea and azidothymidine. Cell Death Dis. 2019;10(6):390.
- Costea T, et al. Alleviation of multidrug resistance by flavonoid and nonflavonoid compounds in breast, lung, colorectal and prostate cancer. Int J Mol Sci. 2020;21(2):401.
- Varela-López A, et al. An update on the mechanisms related to cell death and toxicity of doxorubicin and the protective role of nutrients. Food Chem Toxicol. 2019;134:110834.
- Guo J, et al. Reprogramming of glutamine metabolism via glutamine synthetase silencing induces cisplatin resistance in A2780 ovarian cancer cells. BMC Cancer. 2021;21(1):1-11.
- Wang SF, Chang YL, Fang WL, Li AF, Chen CF, Yeh TS, Hung GY, Huang KH, Lee HC. Growth differentiation factor 15 induces cisplatin resistance through upregulation of xCT expression and glutathione synthesis in gastric cancer. Cancer Sci. 2023;114(8):3301-17. https://doi.org/10. 1111/cas. 15869.
- Housman G, et al. Drug resistance in cancer: an overview. Cancers (Basel). 2014;6(3):1769-92.
- To C, et al. An allosteric inhibitor against the therapy-resistant mutant forms of EGFR in non-small cell lung cancer. Nat Cancer. 2022;3(4):402-17.
- Wu PS, Lin MH, Hsiao JC, Lin PY, Pan SH, Chen YJ. EGFR-T790M MutationDerived Interactome Rerouted EGFR Translocation Contributing to Gefitinib Resistance in Non-Small Cell Lung Cancer. Mol Cell Proteomics. 2023;22(9):100624. https://doi.org/10.1016/j.mcpro.2023.100624.
- Chong CR, Jänne PA. The quest to overcome resistance to EGFR-targeted therapies in cancer. Nat Med. 2013;19(11):1389-400.
- Schulte RR, Ho RH. Organic anion transporting polypeptides: emerging roles in cancer pharmacology. Mol Pharmacol. 2019;95(5):490-506.
- Brecht K, Schäfer AM, Meyer zu Schwabedissen HE. Uptake Transporters of the SLC21, SLC22A, and SLC15A Families in Anticancer TherapyModulators of Cellular Entry or Pharmacokinetics? Cancers (Basel). 2020;12(8):2263.
- Klein I, et al. Glycyrrhizic Acid Prevents Paclitaxel-Induced Neuropathy via Inhibition of OATP-Mediated Neuronal Uptake. Cells. 2023;12(9):1249.
- Zhao D, Long X, Wang J. Transporter-mediated drug-drug interactions involving poly (ADP-ribose) polymerase inhibitors. Oncol Lett. 2023;25(4):1-8.
- Zhou S, Zeng S, Shu Y. Drug-drug interactions at organic cation transporter 1. Front Pharmacol. 2021;12:628705.
- Lin L, Yee SW, Kim RB, Giacomini KM. SLC transporters as therapeutic targets: emerging opportunities. Nat Rev Drug Discov. 2015;14(8):54360. https://doi.org/10.1038/nrd4626.
- Siemer
, et al. Profiling cisplatin resistance in head and neck cancer: A critical role of the VRAC ion channel for chemoresistance. Cancers (Basel). 2021;13(19):4831. - Cabral LKD, Tiribelli C, Sukowati CHJC. Sorafenib resistance in hepatocellular carcinoma: the relevance of genetic heterogeneity. Cancers (Basel). 2020;12(6):1576.
- Otter M , et al. Expression and functional contribution of different organic cation transporters to the cellular uptake of doxorubicin into human breast cancer and cardiac tissue. Int J Mol Sci. 2021;23(1):255.
- Edemir B. Identification of Prognostic Organic Cation and Anion Transporters in Different Cancer Entities by In Silico Analysis. Int J Mol Sci. 2020;21(12):4491. https://doi.org/10.3390/ijms21124491.
- Marin JJ, et al. The role of reduced intracellular concentrations of active drugs in the lack of response to anticancer chemotherapy. Acta Pharmacol Sin. 2014;35(1):1-10.
- Ortiz M, et al. Mechanisms of chemotherapy resistance in ovarian cancer. Cancer Drug Resist. 2022;5(2):304.
- Arnesano F, Natile G. Interference between copper transport systems and platinum drugs. Semin Cancer Biol. 2021;76:173-88. https://doi. org/10.1016/j.semcancer.2021.05.023.
- De Luca A, et al. Copper homeostasis as target of both consolidated and innovative strategies of anti-tumor therapy. J Trace Elem Med Biol. 2019;55:204-13.
- Shiqun, Shao Jingxing, Si Youqing, Shen. Copper as the Target for Anticancer Nanomedicine. Abstr Adv Ther. 2019;2(5):10.1002/adtp.v2.5. https://doi.org/10.1002/adtp. 201800147.
- Mohammad IS, et al. Understanding of human ATP binding cassette superfamily and novel multidrug resistance modulators to overcome MDR. Biomed Pharmacother. 2018;100:335-48.
- Wu Z-X, et al. Elevated ABCB1 expression confers acquired resistance to aurora kinase inhibitor GSK-1070916 in cancer cells. Front Pharmacol. 2021;11:615824.
- Martins-Gomes C, Silva AM. Natural Products as a Tool to Modulate the Activity and Expression of Multidrug Resistance Proteins of Intestinal Barrier. J Xenobiot. 2023;13(2):172-92.
- Barnett-Griness O, Rennert G, Lejbkowicz F, Pinchev M, Saliba W, Gronich N. Association Between ABCG2, ABCB1, ABCC2 Efflux Transporter Single-Nucleotide Variants and Irinotecan Adverse Effects in Patients With Colorectal Cancer: A Real-Life Study. Clin Pharmacol Ther. 2023;113(3):704-11. https://doi.org/10.1002/cpt.2833.
- Petruzzelli R, Polishchuk RSJC. Activity and trafficking of copper-transporting ATPases in tumor development and defense against platinumbased drugs. Cells. 2019;8(9):1080.
- Zhang B, Burke R. Copper homeostasis and the ubiquitin proteasome system. Metallomics. 2023;15(3):mfad010.
- Petruzzelli R, Polishchuk RS. Activity and Trafficking of Copper-Transporting ATPases in Tumor Development and Defense against PlatinumBased Drugs. Cells. 2019;8(9):1080. https://doi.org/10.3390/cells80910 80.
- Zhang Z, et al. Reversing drug resistance of cisplatin by hsp90 inhibitors in human ovarian cancer cells. Int J Clin Exp Med. 2015;8(5):6687.
- Przystupski, D., et al., The modulatory effect of green tea catechin on drug resistance in human ovarian cancer cells. 2019. 28(5): 657-667.
- Hejmady S, Pradhan R, Alexander A, Agrawal M, Singhvi G, Gorain B, Tiwari S, Kesharwani P, Dubey SK. Recent advances in targeted nanomedicine as promising antitumor therapeutics. Drug Discov Today. 2020;25(12):2227-44. https://doi.org/10.1016/j.drudis.2020.09.031.
- Li X, He S, Ma B. Autophagy and autophagy-related proteins in cancer. Mol Cancer. 2020;19(1):12. https://doi.org/10.1186/s12943-020-1138-4.
- Man SM, Jenkins BJ. Context-dependent functions of pattern recognition receptors in cancer. Nat Rev Cancer. 2022;22(7):397-413.
- Manzoor S, et al. Autophagy: A versatile player in the progression of colorectal cancer and drug resistance. Nat Rev Cancer. 2022;12:924290.
- Marsh T, Debnath JJA. Autophagy suppresses breast cancer metastasis by degrading NBR1. Autophagy. 2020;16(6):1164-5.
- Khan SU, Fatima K, Singh U, Singh PP, Malik F. Small molecule “4ab” induced autophagy and endoplasmic reticulum stress-mediated death of aggressive cancer cells grown under adherent and floating conditions. Med Oncol. 2023;40(4):121. https://doi.org/10.1007/ s12032-023-01963-5.
- Assi M, Kimmelman AC. Impact of context-dependent autophagy states on tumor progression. Nat Cancer. 2023;4(5):596-607. https://doi. org/10.1038/s43018-023-00546-7.
- Dower CM, Wills CA, Frisch SM, Wang HG. Mechanisms and context underlying the role of autophagy in cancer metastasis. Autophagy. 2018;14(7):1110-28. https://doi.org/10.1080/15548627.2018.1450020.
- White E. Autophagy and p53. Cold Spring Harb Perspect Med. 2016;6(4):a026120.
- Levy JMM, Towers CG, Thorburn A. Targeting autophagy in cancer. Nat Rev Cancer. 2017;17(9):528-42.
- Rahman MA, et al. Recent Update and Drug Target in Molecular and Pharmacological Insights into Autophagy Modulation in Cancer Treatment and Future Progress. Cells. 2023;12(3):458.
- Anand SK, et al. Entrenching role of cell cycle checkpoints and autophagy for maintenance of genomic integrity. DNA Repair (Amst). 2020;86:102748.
- Ueno S, Sudo T. Hirasawa A, ATM: Functions of ATM kinase and its relevance to hereditary tumors. Int J Mol Sci. 2022;23(1):523.
- Zhang L-H, et al. Enhanced autophagy reveals vulnerability of P-gp mediated epirubicin resistance in triple negative breast cancer cells. Apoptosis. 2016;21(4):473-88.
- Liu ZR, Song Y, Wan LH, Zhang YY, Zhou LM. Over-expression of miR451a can enhance the sensitivity of breast cancer cells to tamoxifen by regulating
, estrogen receptor a, and autophagy. Life Sci. 2016;149:104-13. https://doi.org/10.1016/j.lfs.2016.02.059. - Lee MH, Koh D, Na H, Ka NL, Kim S, Kim HJ, Hong S, Shin YK, Seong JK, Lee MO. MTA1 is a novel regulator of autophagy that induces tamoxifen resistance in breast cancer cells. Autophagy. 2018;14(5):812-24. https:// doi.org/10.1080/15548627.2017.1388476.
- Liu W, et al. Targeting the WEE1 kinase strengthens the antitumor activity of imatinib via promoting KIT autophagic degradation in gastrointestinal stromal tumors. Gastric Cancer. 2020;23:39-51.
- Liu T, et al. Combination of an autophagy inducer and an autophagy inhibitor: a smarter strategy emerging in cancer therapy. Front Pharmacol. 2020;11:408.
- Avril T, Vauléon E, Chevet E. Endoplasmic reticulum stress signaling and chemotherapy resistance in solid cancers. Oncogenesis. 2017;6(8):e373. https://doi.org/10.1038/oncsis.2017.72.
- Pavlović N, Calitz C, Thanapirom K, Mazza G, Rombouts K, Gerwins P, Heindryckx F. Inhibiting IRE1a-endonuclease activity decreases tumor burden in a mouse model for hepatocellular carcinoma. Elife. 2020;9:e55865. https://doi.org/10.7554/eLife.55865.
- Khaled J, Kopsida M, Lennernäs H, Heindryckx F. Drug Resistance and Endoplasmic Reticulum Stress in Hepatocellular Carcinoma. Cells. 2022;11(4):632. https://doi.org/10.3390/cells11040632.
- Hetz C, Zhang K, Kaufman RJ. Mechanisms, regulation and functions of the unfolded protein response. Nat Rev Mol Cell Biol. 2020;21:421-38. https://doi.org/10.1038/s41580-020-0250-z.
- Khaled J, et al. Drug resistance and endoplasmic reticulum stress in hepatocellular carcinoma. Cells. 2022;11(4):632.
- Wang H, Mi K. Emerging roles of endoplasmic reticulum stress in the cellular plasticity of cancer cells. Front Oncol. 2023;13:1110881.
- Zhang C, et al. HMGB1-induced aberrant autophagy contributes to insulin resistance in granulosa cells in PCOS. FASEB J. 2020;34(7):9563-74.
- Li J, Zhou W, Mao Q, Gao D, Xiong L, Hu X, Zheng Y, Xu X. HMGB1 Promotes Resistance to Doxorubicin in Human Hepatocellular Carcinoma Cells by Inducing Autophagy via the AMPK/mTOR Signaling Pathway. Front Oncol. 2021;11:739145. https://doi.org/10.3389/fonc.2021.739145.
- Yu G, Klionsky DJ. Life and Death Decisions-The Many Faces of Autophagy in Cell Survival and Cell Death. Biomolecules. 2022;12(7):866.
- Gupta R, et al. Autophagy and apoptosis cascade: which is more prominent in neuronal death? Cell Mol Life Sci. 2021;78(24):8001-47.
- Booth LA, Roberts JL, Dent P. The role of cell signaling in the crosstalk between autophagy and apoptosis in the regulation of tumor cell survival in response to sorafenib and neratinib. Semin Cancer Biol. 2020;66:129-39. https://doi.org/10.1016/j.semcancer.2019.10.013.
- Anjum J, Mitra S, Das R, Alam R, Mojumder A, Emran TB, Islam F, Rauf A, Hossain MJ, Aljohani ASM, Abdulmonem WA, Alsharif KF, Alzahrani KJ, Khan H. A renewed concept on the MAPK signaling pathway in
cancers: Polyphenols as a choice of therapeutics. Pharmacol Res. 2022;184:106398. https://doi.org/10.1016/j.phrs.2022.106398. - Zhu S, Cao L, Yu Y, Yang L, Yang M, Liu K, Huang J, Kang R, Livesey KM, Tang D. Inhibiting autophagy potentiates the anticancer activity of IFN1@/IFNa in chronic myeloid leukemia cells. Autophagy. 2013;9(3):317-27. https://doi.org/10.4161/auto.22923.
- Balvers RK, et al. ABT-888 enhances cytotoxic effects of temozolomide independent of MGMT status in serum free cultured glioma cells. J Transl Med. 2015;13(1):1-10.
- Christowitz C, et al. Mechanisms of doxorubicin-induced drug resistance and drug resistant tumour growth in a murine breast tumour model. BMC Cancer. 2019;19(1):757.
- Mei Y, et al. Overexpression of RSK4 reverses doxorubicin resistance in human breast cancer cells via PI3K/AKT signalling pathway. J Biochem. 2020;167(6):603-11.
- AI-Malky HS, AI Harthi SE, Osman A-MM. Major obstacles to doxorubicin therapy: Cardiotoxicity and drug resistance. J Oncol Pharm Pract. 2020;26(2):434-44.
- Ramani S, Park S. HSP27 role in cardioprotection by modulating chemotherapeutic doxorubicin-induced cell death. J Mol Med (Berl). 2021;99(6):771-84.
- Panneerpandian P, Rao DB, Ganesan K. Calcium channel blockers lercanidipine and amlodipine inhibit YY1/ERK/TGF-
mediated transcription and sensitize the gastric cancer cells to doxorubicin. Toxicol In Vitro. 2021;74:105152. - Kang
, et al. DUSP4 promotes doxorubicin resistance in gastric cancer through epithelial-mesenchymal transition. Oncotarget. 2017;8(55):94028. - Lee H-J, Choi C-H. Characterization of SN38-resistant T47D breast cancer cell sublines overexpressing BCRP, MRP1, MRP2, MRP3, and MRP4. BMC Cancer. 2022;22(1):446.
- Cho CJ, et al. The modulation study of multiple drug resistance in bladder cancer by curcumin and resveratrol. Oncol Lett. 2019;18(6):6869-76.
- Gupta SK, Singh P, Ali V, Verma M. Role of membrane-embedded drug efflux
transporters in the cancer chemotherapy. Oncol Rev. 2020;14(2):448. https://doi.org/10.4081/oncol.2020. - Monteran L, et al. Chemotherapy-induced complement signaling modulates immunosuppression and metastatic relapse in breast cancer. Nat Commun. 2022;13(1):1-19.
- Makovec T. Cisplatin and beyond: molecular mechanisms of action and drug resistance development in cancer chemotherapy. Radiol Oncol. 2019;53(2):148-58.
- Bulathge AW, et al. Comparative cisplatin reactivity towards human Zn7-metallothionein-2 and MTF-1 zinc fingers: potential implications in anticancer drug resistance. Metallomics. 2022;14(9):mfac061.
- Lee IT-L, et al. Cancer-Derived Extracellular Vesicles as Biomarkers for Cutaneous Squamous Cell Carcinoma: A Systematic Review. Cancers (Basel). 2022;14(20):5098.
- Horibe S, et al. Mitochondrial DNA mutations are involved in the acquisition of cisplatin resistance in human lung cancer A549 cells. Oncol Rep. 2022;47(2):1-12.
- Lilienthal I, Herold N. Targeting Molecular Mechanisms Underlying Treatment Efficacy and Resistance in Osteosarcoma: A Review of Current and Future Strategies. Int J Mol Sci. 2020;21(18):6885. https://doi. org/10.3390/ijms21186885.
- Barbuti AM, Chen Z-S. Paclitaxel through the ages of anticancer therapy: exploring its role in chemoresistance and radiation therapy. Cancers. 2015;7(4):2360-71.
- Germain N, et al. Lipid metabolism and resistance to anticancer treatment. Biology (Basel). 2020;9(12):474.
- Zhang J, Yin D, Li H. hMSH2 expression is associated with paclitaxel resistance in ovarian carcinoma, and inhibition of hMSH2 expression in vitro restores paclitaxel sensitivity. Oncol Rep. 2014;32(5):2199-206.
- Mir MA, Khan SU, Aisha S. Cell Cycle Dysregulation in Breast Cancer. In: Therapeutic potential of Cell Cycle Kinases in Breast Cancer. Springer; 2023. p. 103-31.
- Lopes D, et al. a-Tubulin detyrosination links the suppression of MCAK activity with taxol cytotoxicity. J Cell Biol. 2022;222(2):e202205092.
- Comparetti, E.J., et al., Cancer cell membrane-derived nanoparticles improve the activity of gemcitabine and paclitaxel on pancreatic
cancer cells and coordinate immunoregulatory properties on professional antigen-presenting cells. 2020. 1(6): p. 1775-1787. - Mohiuddin M, Kasahara K. Paclitaxel impedes EGFR-mutated PC9 cell growth via reactive oxygen species-mediated DNA damage and EGFR/ PI3K/AKT/mTOR signaling pathway suppression. Cancer Genomics Proteomics. 2021;18(5):645-59.
- Indini A, et al. NAD/NAMPT and mTOR pathways in melanoma: Drivers of drug resistance and prospective therapeutic targets. Int J Mol Sci. 2022;23(17):9985.
- Dogan E, et al. Cancer stem cells in tumor modeling: Challenges and future directions. Advanced Nanobiomed Res. 2021;1(11):2100017.
- Vasan N, Baselga J, Hyman DM. A view on drug resistance in cancer. Nature. 2019;575(7782):299-309.
- Oshimori N, Guo Y, Taniguchi S. An emerging role for cellular crosstalk in the cancer stem cell niche. J Pathol. 2021;254(4):384-94.
- Li Y, et al. Drug resistance and Cancer stem cells. Cell Commun Signal. 2021;19(1):19.
- Batlle E, Clevers H. Cancer stem cells revisited. Nat Med. 2017;23(10):1124-34.
- Mirzaei S, Gholami MH, Hashemi F, Zabolian A, Farahani MV, Hushmandi K, Zarrabi A, Goldman A, Ashrafizadeh M, Orive G. Advances in understanding the role of P-gp in doxorubicin resistance: Molecular pathways, therapeutic strategies, and prospects. Drug Discov Today. 2022;27(2):436-55. https://doi.org/10.1016/j.drudis.2021.09.020.
Publisher’s Note
Sameer Ullah Khan and Kaneez Fatima contributed equally.
*Correspondence:
Sameer Ullah Khan
muminsameer@gmail.com
Fayaz Malik
fmalik@iiim.res.in
¹ Division of Genitourinary Medical Oncology, The University of Texas MD Anderson Cancer Center, Holcombe Blvd, Houston, TX 77030, USA
Division of Cancer Pharmacology, CSIR-Indian Institute of Integrative Medicine, Srinagar-190005, Jammu and Kashmir, India
Academy of Scientific and Innovative Research (AcSIR), Ghaziabad-201002, India
