DOI: https://doi.org/10.1038/s41598-024-57970-7
PMID: https://pubmed.ncbi.nlm.nih.gov/38538708
تاريخ النشر: 2024-03-27
استخدام التعلم العميق والتعلم الانتقالي للكشف الدقيق عن أورام الدماغ
الملخص
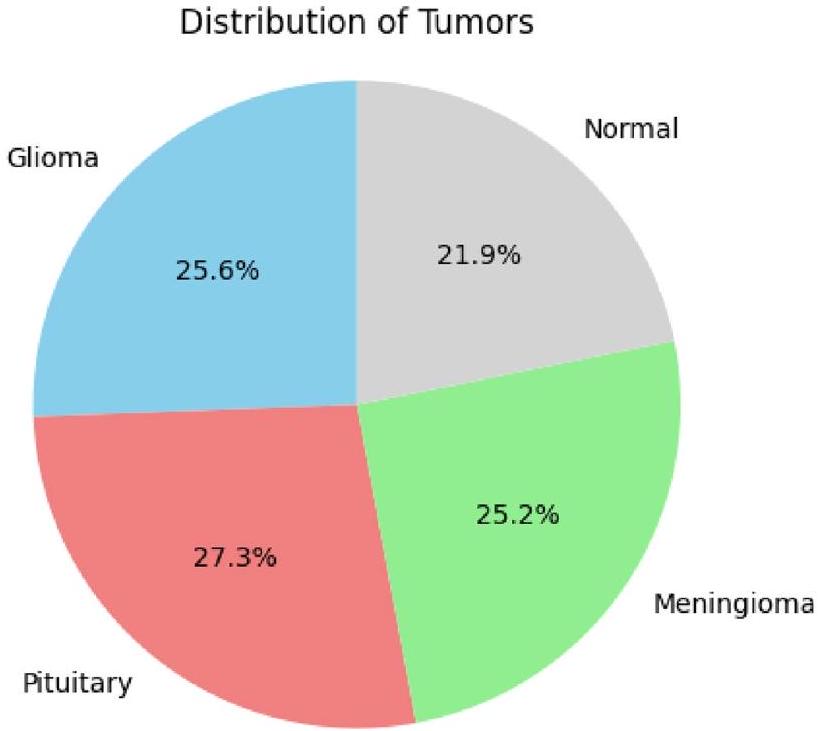
تُستخدم طرق التعلم العميق المدعومة بالذكاء الاصطناعي لتشخيص أورام الدماغ بدقة عالية، وذلك بفضل قدرتها على معالجة كميات كبيرة من البيانات. تُعتبر التصوير بالرنين المغناطيسي المعيار الذهبي لتشخيص أورام الدماغ باستخدام رؤية الآلة، متفوقةً على التصوير المقطعي المحوسب، والأشعة فوق الصوتية، وتصوير الأشعة السينية من حيث الفعالية. على الرغم من ذلك، يبقى تشخيص أورام الدماغ مسعىً صعبًا بسبب التركيبة المعقدة للدماغ. تتناول هذه الدراسة إمكانيات هياكل التعلم العميق للنقل لرفع دقة تشخيص أورام الدماغ. يُعتبر التعلم بالنقل تقنية في التعلم الآلي تسمح لنا بإعادة استخدام النماذج المدربة مسبقًا في مهام جديدة. يمكن أن يكون هذا مفيدًا بشكل خاص لمهام التصوير الطبي، حيث غالبًا ما تكون البيانات المصنفة نادرة. تم تقييم أربعة هياكل مختلفة للتعلم بالنقل في هذه الدراسة: ResNet152، VGG19، DenseNet169، وMobileNetv3. تم تدريب النماذج والتحقق من صحتها على مجموعة بيانات من قاعدة بيانات مرجعية: كاجل. تم اعتماد التحقق المتقاطع بخمس طيات للتدريب والاختبار. لتعزيز توازن مجموعة البيانات وتحسين أداء النماذج، تم تطبيق تقنيات تحسين الصور على البيانات لأربع فئات: الغدة النخامية، الطبيعية، المينينجيوما، والجلوم. حقق MobileNetv3 أعلى دقة بلغت 99.75%، متفوقًا بشكل كبير على الطرق الأخرى الموجودة. يُظهر هذا إمكانيات هياكل التعلم العميق للنقل في إحداث ثورة في مجال تشخيص أورام الدماغ.
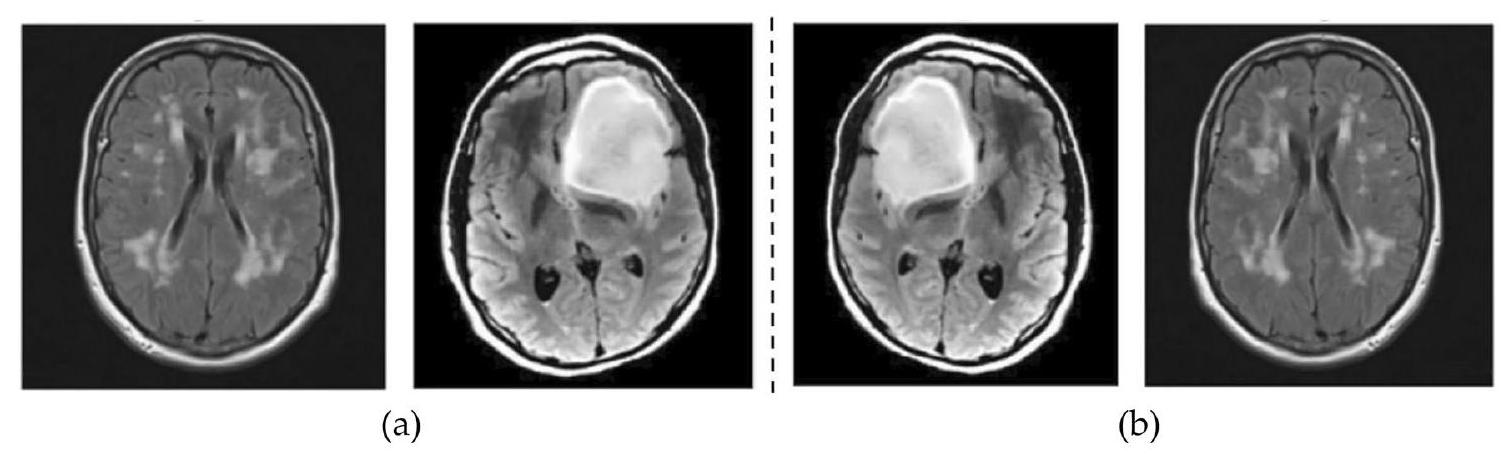
الدماغ البشري، الموجود في الجمجمة، هو عضو حيوي مسؤول عن وظائف متنوعة، تحكمه شبكة من مليارات الخلايا العصبية التي تنسق النبضات الكهربائية والكيميائية، مما يشكل تجاربنا ووجودنا.
(ط) قمنا بتعديل نماذج التعلم بالنقل بعد المعالجة وطبقناها على ثلاثة مجموعات بيانات مرجعية لتحسين أدائها. بالإضافة إلى ذلك، قمنا بتحسين نماذج مثل ResNet152 و VGG19 و DenseNet169 و MobileNetv3 من خلال إضافة طبقة متصلة بالكامل واحدة.
(2) لإجراء مقارنة ذات مغزى، أنشأنا معيارًا يمكن من خلاله تقييم منهجيات التعلم بالنقل المقترحة لدينا مقارنةً بالأبحاث السابقة. النتيجة الرئيسية لدراستنا هي تحقيق أقصى دقة. أظهر MobileNetv3 دقة استثنائية قدرها
(3) يسمح التعلم بالنقل بالاستفادة من النماذج المدربة مسبقًا، وهو مفيد بشكل خاص عند التعامل مع بيانات طبية محدودة معلمة.
(4) حقق MobileNetv3، وهو هيكل محدد للتعلم بالنقل، دقة استثنائية في تشخيص أورام الدماغ.
(5) تسلط هذه النتائج الضوء على فعالية منهجيات التعلم بالنقل لدينا في تصنيف أورام الدماغ، مما يظهر تأثيرها المحتمل على تعزيز دقة التشخيص في تحليل الصور الطبية.
الأعمال ذات الصلة
وتقليل الإفراط في التخصيص. أظهرت التجارب الواسعة باستخدام مجموعة بيانات أورام الدماغ من Figshare أن هيكل VGG16 المحسن حقق دقة تصنيف وكشف مثيرة للإعجاب تصل إلى
حقق أداءً ممتازًا، مع
المواد والطرق
المادة
| المؤلف | السنة | مجموعة البيانات | الطريقة | القيود |
| أرشيا رحمن
|
2019 | Figshare | AlexNet، GoogLeNet، VGGNet | غياب تحليل عميق أو تفسير لقابلية تفسير النموذج |
| تسنيم آزاد عبير
|
2018 | Kaggle | PNN | نقص في التحليل التفصيلي أو المناقشة بشأن التحيزات المحتملة الموجودة في بيانات التدريب |
| جون تشينغ
|
2016 | Figshare | استرجاع الصور القائم على المحتوى | نقص في المناقشة الصريحة أو الاعتبار للقيود المحتملة المتعلقة بتعميم الخوارزمية المقترحة على مجموعات بيانات خارجية أو إعدادات سريرية متنوعة |
| أمينة ناصر
|
2019 | MNIST، PaHaW | ImageNet | غياب مناقشة أو تحليل مفصل بشأن التحيزات المحتملة الموجودة في مجموعات بيانات التدريب، وخاصة ImageNet وMNIST، التي تم استخدامها كمهام مصدر للتعلم بالنقل |
| باكاري بادجي
|
2022 | Kaggle | CNN الخاص بـ AlexNet | نقص في الاعتبار الصريح أو المناقشة حول قابلية تفسير نموذج التعلم العميق |
| راجات مهروترا
|
2020 | Figshare | CNN | غياب تحليل شامل أو مناقشة حول التأثير المحتمل لعدم توازن الفئات في مجموعة البيانات على أداء النموذج |
| نعيم الله
|
2022 | Kaggle | Inceptionresnetv2 | الأداء الضعيف نسبيًا للنماذج المدربة مسبقًا عند استخدامها كتصنيفات مستقلة |
| سارافانان
|
2022 | BRATS، REMBRANDT | CDBLNL | نقص في الوضوح أو المناقشة التفصيلية بشأن القيود أو التحديات المحتملة المرتبطة بنموذج CDBLNL المقترح |
| سارافانان سرينيفاسان
|
2023 | REMBRANDT | RNN التلافيفية | أحد العيوب في العمل المقدم هو نقص التحليل التفصيلي أو المناقشة حول قابلية تفسير نموذج CRNN (الشبكة العصبية التلافيفية المتكررة) المقترح |
| بشتيوان جبار كريم
|
2023 | Figshare | CNN + SVM المعدل | نقص في المناقشة التفصيلية أو استكشاف التحيزات المحتملة في مجموعة بيانات Figshare المستخدمة للتقييم. يمكن أن تؤثر التحيزات في مجموعات البيانات الطبية، خاصة المتعلقة بأورام الدماغ، بشكل كبير على قابلية تعميم طريقة التصنيف المقترحة |
| المرحلة | خبيث (80%) | طبيعي (20%) | الإجمالي |
| تدريب | 4494 | 1124 | 5618 |
| اختبار | 1124 | 281 | 1405 |
| الإجمالي | 5618 | 1405 | 7023 |
طرق

تقييم نموذج التعلم المنقول

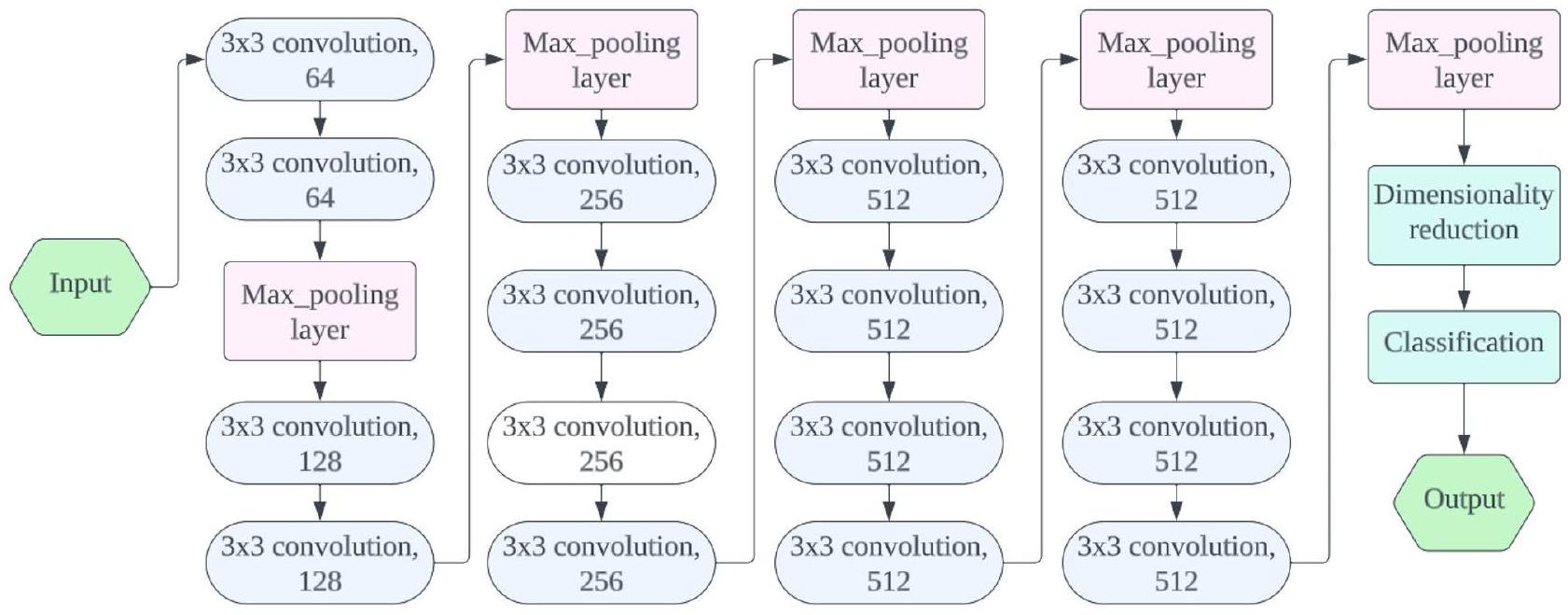

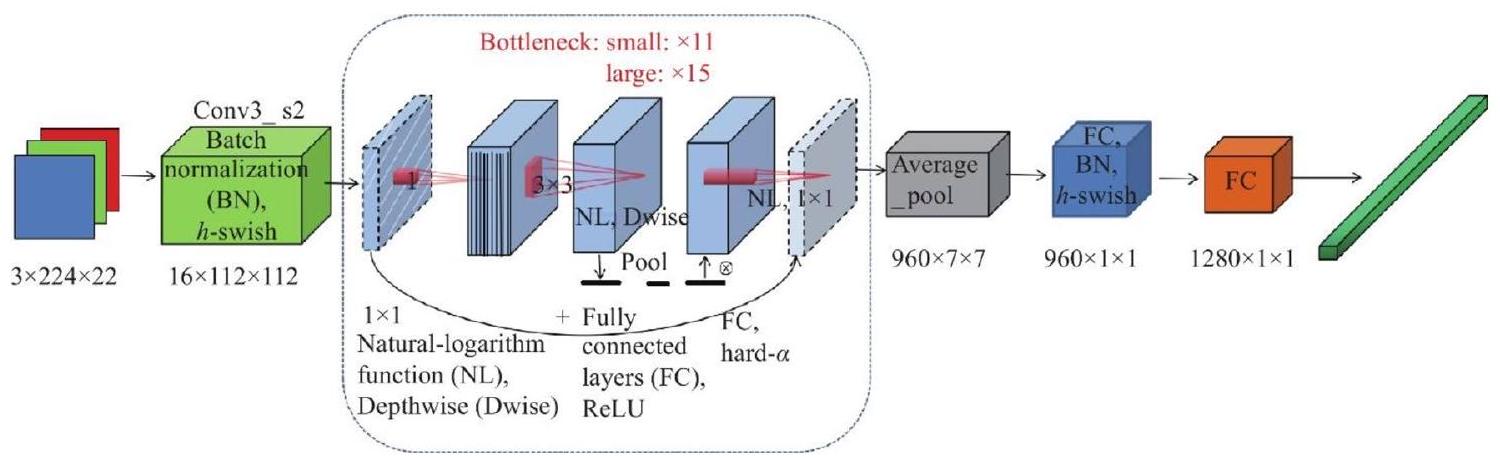
إعداد وتقييم التجارب
| مدخل | مشغل | حجم | الإخراج | خطوة |
|
|
التفاف ثنائي الأبعاد
|
– | 16 | 2 |
|
|
بنك
|
16 | 16 | 2 |
|
|
بنك
|
72 | ٢٤ | ٢ |
|
|
بنك
|
٨٨ | ٢٤ | 1 |
|
|
بنك
|
96 | 40 | 2 |
|
|
بنك
|
٢٤٠ | 40 | 1 |
|
|
بنك
|
٢٤٠ | 40 | 1 |
|
|
بنك
|
١٢٠ | ٤٨ | 1 |
|
|
بنك
|
١٤٤ | ٤٨ | 1 |
|
|
بنك
|
٢٨٨ | 96 | 2 |
|
|
بنك
|
576 | 96 | 1 |
|
|
بنك
|
576 | 96 | 1 |
|
|
التفاف ثنائي الأبعاد
|
– | 576 | 1 |
|
|
مسبح
|
– | – | 1 |
|
|
التفاف ثنائي الأبعاد
|
– | ١٠٢٤ | 1 |
|
|
التفاف ثنائي الأبعاد
|
– | ك | 1 |
| تQuantifying الأداء والتقييم | تقييم نتائج القياس |
| حجم الدفعة | 128 |
| محسّن | آدم |
| عدد العصور | 50 |
| معدل التعلم | 0.001 |
| معيار التقييم | خسارة الانتروبيا المتقاطعة |
| تدريب | التحقق المتقاطع بخمسة أضعاف |
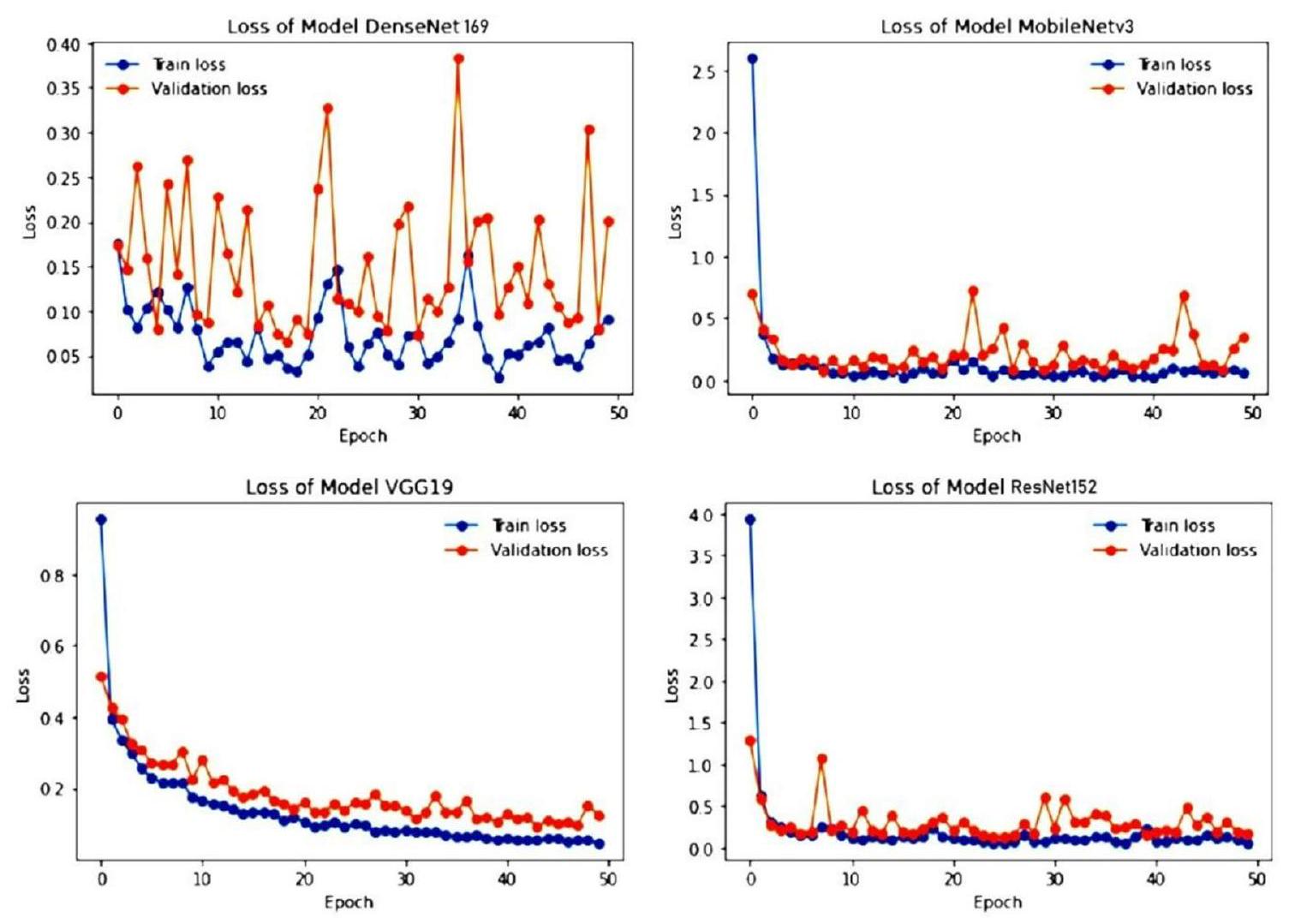
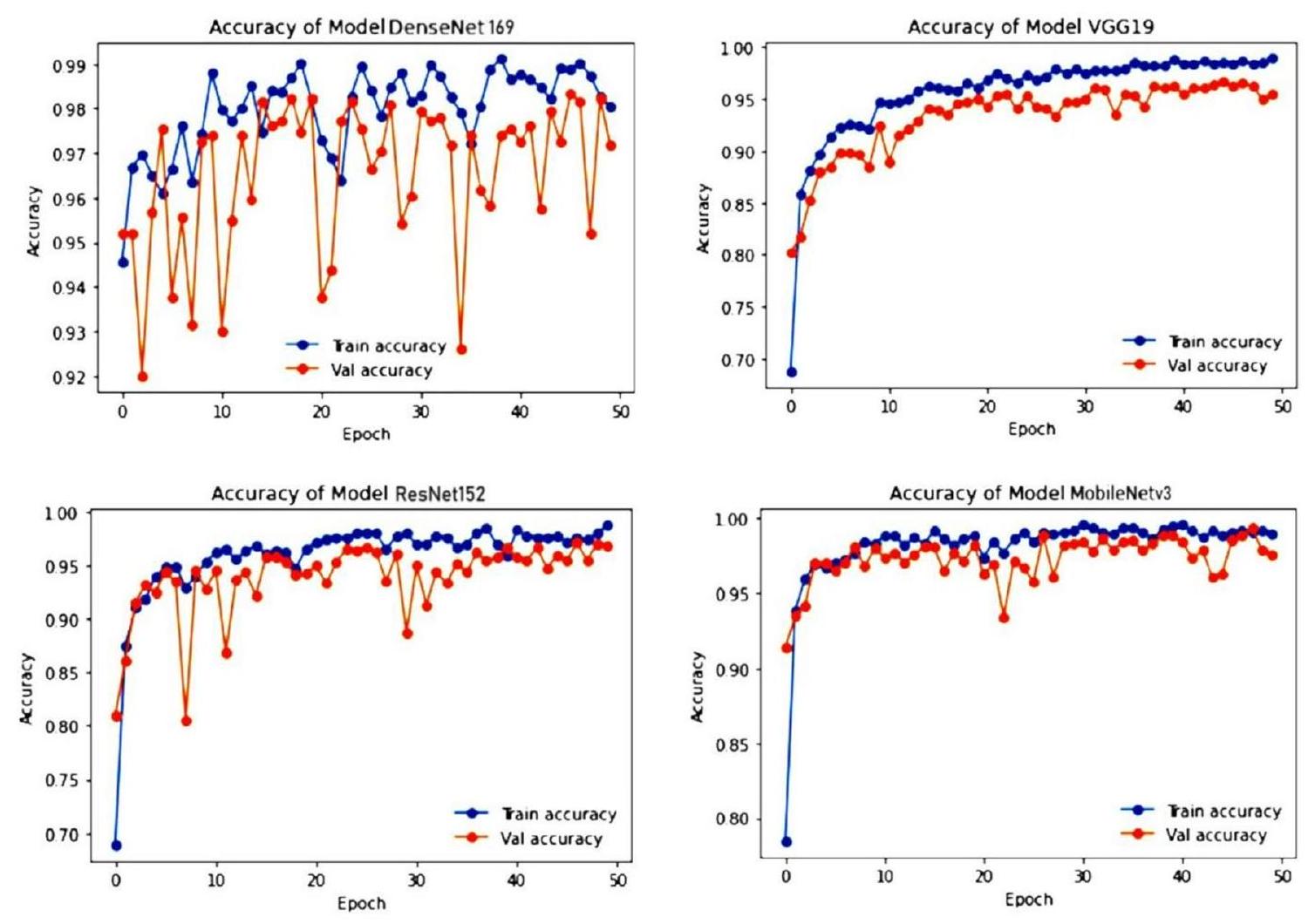
النتائج التجريبية والمناقشة
| العمارة | مرحلة التدريب | مرحلة الاختبار | ||
| الدقة (%) | خسارة | الدقة (%) | خسارة | |
| ريزنت152 | 98.86 | 0.0603 | ٩٦.٩٢ | 0.1854 |
| VGG19 | 99.07 | 0.0451 | 95.62 | 0.1245 |
| دينس نت 169 | 99.22 | 0.0241 | ٩٧.٥٣ | 0.958 |
| موبايل نت في3 | 99.75 | 0.0359 | ٩٨.٥٢ | 0.1272 |
| إيجابي متوقع | سلبي متوقع | |
| إيجابي فعلي | تي بي | FN |
| سلبي فعلي | FP | TN |
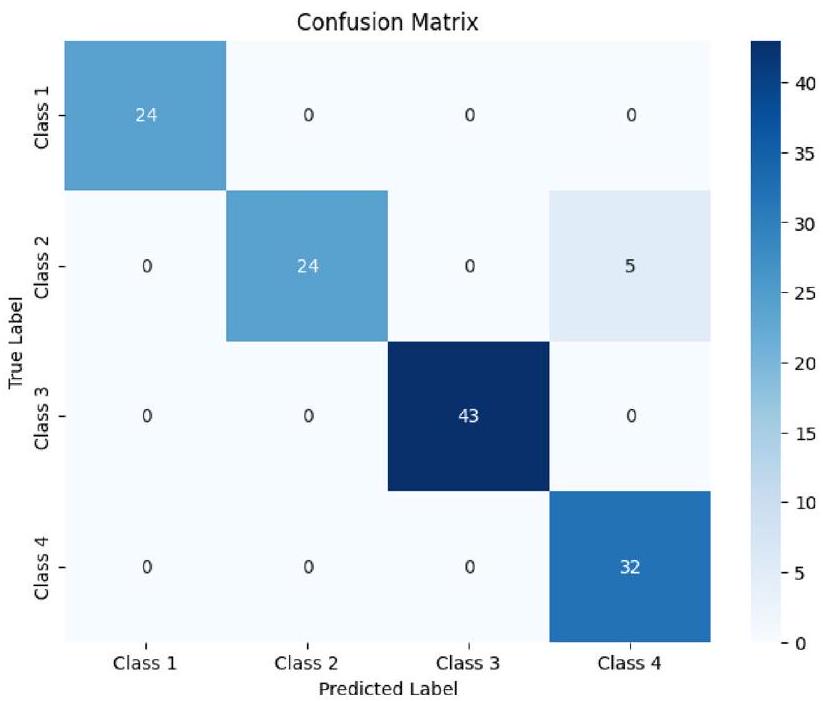

نقاش
| الهندسات | فصل | دقة | استدعاء | درجة F1 | دقة |
| ريزنت152 | الغدة النخامية | 1 | 0.93 | 0.98 | 0.985 |
| عادي | 0.98 | 1 | 0.97 | ||
| ورم السحايا | 1 | 1 | 1 | ||
| ورم دبقي | 0.96 | 1 | 0.99 | ||
| إجمالي | 3.94 | 3.93 | 3.94 | ||
| VGG19 | الغدة النخامية | 1 | 1 | 0.95 | 0.960 |
| عادي | 0.95 | 0.92 | 0.95 | ||
| ورم السحايا | 1 | 1 | 1 | ||
| ورم دبقي | 0.93 | 1 | 0.94 | ||
| إجمالي | 3.88 | 3.92 | 3.84 | ||
| دينس نت 169 | الغدة النخامية | 1 | 0.85 | 0.94 | 0.9675 |
| عادي | 0.88 | 1 | 0.93 | ||
| ورم السحايا | 1 | 1 | 1 | ||
| ورم دبقي | 1 | 1 | 1 | ||
| إجمالي | 3.88 | 3.85 | 3.87 | ||
| موبايل نت في3 | الغدة النخامية | 1 | 1 | 1 | 0.960 |
| عادي | 1 | 0.83 | 0.92 | ||
| ورم السحايا | 1 | 1 | 1 | ||
| ورم دبقي | 0.88 | 1 | 0.92 | ||
| إجمالي | 3.88 | 3.83 | 3.84 |
| نموذج التعلم المنقول | مرحلة | م
|
م
|
مي-أك | مي-إيبي |
| VGG19 | تدريب | ٩٨.٧٨ | 50 | 70.22 | 1 |
| اختبار | ٩٦.٩١ | 50 | 81.45 | 1 | |
| ريزنت152 | تدريب | 98.12 | 50 | 69.34 | 1 |
| اختبار | ٩٧.٧٨ | ٤٥ | 80.96 | ٦ | |
| دينس نت 169 | تدريب | 99.08 | ٣٨ | 95.42 | 1 |
| اختبار | ٩٨.٦٨ | ٤٧ | 90.77 | 2 | |
| موبايل نت في3 | تدريب | 99.75 | 30 | 77.12 | 1 |
| اختبار | 99.52 | ٤٦ | 90.27 | 1 |
| نماذج TL | الجدول الزمني (س:د) |
| ريزنت152 |
|
| VGG19 |
|
| دينس نت 169 |
|
| موبايل نت في3 |
|
| مؤلف | سنة | مجموعة بيانات | طريقة | الدقة (%) |
| تسنيم آزاد عبير
|
2018 | كاجل | PNN | ٨٣.٣٣ |
| باكاري بادجي
|
٢٠٢٢ | كاجل | شبكة أليكس نت العصبية | 99.12 |
| نعيم الله
|
٢٠٢٢ | كاجل | إنسيبشن ريزنت في2 | ٩٨.٩١ |
| سارافانان
|
٢٠٢٠ | كاجل | كانفيس | ٩٨.٧٣ |
| النموذج المقترح | ٢٠٢٣ | كاجل | نهج التعلم بالنقل | 99.75 |
الخاتمة والأعمال المستقبلية

توفر البيانات
نُشر على الإنترنت: 27 مارس 2024
References
- Mockly, S., Houbron, É. & Seitz, H. A rationalized definition of general tumor suppressor micrornas excludes miR-34a. Nucleic Acids Res. 50(8), 4703-4712 (2022).
- Lauko, A., Lo, A., Ahluwalia, M. S. & Lathia, J. D. Cancer cell heterogeneity & plasticity in glioblastoma and brain tumors. Semin. Cancer Biol. 82(1), 162-175 (2022).
- Wang, F. et al. Cerebrospinal fluid-based metabolomics to characterize different types of brain tumors. J. Neurol. 267(1), 984-993 (2020).
- Swati, Z. et al. Content-based brain tumor retrieval for MR images using transfer learning. IEEE Access 7(1), 17809-17822 (2019).
- Chelghoum, R., Ikhlef, A., Hameurlaine, A., & Jacquir, S. Transfer learning using convolutional neural network architectures for brain tumor classification from MRI images, in IFIP International Conference on Artificial Intelligence Applications and Innovations, Vol. 583, 189-200 (Springer, 2020).
- Khan, H., Jue, W., Mushtaq, M. & Mushtaq, M. U. Brain tumor classification in MRI image using convolutional neural network’. Math. Biosci. Eng. 17(5), 6203-6216 (2020).
- Kumar, S. & Mankame, D. P. Optimization driven deep convolution neural network for brain tumor classification. Biocybern. Biomed. Eng. 40(3), 1190-1204 (2020).
- Sharif, J., Amin, M., Raza, M. & Yasmin, S. C. S. An integrated design of particle swarm optimization (PSO) with fusion of features for detection of brain tumor. Pattern Recognit. Lett. 129, 150-157 (2020).
- Amin, J., Sharif, M., Yasmin, M. & Fernandes, S. L. A distinctive approach in brain tumor detection and classification using MRI. Pattern Recognit. Lett. 139, 118-127 (2020).
- Woźniak, M., Siłka, J. & Wieczorek, M. Deep neural network correlation learning mechanism for CT brain tumor detection. Neural Comput. Appl. 35, 14611-14626 (2021).
- Al Rub, S. A., Alaiad, A., Hmeidi, I., Quwaider, M. & Alzoubi, O. Hydrocephalus classification in brain computed tomography medical images using deep learning. Simul. Model. Pract. 123, 102705 (2023).
- Mehnatkesh, H., Jalali, S. M. J., Khosravi, A. & Nahavandi, S. An intelligent driven deep residual learning framework for brain tumor classification using MRI images. Expert Syst. Appl. 213, 119087 (2023).
- Raja, P. S. & Viswasarani, A. Brain tumor classification using a hybrid deep autoencoder with Bayesian fuzzy clustering-based segmentation approach. Biocybern. Biomed. Eng. 40(1), 440-453 (2020).
- Cè, M. et al. Artificial intelligence in brain tumor imaging: A step toward personalized medicine. Curr. Oncol. 30(3), 2673-2701 (2023).
- Badža, M. M. & Barjaktarović, M. Č. Classification of brain tumors from MRI images using a convolutional neural network. Appl. Sci. 10(6), 1999 (2020).
- Ismael, S. A. A., Mohammed, A. & Hefny, H. An enhanced deep learning approach for brain cancer MRI images classification using residual networks. Artif. Intell. Med. 102(1), 101779 (2020).
- Rehman, M. A., Khan, T., Saba, Z., Mehmood, U. & Tariq, N. A. Microscopic brain tumor detection and classification using 3D CNN and feature selection architecture. Microsc. Res. Tech. 84(1), 133-149 (2021).
- Tabatabaei, S., Rezaee, K. & Zhu, M. Attention transformer mechanism and fusion based deep learning architecture for MRI brain tumor classification system. Biomed. Signal Process. Control 86(1), 105119 (2023).
- Rehman, A., Naz, S., Razzak, M. I., Akram, F. & Imran, M. A deep learning-based framework for automatic brain tumors classification using transfer learning. Circuits Syst. Signal Process. 39(1), 757-775 (2019).
- Abir, T. A., Siraji, J. A. & Ahmed, E. Analysis of a novel MRI Based Brain Tumour Classification Using Probabilistic Neural Network (PNN). Int. J. Sci. Res. Sci. Eng. Technol. 4(8), 69-75 (2018).
- Cheng, J. et al. Retrieval of brain tumors by adaptive spatial pooling and fisher vector representation. PLoS ONE 11(6), 1-15 (2016).
- Naseer, A. et al. Refining Parkinson’s neurological disorder identification through deep transfer learning. Neural Comput. Appl. 32(1), 839-854 (2018).
- Badjie, B. & Ülker, E. D. A deep transfer learning based architecture for brain tumor classification using MR images. Inf. Technol. Control 51(2), 333-343 (2022).
- Rajat Mehrotra, M. A., Ansari, R. A. & Anand, R. S. A Transfer Learning approach for AI-based classification of brain tumors. Mach. Learn. Appl. 2(1), 100003 (2020).
- Ullah, N. et al. An effective approach to detect and identify brain tumors using transfer learning. Appl. Sci 12(11), 1-17 (2022).
- Saravanan, S. et al. Computational and mathematical methods in medicine glioma brain tumor detection and classification using convolutional neural network. Comput. Math. Methods Med. 4380901, 1-12 (2022).
- Srinivasan, S. et al. Grade classification of tumors from brain magnetic resonance images using a deep learning technique. Diagnostics 13(6), 1-20 (2023).
- Saravanan, S. & Thirumurugan, P. Performance analysis of glioma brain tumor segmentation using Ridgelet transform and coactive adaptive neuro fuzzy expert system methodology. J. Med. Imaging Health Inf. 10(11), 2642-2648 (2020).
- Karim, P. J., Mahmood, S. R. & Sah, M. Brain tumor classification using fine-tuning based deep transfer learning and support vector machine. Int. J. Comput. Digit. Syst. 13(1), 84-96 (2023).
- Ullah, F. et al. Evolutionary model for brain cancer-grading and classification. IEEE Access 99(1), 1-15 (2023).
- Ullah, F. et al. Enhancing brain tumor segmentation accuracy through scalable federated learning with advanced data privacy and security measures. Mathematics 11(9), 1-27 (2023).
- Ullah, F. et al. Brain tumor segmentation from MRI images using handcrafted convolutional neural network. Diagnostics 13(16), 1-15 (2023).
- Ullah, F., Nadeem, M. & Abrar, M. Revolutionizing brain tumor segmentation in MRI with dynamic fusion of handcrafted features and global pathway-based deep learning. KSII Trans. Internet Info. Syst. 18(1), 105-125 (2024).
- M. Nickparvar, Brain tumor MRI dataset (2023). https://www.kaggle.com/datasets/masoudnickparvar/brain-tumor-mri-dataset.
- Islam, M. M., Uddin, M. R., Ferdous, M. J., Akter, S. & Akhtar, M. N. BdSLW-11: Dataset of Bangladeshi sign language words for recognizing 11 daily useful BdSL words. Data Brief 45, 108747 (2022).
- Xu, X., Li, W. & Duan, Q. Transfer learning and SE-ResNet152 networks-based for small-scale unbalanced fish species identification. Comput. Electron. Agric. 180(1), 1-7 (2023).
- Bansa, M., Kumar, M., Sachdeva, M. & Mittal, A. Transfer learning for image classification using VGG19: Caltech-101 image data set. J. Ambient Intell. Hum. Comput. 14(1), 3609-3620 (2023).
- Al-rimy, B. A. S., Saeed, F., Al-Sarem, M., Albarrak, A. M. & Qasem, S. N. An adaptive early stopping technique for DenseNet169based knee osteoarthritis detection model. Diagnostics 13(11), 1-19 (2023).
- Li, Y., Xiong, X., Xin, W., Huang, J. & Hao, H. MobileNetV3-CenterNet: A target recognition method for avoiding missed detection effectively based on a lightweight network. J. Beijing Inst. Technol. 32(1), 82-94 (2023).
- Chang, Y. L. et al. Consolidated convolutional neural network for hyperspectral image classification. Remote Sens. 14(1), 1571 (2022).
مساهمات المؤلفين
تمويل
المصالح المتنافسة
معلومات إضافية
معلومات إعادة الطباعة والتصاريح متاحة علىwww.nature.com/reprints.
ملاحظة الناشر: تظل شركة سبرينغر ناتشر محايدة فيما يتعلق بالمطالبات القضائية في الخرائط المنشورة والانتماءات المؤسسية.
© المؤلف(ون) 2024
كلية علوم الحاسوب والهندسة، جامعة جالغوتيا، غريتر نويدا 203201، الهند. قسم علوم الحاسوب والهندسة، معهد فيل تيك رانجاراجان د. ساغونثالا للبحث والتطوير في العلوم والتكنولوجيا، تشيناي 600062، الهند. مدرسة علوم الحوسبة والهندسة، جامعة VIT بهوبال، طريق بهوبال-إندور كوثريكالان، سيهور 466114، الهند. جامعة كيبري ديهار، 250، كيبري ديهار، الصومال، إثيوبيا. مركز تأثير وأثر البحث، معهد تشيتكارا للهندسة والتكنولوجيا، جامعة تشيتكارا، راجبورا، البنجاب 140401، الهند. قسم البحث والتطوير، جامعة لوفلي المهنية، فاجوارا، البنجاب 144001، الهند. البريد الإلكتروني:drmohdasifshah@kdu.edu.et
DOI: https://doi.org/10.1038/s41598-024-57970-7
PMID: https://pubmed.ncbi.nlm.nih.gov/38538708
Publication Date: 2024-03-27
Employing deep learning and transfer learning for accurate brain tumor detection
Abstract
Artificial intelligence-powered deep learning methods are being used to diagnose brain tumors with high accuracy, owing to their ability to process large amounts of data. Magnetic resonance imaging stands as the gold standard for brain tumor diagnosis using machine vision, surpassing computed tomography, ultrasound, and X-ray imaging in its effectiveness. Despite this, brain tumor diagnosis remains a challenging endeavour due to the intricate structure of the brain. This study delves into the potential of deep transfer learning architectures to elevate the accuracy of brain tumor diagnosis. Transfer learning is a machine learning technique that allows us to repurpose pre-trained models on new tasks. This can be particularly useful for medical imaging tasks, where labelled data is often scarce. Four distinct transfer learning architectures were assessed in this study: ResNet152, VGG19, DenseNet169, and MobileNetv3. The models were trained and validated on a dataset from benchmark database: Kaggle. Five-fold cross validation was adopted for training and testing. To enhance the balance of the dataset and improve the performance of the models, image enhancement techniques were applied to the data for the four categories: pituitary, normal, meningioma, and glioma. MobileNetv3 achieved the highest accuracy of 99.75%, significantly outperforming other existing methods. This demonstrates the potential of deep transfer learning architectures to revolutionize the field of brain tumor diagnosis.
The human brain, located in the cranium, is a crucial organ responsible for various functions, governed by a network of billions of neurons that coordinate electrical and chemical impulses, shaping our experiences and existence
(i) We fine-tuned the transfer learning models after processing and applied them to three benchmark datasets to optimize their performance. Additionally, we enhanced models like ResNet152, VGG19, DenseNet169, and MobileNetv3 by adding a single fully connected layer.
(ii) To establish a meaningful comparison, we created a benchmark against which our proposed transfer learning methodologies can be evaluated in comparison to previous research. The key outcome of our study is the achievement of maximum precision. MobileNetv3 demonstrated outstanding precision of
(iii) Transfer learning allows leveraging pre-trained models, especially beneficial when dealing with limited labelled medical data.
(iv) MobileNetv3, a specific transfer learning architecture, achieved exceptional accuracy in brain tumor diagnosis.
(v) These results highlight the effectiveness of our transfer learning methodologies in the classification of brain tumors, showcasing their potential impact on advancing diagnostic accuracy in medical image analysis.
Related work
and reduce overfitting. Extensive experimentation using the Figshare MRI brain tumor dataset revealed that the optimized VGG16 architecture achieved an impressive detection and classification accuracy of up to
achieved excellent performance, with
Material and methods
Material
| Author | Year | Dataset | Method | Limitations |
| Arshia Rehman
|
2019 | Figshare | AlexNet, GoogLeNet, VGGNet | Absence of an in-depth analysis or explanation of the interpretability of the model |
| Tasnim Azad Abir
|
2018 | Kaggle | PNN | Lack of detailed analysis or discussion regarding the potential biases present in the training data |
| Jun Cheng
|
2016 | Figshare | Content-based image retrieval | Lack of explicit discussion or consideration of potential limitations related to the generalization of the proposed algorithm to external datasets or diverse clinical settings |
| Amina Naseer
|
2019 | MNIST, PaHaW | ImageNet | The absence of a detailed discussion or analysis regarding the potential biases present in the training datasets, particularly ImageNet and MNIST, which were used as source tasks for transfer learning |
| Bakary Badjie
|
2022 | Kaggle | AlexNet’s CNN | Lack of explicit consideration or discussion about the interpretability of the deep learning mode |
| Rajat Mehrotra
|
2020 | Figshare | CNN | The absence of a comprehensive analysis or discussion about the potential impact of class imbalances in the dataset on the model’s performance |
| Naeem Ullah
|
2022 | Kaggle | Inceptionresnetv2 | The comparatively weak performance of pre-trained deep learning (DL) models when used as stand-alone classifiers |
| Saravanan
|
2022 | BRATS, REMBRANDT | CDBLNL | Lack of clarity or detailed discussion regarding the potential limitations or challenges associated with the proposed CDBLNL model |
| Saravanan Srinivasan
|
2023 | REMBRANDT | Convolutional RNN | One demerit in the presented work is the lack of detailed analysis or discussion about the interpretability of the proposed CRNN (Convolutional Recurrent Neural Network) model |
| Pshtiwan Jabar Karim
|
2023 | Figshare | CNN + fine-tuned SVM | Lack of detailed discussion or exploration of potential biases in the Figshare dataset used for evaluation. Biases in medical datasets, especially related to brain tumors, can significantly impact the generalizability of the proposed classification method |
| Phase | Malignant (80%) | Normal (20%) | Total |
| Train | 4494 | 1124 | 5618 |
| Test | 1124 | 281 | 1405 |
| Total | 5618 | 1405 | 7023 |

Methods


Transfer learning model evaluation




Preparation and evaluation of experiments
| Input | Operator | Size | Output | Stride |
|
|
conv2d,
|
– | 16 | 2 |
|
|
bneck,
|
16 | 16 | 2 |
|
|
bneck,
|
72 | 24 | 2 |
|
|
bneck,
|
88 | 24 | 1 |
|
|
bneck,
|
96 | 40 | 2 |
|
|
bneck,
|
240 | 40 | 1 |
|
|
bneck,
|
240 | 40 | 1 |
|
|
bneck,
|
120 | 48 | 1 |
|
|
bneck,
|
144 | 48 | 1 |
|
|
bneck,
|
288 | 96 | 2 |
|
|
bneck,
|
576 | 96 | 1 |
|
|
bneck,
|
576 | 96 | 1 |
|
|
conv2d,
|
– | 576 | 1 |
|
|
pool,
|
– | – | 1 |
|
|
conv2d,
|
– | 1024 | 1 |
|
|
conv2d,
|
– | k | 1 |
| Quantifying performance and evaluation | Assessing measurement outcomes |
| Size of the batch | 128 |
| Optimizer | Adam |
| No. of epochs | 50 |
| Rate of learning | 0.001 |
| Evaluation criterion | Cross entropy loss |
| Training | Five-fold cross validation |

Experimental results and discussion

| Architecture | Training phase | Testing phase | ||
| Acc (%) | Loss | Acc (%) | Loss | |
| ResNet152 | 98.86 | 0.0603 | 96.92 | 0.1854 |
| VGG19 | 99.07 | 0.0451 | 95.62 | 0.1245 |
| DenseNet169 | 99.22 | 0.0241 | 97.53 | 0.958 |
| MobileNetv3 | 99.75 | 0.0359 | 98.52 | 0.1272 |
| Predicted positive | Predicted negative | |
| Actual positive | TP | FN |
| Actual negative | FP | TN |


Discussion
| Architectures | Class | Precision | Recall | F1 score | Accuracy |
| ResNet152 | Pituitary | 1 | 0.93 | 0.98 | 0.985 |
| Normal | 0.98 | 1 | 0.97 | ||
| Meningioma | 1 | 1 | 1 | ||
| Glioma | 0.96 | 1 | 0.99 | ||
| Total | 3.94 | 3.93 | 3.94 | ||
| VGG19 | Pituitary | 1 | 1 | 0.95 | 0.960 |
| Normal | 0.95 | 0.92 | 0.95 | ||
| Meningioma | 1 | 1 | 1 | ||
| Glioma | 0.93 | 1 | 0.94 | ||
| Total | 3.88 | 3.92 | 3.84 | ||
| DenseNet169 | Pituitary | 1 | 0.85 | 0.94 | 0.9675 |
| Normal | 0.88 | 1 | 0.93 | ||
| Meningioma | 1 | 1 | 1 | ||
| Glioma | 1 | 1 | 1 | ||
| Total | 3.88 | 3.85 | 3.87 | ||
| MobileNetv3 | Pituitary | 1 | 1 | 1 | 0.960 |
| Normal | 1 | 0.83 | 0.92 | ||
| Meningioma | 1 | 1 | 1 | ||
| Glioma | 0.88 | 1 | 0.92 | ||
| Total | 3.88 | 3.83 | 3.84 |
| Transfer learning model | Phase | M
|
M
|
Mi-Acc | Mi-Ep |
| VGG19 | Training | 98.78 | 50 | 70.22 | 1 |
| Testing | 96.91 | 50 | 81.45 | 1 | |
| ResNet152 | Training | 98.12 | 50 | 69.34 | 1 |
| Testing | 97.78 | 45 | 80.96 | 6 | |
| DenseNet169 | Training | 99.08 | 38 | 95.42 | 1 |
| Testing | 98.68 | 47 | 90.77 | 2 | |
| MobileNetv3 | Training | 99.75 | 30 | 77.12 | 1 |
| Testing | 99.52 | 46 | 90.27 | 1 |
| TL models | Timeline (HH:MM) |
| ResNet152 |
|
| VGG19 |
|
| DenseNet169 |
|
| MobileNetv3 |
|

| Author | Year | Dataset | Method | Accuracy (%) |
| Tasnim Azad Abir
|
2018 | Kaggle | PNN | 83.33 |
| Bakary Badjie
|
2022 | Kaggle | AlexNet’s CNN | 99.12 |
| Naeem Ullah
|
2022 | Kaggle | Inceptionresnetv2 | 98.91 |
| Saravanan
|
2020 | Kaggle | CANFES | 98.73 |
| Proposed model | 2023 | Kaggle | Transfer Learning approach | 99.75 |
Conclusion and future work

Data availability
Published online: 27 March 2024
References
- Mockly, S., Houbron, É. & Seitz, H. A rationalized definition of general tumor suppressor micrornas excludes miR-34a. Nucleic Acids Res. 50(8), 4703-4712 (2022).
- Lauko, A., Lo, A., Ahluwalia, M. S. & Lathia, J. D. Cancer cell heterogeneity & plasticity in glioblastoma and brain tumors. Semin. Cancer Biol. 82(1), 162-175 (2022).
- Wang, F. et al. Cerebrospinal fluid-based metabolomics to characterize different types of brain tumors. J. Neurol. 267(1), 984-993 (2020).
- Swati, Z. et al. Content-based brain tumor retrieval for MR images using transfer learning. IEEE Access 7(1), 17809-17822 (2019).
- Chelghoum, R., Ikhlef, A., Hameurlaine, A., & Jacquir, S. Transfer learning using convolutional neural network architectures for brain tumor classification from MRI images, in IFIP International Conference on Artificial Intelligence Applications and Innovations, Vol. 583, 189-200 (Springer, 2020).
- Khan, H., Jue, W., Mushtaq, M. & Mushtaq, M. U. Brain tumor classification in MRI image using convolutional neural network’. Math. Biosci. Eng. 17(5), 6203-6216 (2020).
- Kumar, S. & Mankame, D. P. Optimization driven deep convolution neural network for brain tumor classification. Biocybern. Biomed. Eng. 40(3), 1190-1204 (2020).
- Sharif, J., Amin, M., Raza, M. & Yasmin, S. C. S. An integrated design of particle swarm optimization (PSO) with fusion of features for detection of brain tumor. Pattern Recognit. Lett. 129, 150-157 (2020).
- Amin, J., Sharif, M., Yasmin, M. & Fernandes, S. L. A distinctive approach in brain tumor detection and classification using MRI. Pattern Recognit. Lett. 139, 118-127 (2020).
- Woźniak, M., Siłka, J. & Wieczorek, M. Deep neural network correlation learning mechanism for CT brain tumor detection. Neural Comput. Appl. 35, 14611-14626 (2021).
- Al Rub, S. A., Alaiad, A., Hmeidi, I., Quwaider, M. & Alzoubi, O. Hydrocephalus classification in brain computed tomography medical images using deep learning. Simul. Model. Pract. 123, 102705 (2023).
- Mehnatkesh, H., Jalali, S. M. J., Khosravi, A. & Nahavandi, S. An intelligent driven deep residual learning framework for brain tumor classification using MRI images. Expert Syst. Appl. 213, 119087 (2023).
- Raja, P. S. & Viswasarani, A. Brain tumor classification using a hybrid deep autoencoder with Bayesian fuzzy clustering-based segmentation approach. Biocybern. Biomed. Eng. 40(1), 440-453 (2020).
- Cè, M. et al. Artificial intelligence in brain tumor imaging: A step toward personalized medicine. Curr. Oncol. 30(3), 2673-2701 (2023).
- Badža, M. M. & Barjaktarović, M. Č. Classification of brain tumors from MRI images using a convolutional neural network. Appl. Sci. 10(6), 1999 (2020).
- Ismael, S. A. A., Mohammed, A. & Hefny, H. An enhanced deep learning approach for brain cancer MRI images classification using residual networks. Artif. Intell. Med. 102(1), 101779 (2020).
- Rehman, M. A., Khan, T., Saba, Z., Mehmood, U. & Tariq, N. A. Microscopic brain tumor detection and classification using 3D CNN and feature selection architecture. Microsc. Res. Tech. 84(1), 133-149 (2021).
- Tabatabaei, S., Rezaee, K. & Zhu, M. Attention transformer mechanism and fusion based deep learning architecture for MRI brain tumor classification system. Biomed. Signal Process. Control 86(1), 105119 (2023).
- Rehman, A., Naz, S., Razzak, M. I., Akram, F. & Imran, M. A deep learning-based framework for automatic brain tumors classification using transfer learning. Circuits Syst. Signal Process. 39(1), 757-775 (2019).
- Abir, T. A., Siraji, J. A. & Ahmed, E. Analysis of a novel MRI Based Brain Tumour Classification Using Probabilistic Neural Network (PNN). Int. J. Sci. Res. Sci. Eng. Technol. 4(8), 69-75 (2018).
- Cheng, J. et al. Retrieval of brain tumors by adaptive spatial pooling and fisher vector representation. PLoS ONE 11(6), 1-15 (2016).
- Naseer, A. et al. Refining Parkinson’s neurological disorder identification through deep transfer learning. Neural Comput. Appl. 32(1), 839-854 (2018).
- Badjie, B. & Ülker, E. D. A deep transfer learning based architecture for brain tumor classification using MR images. Inf. Technol. Control 51(2), 333-343 (2022).
- Rajat Mehrotra, M. A., Ansari, R. A. & Anand, R. S. A Transfer Learning approach for AI-based classification of brain tumors. Mach. Learn. Appl. 2(1), 100003 (2020).
- Ullah, N. et al. An effective approach to detect and identify brain tumors using transfer learning. Appl. Sci 12(11), 1-17 (2022).
- Saravanan, S. et al. Computational and mathematical methods in medicine glioma brain tumor detection and classification using convolutional neural network. Comput. Math. Methods Med. 4380901, 1-12 (2022).
- Srinivasan, S. et al. Grade classification of tumors from brain magnetic resonance images using a deep learning technique. Diagnostics 13(6), 1-20 (2023).
- Saravanan, S. & Thirumurugan, P. Performance analysis of glioma brain tumor segmentation using Ridgelet transform and coactive adaptive neuro fuzzy expert system methodology. J. Med. Imaging Health Inf. 10(11), 2642-2648 (2020).
- Karim, P. J., Mahmood, S. R. & Sah, M. Brain tumor classification using fine-tuning based deep transfer learning and support vector machine. Int. J. Comput. Digit. Syst. 13(1), 84-96 (2023).
- Ullah, F. et al. Evolutionary model for brain cancer-grading and classification. IEEE Access 99(1), 1-15 (2023).
- Ullah, F. et al. Enhancing brain tumor segmentation accuracy through scalable federated learning with advanced data privacy and security measures. Mathematics 11(9), 1-27 (2023).
- Ullah, F. et al. Brain tumor segmentation from MRI images using handcrafted convolutional neural network. Diagnostics 13(16), 1-15 (2023).
- Ullah, F., Nadeem, M. & Abrar, M. Revolutionizing brain tumor segmentation in MRI with dynamic fusion of handcrafted features and global pathway-based deep learning. KSII Trans. Internet Info. Syst. 18(1), 105-125 (2024).
- M. Nickparvar, Brain tumor MRI dataset (2023). https://www.kaggle.com/datasets/masoudnickparvar/brain-tumor-mri-dataset.
- Islam, M. M., Uddin, M. R., Ferdous, M. J., Akter, S. & Akhtar, M. N. BdSLW-11: Dataset of Bangladeshi sign language words for recognizing 11 daily useful BdSL words. Data Brief 45, 108747 (2022).
- Xu, X., Li, W. & Duan, Q. Transfer learning and SE-ResNet152 networks-based for small-scale unbalanced fish species identification. Comput. Electron. Agric. 180(1), 1-7 (2023).
- Bansa, M., Kumar, M., Sachdeva, M. & Mittal, A. Transfer learning for image classification using VGG19: Caltech-101 image data set. J. Ambient Intell. Hum. Comput. 14(1), 3609-3620 (2023).
- Al-rimy, B. A. S., Saeed, F., Al-Sarem, M., Albarrak, A. M. & Qasem, S. N. An adaptive early stopping technique for DenseNet169based knee osteoarthritis detection model. Diagnostics 13(11), 1-19 (2023).
- Li, Y., Xiong, X., Xin, W., Huang, J. & Hao, H. MobileNetV3-CenterNet: A target recognition method for avoiding missed detection effectively based on a lightweight network. J. Beijing Inst. Technol. 32(1), 82-94 (2023).
- Chang, Y. L. et al. Consolidated convolutional neural network for hyperspectral image classification. Remote Sens. 14(1), 1571 (2022).
Author contributions
Funding
Competing interests
Additional information
Reprints and permissions information is available at www.nature.com/reprints.
Publisher’s note Springer Nature remains neutral with regard to jurisdictional claims in published maps and institutional affiliations.
© The Author(s) 2024
School of Computer Science and Engineering, Galgotias University, Greater Noida 203201, India. Department of Computer Science and Engineering, Vel Tech Rangarajan Dr.Sagunthala R&D Institute of Science and Technology, Chennai 600062, India. School of Computing Science and Engineering, VIT Bhopal University, Bhopal-Indore Highway Kothrikalan, Sehore 466114, India. Kebri Dehar University, 250, Kebri Dehar, Somali, Ethiopia. Centre of Research Impact and Outcome, Chitkara University Institute of Engineering and Technology, Chitkara University, Rajpura, Punjab 140401, India. Division of Research and Development, Lovely Professional University, Phagwara, Punjab 144001, India. email: drmohdasifshah@kdu.edu.et
