DOI: https://doi.org/10.1007/s00521-023-09366-3
تاريخ النشر: 2024-01-13
تطبيقات التعلم العميق في المعلوماتية الحيوية والطبية المعتمدة على إنترنت الأشياء: مراجعة منهجية للأدبيات
© المؤلفون 2024
الملخص
في الوقت الحاضر، حقق التعلم الآلي (ML) مستوى عالٍ من الإنجاز في العديد من السياقات. نظرًا لأهمية ML في المعلوماتية الطبية والحيوية بسبب دقتها، ناقش العديد من الباحثين حلولًا متعددة لتطوير وظيفة التحديات الطبية والحيوية باستخدام تقنيات التعلم العميق (DL). تكمن أهمية DL في المعلوماتية الحيوية والطبية المعتمدة على إنترنت الأشياء (IoT) في قدرته على تحليل وتفسير كميات كبيرة من البيانات المعقدة والمتنوعة في الوقت الحقيقي، مما يوفر رؤى يمكن أن تحسن نتائج الرعاية الصحية وتزيد من الكفاءة في صناعة الرعاية الصحية. تشمل عدة تطبيقات لـ DL في المعلوماتية الحيوية والطبية المعتمدة على إنترنت الأشياء التشخيص، وتوصية العلاج، ودعم القرار السريري، وتحليل الصور، والمراقبة القابلة للارتداء، واكتشاف الأدوية. تهدف المراجعة إلى تقييم شامل وتجميع الجسم الحالي من الأدبيات حول تطبيق التعلم العميق في تقاطع إنترنت الأشياء مع المعلوماتية الحيوية والطبية. في هذه الورقة، قمنا بتصنيف أحدث حلول DL لمشاكل المعلوماتية الطبية والحيوية إلى خمس فئات بناءً على تقنية DL المستخدمة: الشبكة العصبية التلافيفية، الشبكة العصبية المتكررة، الشبكة التنافسية التوليدية، الإدراك متعدد الطبقات، والأساليب الهجينة. تم تطبيق مراجعة منهجية للأدبيات لدراسة كل واحدة من حيث الخصائص الفعالة، مثل الفكرة الرئيسية، والفوائد، والعيوب، والأساليب، وبيئة المحاكاة، ومجموعات البيانات. بعد ذلك، تم التأكيد على الأبحاث المتقدمة حول أساليب DL وتطبيقاتها لمشاكل المعلوماتية الحيوية. بالإضافة إلى ذلك، تم تناول العديد من التحديات التي ساهمت في تنفيذ DL للمعلوماتية الطبية والحيوية، والتي من المتوقع أن تحفز المزيد من الدراسات لتطوير الأبحاث الطبية والحيوية بشكل تدريجي. وفقًا للنتائج، يتم تقييم معظم المقالات باستخدام ميزات مثل الدقة، والحساسية، والخصوصية،
1 المقدمة
وعلم الأحياء، والرياضيات [10،11]. إنها حاسمة لإدارة البيانات في الطب الحديث وعلم الأحياء [12، 13]. توفر المعلوماتية الحيوية دعمًا كبيرًا للتعامل مع قضايا الوقت والتكلفة في مسارات مختلفة [14، 15]. المعلوماتية الحيوية، المتعلقة بالجينوميات وعلم الوراثة، هي مجال علمي متعدد التخصصات يستخدم تكنولوجيا الكمبيوتر لجمع وتخزين وتقييم وتوزيع البيانات البيولوجية، مثل تسلسلات الحمض النووي والأحماض الأمينية والتعليقات حولها [16، 17].
- إجراء مراجعة منهجية لاستكشاف تطبيقات ML في المعلوماتية الطبية والحيوية المعتمدة على إنترنت الأشياء؛
- تحليل ومقارنة استخدامات DL في المعلوماتية الطبية والحيوية؛
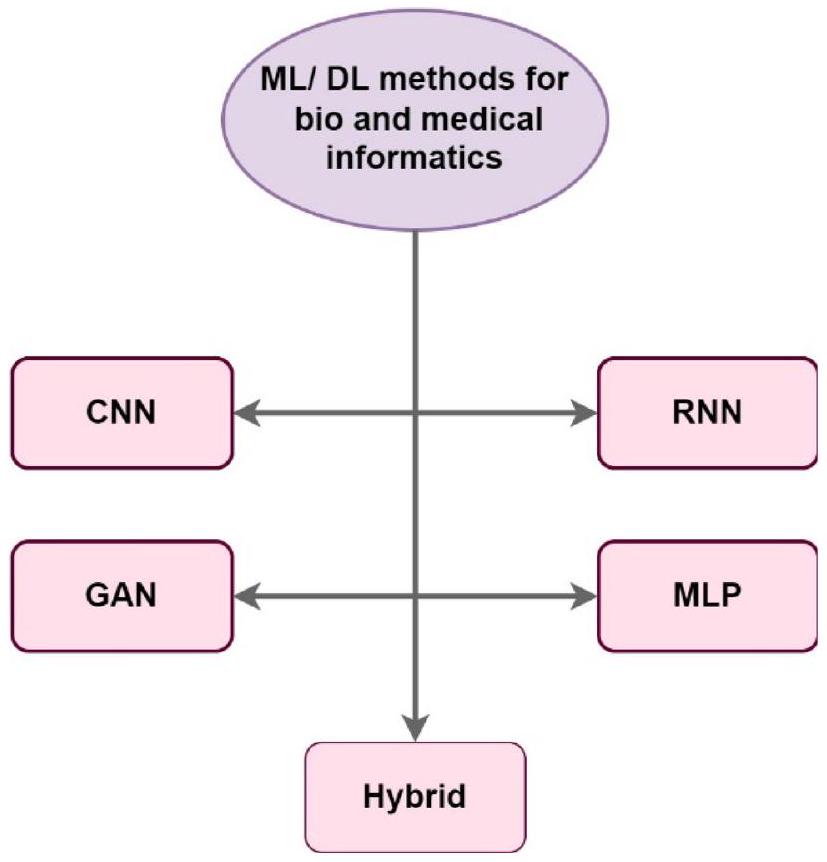
- تصنيف آليات DL إلى خمس مجموعات (CNN، RNN، GAN، MLP، هجينة) وفحص خصائصها؛
- استكشاف منهجيات DL/ML والتطبيقات في المعلوماتية الحيوية؛
- تقديم رؤى للبحوث المستقبلية ومعالجة النقائص الحالية؛
- تقديم تقييم شامل للطرق الحالية لـ ML/DL؛
- المساهمة في فهم أفضل للتحديات والفرص الحالية في هذا المجال;
2 المفاهيم الأساسية والمصطلحات
2.1 مفاهيم التعلم العميق
2.2 تطبيقات المعلوماتية الحيوية
تطبيقات المعلوماتية الحيوية بما في ذلك تسجيل واسترجاع البيانات في العلاج الجيني، والتقييم البيومتري لإدارة المحاصيل، ومكافحة الآفات، والبحث التطوري، واكتشاف الأدوية، والانتفاع الميكروبي [52].
2.3 استخدام التعلم العميق في المعلوماتية الحيوية
2.3.1 اكتشاف الإنزيمات باستخدام الشبكات العصبية متعددة الطبقات
والشبكات العصبية المتكررة. يمكن تدريب هذه الشبكات على مجموعات بيانات كبيرة من بيانات الإنزيمات، ويمكن استخدام النماذج الناتجة لاكتشاف وتصنيف الإنزيمات تلقائيًا في بيانات جديدة [56].
2.3.2 انحدار التعبير الجيني
2.3.3 الشبكات العصبية التلافيفية تتنبأ بنقاط ربط RNA-بروتين
2.3.4 توقع أداء تسلسل DNA باستخدام RNN و CNN
2.3.5 تصنيف الصور الطبية باستخدام ResNet والتعلم الانتقالي
دقة التصنيف مقارنة بخوارزميات التعلم الآلي التقليدية [65].
2.3.6 تضمين الرسوم البيانية باستخدام GCN لتوقع تفاعل البروتين
2.3.7 تحسين دقة الصور باستخدام GAN في علم الأحياء
تستمر هذه العملية التدريبية حتى تنتج الشبكة المولدة صورًا عالية الجودة لا يمكن تمييزها عن الصور الحقيقية عالية الدقة. لتحسين دقة الصور باستخدام GAN في علم الأحياء العديد من التطبيقات، مثل تعزيز دقة الصور المجهرية لتحسين دقة تحليل الصور وتحسين دقة الصور الطبية للمساعدة في التشخيص والعلاج [68].
2.3.8 التشفير التلقائي المتغير لتوليد البيانات البيولوجية عالية الأبعاد وتضمينها
3 مراجعات ذات صلة
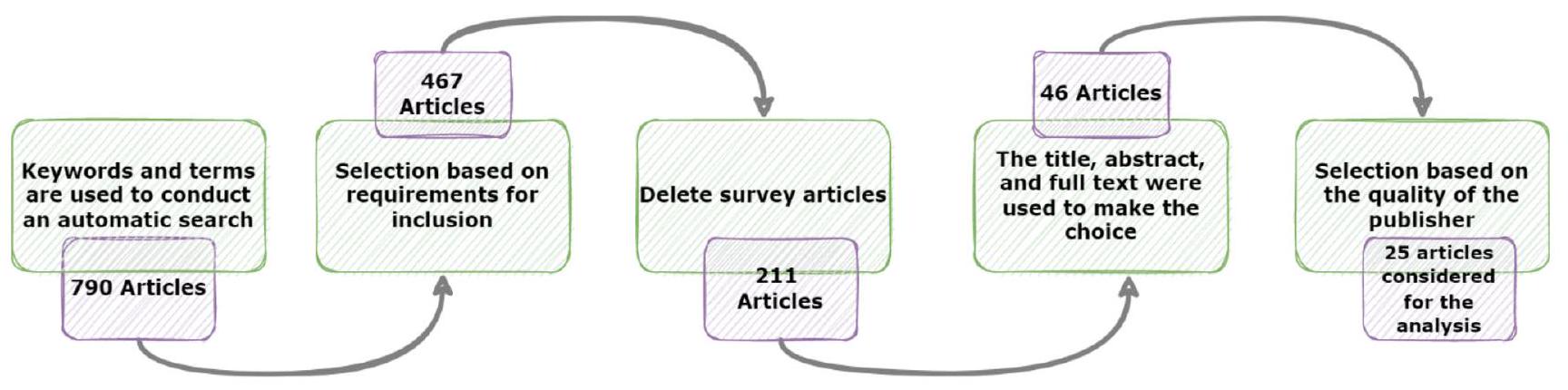
4 منهجية البحث
4.1 صياغة السؤال
- RQ 1: كيف يمكن تصنيف أساليب التعلم العميق في المعلوماتية الحيوية والطبية في الرعاية الصحية الطبية؟ ما هي بعض أمثلتها؟
- RQ 2: ما هي الأعمال الرائدة الأكثر أهمية؟ ما هي فوائدها وعيوبها؟ ما الميزات التي تمتلكها؟
- RQ 3: ما هي التطبيقات والتقنيات والمعايير والعوامل الأخرى الأكثر استخدامًا في المعلوماتية الحيوية والطبية؟
- RQ 4: ما هي الحلول المحتملة الرئيسية والقضايا غير المحلولة في هذا المجال؟
4.2 إجراء استكشاف الأوراق
| المؤلفون | الفكرة الرئيسية | الميزة | العيب |
| لي، هوانغ [19] | تقديم كل من التعريف الخارجي للتعلم العميق ودمج الأمثلة والتنفيذات لاستخداماته التمثيلية في المعلوماتية الحيوية | مقدمة سهلة الفهم للطرق معالجة القضايا من خلال تقديم أمثلة عملية | تم تجاهل بعض المعلمات المهمة للمقارنة بين الطرق |
| ريزيندي، كزافييه [70] | اقتراح دراسة لتعلم تمثيل الرسوم البيانية في المعلوماتية الحيوية، بالإضافة إلى تحديد وتقييم التقنيات | تقديم مسح شامل ومنظم جيدًا لآليات تضمين الرسوم البيانية | مقارنة ضعيفة بين الطرق |
| يي، يو [71] | مقارنة عملية “المحلي لكل مستوى” و”المحلي لكل عقدة” المستخدمة في مجموعتين هيراركية مختلفتين: CATH وBioLip | تقديم مكتبات حسابية لمساعدة المجتمع في عملية اتخاذ القرار لتخطيط البيانات الهرمية | تفاصيل الطرق تم تجاهلها |
| شارما [72] | دمج نتائج متنوعة لتأسيس مجموعات دون الاعتماد على المعايير المستخدمة لتقييم البيانات | مقارنة تخطيطية منظمة جيدًا بين الآليات | تحليل ضعيف للنهج المقترحة |
| سيرا، غالدي [73] | مناقشة تطبيقات التعلم الآلي في المعلوماتية الحيوية وتصوير الأعصاب لحل القضايا ذات الصلة | ذكر عدة أمثلة لتوضيح تطبيق التعلم الآلي في المعلوماتية الحيوية | تجاهل بعض التحديات مثل تفسير نتائج التعلم العميق |
| عملنا | تقديم تصنيف جديد لطرق التعلم العميق/التعلم الآلي في الطب والمعلوماتية الحيوية | مناقشة شاملة لمختلف الدراسات التي تستخدم آليات التعلم العميق في الطب والمعلوماتية الحيوية | عدم توفر الأوراق غير الإنجليزية |
5 طرق تعلم عميق في مجال المعلوماتية الحيوية والطبية
5.1 أساليب الشبكات العصبية التلافيفية في المعلوماتية الحيوية والطبية
| S# | الكلمات الرئيسية ومعايير البحث | S# | الكلمات الرئيسية ومعايير البحث |
| س1 | التعلم العميق” و “المشاكل الطبية | S6 | الذكاء الاصطناعي” و “الرعاية الصحية |
| S2 | التعلم الآلي” و “المعلوماتية الحيوية | S7 | الرعاية الصحية” و “إنترنت الأشياء |
| S3 | التعلم العميق” و “المعلوماتية الحيوية | S8 | طرق التعلم العميق” و “إنترنت الأشياء الطبية |
| S4 | نظام قائم على إنترنت الأشياء” و “المعلوماتية الحيوية | اس9 | طرق التعلم الآلي” و “إنترنت الأشياء الطبية |
| S5 | الذكاء الاصطناعي” و “المعلوماتية الطبية | S10 | طرق الذكاء الاصطناعي” و “إنترنت الأشياء الطبية |

و terminal متنقل لتقييم إمكانياته في الرعاية الصحية السنية المنزلية. علاوة على ذلك، يتم تطوير وترقية معدات الأسنان المتطورة لتشغيل الحصول على صور الأسنان. استنادًا إلى مجموعة بيانات تتكون من 12,600 صورة سريرية تم جمعها بواسطة الجهاز المقدم من 10 عيادات أسنان خاصة، تم تحسين نموذج الكشف التلقائي المدرب بواسطة MASK R-CNN لتحديد وتصنيف 7 أمراض سنية مختلفة، بما في ذلك الأسنان المتدهورة، مرض اللثة، الفلورايد، واللويحات السنية، بدقة كشف تصل إلى
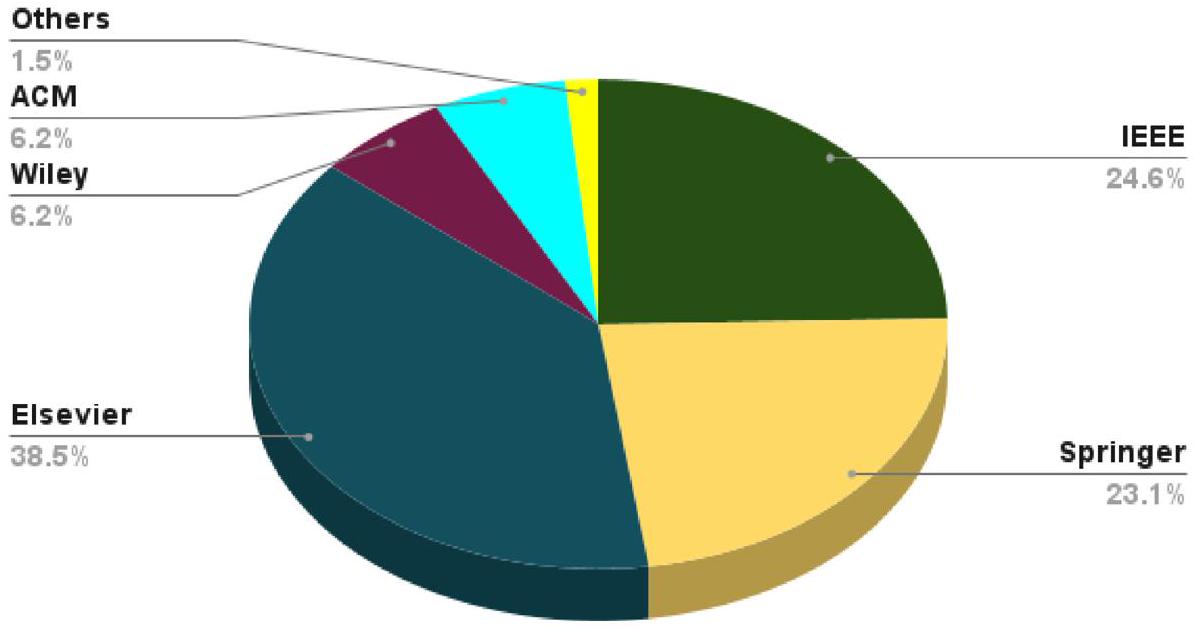



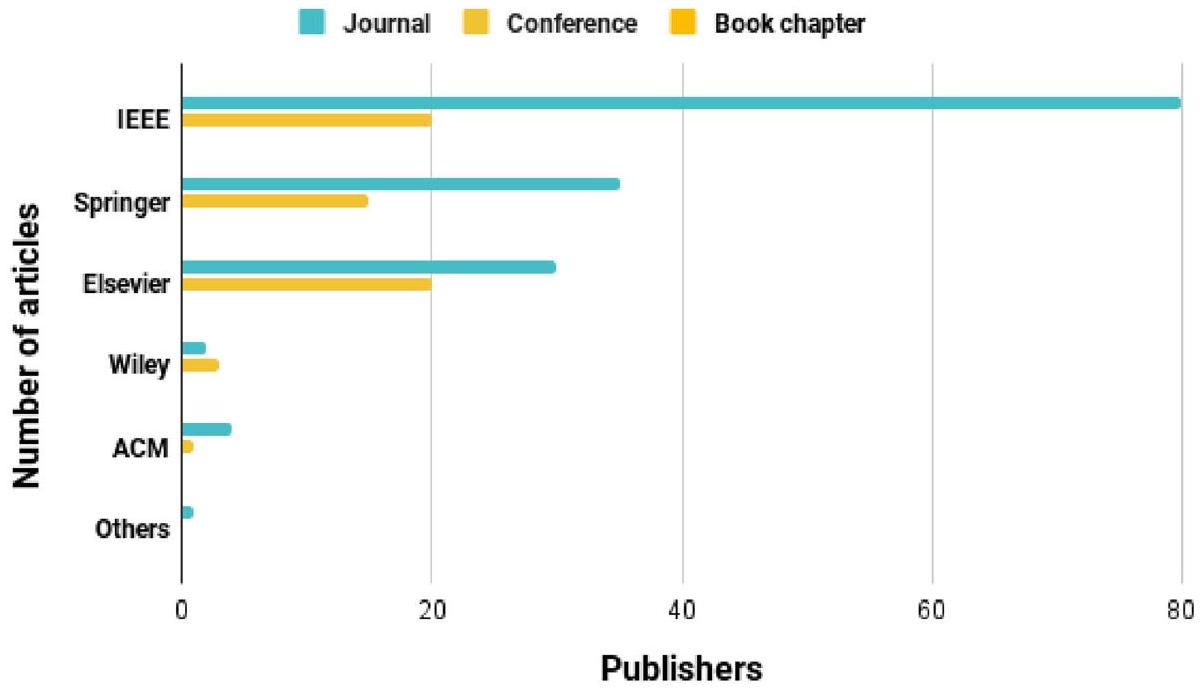
| مؤلف | ناشر | مجلة | استشهاد | ق | بلد | سنة | مؤشر H | |
| 1 | ليو، شو [74] | IEEE | مجلة المعلوماتية الحيوية والصحية | ٥٤ | الربع الأول | الصين | 2019 | ١٣٧ |
| 2 | نيمات زاده، كياني [75] | إلسفير | علم الأحياء الحاسوبي والكيمياء | ١٣ | الربع الثاني | تركيا | ٢٠٢٢ | 61 |
| ٣ | تشن، وانغ [25] | مطبعة جامعة أكسفورد | التقارير في المعلوماتية الحيوية | 14 | الربع الأول | الصين | ٢٠٢١ | 121 |
| ٤ | كومار وشارما [76] | – | المجلة العالمية لتطبيق علم البيانات وإنترنت الأشياء | – | – | روسيا | ٢٠٢١ | – |
| ٥ | جيا، تشين [77] | فرونتيرز ميديا إس. إيه | الحدود في علم الوراثة | ٤ | الربع الأول | الصين | ٢٠٢١ | 93 |
| ٦ | باستورينو وبيسواس [78] | – | المؤتمر الدولي الثالث عشر لجمعية الحوسبة الآلية حول الأنظمة الهجينة: الحوسبة والتحكم | – | – | الولايات المتحدة الأمريكية | 2022 | 14 |
| ٧ | أول، رحمن [79] | مطبعة جامعة أكسفورد | إحاطات في المعلوماتية الحيوية | ٣٨ | الربع الأول | أستراليا | ٢٠٢١ | 121 |
| ٨ | لان، أنت [80] | فرونتيرز ميديا إس. إيه | الحدود في علم الوراثة | ٣٨ | الربع الأول | الصين | 2021 | 121 |
| 9 | هان، رندو [81] | – | طرق الذكاء الحسابي في بيرغامو لعلم المعلومات الحيوية والإحصاء الحيوي | 76 | – | إيطاليا | ٢٠٢١ | – |
| 10 | بالوج، بنسزيك [82] | بيوميد سنترال المحدودة | بي إم سي للمعلوماتية الحيوية | ٥ | الربع الثاني | جائع | 2022 | 218 |
| 11 | جيانسانتي، كاستيلي [83] | – | مؤتمر العلوم والهندسة الحاسوبية الدولي | ٣ | – | إيطاليا | 2019 | – |
| 12 | ليو، تشين [84] | بيوميد سنترال المحدودة | بي إم سي للمعلوماتية الحيوية | 97 | الربع الثاني | الصين | 2017 | 218 |
| ١٣ | العبد، برومبرغ [85] | بيوميد سنترال المحدودة | بي إم سي للمعلوماتية الحيوية | 37 | الربع الثاني | ألمانيا | ٢٠٢٠ | 218 |
| 14 | ليو وقونغ [86] | بيوميد سنترال المحدودة | بي إم سي للمعلوماتية الحيوية | ٢٤ | الربع الثاني | الصين | 2019 | 218 |
| 15 | وانغ، زينغ [87] | IEEE | المؤتمر الدولي IEEE للمعلوماتية الحيوية والطب الحيوي | 161 | – | الصين | 2017 | – |
| 16 | تشاو، شاو [88] | إلسفير | الجينوميات، البروتيوميات والمعلوماتية الحيوية | – | الربع الأول | الولايات المتحدة الأمريكية | ٢٠٢١ | ٥٦ |
| 17 | سوري، غفور [89] | سبرينغر | الحوسبة اللينة | ٥٤ | الربع الثاني | إيران | ٢٠٢٠ | 90 |
| ١٨ | دورازيو، موردوكّا [90] | طبيعة | التقارير العلمية | – | الربع الأول | إيطاليا | ٢٠٢٢ | 242 |
| 19 | كريم، بيان [91] | مطبعة جامعة أكسفورد | التقارير في المعلوماتية الحيوية | ١٠١ | الربع الأول | المملكة المتحدة | ٢٠٢١ | 121 |
| 20 | أيدين [92] | المكتبة العامة للعلوم | بيولوجيا الحوسبة في PLoS | ٣ | الربع الأول | تركيا | ٢٠٢٠ | 191 |
| 21 | محمد شاكيل، باسكار [93] | سبرينغر | مجلة نظم الطب | ٢١٤ | الربع الأول | ماليزيا | 2018 | 89 |
| ٢٢ | هوانغ، شيا [94] | إلسفير | مجلة المعلوماتية الحيوية | 188 | الربع الأول | الصين | 2019 | ١١٢ |
| 23 | وانغ، جيانغ [95] | إلسفير | مجلة المعلوماتية الحيوية | 2 | الربع الأول | الولايات المتحدة الأمريكية | 2021 | ١١٢ |
| ٢٤ | تسوي، زو [96] | إلسفير | مجلة المعلوماتية الحيوية | ٥ | الربع الأول | الولايات المتحدة الأمريكية | ٢٠٢١ | ١١٢ |
| ٢٥ | شاهد، نساجبور [30] | إلسفير | مجلة المعلوماتية الحيوية | ٤٤ | الربع الأول | الولايات المتحدة الأمريكية | ٢٠٢١ | ١١٢ |
مناطق الاهتمام للمرضى الذين ثبتت إصابتهم بفيروس COVID-19، والالتهاب الرئوي البكتيري، والحالات الصحية. كما ناقشوا التحديات التي تواجه تطبيق التعلم العميق في المعلوماتية الحيوية، مثل الحاجة إلى مجموعات بيانات كبيرة، وقابلية التفسير، وجودة البيانات.
5.2 أساليب GAN للمعلوماتية الحيوية والطبية
معلومات من الصور، والتي قد تؤدي إلى تحيز كفاية البيانات. قاموا بتقييم طريقتهم على مجموعة بيانات متاحة للجمهور ووجدوا أنها حققت أداءً قابلاً للمقارنة مع الطرق المتطورة مع الحفاظ على خصوصية البيانات.
| مؤلف | الفكرة الرئيسية | ميزة | عيب | طريقة | بيئة المحاكاة | مجموعة بيانات | ||||
| ليو، شو [74] | اقتراح نظام ذكي لصحة الأسنان يعتمد على الأجهزة الذكية، والتعلم العميق، مما يتيح استكشاف الجدوى | دقة عالية حساسية عالية سبتيكية عالية زمن استجابة منخفض |
|
سي إن إن | تينسورفلو | 10 عيادات أسنان خاصة | ||||
| نيمات زاده، كياني [75] | تقديم استراتيجية لتحسين التعامل مع المعلمات الفائقة لخوارزميات التعلم الآلي |
|
|
سي إن إن | C# | 11 مجموعة بيانات في فئات بيولوجية وطبيعية وطبية حيوية متنوعة | ||||
| تشن، وانغ [25] | استخدام CNNrgb كنموذج حسابي قائم على التعلم العميق لتوقع مواقع nhKcr على البروتينات غير الهيستونية | كفاءة حسابية عالية | مرونة ضعيفة | سي إن إن | بايثون | خادم على الإنترنت يسمى nhKcr | ||||
| كومار وشارما [76] | استخدام تقنية الشبكات العصبية التلافيفية لتشخيص COVID-19 |
|
فقير بشكل مستقل | سي إن إن | بايثون | أشعة الصدر للمرضى المصابين بكوفيد والمرضى غير المصابين بكوفيد | ||||
| جيا، تشين [77] | استخدام بيانات التعبير الجيني من الأومنيبوس وملفات التعبير الجيني من أطلس جينوم السرطان للتمييز بين مرضى سرطان الثدي والأفراد الأصحاء |
|
مرونة ضعيفة | سي إن إن | ر | 1109 مريض سرطان و113 حالة طبيعية |
5.3 أساليب الشبكات العصبية المتكررة للمعلومات الحيوية والطبية
يمكن استخدام نموذج الشبكة لتصنيف الكيانات المسماة بدون الحاجة إلى هندسة ميزات بشرية. استنادًا إلى نتائجهم التجريبية، يمكن استخدام تمثيل الكلمات المدرب مسبقًا الخاص بالنطاق وتمثيل المستوى الحرفي لإنشاء وظيفة نهج LSTM-RNN.
| المؤلف | الفكرة الرئيسية | الميزة | العيب | الطريقة | بيئة المحاكاة | مجموعة البيانات | ||||
| باستورينو وبيسواس [78] | تقديم GAN شبه مشرف مع بيانات معتمة لتطوير عملية التصنيف |
|
تعقيد عالٍ | GAN | بايثون | 1000 دورة من نموذج SGAN | ||||
| أوول، رحمن [79] | تقديم طريقة توزيع بيتا-ثنائي لرسم الإمكانية المناعية للببتيد |
|
|
GAN | بايثون | 9000 اختبارًا لمجموعة جزيئية مناعية | ||||
| لان، يو [80] | استخدام طريقة قائمة على GAN لاستعادة قسم التصوير بالرنين المغناطيسي للدماغ المجاور |
|
إعادة وتعرف عام ضعيف | GAN | تينسور فلو |
|
||||
| هان، رندو [81] | تطوير برنامج يقوم بتشغيل أداة توقع الارتباط لتوقع PPI باستخدام التعلم الآلي |
|
قابلية توسيع ضعيفة | GAN | بايثون | شبكة PPI من قاعدة بيانات STRING | ||||
| بالوغ، بنسزيك [82] | تصميم GAN شبه مشرف مع بيانات معتمة لتحسين عملية التصنيف |
|
مرونة ضعيفة | GAN | بايثون | مجموعة بيانات ChIP-seq وDNase-seq |
5.4 طرق MLP للمعلوماتية الحيوية والطبية
| المؤلف | الفكرة الرئيسية | الميزة | العيب | الطريقة | بيئة المحاكاة | مجموعة البيانات | ||
| جيانسانتي، كاستيلي [83] | تدريب خمسة نماذج من مجالات التعلم الآلي والتعلم العميق لفحص احتمال اكتشاف تفاعلات miRNA-mRNA |
|
توفر ضعيف | RNN | بايثون | TargetScan miRanda RNAhybrid | ||
| ليو، تشين [84] | اقتراح إطار عمل RNN قائم على التضمين وتمثيل الأحرف |
|
مرونة ضعيفة | RNN | C++ | BioCreative GM | ||
| العبد، برومبرغ [85] | استخدام نماذج DL متعددة لإظهار أن التعلم من النهاية إلى النهاية قابل للمقارنة مع الترميز | مرونة عالية |
|
RNN | تينسور فلو | تفاعل PeptideHLA II | ||
| ليو وغونغ [86] | اقتراح LSTM معزز بالانتباه مع نموذج متبقي لمعالجة مشاكل تفاعل البروتينات | دقة عالية | قابلية التكيف ضعيفة | RNN | بايثون | 1H9D | ||
| وانغ وزينغ [87] | تقديم إطار عمل DL لتوقع مواقع الفسفرة العامة والمحددة للكيناز | دقة عالية | قابلية تفسير ضعيفة | RNN | بايثون | NetPhos3.1 |
5.5 الأساليب الهجينة للمعلوماتية الحيوية والطبية
| مؤلف | الفكرة الرئيسية | ميزة | عيب | طريقة | بيئة المحاكاة | مجموعة بيانات | ||||
| تشاو، شاو [88] | اقتراح مجموعة من أساليب التحسين لكل تفسير على معمارية MLP و CNN | دقة عالية | قابلية التوسع الضعيفة | MLP | بايتورتش | 19,241 جين | ||||
| سوري، غفور [89] | اقتراح نمط مراقبة قائم على إنترنت الأشياء لتنظيم علامات الحياة للطلاب بشكل مستمر |
|
ضعف التكيف | MLP | تينسورفلو | 1100 طالب | ||||
| دوارازيو، موردوكّا [90] | اقتراح بأن منصة MLP متاحة لدراسات الفينوميات حول كيفية استجابة خلايا السرطان للعلاج |
|
ضعف التكيف | MLP | ماتلاب | ريسنت101 | ||||
| كريم، بيان [91] | اختيار الجينات المرتبطة بالسرطان لتصنيف السرطان | دقة عالية | قابلية التوسع الضعيفة | MLP | بايتورتش | 400 صورة | ||||
| أيدين [92] | تدريب ستة نماذج تعلم آلي للكشف عن T4SEs |
|
ضعف التكيف | MLP | تينسورفلو | PSI-BLAST HHblits |
المعلومات. كما قدموا تحليلًا نظريًا للمخطط المقترح وأظهروا فعاليته من خلال التجارب باستخدام مجموعات بيانات GWAS الحقيقية. قد تكون طريقتهم مفيدة لضمان موثوقية وأمان حسابات GWAS المعتمدة على الخارج، والتي أصبحت شائعة بشكل متزايد في البحث الطبي الحيوي.
النهج التي تم استخدامها لمعالجة هذه التحديات، مثل تطوير نماذج تنبؤية لانتشار المرض وشدته، وتحديد عوامل الخطر المرتبطة بالمرض، وتطوير طرق لتحليل الصور والبيانات الطبية. تشير الجدول 8 إلى التقنيات والخصائص والسمات لطرق معلومات MLP.
6 النتائج والمقارنات
الفئات. بالإضافة إلى ذلك، نحث الباحثين على التعمق أكثر في هذه المواضيع. أظهر تقييمنا للاستطلاع أن معظم الأبحاث الطبية والبيوانفورماتية تركزت على مزيج مختار من مهام التعلم أو تحسين بروتوكولات التوصيف ومجموعات البيانات الجديدة. لقد حظيت تقنيات التعلم الآلي بشعبية كبيرة وقبول، خاصةً لتطبيقها مع طرق الشبكات العصبية التلافيفية، التي أظهرت نتائج ممتازة. ومع ذلك، هناك بعض القيود التي تعيق تحقيق نفس مستوى الفعالية في التطبيقات الطبية والبيوانفورماتية. بشكل عام، لا يزال البحث في هذا المجال مستمراً. واحدة من أبرز القضايا هي ندرة مجموعات البيانات الكبيرة التي تحتوي على أنماط عالية الجودة لأغراض التدريب. في مثل هذه الحالات، قد يكون دمج البيانات قابلاً للتطبيق لجمع المعلومات من مصادر متعددة. ومن الجدير بالذكر أنه مع زيادة حجم البيانات، تزداد الحاجة إلى مجموعات بيانات أكبر لضمان أن ينتج التعلم الآلي نتائج موثوقة.
6.1 تحليل النتائج
| المؤلف | الفكرة الرئيسية | الميزة | العيب | الطريقة | بيئة المحاكاة | مجموعة البيانات | ||
| محمد شاكيل، باسكار [93] | تطبيق شبكة عصبية عميقة لتطوير فعالية نظام بيانات الصحة المعتمد على إنترنت الأشياء |
|
مرونة ضعيفة | شبكة عصبية عميقة | N2 | محركات معيار ISO/IEC/JTC1/SC 31 | ||
| هوانغ، شيا [94] | اقتراح نظام اتحادي قائم على المجتمع لتصنيف البيانات الموزعة |
|
قابلية التكيف ضعيفة | تعلم الآلة | بايثون | السجلات الطبية الإلكترونية من 50 مستشفى | ||
| وانغ، جيانغ [95] | اقتراح خوارزميتين لتوفير SNPs اصطناعية | دقة عالية | قابلية التكيف ضعيفة | تعلم عميق | НарМар | 89 موضوعًا و83,354 SNPs | ||
| تسوي، زو [96] | تقديم آلية للتدريب في حالة عدم وجود محلل مركزي موثوق | دقة عالية | قابلية التعديل ضعيفة | تعلم الآلة | بايثون | 30,760 بيانات مرضى | ||
| شاهيد، نسا جپور [30] | اقتراح إطار لحماية البيانات الطبية من التهديدات الخارجية |
|
قابلية التعديل ضعيفة | تعلم الآلة | بايثون | – |
من الدراسات قدمت الوصول إلى الشيفرات المصدرية، بشكل رئيسي في MATLAB وPython، لتسهيل إعادة الإنتاج والتجريب الإضافي. كانت MATLAB خيارًا شائعًا، خاصة في الدراسات التي تركز على معالجة الإشارات وتحليل الصور، نظرًا لأدواتها الواسعة المخصصة لهذه المجالات. على العكس من ذلك، كانت بايثون بارزة في الأبحاث التي دمجت أطر تعلم الآلة مثل TensorFlow وKeras، مما يتماشى مع الاتجاه الأوسع في مجتمع تعلم الآلة. من الجدير بالذكر أن العديد من الأوراق التي تمت مراجعتها تضمنت مقتطفات من الشيفرات وجعلت تنفيذاتها الكاملة متاحة على المستودعات العامة، مما يعزز البحث التعاوني ونشر المعرفة في هذا المجال متعدد التخصصات. لعبت هذه التوافر للشيفرات دورًا محوريًا في تعزيز قابلية تطبيق منهجيات التعلم العميق وإمكانية الوصول إليها في سياق المعلوماتية الحيوية والطبية المعتمدة على إنترنت الأشياء.
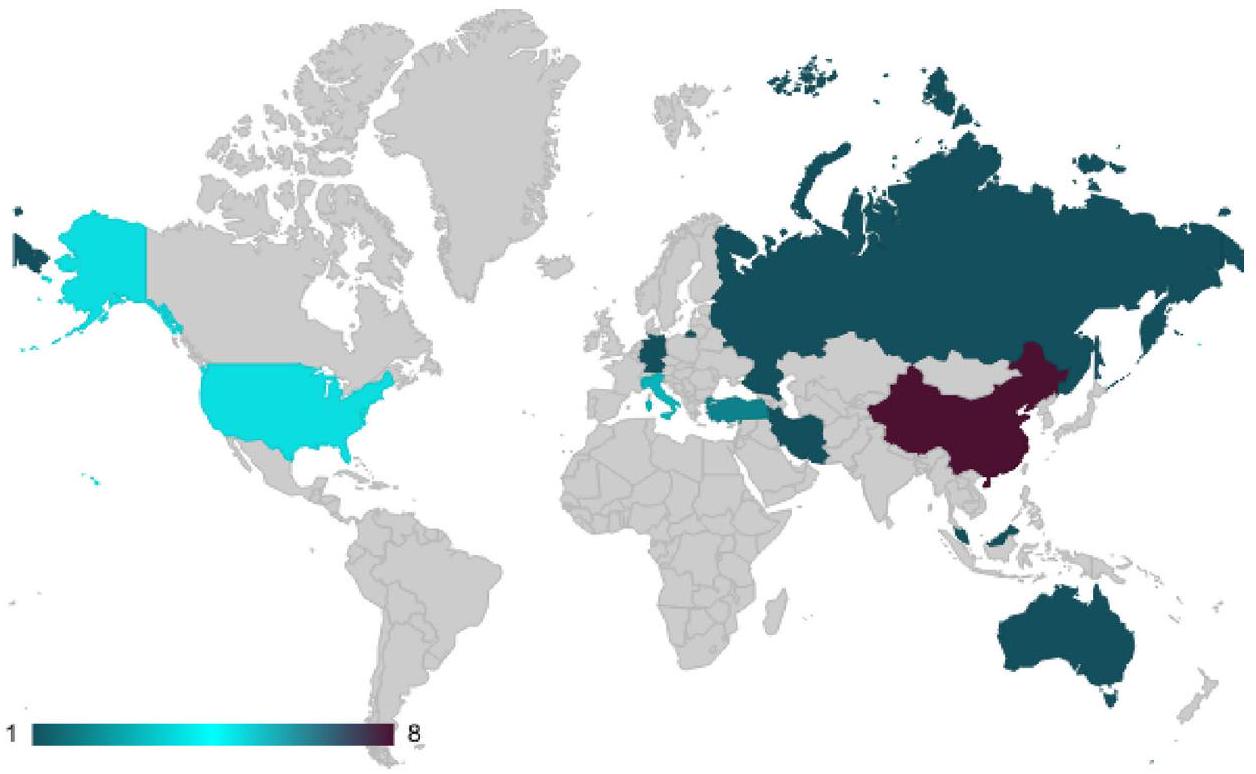
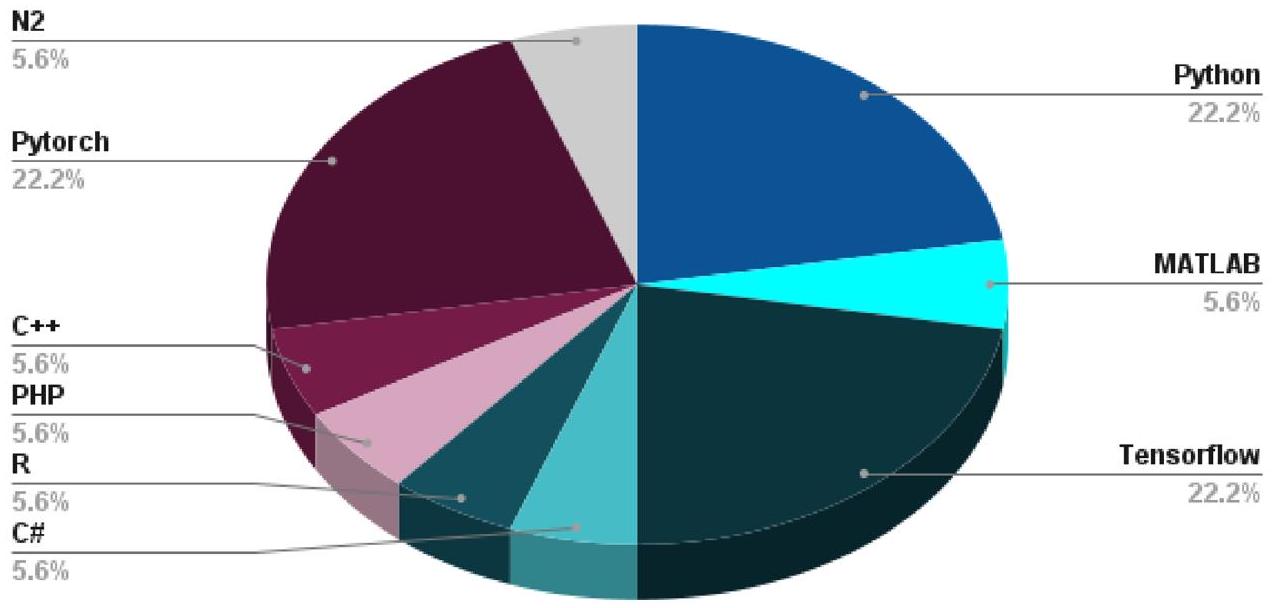

والتي يمكن أن يكون لها آثار كبيرة في بيئات الرعاية الصحية.
| النوع | المؤلفون | قابلية التوسع | الدقة | الدقة | معدل F | الحساسية | الخصوصية | الصلابة | قابلية التكيف |
| شبكة عصبية تلافيفية | ليو، شو [74] |
|
|
|
|
|
|
|
|
| نيمات زاده، كياني [75] |
|
|
|
|
|
|
|
|
|
| تشن، وانغ [25] |
|
|
|
|
|
|
|
|
|
| كومار وشيرما [76] |
|
|
|
|
|
|
|
|
|
| جيا، تشن [77] |
|
|
|
|
|
|
|
|
|
| شبكة عصبية متكررة | باستورينو وبيسواس [78] |
|
|
|
|
|
|
|
|
| أولول، رحمن [79] |
|
|
|
|
|
|
|
|
|
| لان، يو [80] |
|
|
|
|
|
|
|
|
|
| هان، روند [81] |
|
|
|
|
|
|
|
|
|
| بالوغ، بنسزيك [82] |
|
|
|
|
|
|
|
|
|
| شبكة توليد عدائية | جيانسانتي، كاستيلي [97] |
|
|
|
|
|
|
|
|
| ليو، تشن [98] |
|
|
|
|
|
|
|
|
|
| العبيد، برومبرغ [99] |
|
|
|
|
|
|
|
|
|
| ليو وغونغ [100] |
|
|
|
|
|
|
|
|
|
| وانغ، زينغ [87] |
|
|
|
|
|
|
|
|
|
| شبكة عصبية متعددة الطبقات | تشاو، شاو [88] |
|
|
|
|
|
|
|
|
| سوري، غفور [89] |
|
|
|
|
|
|
|
|
|
| دورازيو، موردوككا [90] |
|
|
|
|
|
|
|
|
|
| كريم، بيان [91] |
|
|
|
|
|
|
|
|
|
| أيدين [92] |
|
|
|
|
|
|
|
|
|
| هجين | محمد شاكيل، باسكار [101] |
|
|
|
|
|
|
|
|
| هوانغ، شيا [94] |
|
|
|
|
|
|
|
|
|
| وانغ، جيانغ [95] |
|
|
|
|
|
|
|
|
|
| تسوي، زهو [96] |
|
|
|
|
|
|
|
|
|
| شاهيد، نساجبور [30] |
|
|
|
|
|
|
|
|
6.2 استكشاف دمج ML في التطبيقات الطبية
والذي يجب أخذه في الاعتبار عند تطوير نماذج GAN المدركة للخصوصية [102]. استخدموا نهج المعلوماتية الحيوية وML لتحديد أهداف وطرق الأدوية المحتملة لعلاج COVID-19. تم دمج نهج واحد لمجموعات بيانات متعددة من الأومكس لبناء شبكة جزيئية، والتي استخدمت لتحديد وحدات الجينات المهمة. أظهرت مراجعة شاملة لتطبيقات GAN في المعلوماتية الحيوية إمكانياتها في تحليل الصور الطبية واكتشاف الأدوية [103]. تفوق MADGAN المقترح في القسم 5 على طرق الكشف عن الشذوذ الأخرى ويمكن استخدامه للكشف المبكر عن الأمراض العصبية. تم تطوير نموذج فعال لتوقع الروابط لشبكات تفاعل البروتينات باستخدام معلومات طوبولوجية في إطار GAN. تفوق النموذج على طرق تحليل الشبكات التقليدية ويمكن استخدامه لتحديد أهداف الأدوية المحتملة للأمراض المرتبطة بتفاعلات البروتينات. تسلط هذه الدراسات الضوء على إمكانيات GAN وML في البحث الطبي، لا سيما في اكتشاف الأمراض، واكتشاف الأدوية، وتحليل شبكات تفاعل البروتينات [104].
وقد تؤدي إلى تطوير أدوات أكثر دقة وكفاءة للتطبيقات الطبية والمعلوماتية الحيوية. يمكن أن تؤدي تقنيات DL ودمج مصادر بيانات متعددة في نماذج ML إلى توقعات أكثر دقة وتعزيز أداء هذه النماذج [108].
6.3 معايير التقييم السائدة
الدقة
6.4 تحديات تطبيقات DL في المعلوماتية الحيوية والطبية المعتمدة على إنترنت الأشياء
النطاق، حيث تكون البيانات غالبًا نادرة ومكلفة للحصول عليها. يمكن أن يؤدي ذلك إلى مشكلات مثل التحيز وقلة التنوع في مجموعة البيانات. تم تصميم الشبكات العصبية التلافيفية بشكل أساسي لبيانات الصور، وتطبيقها على أنواع بيانات أخرى، مثل البيانات الزمنية أو النصية، محدود. يمكن أن تكون هذه تحديًا في المعلوماتية الحيوية والطبية المعتمدة على إنترنت الأشياء، حيث قد تكون البيانات غير متجانسة ومتعددة الأنماط. يمكن أن تكون الشبكات العصبية التلافيفية عرضة للهجمات العدائية، حيث يمكن أن تؤدي التغيرات الصغيرة في المدخلات إلى تصنيف خاطئ. يمكن أن يكون هذا مقلقًا بشكل خاص في التطبيقات الطبية، حيث يمكن أن تكون التنبؤات غير الصحيحة لها عواقب وخيمة. باختصار، بينما أظهرت الشبكات العصبية التلافيفية نجاحًا ملحوظًا في تحليل الصور الطبية، إلا أن لديها عدة قيود تحتاج إلى معالجة لتحسين فعاليتها في المعلوماتية الحيوية والطبية المعتمدة على إنترنت الأشياء.
تنبؤات النموذج [118]. يمكن أن تكون البيانات الطبية صاخبة وتحتوي على تباينات بسبب اختلافات في أجهزة الاكتساب، والبروتوكولات، وظروف المرضى. يمكن أن تكون مثل هذه التباينات صعبة الحساب، مما يؤدي إلى انخفاض أداء الشبكات العصبية المتكررة (RNNs). من ناحية أخرى، تفتقر جمع البيانات الطبية والتعليق عليها إلى التوحيد القياسي، مما يجعل من الصعب تطوير الشبكات العصبية المتكررة التي تعمم بشكل جيد عبر المؤسسات. غالبًا ما تعاني البيانات الطبية من عدم توازن الفئات، حيث تحتوي فئة واحدة (مثل، إيجابي المرض) على عدد أقل بكثير من الأمثلة مقارنة بالفئة الأخرى (مثل، سلبي المرض). يمكن أن تؤدي هذه المشكلة إلى أداء ضعيف للشبكات العصبية المتكررة وتتطلب اهتمامًا خاصًا للتعامل معها. يمكن أن تكون الشبكات العصبية المتكررة مكلفة حسابيًا، حيث تتطلب موارد حسابية كبيرة للتدريب والنشر. يمكن أن تعيق محدودية توفر الحوسبة عالية الأداء تطوير ونشر الشبكات العصبية المتكررة في البيئات ذات الموارد المحدودة. يثير استخدام الشبكات العصبية المتكررة في الرعاية الصحية اعتبارات أخلاقية، مثل الموافقة المستنيرة، والخصوصية، والتحيز. إن معالجة هذه القضايا أمر ضروري لضمان أن استخدام الشبكات العصبية المتكررة في التطبيقات الطبية يكون أخلاقيًا وعادلاً [119]. بشكل عام، تسلط هذه التحديات الضوء على الحاجة إلى النظر بعناية في تطبيق الشبكات العصبية المتكررة في المعلوماتية الحيوية والطبية المعتمدة على إنترنت الأشياء وأهمية معالجة التحديات الفريدة في هذا المجال.
يجب معالجة هذه القضايا لجعل الشبكات التنافسية التوليدية (GANs) أكثر فعالية في هذه السياقات، لا سيما في البيئات السحابية. ومع ذلك، تعتمد الشبكات التنافسية التوليدية بشكل كبير على بيانات عالية الجودة للتدريب، وتكون جودة البيانات حاسمة بشكل خاص في المعلوماتية الحيوية والطبية. في المعلوماتية الحيوية والطبية المعتمدة على إنترنت الأشياء، يمكن أن تكون البيانات مشوشة وغير مكتملة ومتحيزة، مما يجعل من الصعب تدريب الشبكات التنافسية التوليدية بدقة. تتضمن المعلوماتية الحيوية والطبية المعتمدة على إنترنت الأشياء بيانات حساسة للمرضى، والتي يجب أن تظل خاصة وآمنة. ومع ذلك، تتطلب الشبكات التنافسية التوليدية كميات كبيرة من البيانات للتدريب، مما يشكل خطرًا على خصوصية المرضى وأمانهم. لذلك، يجب أن تكون هناك تدابير قوية لأمان البيانات عند استخدام الشبكات التنافسية التوليدية في هذا السياق. يجب على الباحثين إيجاد طرق لجعل نماذج الشبكات التنافسية التوليدية أكثر قابلية للتفسير. واحدة من التحديات الكبيرة في المعلوماتية الحيوية والطبية هي توفر مجموعة بيانات محدودة. يمكن أن تؤثر البيانات المحدودة على دقة نتائج نموذج الشبكات التنافسية التوليدية، وفي بعض الحالات، قد لا يكون من الممكن تدريب نموذج الشبكات التنافسية التوليدية بمجموعة بيانات محدودة. صناعة الرعاية الصحية تخضع لتنظيمات صارمة، ويجب أن تمتثل الشبكات التنافسية التوليدية للمتطلبات التنظيمية لتكون معتمدة للاستخدام. يمكن أن يكون ضمان الامتثال للوائح تحديًا عند العمل مع الشبكات التنافسية التوليدية، خاصة عند التعامل مع المعلوماتية الحيوية والطبية المعتمدة على إنترنت الأشياء، حيث تكون مخاوف أمان البيانات وخصوصيتها مرتفعة. باختصار، بينما تقدم الشبكات التنافسية التوليدية إمكانات كبيرة للمعلوماتية الحيوية والطبية المعتمدة على إنترنت الأشياء، لا تزال هناك بعض التحديات التي يجب معالجتها لجعلها أكثر فعالية وقبولًا للاستخدام في هذا السياق. تشمل هذه التحديات جودة البيانات، وخصوصية وأمان البيانات، وقابلية التفسير، ومجموعات البيانات المحدودة، والامتثال التنظيمي.
البيانات متعددة الأبعاد. يمكن أن تكون هذه قيودًا في المعلوماتية الحيوية والطبية، حيث يمكن أن تكون البيانات عالية الأبعاد. بينما أظهرت الشبكات العصبية متعددة الطبقات نتائج متفوقة في المعلوماتية الحيوية والطبية المعتمدة على إنترنت الأشياء، من الضروري مراعاة قيودها واستكشاف نماذج بديلة يمكن أن تعالج هذه التحديات بشكل أفضل.
تثير خوارزميات التعلم العميق في الرعاية الصحية اعتبارات أخلاقية، مثل الموافقة المستنيرة، والخصوصية، والتحيز. إن معالجة هذه القضايا أمر ضروري لضمان أن استخدام نماذج التعلم العميق في التطبيقات الطبية يكون أخلاقياً وعادلاً. يمكن أن يكون تطوير ونشر نماذج التعلم العميق مكلفاً، مما يجعل من الصعب تنفيذها في البيئات ذات الموارد المحدودة. علاوة على ذلك، قد تتطلب نماذج التعلم العميق أجهزة وبرامج متخصصة، مما يزيد من تكلفتها.
6.5 مجموعة البيانات في المعلوماتية الطبية والبيولوجية باستخدام أساليب التعلم العميق
تشخيص الأمراض بدقة، وتوقع نتائج العلاج، وتحديد أهداف الأدوية المحتملة. علاوة على ذلك، يعتمد نجاح نماذج التعلم العميق بشكل كبير على جودة وتنوع البيانات المستخدمة لتدريبها. يمكن أن تؤدي مجموعة بيانات متحيزة أو غير مكتملة أو غير تمثيلية للسكان المستهدفين إلى نتائج متحيزة أو غير دقيقة. لذلك، من الضروري التأكد من أن مجموعات بيانات المعلوماتية الطبية الحيوية متنوعة وتمثل السكان المستهدفين وعالية الجودة. علاوة على ذلك، فإن استخدام مجموعات بيانات موحدة أمر حاسم لتسهيل المقارنة وإعادة إنتاج نتائج البحث عبر دراسات مختلفة. تتيح مجموعات البيانات الموحدة للباحثين تقييم أداء نماذجهم مقابل الآخرين باستخدام نفس البيانات، مما يسهل تطوير خوارزميات ومنهجيات جديدة ومحسنة. باختصار، تعتبر مجموعات البيانات عالية الجودة وشاملة ومتنوعة وموحدة ضرورية لتطوير وتقييم نماذج التعلم العميق في المعلوماتية الطبية الحيوية المعتمدة على إنترنت الأشياء. إنها توفر الأساس للتشخيص الدقيق وعلاج الأمراض وتحديد أهداف الأدوية الجديدة. إن تطبيق مجموعات البيانات في مجال التعلم العميق للمعلوماتية الطبية الحيوية المعتمدة على إنترنت الأشياء أمر حاسم لتطوير نماذج دقيقة وفعالة. بدون مجموعات بيانات موحدة، لا يمكن للنماذج التعلم وإجراء توقعات دقيقة. واحدة من التحديات الرئيسية في تطوير نماذج التعلم العميق للمعلوماتية الطبية الحيوية هي توفر مجموعات البيانات المعلّمة. تعتبر مجموعات البيانات المعلّمة حاسمة للتعلم تحت الإشراف، وهو النهج الأكثر شيوعًا في التعلم العميق. وذلك لأن نماذج التعلم العميق تحتاج إلى كميات كبيرة من البيانات المعلّمة لتعلم الأنماط والعلاقات المعقدة في البيانات. في المعلوماتية الطبية الحيوية، غالبًا ما يتم إنشاء هذه المجموعات المعلّمة من خلال التوصيف اليدوي أو من قبل خبراء في المجال. يمكن استخدام العديد من مجموعات البيانات المتاحة للجمهور في المعلوماتية الطبية الحيوية لتطبيقات التعلم العميق، مثل مجموعة بيانات MIMIC-III للسجلات الصحية الإلكترونية، ومجموعة بيانات ImageNet للتصوير الطبي، ومجموعة بيانات PhysioNet للإشارات الفسيولوجية. تم استخدام هذه المجموعات لتطوير نماذج لمجموعة متنوعة من التطبيقات مثل تشخيص الأمراض، واكتشاف الأدوية، والطب الشخصي. يتطلب استخدام مجموعات البيانات في تطبيقات التعلم العميق للمعلوماتية الطبية الحيوية أيضًا اهتمامًا دقيقًا بخصوصية البيانات وأمانها. تعتبر بيانات المرضى حساسة للغاية ويجب التعامل معها بعناية لحماية خصوصية المرضى. يجب على الباحثين التأكد من أن مجموعات البيانات المستخدمة لتدريب نماذجهم تتوافق مع المتطلبات الأخلاقية والقانونية وأن البيانات تم إزالة تعريفها قبل الاستخدام. يجب على الباحثين اختيار مجموعات البيانات ومعالجتها بعناية، والامتثال للمتطلبات الأخلاقية والقانونية، والتعامل مع بيانات المرضى بعناية كبيرة لحماية خصوصية المرضى.
تسجيلات تخطيط القلب الكهربائي (ECG)، كل منها يمتد على 10 ثوانٍ ومأخوذة بمعدل 500 هرتز، مما أسفر عن إجمالي 50,000 نقطة بيانات لكل تسجيل. تتكون مجموعة بيانات ملحوظة أخرى في أبحاث الأعصاب من 500 مريض مصاب بمرض باركنسون، مما أسفر عن أكثر من 150,000 نقطة بيانات لكل مريض عبر قراءات مستشعرات مختلفة. بالإضافة إلى ذلك، دمجت مجموعة بيانات شاملة لتوقع مرض الزهايمر بيانات متعددة الأنماط، بما في ذلك صور الرنين المغناطيسي الهيكلي من 1000 موضوع، جنبًا إلى جنب مع التقييمات الديموغرافية والمعرفية. توضح هذه التفاصيل الكمية الطبيعة الغنية والمتنوعة لمجموعات البيانات في هذا المجال، والتي تلعب دورًا محوريًا في تدريب وتقييم نماذج التعلم العميق لتطبيقات المعلوماتية الطبية الحيوية ضمن إطار إنترنت الأشياء.
عدد أمثلة المعلومات مثل عدد خصائص البيانات.
6.6 تطبيقات إنترنت الأشياء باستخدام طرق التعلم العميق في المعلوماتية الحيوية والطبية
6.7 قضايا الأمان، التحديات، المخاطر، إنترنت الأشياء، واستخدام البلوكشين
| الاسم | الأوصاف |
| MNIST | مجموعة بيانات MNIST شائعة في تطبيقات رؤية الكمبيوتر، بما في ذلك التعلم العميق. تتكون من مجموعة تضم 70,000 رقم مكتوب بخط اليد، كل منها مع
|
| سيفار-10 وسيفار-100 | تستخدم هذان المجموعتان من البيانات بشكل شائع في مهام تصنيف الصور في التعلم العميق. تتكون مجموعة بيانات CIFAR-10 من
|
| إيميج نت | ImageNet هو تحدٍ كبير في التعرف على الصور يتكون من أكثر من 14 مليون صورة في 21,000 فئة. تم استخدام هذه المجموعة من البيانات في تطبيقات التعلم العميق المختلفة، بما في ذلك المعلوماتية الحيوية والطبية. على سبيل المثال، تم استخدامها لتدريب نماذج التعلم العميق لتصنيف آفات الجلد أو تشخيص الأمراض بناءً على الصور الطبية. |
| فيزيو نت | PhysioNet هو مجموعة بيانات للإشارات الفسيولوجية تشمل تخطيط القلب الكهربائي، وتخطيط الدماغ الكهربائي، وعلامات الحياة. تم استخدام هذه المجموعة في تطبيقات التعلم العميق في المعلوماتية الحيوية والطبية لمهام مثل تشخيص الأمراض، وتوقع نتائج المرضى، واكتشاف الأنماط غير الطبيعية في الإشارات الفسيولوجية. |
| ميميك-III | MIMIC-III هو قاعدة بيانات للرعاية الحرجة متاحة للجمهور تحتوي على بيانات صحية مجهولة الهوية لأكثر من 40,000 مريض. تتضمن هذه المجموعة من البيانات معلومات مثل العلامات الحيوية، ونتائج المختبر، والتاريخ الطبي. وقد تم استخدامها في تطبيقات التعلم العميق للتنبؤ بنتائج المرضى، وتحديد عوامل خطر الأمراض، وتحسين اتخاذ القرارات السريرية. |
| TCGA | أطلس جينوم السرطان (TCGA) هو مجموعة من البيانات الجينومية، والإبيجينومية، والترانسكريبتومية من أكثر من 30 نوعًا من السرطان. تم استخدام هذه المجموعة من البيانات في تطبيقات التعلم العميق لتشخيص السرطان، وتوقع نتائج المرضى، وتحديد العلاجات الجديدة. |
| الأهداف. MIMIC-III | قاعدة بيانات المعلومات الطبية للعناية المركزة (MIMIC-III) هي مجموعة بيانات كبيرة متاحة مجانًا تتكون من سجلات صحية إلكترونية مجهولة الهوية لأكثر من 50,000 مريض تم إدخالهم إلى وحدات العناية الحرجة في مستشفى كبير للرعاية الثلاثية. تحتوي مجموعة البيانات على بيانات سريرية مثل العلامات الحيوية، ونتائج المختبر، والأدوية، والبيانات السكانية، مما يجعلها موردًا قيمًا للبحث في العناية الحرجة واتخاذ القرارات السريرية. |
| مجموعة بيانات الأشعة السينية للصدر من المعهد الوطني للصحة | مجموعة بيانات الأشعة السينية للصدر من المعاهد الوطنية للصحة هي مجموعة تضم أكثر من 100,000 صورة أشعة سينية للصدر مصنفة مع مجموعة متنوعة من الأمراض الصدرية مثل الالتهاب الرئوي، والسل، وسرطان الرئة. تعتبر مجموعة البيانات هذه موردًا قيمًا للبحث في التشخيص المدعوم بالحاسوب، وتصنيف الأمراض، وتحليل الصور. |
| فيزيو نت | مجموعة بيانات فيزيو نت هي مجموعة من الإشارات الفسيولوجية والبيانات السريرية ذات الصلة مثل تخطيط القلب الكهربائي (ECG) وتخطيط الدماغ الكهربائي (EEG) وتسجيلات ضغط الدم. تعتبر مجموعة البيانات هذه موردًا قيمًا لأبحاث تشخيص الأمراض والمراقبة والتنبؤ. |
| أدني | مجموعة بيانات مبادرة تصوير الأعصاب لمرض الزهايمر (ADNI) هي مجموعة من بيانات التصوير العصبي الطولية والبيانات السريرية وبيانات العلامات الحيوية من الأفراد المصابين بمرض الزهايمر، والضعف الإدراكي الخفيف، والأشخاص الأصحاء. تعتبر مجموعة البيانات هذه موردًا قيمًا لتشخيص المرض، والتنبؤ، وأبحاث العلاج. |
| رائي | مجموعة بيانات المراقبة، الوبائيات، والنتائج النهائية (SEER) هي سجل سرطان قائم على السكان يجمع البيانات السريرية والديموغرافية وبيانات البقاء على قيد الحياة من مرضى السرطان في الولايات المتحدة. تعتبر مجموعة البيانات هذه موردًا قيمًا لأبحاث تشخيص السرطان وعلاجه وتحليل البقاء على قيد الحياة. |
تقدم تقنية السجل طريقة آمنة وغير قابلة للتلاعب لتخزين ومشاركة البيانات. تحقق ذلك من خلال استخدام خوارزميات التشفير واللامركزية لضمان أن البيانات المخزنة على سلسلة الكتل غير قابلة للتغيير وشفافة. في سياق المعلومات الحيوية والطبية المعتمدة على إنترنت الأشياء، يمكن استخدام سلسلة الكتل لتأمين البيانات التي تولدها أجهزة إنترنت الأشياء وضمان سلامتها وموثوقيتها وخصوصيتها. على سبيل المثال، يمكن استخدام سلسلة الكتل لإنشاء سجل آمن وغير قابل للتلاعب لجميع البيانات التي تولدها أجهزة إنترنت الأشياء، والتي يمكن الوصول إليها فقط من قبل الأطراف المصرح لها. بالإضافة إلى ذلك، يمكن أن تنفذ سلسلة الكتل آليات مشاركة بيانات آمنة وتحافظ على الخصوصية بين مقدمي الرعاية الصحية والباحثين. ومع ذلك، فإن استخدام سلسلة الكتل في هذا السياق يأتي أيضًا مع تحدياته ومخاطره الخاصة. على سبيل المثال، قد لا تكون متطلبات سلسلة الكتل العالية من حيث الحوسبة والتخزين قابلة للتطبيق على أجهزة إنترنت الأشياء ذات الموارد المحدودة. بالإضافة إلى ذلك،
يمكن أن تجعل عدم قابلية التغيير في تقنية البلوكشين من الصعب تصحيح الأخطاء أو تحديث البيانات، مما قد يكون مشكلة في سياق البيانات الطبية التي قد تحتاج إلى تحديث أو تصحيح مع مرور الوقت. أخيرًا، يثير استخدام البلوكشين أيضًا مخاوف بشأن خصوصية البيانات وسرية المعلومات، حيث يمكن أن يكون من الصعب ضمان عدم مشاركة البيانات الطبية الحساسة أو الوصول إليها من قبل أطراف غير مصرح لها. لذلك، بينما تقدم تقنية البلوكشين حلاً واعدًا لتأمين تطبيقات المعلوماتية الحيوية والطبية المعتمدة على إنترنت الأشياء، من المهم النظر بعناية في تطبيقها وتقييم المخاطر والفوائد قبل التنفيذ.
البيانات بأمان، مما يسمح للمرضى بالتحكم في من لديه حق الوصول إلى بياناتهم ومنح الإذن لمقدمي الرعاية الصحية للوصول إليها، وبالتالي حماية خصوصيتهم. ومع ذلك، فإن تقنية البلوكشين في الرعاية الصحية تقدم أيضًا تحديات مثل قابلية التوسع، التي تتطلب قوة حسابية كبيرة وسعة تخزين لإدارة كميات كبيرة من البيانات التي تنتجها أجهزة إنترنت الأشياء. بالإضافة إلى ذلك، فإن نقص معايير التوافق بين شبكات البلوكشين المختلفة والأجهزة الطبية يجعل من الصعب دمجها.
6.8 نماذج التعلم العميق القادمة
7 قضايا مفتوحة وتحديات رئيسية
التوصيات. تركز الأبحاث الجارية على تطوير نماذج التعلم العميق الأكثر قابلية للتفسير. علاوة على ذلك، تولد المعلومات الحيوية والطبية المعتمدة على إنترنت الأشياء كميات هائلة من البيانات الحساسة، مما يعرضها لخطر كبير من خروقات البيانات عند استخدام نماذج التعلم العميق، مما يستلزم اتخاذ تدابير أمنية قوية لمنع الوصول غير المصرح به أو السرقة أو التلاعب بالبيانات. ومع ذلك، فإن تطوير واختبار نماذج التعلم العميق مقيد بنقص في مجموعات البيانات الطبية عالية الجودة. علاوة على ذلك، تثار مخاوف أخلاقية تتعلق بخصوصية المرضى، والموافقة المستنيرة، والتحيز عند استخدام نماذج التعلم العميق في التطبيقات الطبية، مما يستلزم تطوير إرشادات وتنظيمات لضمان الاستخدام الأخلاقي. بالإضافة إلى ذلك، فإن دمج هذه النماذج في سير العمل السريري وتثقيف الأطباء حول استخدامها الفعال وتفسير النتائج يمثل تحديًا كبيرًا لاعتمادها في البيئات السريرية. قضية أخرى هي صعوبة نماذج التعلم العميق في التعميم على بيانات جديدة تتجاوز بيانات التدريب، وهو أمر حاسم في التطبيقات الطبية للتعميم على مجموعات المرضى الجديدة أو أنواع الأمراض. يتطلب معالجة هذه القضايا المفتوحة التعاون بين الباحثين والأطباء وصانعي السياسات. إذا تم التعامل معها بشكل مناسب، يمكن أن تحدث نماذج التعلم العميق ثورة في مجال المعلومات الحيوية والطبية المعتمدة على إنترنت الأشياء، مما يؤدي إلى تحسين نتائج المرضى.
7.1 التحديات الرئيسية في البحث
7.1.1 القابلية للتفسير
لتوصيف [157]. إن نقص القابلية للتفسير هو عقبة كبيرة في علم الأحياء الحاسوبي، حيث إن موثوقية نموذج التعلم العميق ضرورية لتطبيقات اتخاذ القرارات السريرية الحساسة. من المهم بنفس القدر أن نفهم لماذا يمكن لنموذج ما أن يقدم توقعات دقيقة كما هو مهم أن نفهم كيف يقوم بإجراء تلك التوقعات في علم الأحياء. على سبيل المثال، في توقع وظيفة البروتين وبنيته، يجب أن نفهم السياسات التي تتحكم في هندسة البروتين ثلاثية الأبعاد وخصائصه. إن معالجة هذه المشكلات أمر حاسم لتوفير رؤى بيولوجية واتخاذ قرارات عملية في البيئات السريرية.
7.1.2 التدريب الفعال
7.1.3 أمان البيانات والخصوصية
7.1.4 التشغيل البيني وتكامل البيانات
7.1.5 المراقبة والتشخيص في الوقت الحقيقي
جمع البيانات وتحليلها بشكل مستمر، مما يسمح بالتشخيص والعلاج في الوقت المناسب للحالات الطبية. على سبيل المثال، يمكن أن تراقب المستشعرات القابلة للارتداء العلامات الحيوية مثل معدل ضربات القلب، ضغط الدم، وتشبع الأكسجين في الوقت الحقيقي، مما يوفر تدفقات بيانات مستمرة لتحليلها بواسطة نماذج التعلم العميق. يمكن أن تحدد هذه النماذج بعد ذلك الأنماط والشذوذ التي قد تشير إلى مشكلة طبية محتملة، مما يسمح بالتدخل والعلاج المبكر. يمكن أن تحسن المراقبة والتشخيص في الوقت الحقيقي أيضًا نتائج المرضى من خلال تمكين خطط علاج شخصية. من خلال جمع وتحليل البيانات باستمرار حول حالة المريض، يمكن لنماذج التعلم العميق تحديد أساليب علاج فردية تتناسب مع احتياجات المريض المحددة. ومع ذلك، هناك أيضًا تحديات في تنفيذ المراقبة والتشخيص في الوقت الحقيقي في المعلوماتية الطبية والبيولوجية المعتمدة على إنترنت الأشياء. تشمل هذه الحاجة إلى نقل بيانات آمن وموثوق، دمج البيانات من مصادر متعددة، وتطوير نماذج تعلم عميق فعالة وقابلة للتفسير يمكن أن توفر تشخيصات دقيقة وفي الوقت المناسب. تعتبر المراقبة والتشخيص في الوقت الحقيقي منطقة تطبيق حاسمة للتعلم العميق في المعلوماتية الطبية والبيولوجية المعتمدة على إنترنت الأشياء. يتضمن ذلك جمع البيانات باستمرار من مستشعرات وأجهزة متنوعة، معالجتها في الوقت الحقيقي باستخدام نماذج التعلم العميق وتقديم ملاحظات فورية للمهنيين الطبيين أو المرضى. أحد الأمثلة على المراقبة والتشخيص في الوقت الحقيقي هو في الأجهزة القابلة للارتداء التي تجمع بيانات عن معدل ضربات القلب، ضغط الدم، وغيرها من العلامات الحيوية. يمكن لنماذج التعلم العميق تحليل هذه البيانات في الوقت الحقيقي وتنبيه المهنيين الطبيين إذا تم اكتشاف أي شذوذ أو anomalies. يمكن أن يساعد ذلك المهنيين الطبيين في اتخاذ تدخلات في الوقت المناسب ومنع النتائج الصحية السلبية. مثال آخر هو في التصوير الطبي، حيث يمكن لنماذج التعلم العميق تحليل الصور الطبية في الوقت الحقيقي وتقديم تشخيصات سريعة ودقيقة. يمكن أن يكون هذا مفيدًا بشكل خاص في حالات الطوارئ حيث يجب اتخاذ قرارات سريعة بناءً على معلومات محدودة. تمتلك المراقبة والتشخيص في الوقت الحقيقي القدرة على تحسين نتائج المرضى وتقليل تكاليف الرعاية الصحية من خلال تمكين التدخلات المبكرة ومنع الأحداث السلبية. ومع ذلك، فإنه يقدم أيضًا تحديات تتعلق بخصوصية البيانات وأمانها والحاجة إلى نماذج تعلم عميق قوية وموثوقة يمكن أن تعمل في الوقت الحقيقي. يتطلب ذلك استخدام الحوسبة عالية الأداء وتقنيات التعلم الآلي المتقدمة.
7.1.6 التحليلات التنبؤية
تحديد الأنماط وإجراء توقعات حول الأحداث الصحية المستقبلية من خلال تحليل البيانات من مصادر متنوعة، مثل الأجهزة الطبية، السجلات الصحية الإلكترونية، وبيانات المرضى التي تم إنشاؤها. على سبيل المثال، يمكن استخدام التحليلات التنبؤية لتحديد المرضى الذين هم في خطر مرتفع لتطوير مرض أو حالة معينة، مما يسمح للأطباء بالتدخل مبكرًا ومنع ظهور المرض. بالإضافة إلى توقع الأحداث الصحية المستقبلية، يمكن أيضًا استخدام التحليلات التنبؤية لتحسين خطط العلاج وتحسين نتائج المرضى. من خلال تحليل البيانات من مرضى سابقين لديهم حالات مشابهة، يمكن لنماذج التعلم العميق تحديد أكثر خيارات العلاج فعالية للمرضى الفرديين وتقديم توصيات علاج شخصية. يمكن أن تستفيد المراقبة والتشخيص في الوقت الحقيقي بشكل كبير من استخدام التحليلات التنبؤية، حيث يسمح للأطباء باتخاذ تدابير استباقية لمنع الأحداث الصحية السلبية وتحسين نتائج المرضى. ومع ذلك، من المهم أن نلاحظ أن التحليلات التنبؤية دقيقة فقط بقدر دقة البيانات التي تستند إليها. لذلك، من الضروري ضمان أن البيانات المستخدمة لتدريب واختبار نماذج التعلم العميق دقيقة وتمثل وتكون خالية من التحيز.
7.1.7 الاعتبارات الأخلاقية والقانونية
7.1.8 التفاعل بين الإنسان والحاسوب
7.1.9 قابلية التوسع والعمومية
تحسن قابلية التوسع والعمومية للنماذج. على سبيل المثال، يُعتبر التعلم بالنقل تقنية تسمح للنماذج بإعادة استخدام الميزات المتعلمة من مهمة إلى أخرى، مما يقلل من كمية البيانات المطلوبة للتدريب ويحسن العمومية. بالإضافة إلى ذلك، يُعتبر التعلم الفيدرالي تقنية تسمح بتدريب النماذج على مجموعات بيانات موزعة، مما يقلل من كمية البيانات التي تحتاج إلى النقل ويحسن قابلية التوسع. إن معالجة قضايا قابلية التوسع والعمومية أمر حاسم لنشر نماذج التعلم العميق بنجاح في المعلوماتية الحيوية والطبية المعتمدة على إنترنت الأشياء [163].
7.2 الأعمال المستقبلية
7.2.1 دمج البيانات متعددة الأنماط
7.2.2 التعلم الفيدرالي
مدى أدائه في تطبيقات أخرى، مثل علم الجينوم أو اتخاذ القرارات السريرية. يمكن أن تبحث الأبحاث المستقبلية في أداء التعلم الفيدرالي في تطبيقات طبية مختلفة. يجب أن تكون الاتصالات بين الأجهزة في إعداد التعلم الفيدرالي آمنة لضمان خصوصية المرضى ومنع تسرب البيانات. يمكن أن تركز الأبحاث المستقبلية على تطوير بروتوكولات اتصال تكون آمنة وفعالة، مما يسمح بالتعلم الفيدرالي الفعال عبر مجموعة واسعة من التطبيقات الطبية. غالبًا ما تأتي البيانات الطبية من مجموعة متنوعة من المصادر وبأشكال مختلفة، مما يجعل من الصعب دمجها للاستخدام في التعلم الفيدرالي. يمكن أن تركز الأبحاث المستقبلية على تطوير تقنيات لمعالجة تباين البيانات، مثل تطبيع البيانات وزيادة البيانات، لتحسين فعالية التعلم الفيدرالي. الهدف النهائي من التعلم الفيدرالي في المعلوماتية الحيوية والطبية المعتمدة على إنترنت الأشياء هو تحسين نتائج المرضى. يمكن أن تركز الأبحاث المستقبلية على تطوير أطر لنشر نماذج التعلم الفيدرالي في الممارسة السريرية، بما في ذلك كيفية دمجها بفعالية في سير العمل السريري الحالي.
7.2.3 الذكاء الاصطناعي القابل للتفسير
7.2.4 التعلم المنقول
7.2.5 مراقبة الرعاية الصحية الشخصية
مصادر متعددة مثل الأجهزة القابلة للارتداء، وأجهزة الاستشعار الطبية، والسجلات الصحية الإلكترونية. يمكن استخدام تقنيات التعلم العميق لدمج هذه البيانات لرؤية شاملة لحالة صحة الفرد. يمكن أن يساعد دمج البيانات متعددة الأنماط باستخدام التعلم العميق في تحسين دقة وموثوقية أنظمة رصد الرعاية الصحية المخصصة. يعد اكتشاف الشذوذ جانبًا مهمًا من رصد الرعاية الصحية المخصصة حيث يساعد في تحديد الأنماط غير العادية في حالة صحة الفرد. يمكن استخدام تقنيات التعلم العميق لتحديد هذه الأنماط وإطلاق الإنذارات إذا لزم الأمر. يمكن أن يكون هذا مفيدًا بشكل خاص في اكتشاف الأمراض المزمنة أو الطوارئ الصحية المفاجئة. يمكن تحقيق رصد حالة صحة الفرد في الوقت الحقيقي باستخدام الأجهزة القابلة للارتداء وأجهزة الاستشعار المدعومة من إنترنت الأشياء. يمكن نشر نماذج التعلم العميق على هذه الأجهزة لمراقبة حالة صحة الفرد باستمرار وتقديم تنبيهات في الوقت الحقيقي إذا لزم الأمر. يمكن أن يكون هذا مفيدًا بشكل خاص للمرضى المسنين أو ذوي المخاطر العالية.
7.2.6 التشخيص الفوري وتخطيط العلاج
لتقديم تشخيص فوري. يمكن نشر هذه النماذج على الأجهزة المدعومة من إنترنت الأشياء لتقديم ملاحظات فورية لمقدمي الرعاية الصحية. يمكن أن يكون هذا مفيدًا بشكل خاص في حالات الطوارئ حيث يكون التشخيص السريع أمرًا حاسمًا. يمكن استخدام نماذج التعلم العميق لتطوير خطط علاج مخصصة للمرضى. يمكن أن تأخذ هذه النماذج في الاعتبار التاريخ الطبي للفرد، والمعلومات الجينية، وعوامل أخرى لتقديم توصيات علاجية مخصصة. يمكن استخدام الأجهزة المدعومة من إنترنت الأشياء لمراقبة استجابة المريض للعلاج وضبط خطة العلاج وفقًا لذلك. يمكن أن تساعد أنظمة دعم القرار باستخدام التعلم العميق مقدمي الرعاية الصحية في اتخاذ قرارات مستنيرة بشأن التشخيص والعلاج. يمكن أن تقدم هذه الأنظمة توصيات بناءً على بيانات المرضى، والإرشادات الطبية، ومعلومات أخرى ذات صلة. يمكن أن تساعد التحليلات التنبؤية باستخدام التعلم العميق في التنبؤ باستجابة المريض للعلاج والمخاطر الصحية المحتملة. يمكن تدريب هذه النماذج على مجموعات بيانات كبيرة من السجلات الطبية لتحديد الأنماط والتنبؤ بالمشكلات الصحية المحتملة. يمكن أن يكون هذا مفيدًا بشكل خاص في الرعاية الصحية الوقائية. يمكن تدريب نماذج التعلم العميق لتحليل الصور الطبية مثل الأشعة السينية، والرنين المغناطيسي، والأشعة المقطعية. يمكن أن تساعد هذه النماذج مقدمي الرعاية الصحية في تحديد الشذوذ وتشخيص الأمراض. يمكن استخدام الأجهزة المدعومة من إنترنت الأشياء لالتقاط ونقل هذه الصور في الوقت الحقيقي، مما يمكّن من التشخيص عن بُعد وتخطيط العلاج. تعتبر الخصوصية والأمان من القضايا الرئيسية في التشخيص الفوري وتخطيط العلاج. يمكن استخدام نماذج التعلم العميق لضمان خصوصية وأمان بيانات المرضى. يمكن استخدام تقنيات مثل التعلم الفيدرالي لتدريب النماذج على مجموعات بيانات موزعة دون المساس بالخصوصية. في التشخيص الفوري وتخطيط العلاج، من المهم تقديم نماذج قابلة للتفسير لفوز ثقة المرضى ومقدمي الرعاية الصحية. يمكن استخدام تقنيات الذكاء الاصطناعي القابلة للتفسير لتقديم رؤى حول كيفية عمل هذه النماذج. هذه مجرد بعض من الأعمال والأفكار المستقبلية التي يمكن استكشافها في التشخيص الفوري وتخطيط العلاج باستخدام التعلم العميق والمعلوماتية الحيوية والطبية المعتمدة على إنترنت الأشياء. مع تزايد توفر بيانات الرعاية الصحية وتقدم تقنيات التعلم العميق، فإن التشخيص الفوري وتخطيط العلاج لديه القدرة على تحويل الطريقة التي نقدم بها الرعاية الصحية [165].
7.2.7 الصيانة التنبؤية للأجهزة الطبية
تحليل البيانات من أجهزة الاستشعار مثل درجة الحرارة، والضغط، والاهتزاز لاكتشاف الشذوذ والفشل المحتمل. يمكن أن يساعد اكتشاف الشذوذ باستخدام التعلم العميق في تحديد الأنماط غير العادية في بيانات الأجهزة الطبية. يمكن أن تساعد هذه النماذج في اكتشاف المشكلات التي قد لا تكون واضحة على الفور للعين البشرية وإطلاق الإنذارات إذا لزم الأمر. يمكن استخدام نماذج التنبؤ باستخدام التعلم العميق للتنبؤ بالعمر المتبقي المفيد للأجهزة الطبية. يمكن أن تساعد هذه النماذج مقدمي الرعاية الصحية في التخطيط لصيانة واستبدال الأجهزة الطبية قبل أن تفشل. يمكن أن تساعد جدولة الصيانة التنبؤية باستخدام التعلم العميق مقدمي الرعاية الصحية في تحسين جداول الصيانة بناءً على معدلات الفشل المتوقعة للأجهزة الطبية. يمكن أن يساعد ذلك في تقليل وقت التوقف وتحسين موثوقية الأجهزة الطبية. يمكن أن تساعد تشخيص الأعطال باستخدام التعلم العميق مقدمي الرعاية الصحية في تحديد وتشخيص المشكلات بسرعة مع الأجهزة الطبية. يمكن أن تحلل هذه النماذج بيانات الاستشعار وتقدم توصيات بالإصلاح أو الاستبدال. يمكن دمج نماذج الصيانة التنبؤية مع السجلات الصحية الإلكترونية لرؤية أداء الأجهزة الطبية ونتائج المرضى بشكل شامل. يمكن أن يساعد ذلك مقدمي الرعاية الصحية في اتخاذ قرارات مستنيرة بشأن إدارة الأجهزة الطبية ورعاية المرضى. هذه مجرد بعض من الأعمال والأفكار المستقبلية التي يمكن استكشافها في الصيانة التنبؤية للأجهزة الطبية باستخدام التعلم العميق والمعلوماتية الحيوية والطبية المعتمدة على إنترنت الأشياء. مع تزايد استخدام الأجهزة الطبية والحاجة إلى تقديم رعاية صحية موثوقة وآمنة، يمكن أن تحسن الصيانة التنبؤية من كفاءة وفعالية أنظمة الرعاية الصحية.
7.2.8 تحسين اكتشاف الأدوية
استجابة المريض للعلاج وتحديد المجموعات الفرعية التي من المرجح أن تستفيد من دواء ما. يمكن استخدام نماذج التعلم العميق لتطوير خطط علاج شخصية بناءً على المعلومات الجينية للفرد، والتاريخ الطبي، وعوامل أخرى. يمكن أن تتنبأ هذه النماذج بفعالية الأدوية المختلفة وتساعد مقدمي الرعاية الصحية في اتخاذ قرارات علاج مستنيرة. يمكن دمج نماذج التعلم العميق مع السجلات الصحية الإلكترونية لتوفير رؤية شاملة لصحة المريض ونتائج العلاج. يمكن أن يساعد ذلك مقدمي الرعاية الصحية في اتخاذ قرارات مستنيرة بشأن علاج الأدوية ورعاية المرضى. هذه مجرد بعض الأعمال والأفكار المستقبلية التي يمكن استكشافها في تحسين اكتشاف الأدوية باستخدام التعلم العميق والمعلوماتية الحيوية والطبية المعتمدة على إنترنت الأشياء. مع الطلب المتزايد على أدوية جديدة وفعالة، فإن تحسين اكتشاف الأدوية لديه القدرة على تحويل صناعة الأدوية وتحسين نتائج المرضى.
7.2.9 تحليل الصور الطبية
المعلوماتية الطبية المعتمدة على إنترنت الأشياء. مع الاستخدام المتزايد للتصوير الطبي في الرعاية الصحية، فإن تحليل الصور الطبية لديه القدرة على تحسين دقة وكفاءة التشخيص وتخطيط العلاج.
7.2.10 مراقبة الصحة باستخدام أجهزة إنترنت الأشياء القابلة للارتداء والتعلم العميق
7.2.11 الطب عن بُعد
يمكن أن تساعد المعلومات الحيوية والطبية في تحسين جودة خدمات الطب عن بُعد وتعزيز نتائج المرضى. يمكن تدريب نماذج التعلم العميق لتحليل بيانات المرضى عن بُعد مثل الصور الطبية ونتائج المختبرات والعلامات الحيوية. يمكن أن يساعد ذلك في تحسين دقة وسرعة التشخيص، خاصة في المناطق التي تعاني من نقص في الوصول إلى المتخصصين في الرعاية الصحية. يمكن استخدام نماذج التعلم العميق لتطوير روبوتات محادثة ومساعدين افتراضيين يمكنهم التواصل مع المرضى وتقديم المشورة الطبية. يمكن أن يساعد ذلك في تحسين وصول المرضى إلى خدمات الرعاية الصحية وتقليل عبء العمل على المتخصصين في الرعاية الصحية. يمكن استخدام الأجهزة القابلة للارتداء المعتمدة على إنترنت الأشياء لمراقبة بيانات صحة المرضى عن بُعد مثل معدل ضربات القلب وضغط الدم ومعدل التنفس. يمكن لنماذج التعلم العميق تحليل هذه البيانات في الوقت الفعلي وتنبيه المتخصصين في الرعاية الصحية إذا كانت هناك أي تغييرات تتطلب الانتباه. يمكن استخدام نماذج التعلم العميق لتحليل بيانات المرضى لتحديد المرضى الذين هم في خطر تطوير أمراض معينة. يمكن أن يساعد ذلك المتخصصين في الرعاية الصحية على تقديم رعاية استباقية ومنع تقدم المرض. يمكن استخدام نماذج التعلم العميق لتطوير خطط علاج شخصية بناءً على بيانات المرضى. يمكن أن يساعد ذلك في تحسين نتائج العلاج وتقليل تكاليف الرعاية الصحية من خلال تجنب العلاجات غير الضرورية. يمكن استخدام نماذج التعلم العميق لتطوير أنظمة فرز آلية يمكنها تحديد المرضى الذين يحتاجون إلى رعاية عاجلة. يمكن أن يساعد ذلك في تقليل أوقات الانتظار للمرضى الذين يحتاجون إلى اهتمام فوري. يمكن دمج خدمات الطب عن بُعد مع السجلات الصحية الإلكترونية لتوفير رؤية شاملة لصحة المرضى ونتائج العلاج. يمكن أن يساعد ذلك مقدمي الرعاية الصحية في اتخاذ قرارات مستنيرة بشأن رعاية المرضى. هذه بعض من الأعمال والأفكار المستقبلية التي يمكن استكشافها في الطب عن بُعد مع دمج التعلم العميق والمعلومات الحيوية والطبية المعتمدة على إنترنت الأشياء. مع الطلب المتزايد على خدمات الطب عن بُعد، فإن دمج هذه التقنيات لديه القدرة على تحسين الوصول إلى خدمات الرعاية الصحية وتعزيز نتائج المرضى.
7.2.12 التحليلات التنبؤية للرعاية الصحية
خطط علاج شخصية بناءً على بيانات المرضى. يمكن أن يساعد ذلك في تحسين نتائج العلاج وتقليل تكاليف الرعاية الصحية من خلال تجنب العلاجات غير الضرورية. يمكن استخدام التحليلات التنبؤية لتحسين موارد الرعاية الصحية مثل أسرة المستشفيات والموظفين والمعدات. يمكن استخدام نماذج التعلم العميق للتنبؤ بطلب المرضى وتحسين تخصيص الموارد وفقًا لذلك. يمكن استخدام نماذج التعلم العميق لتحليل بيانات المرضى لتحديد التفاعلات المحتملة بين الأدوية والأحداث السلبية. يمكن أن يساعد ذلك مقدمي الرعاية الصحية في تقديم علاجات دوائية أكثر أمانًا وفعالية. يمكن استخدام نماذج التعلم العميق لتطوير أنظمة دعم القرار السريري التي يمكن أن تساعد مقدمي الرعاية الصحية في اتخاذ قرارات مستنيرة بشأن رعاية المرضى. يمكن أن يساعد ذلك في تحسين نتائج المرضى وتقليل تكاليف الرعاية الصحية من خلال تجنب الاختبارات والعلاجات غير الضرورية. يمكن استخدام نماذج التعلم العميق لتحليل بيانات صحة السكان لتحديد الاتجاهات الصحية وتفشي الأمراض. يمكن أن يساعد ذلك مقدمي الرعاية الصحية في تطوير تدخلات مستهدفة لمنع انتشار الأمراض. هذه بعض من الأعمال والأفكار المستقبلية التي يمكن استكشافها في التحليلات التنبؤية للرعاية الصحية مع دمج التعلم العميق والمعلومات الحيوية والطبية المعتمدة على إنترنت الأشياء. مع الطلب المتزايد على التحليلات التنبؤية في الرعاية الصحية، فإن دمج هذه التقنيات لديه القدرة على تحسين نتائج المرضى وتقليل تكاليف الرعاية الصحية.
8 الخاتمة والقيود
الدقة، والحساسية، والخصوصية، ودرجة F، والقدرة على التكيف، وقابلية التوسع، والزمن. ومع ذلك، فإن بعض الميزات، مثل الأمان ووقت التقارب، لا تُستخدم بشكل كافٍ. لتقييم وتنفيذ الطرق المقترحة، يتم استخدام لغات برمجة متنوعة. علاوة على ذلك، نتوقع أن يوفر تحقيقنا دليلًا قيمًا لمزيد من الأبحاث حول استخدام التعلم العميق في القضايا الطبية والمعلومات الحيوية.
الإعلانات
الوصول المفتوح هذه المقالة مرخصة بموجب رخصة المشاع الإبداعي للاستخدام والمشاركة والتكيف والتوزيع وإعادة الإنتاج في أي وسيلة أو صيغة، طالما أنك تعطي الائتمان المناسب للمؤلفين الأصليين والمصدر، وتوفر رابطًا لرخصة المشاع الإبداعي، وتوضح ما إذا كانت هناك تغييرات قد تم إجراؤها. الصور أو المواد الأخرى من طرف ثالث في هذه المقالة مشمولة في رخصة المشاع الإبداعي للمقالة، ما لم يُشار إلى خلاف ذلك في سطر ائتمان للمادة. إذا لم تكن المادة مشمولة في رخصة المشاع الإبداعي للمقالة واستخدامك المقصود غير مسموح به بموجب اللوائح القانونية أو يتجاوز الاستخدام المسموح به، فستحتاج إلى الحصول على إذن مباشرة من صاحب حقوق الطبع والنشر. لعرض نسخة من هذه الرخصة، قم بزيارةhttp://creativecommons. org/licenses/by/4.0/.
References
- Muhammad AN et al (2021) Deep learning application in smart cities: recent development, taxonomy, challenges and research prospects. Neural Comput Appl 33(7):2973-3009
- Nosratabadi S et al (2020) State of the art survey of deep learning and machine learning models for smart cities and urban sustainability. In: International conference on global research and education. Springer
- Shafqat
et al (2022) Standard ner tagging scheme for big data healthcare analytics built on unified medical corpora. J Artif Intell Technol 2(4):152-157 - Atitallah SB et al (2020) Leveraging deep learning and iot big data analytics to support the smart cities development: review and future directions. Comput Sci Rev 38:100303
- Kök I, Şimşek MU, Özdemir (2017) A deep learning model for air quality prediction in smart cities. In: 2017 IEEE international conference on big data (Big Data). 2017. IEEE
- Bolhasani H, Mohseni M, Rahmani AM (2021) Deep learning applications for IoT in health care: a systematic review. Inform Med Unlocked 23:100550
- Rastogi R, Chaturvedi DK, Sagar S, Tandon N, Rastogi AR (2022) Brain tumor analysis using deep learning: sensor and iotbased approach for futuristic healthcare. In: Bioinformatics and medical applications: big data using deep learning algorithms, pp 171-190
- Roopashree
et al (2022) An IoT based authentication system for therapeutic herbs measured by local descriptors using machine learning approach. Measurement 200:111484 - Bharadwaj HK et al (2021) A review on the role of machine learning in enabling IoT based healthcare applications. IEEE Access 9:38859-38890
- Awotunde JB et al (2021) Disease diagnosis system for IoTbased wearable body sensors with machine learning algorithm. In: Hybrid artificial intelligence and IoT in healthcare. 2021. Springer, pp 201-222
- Alansari Z et al (2017) Computational intelligence tools and databases in bioinformatics. In: 2017 4th IEEE international conference on engineering technologies and applied sciences (ICETAS). 2017. IEEE
- Daoud H, Williams P, Bayoumi M (2020) IoT based efficient epileptic seizure prediction system using deep learning. In: 2020 IEEE 6th world forum on internet of things (WF-IoT). 2020. IEEE
- Wu Y et al (2021) Deep learning for big data analytics. Mobile Netw Appl 26(6):2315-2317
- Ambika N (2022) An economical machine learning approach for anomaly detection in IoT environment. In: Bioinformatics and medical applications: big data using deep learning algorithms, 2022: pp 215-234
- Srivastava M (2020) A Surrogate data-based approach for validating deep learning model used in healthcare. In: Applications of deep learning and big IoT on personalized healthcare services. 2020. IGI Global, pp 132-146
- da Costa KA et al (2019) Internet of things: a survey on machine learning-based intrusion detection approaches. Comput Netw 151:147-157
- Min S, Lee B, Yoon S (2017) Deep learning in bioinformatics. Brief Bioinform 18(5):851-869
- Aminizadeh
et al (2023) The applications of machine learning techniques in medical data processing based on distributed computing and the internet of things. In: Computer methods and programs in biomedicine, 2023, p 107745 - Li Y et al (2019) Deep learning in bioinformatics: introduction, application, and perspective in the big data era. Methods 166:4-21
- Cao
et al (2020) Ensemble deep learning in bioinformatics. Nat Mach Intell 2(9):500-508 - Tang B et al (2019) Recent advances of deep learning in bioinformatics and computational biology. Front Genet 10:214
- Koumakis L (2020) Deep learning models in genomics; are we there yet? Comput Struct Biotechnol J 18:1466-1473
- Dhombres F, Charlet J (2019) Formal medical knowledge representation supports deep learning algorithms, bioinformatics pipelines, genomics data analysis, and big data processes. Yearb Med Inform 28(01):152-155
- Peng
et al (2018) The advances and challenges of deep learning application in biological big data processing. Curr Bioinform 13(4):352-359 - Chen Y-Z et al (2021) nhKcr: a new bioinformatics tool for predicting crotonylation sites on human nonhistone proteins based on deep learning. Brief Bioinform 22(6):bbab146
- Chen Y et al (2016) Gene expression inference with deep learning. Bioinformatics 32(12):1832-1839
- Jabbar MA (2022) An insight into applications of deep learning in bioinformatics. In: Deep learning, machine learning and IoT in biomedical and health informatics. CRC Press, pp 175-197
- Khurana S et al (2018) DeepSol: a deep learning framework for sequence-based protein solubility prediction. Bioinformatics 34(15):2605-2613
- Baranwal M et al (2020) A deep learning architecture for metabolic pathway prediction. Bioinformatics 36(8):2547-2553
- Shahid O et al (2021) Machine learning research towards combating COVID-19: Virus detection, spread prevention, and medical assistance. J Biomed Inform 117:103751
- Roy PK et al (2023) Analysis of community question-answering issues via machine learning and deep learning: state-of-the-art review. CAAI Trans Intell Technol 8(1):95-117
- Samanta RK et al (2022) Scope of machine learning applications for addressing the challenges in next-generation wireless networks. CAAI Trans Intell Technol 7(3):395-418
- Wang W et al (2023) Fully Bayesian analysis of the relevance vector machine classification for imbalanced data problem. CAAI Trans Intell Technol 8(1):192-205
- Ashrafuzzaman M (2021) Artificial intelligence, machine learning and deep learning in ion channel bioinformatics. Membranes 11(9):672
- Fiannaca A et al (2018) Deep learning models for bacteria taxonomic classification of metagenomic data. BMC Bioinform 19(7):61-76
- Li F et al (2020) DeepCleave: a deep learning predictor for caspase and matrix metalloprotease substrates and cleavage sites. Bioinformatics 36(4):1057-1065
- Meher J (2021) Potential applications of deep learning in bioinformatics big data analysis. In: Advanced deep learning for engineers and scientists, 2021, pp 183-193
- Preuer K et al (2018) DeepSynergy: predicting anti-cancer drug synergy with Deep Learning. Bioinformatics 34(9):1538-1546
- Xia Z et al (2019) DeeReCT-PolyA: a robust and generic deep learning method for PAS identification. Bioinformatics 35(14):2371-2379
- Fang B et al (2022) Deep generative inpainting with comparative sample augmentation. J Comput Cogn Eng 1(4):174-180
- Wang
et al (2020) Block switching: a stochastic approach for deep learning security. arXiv preprint arXiv:2002.07920, 2020 - Kumar I, Singh SP (2022) Machine learning in bioinformatics. In: Bioinformatics. Academic Press, pp 443-456
- Yu L et al (2018) Drug and nondrug classification based on deep learning with various feature selection strategies. Curr Bioinform 13(3):253-259
- Jurtz VI et al (2017) An introduction to deep learning on biological sequence data: examples and solutions. Bioinformatics 33(22):3685-3690
- Deng Y et al (2020) A multimodal deep learning framework for predicting drug-drug interaction events. Bioinformatics 36(15):4316-4322
- Shakeel N, Shakeel S (2022) Context-free word importance scores for attacking neural networks. J Comput Cogn Eng 1(4):187-192
- Oubounyt M et al (2019) DeePromoter: robust promoter predictor using deep learning. Front Genet 10:286
- Leung MK et al (2014) Deep learning of the tissue-regulated splicing code. Bioinformatics 30(12):i121-i129
- Dai B, Bailey-Kellogg C (2021) Protein interaction interface region prediction by geometric deep learning. Bioinformatics 37(17):2580-2588
- Luo F et al (2019) DeepPhos: prediction of protein phosphorylation sites with deep learning. Bioinformatics 35(16):2766-2773
- Liu X (2022) Real-world data for the drug development in the digital era. J Artif Intell Technol 2(2):42-46
- Wei L et al (2018) Prediction of human protein subcellular localization using deep learning. J Parallel Distrib Comput 117:212-217
- Heidari A et al (2023) A new lung cancer detection method based on the chest CT images using federated learning and blockchain systems. Artif Intell Med 141:102572
- Cai Q et al (2023) Image neural style transfer: a review. Comput Electr Eng 108:108723
- Ai Q et al (2021) Editorial for FGCS special issue: intelligent IoT systems for healthcare and rehabilitation. Elsevier, New York, pp 770-773
- Niu L-Y, Wei Y, Liu W-B, Long JY, Xue T-H (2023) Research Progress of spiking neural network in image classification: a review. In: Applied intelligence, pp 1-25
- Karnati M et al (2022) A novel multi-scale based deep convolutional neural network for detecting COVID-19 from X-rays. Appl Soft Comput 125:109109
- Ravindran U, Gunavathi C (2023) A survey on gene expression data analysis using deep learning methods for cancer diagnosis. Prog Biophys Mol Biol 177:1-13
- Zheng M et al (2022) A hybrid CNN for image denoising. J Artif Intell Technol 2(3):93-99
- Togneri R, Prati R, Nagano H, Kamienski C (2023) Data-driven water need estimation for IoT-based smart irrigation: a survey. Expert Syst Appl 225:120194
- Sheng N, Huang L, Lu Y, Wang H, Yang L, Gao L, Xie X, Fu Y, Wang Y (2023) Data resources and computational methods for lncRNA-disease association prediction. Comput Biol Med 153:106527
- Sharan RV, Rahimi-Ardabili H (2023) Detecting acute respiratory diseases in the pediatric population using cough sound features and machine learning: a systematic review. Int J Med Inform 176:105093
- Bhosale YH, Patnaik KS (2023) Bio-medical imaging (X-ray, CT, ultrasound, ECG), genome sequences applications of deep neural network and machine learning in diagnosis, detection, classification, and segmentation of COVID-19: a meta-analysis & systematic review. Multimed Tools Appl 82:39157-39210. https://doi.org/10.1007/s11042-023-15029-1
- Azhari F, Sennersten CC, Lindley CA et al (2023) Deep learning implementations in mining applications: a compact critical review. Artif Intell Rev 56:14367-14402. https://doi.org/10. 1007/s10462-023-10500-9
- Nazir S, Dickson DM, Akram MU (2023) Survey of explainable artificial intelligence techniques for biomedical imaging with deep neural networks. Comput Biol Med 156:106668
- Jacob TP, Pravin A, Kumar RR (2022) A secure IoT based healthcare framework using modified RSA algorithm using an artificial hummingbird based CNN. Trans Emerg Tel Tech 33(12):e4622. https://doi.org/10.1002/ett. 4622
- Phan HT, Nguyen NT, Hwang D (2023) Aspect-level sentiment analysis: a survey of graph convolutional network methods. Inf Fusion 91:149-172
- Qiu D, Cheng Y, Wang X (2023) Medical image super-resolution reconstruction algorithms based on deep learning: a survey. Comput Methods Prog Biomed 238:107590
- Sanders LM et al (2023) Biological research and self-driving labs in deep space supported by artificial intelligence. Nat Mach Intell 5(3):208-219
- Rezende PM et al (2022) Evaluating hierarchical machine learning approaches to classify biological databases. Brief Bioinform 23(4):bbac216
- Yi H-C et al (2022) Graph representation learning in bioinformatics: trends, methods and applications. Brief Bioinform 23(1):bbab340
- Sharma S (2021) The bioinformatics: detailed review of various applications of cluster analysis. Glob J Appl Data Sci Internet Things 5:1-2021
- Serra A, Galdi P, Tagliaferri R (2018) Machine learning for bioinformatics and neuroimaging. Wiley Interdiscip Rev Data Min Knowl Discov 8(5):e1248
- Liu L et al (2019) A smart dental health-IoT platform based on intelligent hardware, deep learning, and mobile terminal. IEEE J Biomed Health Inform 24(3):898-906
- Nematzadeh S et al (2022) Tuning hyperparameters of machine learning algorithms and deep neural networks using metaheuristics: a bioinformatics study on biomedical and biological cases. Comput Biol Chem 97:107619
- Kumar H, Sharma S (2021) Contribution of deep learning in bioinformatics. Glob J Appl Data Sci Internet Things 5:1-202
- Jia D et al (2021) Breast cancer case identification based on deep learning and bioinformatics analysis. Front Genet 12:628136
- Pastorino J, Biswas AK (2022) Data adequacy bias impact in a data-blinded semi-supervised GAN for privacy-aware COVID19 chest X-ray classification. In: Proceedings of the 13th ACM international conference on bioinformatics, computational biology and health informatics, 2022
- Auwul MR et al (2021) Bioinformatics and machine learning approach identifies potential drug targets and pathways in COVID-19. Brief Bioinform 22(5):bbab120
- Lan L et al (2020) Generative adversarial networks and its applications in biomedical informatics. Front Public Health 8:164
- Han C et al (2021) MADGAN: Unsupervised medical anomaly detection GAN using multiple adjacent brain MRI slice reconstruction. BMC Bioinform 22(2):1-20
- Balogh OM et al (2022) Efficient link prediction in the proteinprotein interaction network using topological information in a generative adversarial network machine learning model. BMC Bioinform 23(1):1-19
- Giansanti V et al (2019) Comparing deep and machine learning approaches in bioinformatics: a miRNA-target prediction case study. In: International conference on computational science. 2019. Springer
- Lyu C et al (2017) Long short-term memory RNN for biomedical named entity recognition. BMC Bioinform 18(1):1-11
- ElAbd H et al (2020) Amino acid encoding for deep learning applications. BMC Bioinform 21(1):1-14
- Liu J, Gong X (2019) Attention mechanism enhanced LSTM with residual architecture and its application for protein-protein interaction residue pairs prediction. BMC Bioinform 20(1):1-11
- Wang D et al (2017) MusiteDeep: a deep-learning framework for general and kinase-specific phosphorylation site prediction. Bioinformatics 33(24):3909-3916
- Zhao Y, Shao J, Asmann YW (2022) Assessment and optimization of explainable machine learning models applied to transcriptomic data. Genom Proteom Bioinform 20:899-911
- Souri A et al (2020) A new machine learning-based healthcare monitoring model for student’s condition diagnosis in Internet of Things environment. Soft Comput 24(22):17111-17121
- D’Orazio M et al (2022) Machine learning phenomics (MLP) combining deep learning with time-lapse-microscopy for monitoring colorectal adenocarcinoma cells gene expression and drug-response. Sci Rep 12(1):1-14
- Karim MR et al (2021) Deep learning-based clustering approaches for bioinformatics. Brief Bioinform 22(1):393-415
- Aydin Z (2020) Performance analysis of machine learning and bioinformatics applications on high performance computing systems. Acad Platf J Eng Sci 8(1):1-14
- Mohamed Shakeel P et al (2018) Maintaining security and privacy in health care system using learning based deep-Q-networks. J Med Syst 42(10):1-10
- Huang L et al (2019) Patient clustering improves efficiency of federated machine learning to predict mortality and hospital stay time using distributed electronic medical records. J Biomed Inform 99:103291
- Wang X, Jiang X, Vaidya J (2021) Efficient verification for outsourced genome-wide association studies. J Biomed Inform 117:103714
- Cui J et al (2021) FeARH: Federated machine learning with anonymous random hybridization on electronic medical records. J Biomed Inform 117:103735
- Giansanti V et al (2019) Comparing deep and machine learning approaches in bioinformatics: a miRNA-target prediction case study. In: Computational science-ICCS 2019: 19th international conference, Faro, Portugal, June 12-14, 2019, proceedings, part III, vol 19, 2019. Springer
- Lyu C et al (2017) Long short-term memory RNN for biomedical named entity recognition. BMC Bioinform 18:1-11
- ElAbd H et al (2020) Amino acid encoding for deep learning applications. BMC Bioinform 21:1-14
- Liu J, Gong X (2019) Attention mechanism enhanced LSTM with residual architecture and its application for protein-protein interaction residue pairs prediction. BMC Bioinform 20:1-11
- Mohamed Shakeel P et al (2018) Maintaining security and privacy in health care system using learning based deep-Q-networks. J Med Syst 42:1-10
- Sarbaz M et al (2022) Adaptive optimal control of chaotic system using backstepping neural network concept. In: 2022 8th international conference on control, instrumentation and automation (ICCIA). 2022. IEEE
- Bagheri M et al (2020) Data conditioning and forecasting methodology using machine learning on production data for a well pad. In: Offshore technology conference. 2020. OTC
- Soleimani R, Lobaton E (2022) Enhancing inference on physiological and kinematic periodic signals via phase-based interpretability and multi-task learning. Information 13(7):326
- Mirzaeibonehkhater M (2018) Developing a dynamic recommendation system for personalizing educational content within an e-learning network. 2018: Purdue University
- Morteza A et al (2023) Deep learning hyperparameter optimization: application to electricity and heat demand prediction for buildings. Energy Build 289:113036
- Webber J et al (2017) Study on idle slot availability prediction for WLAN using a probabilistic neural network. In: 2017 23rd Asia-Pacific conference on communications (APCC). 2017. IEEE
- Webber J et al (2022) Improved human activity recognition using majority combining of reduced-complexity sensor branch classifiers. Electronics 11(3):392
- Gera T et al (2021) Dominant feature selection and machine learning-based hybrid approach to analyze android ransomware. Secur Commun Netw 2021:1-22
- Bukhari SNH, Webber J, Mehbodniya A (2022) Decision tree based ensemble machine learning model for the prediction of
111. Heidari A et al (2023) Machine learning applications in internet-of-drones: systematic review, recent deployments, and open issues. ACM Comput Surv 55(12):1-45
112. Singh R et al (2022) Analysis of network slicing for management of 5G networks using machine learning techniques. Wirel Commun Mobile Comput 2022:9169568
113. He P et al (2022) Towards green smart cities using Internet of Things and optimization algorithms: a systematic and bibliometric review. Sustain Comput Inform Syst 36:100822
114. Sadi M et al (2022) Special session: on the reliability of conventional and quantum neural network hardware. In: 2022 IEEE 40th VLSI test symposium (VTS). 2022. IEEE
115. Moradi M, Weng Y, Lai Y-C (2022) Defending smart electrical power grids against cyberattacks with deep Q-learning. P R X Energy 1:033005
116. Zhai Z-M et al (2023) Detecting weak physical signal from noise: a machine-learning approach with applications to mag-netic-anomaly-guided navigation. Phys Rev Appl 19(3):034030
117. Li Z, Han C, Coit DW (2023) System reliability models with dependent degradation processes. In: Advances in reliability and maintainability methods and engineering applications: essays in honor of professor Hong-Zhong Huang on his 60th birthday. 2023. Springer, pp 475-497
118. Zhang Y et al (2019) Fault diagnosis strategy of CNC machine tools based on cascading failure. J Intell Manuf 30:2193-2202
119. Shen G, Zeng W, Han C, Liu P, Zhang Y (2017) Determination of the average maintenance time of CNC machine tools based on type II failure correlation. Eksploatacja i Niezawodność 19(4)
120. Shen G et al (2018) Fault analysis of machine tools based on grey relational analysis and main factor analysis. J Phys Conf Ser. IOP Publishing
121. Han C, Fu X (2023) Challenge and opportunity: deep learningbased stock price prediction by using Bi-directional LSTM model. Front Bus Econ Manag 8(2):51-54
122. Darbandi M (2017) Proposing new intelligent system for suggesting better service providers in cloud computing based on Kalman filtering. Int J Technol Innov Res 24(1):1-9
123. Dehghani F, Larijani A (2023) Average portfolio optimization using multi-layer neural networks with risk consideration. Available at SSRN, 2023
124. Rezaei M, Rastgoo R, Athitsos V (2023) TriHorn-Net: a model for accurate depth-based 3D hand pose estimation. Expert Syst Appl 223:119922
125. Ahmadi SS, Khotanlou H (2022) A hybrid of inference and stacked classifiers to indoor scenes classification of rgb-d images. In: 2022 International conference on machine vision and image processing (MVIP). 2022. IEEE
126. Mirzapour O, Arpanahi SK (2017) Photovoltaic parameter estimation using heuristic optimization. In: 2017 IEEE 4th international conference on knowledge-based engineering and innovation (KBEI). 2017. IEEE
127. Khorshidi M, Ameri M, Goli A (2023) Cracking performance evaluation and modelling of RAP mixtures containing different recycled materials using deep neural network model. Road Mater Pavement Des 1-20
128. Rastegar RM et al (2022) From evidence to assessment: DEVELOPING a scenario-based computational design algorithm to support informed decision-making in primary care clinic design workflow. Int J Archit Comput 20(3):567-586
129. Esmaeili N, Bamdad Soofi J (2022) Expounding the knowledge conversion processes within the occupational safety and health management system (OSH-MS) using concept mapping. Int J Occup Saf Ergon 28(2):1000-1015
130. Akyash M, Mohammadzade H, Behroozi H (2021) Dtw-merge: a novel data augmentation technique for time series classification. arXiv preprint arXiv:2103.01119
131. Darbandi M (2017) Proposing new intelligence algorithm for suggesting better services to cloud users based on Kalman filtering. J Comput Sci Appl 5(1):11-16
132. Darbandi M (2017) Kalman filtering for estimation and prediction servers with lower traffic loads for transferring highlevel processes in cloud computing. Int J Technol Innov Res 23(1):10-20
133. Liu H et al (2023) MEMS piezoelectric resonant microphone array for lung sound classification. J Micromech Microeng 33(4):044003
134. Loghmani N, Moqadam R, Allahverdy A (2022) Brain tumor segmentation using multimodal mri and convolutional neural network. In: 2022 30th international conference on electrical engineering (ICEE). 2022. IEEE
135. Niknejad N, Caro JL, Bidese-Puhl R, Bao Y, Staiger EA (2023) Equine kinematic gait analysis using stereo videography and deep learning: stride length and stance duration estimation. J ASABE 66(4):865-877
136. Amiri Z et al (2023) Resilient and dependability management in distributed environments: a systematic and comprehensive literature review. Clust Comput 26(2):1565-1600
137. Zeng Q et al (2020) Hyperpolarized Xe NMR signal advancement by metal-organic framework entrapment in aqueous solution. Proc Natl Acad Sci 117(30):17558-17563
138. Liu N et al (2021) An eyelid parameters auto-measuring method based on 3D scanning. Displays 69:102063
139. Li C et al (2021) Long noncoding RNA p21 enhances autophagy to alleviate endothelial progenitor cells damage and promote endothelial repair in hypertension through SESN2/AMPK/TSC2 pathway. Pharmacol Res 173:105920
140. Li B et al (2022) Dynamic event-triggered security control for networked control systems with cyber-attacks: a model predictive control approach. Inf Sci 612:384-398
141. Li H, Peng R, Wang Z-A (2018) On a diffusive susceptible-infected-susceptible epidemic model with mass action mechanism and birth-death effect: analysis, simulations, and comparison with other mechanisms. SIAM J Appl Math 78(4):2129-2153
142. Amiri Z et al (2023) The personal health applications of machine learning techniques in the internet of behaviors. Sustainability 15(16):12406
143. Zhu Y et al (2021) Deep learning-based predictive identification of neural stem cell differentiation. Nat Commun 12(1):2614
144. Yang S et al (2022) Dual-level representation enhancement on characteristic and context for image-text retrieval. IEEE Trans Circuits Syst Video Technol 32(11):8037-8050
145. Yan L et al (2023) Multi-feature fusing local directional ternary pattern for facial expressions signal recognition based on video communication system. Alex Eng J 63:307-320
146. Dai
147. Yan L et al (2021) Method of reaching consensus on probability of food safety based on the integration of finite credible data on block chain. IEEE access 9:123764-123776
148. Jiang H et al (2020) An energy-efficient framework for internet of things underlaying heterogeneous small cell networks. IEEE Trans Mob Comput 21(1):31-43
149. Sun L, Zhang M, Wang B, Tiwari P (2023) Few-shot classincremental learning for medical time series classification. IEEE J Biomed Health Inform. https://doi.org/10.1109/JBHI.2023. 3247861
150. Gao Z, Pan X, Shao J, Jiang X, Su Z, Jin K, Ye J (2023) Automatic interpretation and clinical evaluation for fundus fluorescein angiography images of diabetic retinopathy patients by deep learning. Br J Ophthalmol 107(12):1852-1858
151. Wang H et al (2022) Transcranial alternating current stimulation for treating depression: a randomized controlled trial. Brain 145(1):83-91
152. Luan D et al (2022) Robust two-stage location allocation for emergency temporary blood supply in postdisaster. Discret Dyn Nat Soc 2022:1-20
153. Chen G et al (2022) Continuance intention mechanism of middle school student users on online learning platform based on qualitative comparative analysis method. Math Probl Eng 2022:1-12
154. Cui G et al (2013) Synthesis and characterization of Eu (III) complexes of modified cellulose and poly (N-isopropylacrylamide). Carbohyd Polym 94(1):77-81
155. Cheng B et al (2016) Situation-aware IoT service coordination using the event-driven SOA paradigm. IEEE Trans Netw Serv Manag 13(2):349-361
156. Cheng B et al (2017) Situation-aware dynamic service coordination in an IoT environment. IEEE/ACM Trans Netw 25(4):2082-2095
157. Zhuang Y, Jiang N, Xu Y (2022) Progressive distributed and parallel similarity retrieval of large CT image sequences in mobile telemedicine networks. Wirel Commun Mob Comput 2022:1-13
158. Tang Y et al (2021) An improved method for soft tissue modeling. Biomed Signal Process Control 65:102367
159. Zhang Z et al (2022) Endoscope image mosaic based on pyramid ORB. Biomed Signal Process Control 71:103261
160. Lu S et al (2023) Iterative reconstruction of low-dose CT based on differential sparse. Biomed Signal Process Control 79:104204
161. Lu S et al (2023) Soft tissue feature tracking based on deepmatching network. CMES Comput Model Eng Sci 136(1):363
162. Liu M et al (2023) Three-dimensional modeling of heart soft tissue motion. Appl Sci 13(4):2493
163. Heidari A et al (2023) A hybrid approach for latency and battery lifetime optimization in IoT devices through offloading and CNN learning. Sustain Comput Inform Syst 39:100899
164. Heidari A, Jafari Navimipour N, Unal M (2022) The history of computing in Iran (Persia)-since the achaemenid empire. Technologies 10(4):94
165. Ahmadpour S-S, Heidari A, Navimpour NJ, Asadi M-A, Yalcin S (2023) An efficient design of multiplier for using in nano-scale IoT systems using atomic silicon. IEEE Internet Things J 10(16):14908-14909. https://doi.org/10.1109/JIOT.2023. 3267165
166. Amiri Z, Heidari A, Navimipour NJ et al (2023) Adventures in data analysis: a systematic review of deep learning techniques for pattern recognition in cyber-physical-social systems. Multimed Tools Appl. https://doi.org/10.1007/s11042-023-16382-x
- Arash Heidari
arash_heidari@ieee.org
Nima Jafari Navimipour
nima.navimipour@khas.edu.tr; jnnima@khas.edu.tw
1 Department of Computer Engineering, Tabriz Branch, Islamic Azad University, Tabriz, Iran2 Department of Software Engineering, Haliç University, 34060 Istanbul, Turkey3 Department of Computer Engineering, Kadir Has University, Istanbul, Turkey
4 Future Technology Research Center, National Yunlin University of Science and Technology, 64002 Douliou, Yunlin, Taiwan5 Computer Engineering Department, Hamedan Branch, Islamic Azad University, Hamedan, Iran
Immunology Research Center, Tabriz University of Medical Sciences, Tabriz, Iran
DOI: https://doi.org/10.1007/s00521-023-09366-3
Publication Date: 2024-01-13
The deep learning applications in loT-based bio- and medical informatics: a systematic literature review
© The Author(s) 2024
Abstract
Nowadays, machine learning (ML) has attained a high level of achievement in many contexts. Considering the significance of ML in medical and bioinformatics owing to its accuracy, many investigators discussed multiple solutions for developing the function of medical and bioinformatics challenges using deep learning (DL) techniques. The importance of DL in Internet of Things (IoT)-based bio- and medical informatics lies in its ability to analyze and interpret large amounts of complex and diverse data in real time, providing insights that can improve healthcare outcomes and increase efficiency in the healthcare industry. Several applications of DL in IoT-based bio- and medical informatics include diagnosis, treatment recommendation, clinical decision support, image analysis, wearable monitoring, and drug discovery. The review aims to comprehensively evaluate and synthesize the existing body of the literature on applying deep learning in the intersection of the IoT with bio- and medical informatics. In this paper, we categorized the most cutting-edge DL solutions for medical and bioinformatics issues into five categories based on the DL technique utilized: convolutional neural network, recurrent neural network, generative adversarial network, multilayer perception, and hybrid methods. A systematic literature review was applied to study each one in terms of effective properties, like the main idea, benefits, drawbacks, methods, simulation environment, and datasets. After that, cutting-edge research on DL approaches and applications for bioinformatics concerns was emphasized. In addition, several challenges that contributed to DL implementation for medical and bioinformatics have been addressed, which are predicted to motivate more studies to develop medical and bioinformatics research progressively. According to the findings, most articles are evaluated using features like accuracy, sensitivity, specificity,
1 Introduction
biology, and mathematics [10,11]. It is critical for data management in modern medicine and biology [12, 13]. Bioinformatics provides considerable support to deal with time context and cost issues in different tracks [14, 15]. Bioinformatics, as pertinent to genomics and genetics, is a scientific interdisciplinary field that utilizes computer technology to gather, store, evaluate, and distribute biological data, like DNA and amino acid sequences and annotations about them [16, 17].
- Conducting an SLR to explore ML applications in IoTbased medical and bioinformatics;
- Analyzing and comparing DL uses in medical and bioinformatics;
- Categorizing DL mechanisms into five groups (CNN, RNN, GAN, MLP, hybrids) and examining their properties;
- Investigating DL/ML methodologies and applications in bioinformatics;
- Providing insights for future research and addressing existing shortcomings;
- Offering a comprehensive evaluation of existing ML/ DL methods;
- Contributing to a better understanding of current challenges and opportunities in the field;
2 Fundamental concepts and terminology
2.1 Deep learning concepts
2.2 Bioinformatics applications
applications of bioinformatics including recording and retrieval of data in gene therapy, biometrical evaluation for crop management, pest control, evolutionary research, drug discovery, and microbial utilitarianism [52].
2.3 Deep learning usage in bioinformatics
2.3.1 Detecting enzymes applying multilayer neural networks
and RNN. These networks can be trained on large datasets of enzyme data, and the resulting models can be used to automatically detect and classify enzymes in new data [56].
2.3.2 Gene expression regression
2.3.3 CNN predicting RNA-protein linking points
2.3.4 DNA sequence performance anticipation with RNN and CNN
2.3.5 Biomedical image classification applying ResNet and transfer learning
the accuracy of classification compared to traditional ML algorithms [65].
2.3.6 Graph embedding using GCN for protein interaction prediction
2.3.7 GAN image super-resolution in biology
resolution images. This training continues until the generator network produces high-quality images indistinguishable from real high-resolution images. GAN image superresolution in biology has many applications, such as enhancing the resolution of microscopy images to improve the accuracy of image analysis and improving the resolution of medical images to aid in diagnosis and treatment [68].
2.3.8 Variational autoencoder high-dimensional biological generative and data embedding
3 Relevant reviews
4 Methodology of research
4.1 Formalization of question
- RQ 1: How may DL approaches in bio- and medical informatics be classified in medical healthcare? What are some of their examples?
- RQ 2: What are the most significant cutting-edge works? What are their benefits and drawbacks? What features do they have?
- RQ 3: What are the most widely utilized applications, techniques, criteria, and other factors in bio- and medical informatics?
- RQ 4: What are the key potential solutions and unanswered issues in this field?
4.2 The procedure of paper exploration
| Authors | Main idea | Advantage | Disadvantage |
| Li, Huang [19] | Presenting both the exoteric definition of DL and integrating instances and executions of its representative uses in bioinformatics | Easy-to-understand introduction of methods Addressing the issues via providing practical examples | Some important parameters for comparison between methods have been overlooked |
| Rezende, Xavier [70] | Proposing a study of graph representation learning in bioinformatics, as well as identifying and evaluating techniques | Providing a comprehensive well-structured survey of graph embedding mechanisms | Poor comparison among methods |
| Yi, You [71] | Contrasting the operation of “Local per Level” and “Local per Node” methods employed to two various hierarchical datasets: CATH and BioLip | Providing computational libraries to assist the community in the decision-making process for planning hierarchical data | Details of methods overlooked |
| Sharma [72] | Integrating various results to establish clusters without depending on the criteria utilized to evaluate data | Well-organized schematic comparison between mechanisms | Poor analysis of proposed approaches |
| Serra, Galdi [73] | Discussing applications of ML in bioinformatics and neuroimaging to solve related issues | Stating several examples to clarify the application of ML in bioinformatics | Overlooked some challenges like DL results interpretation |
| Our work | Providing a new taxonomy of DL/ML method in medical and bioinformatics | Comprehensively discussing various studies using DL mechanisms in medical and bioinformatics | Unavailability of non-English papers |
5 DL approaches in the field of bioand medical informatics
5.1 CNN approaches for bio- and medical informatics

| S# | Keywords and search criteria | S# | Keywords and search criteria |
| S1 | “Deep learning” and “Medical issues” | S6 | “AI” and “Healthcare” |
| S2 | “Machine learning” and “Bioinformatics” | S7 | “Healthcare” and “IoT” |
| S3 | “Deep learning” and “Bioinformatics” | S8 | “DL methods” and “Medical Internet of Things” |
| S4 | “IoT-based system” and “Bioinformatics” | S9 | “ML methods” and “Medical Internet of Things” |
| S5 | “AI” and “Medical informatics” | S10 | “AI methods” and “Medical Internet of Things” |


and a mobile terminal to assess its potential in in-home dental healthcare. Moreover, sophisticated dental equipment is being developed and upgraded to operate the image attainment of teeth. Based on a dataset of 12,600 clinical images collected by the presented device from 10 private dental clinics, an automatic detection model trained by MASK R-CNN was improved for the identification and classification of 7 different dental diseases, including deteriorated teeth, periodontal disease, fluorosis, and dental plaque, with detection precision of up to




| Author | Publisher | Journal | Citation | Q | Country | Year | Hindex | |
| 1 | Liu, Xu [74] | IEEE | Journal of Biomedical and Health Informatics | 54 | Q1 | China | 2019 | 137 |
| 2 | Nematzadeh, Kiani [75] | Elsevier | Computational Biology and Chemistry | 13 | Q2 | Turkey | 2022 | 61 |
| 3 | Chen, Wang [25] | Oxford University Press | Briefings in Bioinformatics | 14 | Q1 | China | 2021 | 121 |
| 4 | Kumar and Sharma [76] | – | Global Journal on Application of Data Science and Internet of Things | – | – | Russia | 2021 | – |
| 5 | Jia, Chen [77] | Frontiers Media S.A | Frontiers in Genetics | 4 | Q1 | China | 2021 | 93 |
| 6 | Pastorino and Biswas [78] | – | The 13th ACM international conference on hybrid systems: computation and control | – | – | USA | 2022 | 14 |
| 7 | Auwul, Rahman [79] | Oxford University Press | Briefings in Bioinformatics | 38 | Q1 | Australia | 2021 | 121 |
| 8 | Lan, You [80] | Frontiers Media S.A | Frontiers in Genetics | 38 | Q1 | China | 2021 | 121 |
| 9 | Han, Rundo [81] | – | Bergamo Computational Intelligence Methods for Bioinformatics and Biostatistics | 76 | – | Italy | 2021 | – |
| 10 | Balogh, Benczik [82] | BioMed Central Ltd | BMC Bioinformatics | 5 | Q2 | Hungry | 2022 | 218 |
| 11 | Giansanti, Castelli [83] | – | International computational science and engineering conference | 3 | – | Italy | 2019 | – |
| 12 | Lyu, Chen [84] | BioMed Central Ltd | BMC Bioinformatics | 97 | Q2 | China | 2017 | 218 |
| 13 | ElAbd, Bromberg [85] | BioMed Central Ltd | BMC Bioinformatics | 37 | Q2 | Germany | 2020 | 218 |
| 14 | Liu and Gong [86] | BioMed Central Ltd | BMC Bioinformatics | 24 | Q2 | China | 2019 | 218 |
| 15 | Wang, Zeng [87] | IEEE | IEEE International Conference on Bioinformatics and Biomedicine | 161 | – | China | 2017 | – |
| 16 | Zhao, Shao [88] | Elsevier | Genomics, proteomics & Bioinformatics | – | Q1 | USA | 2021 | 56 |
| 17 | Souri, Ghafour [89] | Springer | Soft computing | 54 | Q2 | Iran | 2020 | 90 |
| 18 | D’Orazio, Murdocca [90] | Nature | Scientific reports | – | Q1 | Italy | 2022 | 242 |
| 19 | Karim, Beyan [91] | Oxford university press | Briefings in bioinformatics | 101 | Q1 | UK | 2021 | 121 |
| 20 | AYDIN [92] | The public library of science | PLoS Computational Biology | 3 | Q1 | Turkey | 2020 | 191 |
| 21 | Mohamed Shakeel, Baskar [93] | Springer | Journal of Medical Systems | 214 | Q1 | Malaysia | 2018 | 89 |
| 22 | Huang, Shea [94] | Elsevier | Journal of Biomedical Informatics | 188 | Q1 | China | 2019 | 112 |
| 23 | Wang, Jiang [95] | Elsevier | Journal of Biomedical Informatics | 2 | Q1 | USA | 2021 | 112 |
| 24 | Cui, Zhu [96] | Elsevier | Journal of Biomedical Informatics | 5 | Q1 | USA | 2021 | 112 |
| 25 | Shahid, Nasajpour [30] | Elsevier | Journal of Biomedical Informatics | 44 | Q1 | USA | 2021 | 112 |
regions of interest for proven COVID-19-positive patients, bacterial pneumonia, and healthy cases. They also discussed the challenges faced in applying DL in bioinformatics, such as the need for large datasets, interpretability, and data quality.

5.2 GAN approaches for bio- and medical informatics
information from the images, which may lead to data adequacy bias. They evaluated their method on a publicly available dataset and found that it achieved comparable performance to state-of-the-art methods while preserving data privacy.
| Author | Main idea | Advantage | Drawback | Method | Simulation environment | Dataset | ||||
| Liu, Xu [74] | Proposing an intelligent dental Health-IoT system relied on smart hardware, DL, enabling exploration of the viability | High accuracy High sensitivity High septicity Low latency |
|
CNN | TensorFlow | 10 private dental clinics | ||||
| Nematzadeh, Kiani [75] | Presenting a strategy for optimizing the handling hyperparameters of ML algorithms |
|
|
CNN | C# | 11 datasets in various biological, natural, and biomedical categories | ||||
| Chen, Wang [25] | Using CNNrgb as a DL-based computational paradigm for nhKcr site anticipation on nonhistone proteins | High computational efficiency | Poor flexibility | CNN | Python | An online server named nhKcr | ||||
| Kumar and Sharma [76] | Using the CNN technique to diagnose COVID-19 |
|
Poor autonomously | CNN | Python | COVID and nonCOVID patients’ chest X-rays | ||||
| Jia, Chen [77] | Utilizing gene expression omnibus and cancer genome atlas gene expression profiles to differentiate between breast cancer patients and healthy individuals |
|
Poor flexibility | CNN | R | 1109 cancer patients and 113 normal cases |
5.3 RNN approaches for bio- and medical informatics
network model could be used for BNER without the need for human feature engineering. Based on their experimental findings, the domain-specific pre-trained word embedding and character-level representation may be used to create the function of the LSTM-RNN approaches.
| Author | Main idea | Advantage | Drawback | Method | Simulation environment | Dataset | ||||
| Pastorino and Biswas [78] | Introducing data-blinded semisupervised GAN to develop classification operation |
|
High complexity | GAN | Python | 1000 epochs of SGAN model | ||||
| Auwul, Rahman [79] | Presenting a beta-binomial distribution method to draw peptide immunogenic potential |
|
|
GAN | Python | 9000 tested immunogenicity molecular assays | ||||
| Lan, You [80] | Using GAN-based method for neighboring brain MRI section restoration |
|
Poor generalizable recreation and detection | GAN | TensorFlow |
|
||||
| Han, Rundo [81] | Developing software that runs a link anticipation tool for PPI forecasting utilizing ML |
|
Poor scalability | GAN | Python | PPI network from the STRING database | ||||
| Balogh, Benczik [82] | Designing a data-blind semisupervised GAN to improve classification operation |
|
Poor flexibility | GAN | Python | ChIP-seq and DNase-seq datasets |
5.4 MLP approaches for bio- and medical informatics
| Author | Main idea | Advantage | Drawback | Method | Simulation environment | Dataset | ||
| Giansanti, Castelli [83] | Training five models from ML and DL domains to examine the probability of detecting miRNA-mRNA interactions |
|
Poor availability | RNN | Python | TargetScan miRanda RNAhybrid | ||
| Lyu, Chen [84] | Proposing an RNN framework based on embedding and character representation |
|
Poor flexibility | RNN | C+ + | BioCreative GM | ||
| ElAbd, Bromberg [85] | Using multiple DL models to demonstrate that end-toend learning is comparable to encoding | High flexibility |
|
RNN | TensorFlow | PeptideHLA II interaction | ||
| Liu and Gong [86] | Proposing an attention-enhanced LSTM with a residual model to address protein-protein interaction problems | High accuracy | Poor adaptability | RNN | Python | 1H9D | ||
| Wang, Zeng [87] | Presenting DL framework for anticipating general and kinase-specific phosphorylation sites | High accuracy | Poor interpretability | RNN | Python | NetPhos3.1 |
5.5 Hybrid approaches for bio- and medical informatics
| Author | Main idea | Advantage | Drawback | Method | Simulation environment | Dataset | ||||
| Zhao, Shao [88] | Proposing a set of optimization approaches for each explanation on two architectures of MLP and CNN | High accuracy | Poor scalability | MLP | PyTorch | 19,241 genes | ||||
| Souri, Ghafour [89] | Suggesting an IoT-based monitoring pattern to continually regulate student vital signs |
|
Poor adaptability | MLP | TensorFlow | 1100 students | ||||
| D’Orazio, Murdocca [90] | Suggesting An MLP platform is being made available for phenomics studies on how cancer cells react to therapy |
|
Poor adaptability | MLP | MATLAB | RESNET101 | ||||
| Karim, Beyan [91] | Choosing related genes to cancer to classify cancer | High accuracy | Poor scalability | MLP | PyTorch | 400 images | ||||
| AYDIN [92] | Training six ML models for detecting T4SEs |
|
poor adaptability | MLP | TensorFlow | PSI-BLAST HHblits |
information. They also provided a theoretical analysis of the proposed scheme and demonstrated its effectiveness through experiments using real GWAS datasets. Their method could be useful for ensuring the reliability and security of outsourced GWAS computations, which are becoming increasingly common in biomedical research.
approaches that have been used to address these challenges, such as developing predictive models for disease spread and severity, identifying risk factors associated with the disease, and developing methods for analyzing medical images and data. Table 8 indicates the techniques, properties, and characteristics of MLP-informatics methods.
6 Results and comparisons
categories. Additionally, we urge scholars to delve deeper into these subjects. Our survey evaluation revealed that most medical and bioinformatics investigations focused on a select blend of learning tasks or the improvement of annotation protocols and new datasets. ML has garnered significant popularity and acceptance, particularly for its implementation with CNN methods, which have demonstrated excellent results. However, there exist certain limitations to achieving the same level of efficacy in medical and bioinformatics applications. In general, research in this area is still ongoing. One of the most salient issues is the scarcity of large datasets containing high-quality patterns for training purposes. In such cases, data integration may be viable for amalgamating information from multiple sources. It is noteworthy that as the scale of data increases, so does the necessity for larger datasets to ensure that ML produces dependable results.
6.1 Analysis of results
| Author | Main idea | Advantage | drawback | Method | Simulation environment | Dataset | ||
| Mohamed Shakeel, Baskar [93] | Applying deep CNN to develop the effectiveness of the IoT-health data system |
|
Poor flexibility | CNN | N2 | ISO/IEC/JTC1/SC 31 standard drivers | ||
| Huang, Shea [94] | Proposing a community-based federated to classify the distributed data |
|
Poor adaptability | ML | Python | EMRs from 50 hospitals | ||
| Wang, Jiang [95] | Proposing two algorithms to provide synthetic SNPs | High accuracy | Poor adaptability | DL | НарМар | 89 subjects and 83,354 SNPs | ||
| Cui, Zhu [96] | Presenting a mechanism for training in a condition without a confident central analyzer | High accuracy | Poor adjustability | ML | Python | 30,760 patients data | ||
| Shahid, Nasajpour [30] | Suggesting a framework to protect medical data from exterior threats |
|
Poor adjustability | ML | Python | – |
portion of the studies provided access to the source codes, predominantly in MATLAB and Python, to facilitate reproducibility and further experimentation. MATLAB was a prevalent choice, particularly in studies emphasizing signal processing and image analysis, owing to its extensive toolboxes tailored for these domains. Conversely, Python was prominently featured in research that incorporated machine learning frameworks like TensorFlow and Keras, aligning with the broader trend in the machine learning community. Notably, several reviewed papers included code snippets and made their complete implementations available on public repositories, fostering collaborative research and knowledge dissemination in this interdisciplinary field. This availability of codes played a pivotal role in advancing the applicability and accessibility of deep learning methodologies in the context of IoT-based bio- and medical informatics.



which can have significant implications in healthcare settings.
| Type | Authors | Scalability | Accuracy | Precision | Fscore | Sensitivity | Specificity | Robustness | Adaptability |
| CNN | Liu, Xu [74] |
|
|
|
|
|
|
|
|
| Nematzadeh, Kiani [75] |
|
|
|
|
|
|
|
|
|
| Chen, Wang [25] |
|
|
|
|
|
|
|
|
|
| Kumar and Sharma [76] |
|
|
|
|
|
|
|
|
|
| Jia, Chen [77] |
|
|
|
|
|
|
|
|
|
| RNN | Pastorino and Biswas [78] |
|
|
|
|
|
|
|
|
| Auwul, Rahman [79] |
|
|
|
|
|
|
|
|
|
| Lan, You [80] |
|
|
|
|
|
|
|
|
|
| Han, Rundo [81] |
|
|
|
|
|
|
|
|
|
| Balogh, Benczik [82] |
|
|
|
|
|
|
|
|
|
| GAN | Giansanti, Castelli [97] |
|
|
|
|
|
|
|
|
| Lyu, Chen [98] |
|
|
|
|
|
|
|
|
|
| ElAbd, Bromberg [99] |
|
|
|
|
|
|
|
|
|
| Liu and Gong [100] |
|
|
|
|
|
|
|
|
|
| Wang, Zeng [87] |
|
|
|
|
|
|
|
|
|
| MLP | Zhao, Shao [88] |
|
|
|
|
|
|
|
|
| Souri, Ghafour [89] |
|
|
|
|
|
|
|
|
|
| D’Orazio, Murdocca [90] |
|
|
|
|
|
|
|
|
|
| Karim, Beyan [91] |
|
|
|
|
|
|
|
|
|
| AYDIN [92] |
|
|
|
|
|
|
|
|
|
| Hybrid | Mohamed Shakeel, Baskar [101] |
|
|
|
|
|
|
|
|
| Huang, Shea [94] |
|
|
|
|
|
|
|
|
|
| Wang, Jiang [95] |
|
|
|
|
|
|
|
|
|
| Cui, Zhu [96] |
|
|
|
|
|
|
|
|
|
| Shahid, Nasajpour [30] |
|
|
|
|
|
|
|
|
6.2 Exploring the Integration of ML in medical applications
which must be considered when developing privacy-aware GAN models [102]. They utilized a bioinformatics and ML approach to identify potential drug targets and pathways for COVID-19 treatment. One approach integrated multiple omics data sets to construct a molecular network, which was used to identify significant gene modules. A comprehensive review of the applications of GANs in biomedical informatics showed their potential in medical image analysis and drug discovery [103]. The proposed MADGAN in Sect. 5 outperformed other anomaly detection methods and could be used for the early detection of neurological diseases. An efficient link prediction model for protein-protein interaction networks was developed using topological information in a GAN framework. The model outperformed traditional network analysis methods and could be used to identify potential drug targets for diseases associated with protein-protein interactions. These studies highlight the potential of GANs and ML in medical research, particularly in disease detection, drug discovery, and protein-protein interaction network analysis [104].
results and could potentially lead to the development of more accurate and efficient tools for medical and bioinformatics applications. DL techniques and the incorporation of multiple data sources in ML models can lead to more accurate predictions and enhance the performance of these models [108].
6.3 Prevalent evaluation criteria
Precision
6.4 Challenges of The DL applications in IoTbased bio- and medical informatics
domain, where data are often scarce and expensive to obtain. This can lead to issues such as bias and limited diversity in the data set. CNNs are primarily designed for image data, and their application to other data types, such as time-series or text-based data, is limited [115]. This can be a challenge in IoT-based bio- and medical informatics, where data may be heterogeneous and multimodal. CNNs can be susceptible to adversarial attacks, where small perturbations to the input can lead to misclassification. This can be particularly concerning in medical applications, where incorrect predictions can have serious consequences. In summary, while CNNs have shown remarkable success in medical image analysis, they have several limitations that need to be addressed to improve their efficacy in IoTbased bio- and medical informatics.
model’s predictions [118]. Medical data can be noisy and contain variations due to differences in acquisition devices, protocols, and patient conditions. Such variations can be challenging to account for, leading to decreased performance of RNNs. On the other hand, medical data collection and annotation lack standardization, making it challenging to develop RNNs that generalize well across institutions. Medical data often suffer from class imbalance, where one class (e.g., disease-positive) has significantly fewer examples than the other class (e.g., disease-negative). This issue can lead to poor performance of RNNs and requires special attention to handle. RNNs can be computationally expensive, requiring significant computational resources to train and deploy. The limited availability of high-performance computing can hinder the development and deployment of RNNs in resource-constrained settings. The use of RNNs in healthcare raises ethical considerations, such as informed consent, privacy, and bias. Addressing these issues is essential to ensure that the use of RNNs in medical applications is ethical and fair [119]. Overall, these challenges highlight the need for careful consideration of RNNs’ application in IoT-based bio- and medical informatics and the importance of addressing the unique challenges of this domain.
need to be addressed to make GANs more effective in these contexts, particularly in cloud-based settings [122]. However, GANs rely heavily on high-quality data for training, and data quality is especially critical in bio- and medical informatics. In IoT-based bio- and medical informatics, data can be noisy, incomplete, and biased, making it challenging to train GANs accurately [123]. IoT-based bioand medical informatics involve sensitive patient data, which must be kept private and secure. However, GANs require large amounts of data to train, which poses a risk to patient privacy and security. Therefore, robust data security measures must be in place when using GANs in this context. Researchers must find ways to make GAN models more interpretable. One of the significant challenges in bioand medical informatics is the availability of a limited dataset. The limited data can affect the accuracy of the GAN model’s results, and in some cases, it may not be possible to train a GAN model with a limited dataset. The healthcare industry is highly regulated, and GANs must comply with regulatory requirements to be approved for use. Ensuring compliance with regulations can be challenging when working with GANs, especially when dealing with IoT-based bio- and medical informatics, where data security and privacy concerns are high. In summary, while GANs offer significant potential for IoT-based bioand medical informatics, some challenges still need to be addressed to make them more effective and acceptable for use in this context [124]. These challenges include data quality, data privacy and security, interpretability, limited datasets, and regulatory compliance.
dimensional data. This can be a limitation in bio- and medical informatics, where data can be high-dimensional. While MLPs have shown outperformed results in IoTbased bio- and medical informatics, it is essential to consider their limitations and explore alternative models that can better address these challenges.
of DL algorithms in healthcare raises ethical considerations, such as informed consent, privacy, and bias. Addressing these issues is essential to ensure that the use of DL models in medical applications is ethical and fair. The development and deployment of DL models can be expensive, making it difficult to implement them in resource-limited settings. Moreover, DL models may require specialized hardware and software, further increasing their cost.
6.5 Dataset in medical and bioinformatics using DL approaches
accurately diagnose diseases, predict treatment outcomes, and identify potential drug targets. Furthermore, the success of DL models depends heavily on the quality and diversity of the data used to train them. A biased, incomplete, or unrepresentative dataset of the target population can lead to biased or inaccurate results [133]. Therefore, it is essential to ensure that bio- and medical informatics datasets are diverse, representative, and of high quality. Moreover, using standardized datasets is critical for facilitating comparison and reproducibility of research results across different studies. Standardized datasets enable researchers to evaluate the performance of their models against others using the same data, facilitating the development of new and improved algorithms and methodologies. In summary, high-quality, comprehensive, diverse, and standardized datasets are essential for developing and evaluating DL models in IoT-based bio- and medical informatics [134]. They provide the foundation for the accurate diagnosis and treatment of diseases and the identification of new drug targets. The application of datasets in the field of DL for IoT-based bio- and medical informatics is crucial for developing accurate and efficient models. Without standard datasets, the models cannot learn and make accurate predictions. One of the key challenges in developing DL models for bio- and medical informatics is the availability of labeled datasets. Labeled datasets are critical for supervised learning, which is the most common approach in DL. This is because DL models need large amounts of labeled data to learn complex patterns and relationships in the data. In bio- and medical informatics, these labeled datasets are often created through manual annotation or by experts in the field. Many publicly available bio- and medical informatics datasets can be used for DL applications, such as the MIMIC-III dataset for EHR, the ImageNet dataset for medical imaging, and the PhysioNet dataset for physiological signals. These datasets have been used to develop models for various applications such as disease diagnosis, drug discovery, and personalized medicine. The usage of datasets in DL applications for bioand medical informatics also requires careful attention to data privacy and security. Patient data are highly sensitive and must be handled carefully to protect patient privacy [135]. Researchers must ensure that the datasets used for training their models comply with ethical and legal requirements and that the data are de-identified before use. Researchers must carefully select and preprocess datasets, comply with ethical and legal requirements, and handle patient data with great care to protect patient privacy.
electrocardiogram (ECG) recordings, each spanning 10 s and sampled at a rate of 500 Hz , resulting in a total of 50,000 data points per recording. Another noteworthy dataset in neurology research consisted of 500 patients with Parkinson’s disease, yielding over 150,000 data points per patient across various sensor readings. Additionally, a comprehensive dataset for Alzheimer’s disease prediction integrated multimodal data, including structural MRI images from 1000 subjects, alongside demographic and cognitive assessments. These quantitative specifics exemplify the rich and varied nature of datasets in this field, which play a pivotal role in training and evaluating deep learning models for bio- and medical informatics applications within the IoT framework.
as many information examples as there are data characteristics.
6.6 IoT applications using DL methods in bioand medical informatics
6.7 Security issues, challenges, risks, IoT, and blockchain usage
| Name | Descriptions |
| MNIST | The MNIST dataset is popular in computer vision applications, including DL. It consists of a set of 70,000 handwritten digits, each with a
|
| CIFAR-10 and CIFAR-100 | These two datasets are commonly used in image classification tasks in DL. CIFAR-10 consists of
|
| ImageNet | ImageNet is a large-scale visual recognition challenge comprising over 14 million images in 21,000 categories. This dataset has been used in various DL applications, including bio- and medical informatics. For example, it has been used to train DL models to classify skin lesions or diagnose diseases based on medical images |
| PhysioNet | PhysioNet is a physiological signal dataset collection that includes electrocardiograms, electroencephalograms, and vital signs. This dataset has been used in DL applications in bio- and medical informatics for tasks such as disease diagnosis, predicting patient outcomes, and detecting abnormal patterns in physiological signals |
| MIMIC-III | MIMIC-III is a publicly available critical care database that contains de-identified health data of over 40,000 patients. This dataset includes information such as vital signs, laboratory results, and medical histories. It has been used in DL applications to predict patient outcomes, identify disease risk factors, and improve clinical decision-making |
| TCGA | The Cancer Genome Atlas (TCGA) is a collection of genomic, epigenomic, and transcriptomic data from over 30 cancer types. This dataset has been used in DL applications for cancer diagnosis, predicting patient outcomes, and identifying novel therapeutics |
| Targets.MIMIC-III | The Medical Information Mart for Intensive Care (MIMIC-III) is a large, freely available dataset consisting of deidentified electronic health records of more than 50,000 patients admitted to the critical care units of a large tertiary care hospital. The dataset contains clinical data such as vital signs, laboratory results, medications, and demographics, making it a valuable resource for research in critical care and clinical decision-making |
| NIH Chest X-ray Dataset | The National Institutes of Health Chest X-ray dataset is a collection of over 100,000 chest X-ray images labeled with various thoracic pathologies such as pneumonia, tuberculosis, and lung cancer. The dataset is a valuable resource for research in computer-aided diagnosis, disease classification, and image analysis |
| PhysioNet | The PhysioNet dataset is a collection of physiological signals and related clinical data such as ECG, electroencephalogram (EEG), and blood pressure recordings. The dataset is a valuable resource for disease diagnosis, monitoring, and prediction research |
| ADNI | The Alzheimer’s Disease Neuroimaging Initiative (ADNI) dataset is a collection of longitudinal neuroimaging, clinical, and biomarker data from individuals with Alzheimer’s disease, mild cognitive impairment, and healthy controls. The dataset is a valuable resource for disease diagnosis, prediction, and treatment research |
| SEER | The Surveillance, Epidemiology, and End Results (SEER) dataset is a population-based cancer registry that collects clinical, demographic, and survival data from cancer patients in the United States. The dataset is a valuable resource for cancer diagnosis, treatment, and survival analysis research |
ledger technology offering a secure and tamper-proof way to store and share data. It achieves this by using cryptographic algorithms and decentralization to ensure that the data stored on the blockchain is immutable and transparent. In the context of IoT-based bio- and medical informatics, blockchain can be used to secure the data generated by IoT devices and ensure its integrity, authenticity, and privacy. For example, blockchain can be used to create a secure and tamper-proof log of all the data generated by IoT devices, which can be accessed only by authorized parties [145]. Additionally, blockchain can implement secure and pri-vacy-preserving data sharing mechanisms between healthcare providers and researchers. However, the use of blockchain in this context also comes with its own challenges and risks [146]. For instance, blockchain’s high computational and storage requirements may not be feasible for resource-constrained IoT devices. Additionally,
blockchain’s immutability can make it difficult to correct errors or update data, which can be problematic in the context of medical data that may need to be updated or corrected over time [147]. Finally, the use of blockchain also raises concerns about data privacy and confidentiality, as it can be challenging to ensure that sensitive medical data are not shared or accessed by unauthorized parties. Therefore, while blockchain technology offers a promising solution for securing IoT-based bio- and medical informatics applications, it is important to carefully consider its application and weigh the risks and benefits before implementation.
data securely, allowing patients to control who has access to their data and grant permission to healthcare providers to access it, thus protecting their privacy. However, blockchain technology in healthcare also presents challenges such as scalability, which requires significant computational power and storage capacity to manage large volumes of data generated by IoT devices. Additionally, the lack of blockchain interoperability standards makes it difficult to integrate different blockchain networks and medical devices.
6.8 Upcoming deep learning models
7 Open issues and key challenges
recommendations. Ongoing research is focused on developing more interpretable DL models. Moreover, IoT-based bio- and medical informatics generate vast amounts of sensitive data, thereby presenting a significant risk of data breaches when using DL models, necessitating robust security measures to prevent unauthorized access, theft, or alteration of the data. However, the development and testing of DL models are restricted by a shortage of highquality medical datasets. Furthermore, ethical concerns related to patient privacy, informed consent, and bias arise with the use of DL models in medical applications, necessitating the development of guidelines and regulations to guarantee ethical usage. In addition, integrating these models into clinical workflows and educating clinicians on their effective usage and result interpretation presents a significant challenge to their adoption in clinical settings. Another issue is the difficulty of DL models to generalize to new data beyond the training data, which is crucial in medical applications for generalizing to new patient populations or disease types. Addressing these open issues demands collaboration among researchers, clinicians, and policymakers [155]. If adequately addressed, DL models can revolutionize the field of IoT-based bio- and medical informatics, leading to better patient outcomes.
7.1 Key research challenges
7.1.1 Explainability
to characterize [157]. This lack of explainability is a significant obstacle in computational biology, where the trustworthiness of a DL model is essential for sensitive clinical decision-making applications. It is equally important to understand why a model can make accurate predictions as it is to understand how it makes those predictions in biology. For instance, in protein function and structure prediction, we must understand the policies controlling a protein’s 3D geometry and attributes. Addressing these problems is crucial for providing biological insights and making practical decisions in clinical settings.
7.1.2 Effective training
7.1.3 Data security and privacy
7.1.4 Interoperability and data integration
7.1.5 Real-time monitoring and diagnosis
continuous collection and analysis of data, allowing for timely diagnosis and treatment of medical conditions. For example, wearable sensors can monitor vital signs such as heart rate, blood pressure, and oxygen saturation in real time, providing continuous data streams for DL models to analyze. These models can then identify patterns and anomalies that may indicate a potential medical issue, allowing for early intervention and treatment. Real-time monitoring and diagnosis can also improve patient outcomes by enabling personalized treatment plans. By continuously collecting and analyzing data on a patient’s condition, DL models can identify individualized treatment approaches that are tailored to a patient’s specific needs. However, there are also challenges to implementing realtime monitoring and diagnosis in IoT-based bio- and medical informatics. These include the need for secure and reliable data transmission, integrating data from multiple sources, and developing effective and interpretable DL models that can provide accurate and timely diagnoses. Real-time monitoring and diagnosis is a critical application area of DL in IoT-based bio- and medical informatics. This involves continuously collecting data from various sensors and devices, processing it in real time using DL models and providing real-time feedback to medical professionals or patients. One example of real-time monitoring and diagnosis is in wearable devices that collect data on heart rate, blood pressure, and other vital signs. DL models can analyze this data in real time and alert medical professionals if any abnormalities or anomalies are detected. This can help medical professionals make timely interventions and prevent adverse health outcomes. Another example is in medical imaging, where DL models can analyze medical images in real time and provide quick and accurate diagnoses. This can be especially useful in emergencies where quick decisions must be made based on limited information. Real-time monitoring and diagnosis have the potential to improve patient outcomes and reduce healthcare costs by enabling early interventions and preventing adverse events. However, it also presents challenges related to data privacy and security and the need for robust and reliable DL models that can operate in real time. This requires the use of high-performance computing and advanced ML techniques.
7.1.6 Predictive analytics
can identify patterns and make predictions about future health events by analyzing data from various sources, such as medical devices, electronic health records, and patientgenerated data. For example, predictive analytics can be used to identify patients who are at high risk of developing a particular disease or condition, allowing clinicians to intervene early and prevent the onset of the disease. In addition to predicting future health events, predictive analytics can also be used to optimize treatment plans and improve patient outcomes. By analyzing data from previous patients with similar conditions, DL models can identify the most effective treatment options for individual patients and provide personalized treatment recommendations. Real-time monitoring and diagnosis can benefit greatly from the use of predictive analytics, as it allows clinicians to take proactive measures to prevent adverse health events and improve patient outcomes. However, it is important to note that predictive analytics is only as accurate as the data it is based on. Therefore, it is crucial to ensure that the data used for training and testing DL models is accurate, representative, and unbiased.
7.1.7 Ethical and legal considerations
7.1.8 Human-computer interaction
7.1.9 Scalability and generalizability
improve the scalability and generalizability of models. For example, transfer learning is a technique that allows models to reuse learned features from one task to another, reducing the amount of data required for training and improving generalizability. Additionally, federated learning is a technique that allows models to be trained on distributed datasets, reducing the amount of data that needs to be transferred and improving scalability. Addressing scalability and generalizability issues is crucial for successfully deploying DL models in IoT-based bio- and medical informatics [163].
7.2 Future works
7.2.1 Multimodal data integration
7.2.2 Federated learning
how well it will perform in other applications, such as genomics or clinical decision-making. Future research could investigate the performance of federated learning in different medical applications. Communication between devices in a federated learning setup must be secure to ensure patient privacy and prevent data breaches. Future research could focus on developing communication protocols that are both secure and efficient, allowing for effective federated learning across a wide range of medical applications. Medical data often come from a variety of sources and in different formats, making it challenging to integrate for use in federated learning. Future research could focus on developing techniques to address data heterogeneity, such as data normalization and data augmentation, to improve the effectiveness of federated learning. The ultimate goal of federated learning in IoTbased bio- and medical informatics is to improve patient outcomes. Future research could focus on developing frameworks for the deployment of federated learning models in clinical practice, including how to integrate them into existing clinical workflows effectively.
7.2.3 Explainable AI
7.2.4 Transfer learning
7.2.5 Personalized healthcare monitoring
multiple sources such as wearable devices, medical sensors, and electronic health records. DL techniques can be used to fuse this data to comprehensively view an individual’s health status. Multimodal data fusion using DL can help improve the accuracy and reliability of personalized healthcare monitoring systems. Anomaly detection is an important aspect of personalized healthcare monitoring as it helps in identifying unusual patterns in an individual’s health status. DL techniques can be used to identify these patterns and raise alarms if necessary. This can be particularly useful in detecting chronic diseases or sudden health emergencies. Real-time monitoring of an individual’s health status can be achieved using wearable devices and IoT-enabled sensors. DL models can be deployed on these devices to continuously monitor an individual’s health status and provide real-time alerts if necessary. This can be particularly useful for elderly or high-risk patients.
7.2.6 Real-time diagnosis and treatment planning
records to provide a real-time diagnosis. These models can be deployed on IoT-enabled devices to provide immediate feedback to healthcare providers. This can be particularly useful in emergencies where quick diagnosis is critical. DL models can be used to develop personalized treatment plans for patients. These models can take into account an individual’s medical history, genetic information, and other factors to provide tailored treatment recommendations. IoT-enabled devices can be used to monitor a patient’s response to treatment and adjust the treatment plan accordingly. Decision support systems using DL can help healthcare providers make informed decisions about diagnosis and treatment. These systems can provide recommendations based on patient data, medical guidelines, and other relevant information. Predictive analytics using DL can help in predicting a patient’s response to treatment and potential health risks. These models can be trained on large datasets of medical records to identify patterns and predict potential health issues. This can be particularly useful in preventive healthcare. DL models can be trained to analyze medical images such as X-rays, MRIs, and CT scans. These models can help healthcare providers identify abnormalities and diagnose diseases. IoT-enabled devices can be used to capture and transmit these images in real time, enabling remote diagnosis and treatment planning. Privacy and security are major concerns in real-time diagnosis and treatment planning. DL models can be used to ensure the privacy and security of patient data. Techniques such as federated learning can be used to train models on distributed datasets without compromising privacy. In realtime diagnosis and treatment planning, it is important to provide explainable and interpretable models to gain the trust of patients and healthcare providers. Explainable AI techniques can be used to provide insights into the inner workings of these models. These are just some of the future works and ideas that can be explored in real-time diagnosis and treatment planning using DL and IoT-based bio- and medical informatics. With the increasing availability of healthcare data and the advancement of DL techniques, real-time diagnosis, and treatment planning has the potential to transform the way we deliver healthcare [165].
7.2.7 Predictive maintenance of medical devices
analyze data from sensors such as temperature, pressure, and vibration to detect abnormalities and potential failures. Anomaly detection using DL can help identify unusual patterns in medical device data. These models can help detect issues that may not be immediately apparent to the human eye and raise alarms if necessary. Prognostic models using DL can be used to predict the remaining useful life of medical devices. These models can help healthcare providers plan for the maintenance and replacement of medical devices before they fail. Predictive maintenance scheduling using DL can help healthcare providers optimize maintenance schedules based on the predicted failure rates of medical devices. This can help reduce downtime and improve the reliability of medical devices. Fault diagnosis using DL can help healthcare providers quickly identify and diagnose issues with medical devices. These models can analyze sensor data and provide repair or replacement recommendations. Predictive maintenance models can be integrated with electronic health records to view medical device performance and patient outcomes comprehensively. This can help healthcare providers make informed medical device management and patient care decisions. These are just some of the future works and ideas that can be explored in the predictive maintenance of medical devices using DL and IoT-based bio- and medical informatics. With the increasing use of medical devices and the need for reliable and safe healthcare delivery, predictive maintenance can potentially improve healthcare systems’ efficiency and effectiveness.
7.2.8 Optimization of drug discovery
patient response to treatment and identify subgroups that are more likely to benefit from a drug. DL models can be used to develop personalized treatment plans based on an individual’s genetic information, medical history, and other factors. These models can predict the effectiveness of different drugs and help healthcare providers make informed treatment decisions. DL models can be integrated with electronic health records to provide a comprehensive view of patient health and treatment outcomes. This can help healthcare providers make informed drug treatment and patient care decisions. These are just some of the future works and ideas that can be explored in the optimization of drug discovery using DL and IoT-based bio- and medical informatics. With the increasing demand for new and effective drugs, drug discovery optimization has the potential to transform the pharmaceutical industry and improve patient outcomes.
7.2.9 Medical imaging analysis
medical informatics. With the increasing use of medical imaging in healthcare, medical imaging analysis has the potential to improve the accuracy and efficiency of diagnosis and treatment planning.
7.2.10 Health monitoring with wearable IoT devices and DL
7.2.11 Telemedicine
bio- and medical informatics can help improve the quality of telemedicine services and enhance patient outcomes. DL models can be trained to remotely analyze patient data such as medical images, laboratory results, and vital signs. This can help improve the accuracy and speed of diagnosis, especially in areas with limited access to healthcare professionals. DL models can be used to develop chatbots and virtual assistants that can communicate with patients and provide medical advice. This can help improve patient access to healthcare services and reduce the workload of healthcare professionals. IoT-based wearable devices can be used to remotely monitor patient health data such as heart rate, blood pressure, and respiratory rate. DL models can analyze this data in real time and alert healthcare professionals if any changes require attention. DL models can be used to analyze patient data to identify patients who are at risk of developing certain diseases. This can help healthcare professionals to provide proactive care and prevent disease progression. DL models can be used to develop personalized treatment plans based on patient data. This can help improve treatment outcomes and reduce healthcare costs by avoiding unnecessary treatments. DL models can be used to develop automated triage systems that can identify patients who require urgent care. This can help reduce wait times for patients who require immediate attention. Telemedicine services can be integrated with electronic health records to provide a comprehensive view of patient health and treatment outcomes. This can help healthcare providers make informed decisions about patient care. These are just some of the future works and ideas that can be explored in telemedicine with the integration of DL and IoT-based bio- and medical informatics. With the increasing demand for telemedicine services, the integration of these technologies has the potential to improve access to healthcare services and enhance patient outcomes.
7.2.12 Predictive analytics for healthcare
personalized treatment plans based on patient data. This can help improve treatment outcomes and reduce healthcare costs by avoiding unnecessary treatments. Predictive analytics can be used to optimize healthcare resources such as hospital beds, staff, and equipment. DL models can be used to predict patient demand and optimize resource allocation accordingly. DL models can be used to analyze patient data to identify potential drug interactions and adverse events. This can help healthcare providers to provide safer and more effective drug therapies. DL models can be used to develop clinical decision support systems that can assist healthcare providers in making informed decisions about patient care. This can help improve patient outcomes and reduce healthcare costs by avoiding unnecessary tests and treatments. DL models can be used to analyze population health data to identify health trends and disease outbreaks. This can help healthcare providers to develop targeted interventions to prevent the spread of disease. These are just some of the future works and ideas that can be explored in predictive analytics for healthcare with the integration of DL and IoT-based bio- and medical informatics. With the increasing demand for predictive analytics in healthcare, integrating these technologies has the potential to improve patient outcomes and reduce healthcare costs.
8 Conclusion and limitation
accuracy, sensitivity, specificity, F-score, adaptability, scalability, and latency. However, certain features, such as security and convergence time, are underutilized. To evaluate and perform the proposed methods, various programming languages are used. Furthermore, we anticipate that our investigation will provide a valuable guide for further research on DL and medical usage in medical and bioinformatics issues.
Declarations
Open Access This article is licensed under a Creative Commons Attribution 4.0 International License, which permits use, sharing, adaptation, distribution and reproduction in any medium or format, as long as you give appropriate credit to the original author(s) and the source, provide a link to the Creative Commons licence, and indicate if changes were made. The images or other third party material in this article are included in the article’s Creative Commons licence, unless indicated otherwise in a credit line to the material. If material is not included in the article’s Creative Commons licence and your intended use is not permitted by statutory regulation or exceeds the permitted use, you will need to obtain permission directly from the copyright holder. To view a copy of this licence, visit http://creativecommons. org/licenses/by/4.0/.
References
- Muhammad AN et al (2021) Deep learning application in smart cities: recent development, taxonomy, challenges and research prospects. Neural Comput Appl 33(7):2973-3009
- Nosratabadi S et al (2020) State of the art survey of deep learning and machine learning models for smart cities and urban sustainability. In: International conference on global research and education. Springer
- Shafqat
et al (2022) Standard ner tagging scheme for big data healthcare analytics built on unified medical corpora. J Artif Intell Technol 2(4):152-157 - Atitallah SB et al (2020) Leveraging deep learning and iot big data analytics to support the smart cities development: review and future directions. Comput Sci Rev 38:100303
- Kök I, Şimşek MU, Özdemir (2017) A deep learning model for air quality prediction in smart cities. In: 2017 IEEE international conference on big data (Big Data). 2017. IEEE
- Bolhasani H, Mohseni M, Rahmani AM (2021) Deep learning applications for IoT in health care: a systematic review. Inform Med Unlocked 23:100550
- Rastogi R, Chaturvedi DK, Sagar S, Tandon N, Rastogi AR (2022) Brain tumor analysis using deep learning: sensor and iotbased approach for futuristic healthcare. In: Bioinformatics and medical applications: big data using deep learning algorithms, pp 171-190
- Roopashree
et al (2022) An IoT based authentication system for therapeutic herbs measured by local descriptors using machine learning approach. Measurement 200:111484 - Bharadwaj HK et al (2021) A review on the role of machine learning in enabling IoT based healthcare applications. IEEE Access 9:38859-38890
- Awotunde JB et al (2021) Disease diagnosis system for IoTbased wearable body sensors with machine learning algorithm. In: Hybrid artificial intelligence and IoT in healthcare. 2021. Springer, pp 201-222
- Alansari Z et al (2017) Computational intelligence tools and databases in bioinformatics. In: 2017 4th IEEE international conference on engineering technologies and applied sciences (ICETAS). 2017. IEEE
- Daoud H, Williams P, Bayoumi M (2020) IoT based efficient epileptic seizure prediction system using deep learning. In: 2020 IEEE 6th world forum on internet of things (WF-IoT). 2020. IEEE
- Wu Y et al (2021) Deep learning for big data analytics. Mobile Netw Appl 26(6):2315-2317
- Ambika N (2022) An economical machine learning approach for anomaly detection in IoT environment. In: Bioinformatics and medical applications: big data using deep learning algorithms, 2022: pp 215-234
- Srivastava M (2020) A Surrogate data-based approach for validating deep learning model used in healthcare. In: Applications of deep learning and big IoT on personalized healthcare services. 2020. IGI Global, pp 132-146
- da Costa KA et al (2019) Internet of things: a survey on machine learning-based intrusion detection approaches. Comput Netw 151:147-157
- Min S, Lee B, Yoon S (2017) Deep learning in bioinformatics. Brief Bioinform 18(5):851-869
- Aminizadeh
et al (2023) The applications of machine learning techniques in medical data processing based on distributed computing and the internet of things. In: Computer methods and programs in biomedicine, 2023, p 107745 - Li Y et al (2019) Deep learning in bioinformatics: introduction, application, and perspective in the big data era. Methods 166:4-21
- Cao
et al (2020) Ensemble deep learning in bioinformatics. Nat Mach Intell 2(9):500-508 - Tang B et al (2019) Recent advances of deep learning in bioinformatics and computational biology. Front Genet 10:214
- Koumakis L (2020) Deep learning models in genomics; are we there yet? Comput Struct Biotechnol J 18:1466-1473
- Dhombres F, Charlet J (2019) Formal medical knowledge representation supports deep learning algorithms, bioinformatics pipelines, genomics data analysis, and big data processes. Yearb Med Inform 28(01):152-155
- Peng
et al (2018) The advances and challenges of deep learning application in biological big data processing. Curr Bioinform 13(4):352-359 - Chen Y-Z et al (2021) nhKcr: a new bioinformatics tool for predicting crotonylation sites on human nonhistone proteins based on deep learning. Brief Bioinform 22(6):bbab146
- Chen Y et al (2016) Gene expression inference with deep learning. Bioinformatics 32(12):1832-1839
- Jabbar MA (2022) An insight into applications of deep learning in bioinformatics. In: Deep learning, machine learning and IoT in biomedical and health informatics. CRC Press, pp 175-197
- Khurana S et al (2018) DeepSol: a deep learning framework for sequence-based protein solubility prediction. Bioinformatics 34(15):2605-2613
- Baranwal M et al (2020) A deep learning architecture for metabolic pathway prediction. Bioinformatics 36(8):2547-2553
- Shahid O et al (2021) Machine learning research towards combating COVID-19: Virus detection, spread prevention, and medical assistance. J Biomed Inform 117:103751
- Roy PK et al (2023) Analysis of community question-answering issues via machine learning and deep learning: state-of-the-art review. CAAI Trans Intell Technol 8(1):95-117
- Samanta RK et al (2022) Scope of machine learning applications for addressing the challenges in next-generation wireless networks. CAAI Trans Intell Technol 7(3):395-418
- Wang W et al (2023) Fully Bayesian analysis of the relevance vector machine classification for imbalanced data problem. CAAI Trans Intell Technol 8(1):192-205
- Ashrafuzzaman M (2021) Artificial intelligence, machine learning and deep learning in ion channel bioinformatics. Membranes 11(9):672
- Fiannaca A et al (2018) Deep learning models for bacteria taxonomic classification of metagenomic data. BMC Bioinform 19(7):61-76
- Li F et al (2020) DeepCleave: a deep learning predictor for caspase and matrix metalloprotease substrates and cleavage sites. Bioinformatics 36(4):1057-1065
- Meher J (2021) Potential applications of deep learning in bioinformatics big data analysis. In: Advanced deep learning for engineers and scientists, 2021, pp 183-193
- Preuer K et al (2018) DeepSynergy: predicting anti-cancer drug synergy with Deep Learning. Bioinformatics 34(9):1538-1546
- Xia Z et al (2019) DeeReCT-PolyA: a robust and generic deep learning method for PAS identification. Bioinformatics 35(14):2371-2379
- Fang B et al (2022) Deep generative inpainting with comparative sample augmentation. J Comput Cogn Eng 1(4):174-180
- Wang
et al (2020) Block switching: a stochastic approach for deep learning security. arXiv preprint arXiv:2002.07920, 2020 - Kumar I, Singh SP (2022) Machine learning in bioinformatics. In: Bioinformatics. Academic Press, pp 443-456
- Yu L et al (2018) Drug and nondrug classification based on deep learning with various feature selection strategies. Curr Bioinform 13(3):253-259
- Jurtz VI et al (2017) An introduction to deep learning on biological sequence data: examples and solutions. Bioinformatics 33(22):3685-3690
- Deng Y et al (2020) A multimodal deep learning framework for predicting drug-drug interaction events. Bioinformatics 36(15):4316-4322
- Shakeel N, Shakeel S (2022) Context-free word importance scores for attacking neural networks. J Comput Cogn Eng 1(4):187-192
- Oubounyt M et al (2019) DeePromoter: robust promoter predictor using deep learning. Front Genet 10:286
- Leung MK et al (2014) Deep learning of the tissue-regulated splicing code. Bioinformatics 30(12):i121-i129
- Dai B, Bailey-Kellogg C (2021) Protein interaction interface region prediction by geometric deep learning. Bioinformatics 37(17):2580-2588
- Luo F et al (2019) DeepPhos: prediction of protein phosphorylation sites with deep learning. Bioinformatics 35(16):2766-2773
- Liu X (2022) Real-world data for the drug development in the digital era. J Artif Intell Technol 2(2):42-46
- Wei L et al (2018) Prediction of human protein subcellular localization using deep learning. J Parallel Distrib Comput 117:212-217
- Heidari A et al (2023) A new lung cancer detection method based on the chest CT images using federated learning and blockchain systems. Artif Intell Med 141:102572
- Cai Q et al (2023) Image neural style transfer: a review. Comput Electr Eng 108:108723
- Ai Q et al (2021) Editorial for FGCS special issue: intelligent IoT systems for healthcare and rehabilitation. Elsevier, New York, pp 770-773
- Niu L-Y, Wei Y, Liu W-B, Long JY, Xue T-H (2023) Research Progress of spiking neural network in image classification: a review. In: Applied intelligence, pp 1-25
- Karnati M et al (2022) A novel multi-scale based deep convolutional neural network for detecting COVID-19 from X-rays. Appl Soft Comput 125:109109
- Ravindran U, Gunavathi C (2023) A survey on gene expression data analysis using deep learning methods for cancer diagnosis. Prog Biophys Mol Biol 177:1-13
- Zheng M et al (2022) A hybrid CNN for image denoising. J Artif Intell Technol 2(3):93-99
- Togneri R, Prati R, Nagano H, Kamienski C (2023) Data-driven water need estimation for IoT-based smart irrigation: a survey. Expert Syst Appl 225:120194
- Sheng N, Huang L, Lu Y, Wang H, Yang L, Gao L, Xie X, Fu Y, Wang Y (2023) Data resources and computational methods for lncRNA-disease association prediction. Comput Biol Med 153:106527
- Sharan RV, Rahimi-Ardabili H (2023) Detecting acute respiratory diseases in the pediatric population using cough sound features and machine learning: a systematic review. Int J Med Inform 176:105093
- Bhosale YH, Patnaik KS (2023) Bio-medical imaging (X-ray, CT, ultrasound, ECG), genome sequences applications of deep neural network and machine learning in diagnosis, detection, classification, and segmentation of COVID-19: a meta-analysis & systematic review. Multimed Tools Appl 82:39157-39210. https://doi.org/10.1007/s11042-023-15029-1
- Azhari F, Sennersten CC, Lindley CA et al (2023) Deep learning implementations in mining applications: a compact critical review. Artif Intell Rev 56:14367-14402. https://doi.org/10. 1007/s10462-023-10500-9
- Nazir S, Dickson DM, Akram MU (2023) Survey of explainable artificial intelligence techniques for biomedical imaging with deep neural networks. Comput Biol Med 156:106668
- Jacob TP, Pravin A, Kumar RR (2022) A secure IoT based healthcare framework using modified RSA algorithm using an artificial hummingbird based CNN. Trans Emerg Tel Tech 33(12):e4622. https://doi.org/10.1002/ett. 4622
- Phan HT, Nguyen NT, Hwang D (2023) Aspect-level sentiment analysis: a survey of graph convolutional network methods. Inf Fusion 91:149-172
- Qiu D, Cheng Y, Wang X (2023) Medical image super-resolution reconstruction algorithms based on deep learning: a survey. Comput Methods Prog Biomed 238:107590
- Sanders LM et al (2023) Biological research and self-driving labs in deep space supported by artificial intelligence. Nat Mach Intell 5(3):208-219
- Rezende PM et al (2022) Evaluating hierarchical machine learning approaches to classify biological databases. Brief Bioinform 23(4):bbac216
- Yi H-C et al (2022) Graph representation learning in bioinformatics: trends, methods and applications. Brief Bioinform 23(1):bbab340
- Sharma S (2021) The bioinformatics: detailed review of various applications of cluster analysis. Glob J Appl Data Sci Internet Things 5:1-2021
- Serra A, Galdi P, Tagliaferri R (2018) Machine learning for bioinformatics and neuroimaging. Wiley Interdiscip Rev Data Min Knowl Discov 8(5):e1248
- Liu L et al (2019) A smart dental health-IoT platform based on intelligent hardware, deep learning, and mobile terminal. IEEE J Biomed Health Inform 24(3):898-906
- Nematzadeh S et al (2022) Tuning hyperparameters of machine learning algorithms and deep neural networks using metaheuristics: a bioinformatics study on biomedical and biological cases. Comput Biol Chem 97:107619
- Kumar H, Sharma S (2021) Contribution of deep learning in bioinformatics. Glob J Appl Data Sci Internet Things 5:1-202
- Jia D et al (2021) Breast cancer case identification based on deep learning and bioinformatics analysis. Front Genet 12:628136
- Pastorino J, Biswas AK (2022) Data adequacy bias impact in a data-blinded semi-supervised GAN for privacy-aware COVID19 chest X-ray classification. In: Proceedings of the 13th ACM international conference on bioinformatics, computational biology and health informatics, 2022
- Auwul MR et al (2021) Bioinformatics and machine learning approach identifies potential drug targets and pathways in COVID-19. Brief Bioinform 22(5):bbab120
- Lan L et al (2020) Generative adversarial networks and its applications in biomedical informatics. Front Public Health 8:164
- Han C et al (2021) MADGAN: Unsupervised medical anomaly detection GAN using multiple adjacent brain MRI slice reconstruction. BMC Bioinform 22(2):1-20
- Balogh OM et al (2022) Efficient link prediction in the proteinprotein interaction network using topological information in a generative adversarial network machine learning model. BMC Bioinform 23(1):1-19
- Giansanti V et al (2019) Comparing deep and machine learning approaches in bioinformatics: a miRNA-target prediction case study. In: International conference on computational science. 2019. Springer
- Lyu C et al (2017) Long short-term memory RNN for biomedical named entity recognition. BMC Bioinform 18(1):1-11
- ElAbd H et al (2020) Amino acid encoding for deep learning applications. BMC Bioinform 21(1):1-14
- Liu J, Gong X (2019) Attention mechanism enhanced LSTM with residual architecture and its application for protein-protein interaction residue pairs prediction. BMC Bioinform 20(1):1-11
- Wang D et al (2017) MusiteDeep: a deep-learning framework for general and kinase-specific phosphorylation site prediction. Bioinformatics 33(24):3909-3916
- Zhao Y, Shao J, Asmann YW (2022) Assessment and optimization of explainable machine learning models applied to transcriptomic data. Genom Proteom Bioinform 20:899-911
- Souri A et al (2020) A new machine learning-based healthcare monitoring model for student’s condition diagnosis in Internet of Things environment. Soft Comput 24(22):17111-17121
- D’Orazio M et al (2022) Machine learning phenomics (MLP) combining deep learning with time-lapse-microscopy for monitoring colorectal adenocarcinoma cells gene expression and drug-response. Sci Rep 12(1):1-14
- Karim MR et al (2021) Deep learning-based clustering approaches for bioinformatics. Brief Bioinform 22(1):393-415
- Aydin Z (2020) Performance analysis of machine learning and bioinformatics applications on high performance computing systems. Acad Platf J Eng Sci 8(1):1-14
- Mohamed Shakeel P et al (2018) Maintaining security and privacy in health care system using learning based deep-Q-networks. J Med Syst 42(10):1-10
- Huang L et al (2019) Patient clustering improves efficiency of federated machine learning to predict mortality and hospital stay time using distributed electronic medical records. J Biomed Inform 99:103291
- Wang X, Jiang X, Vaidya J (2021) Efficient verification for outsourced genome-wide association studies. J Biomed Inform 117:103714
- Cui J et al (2021) FeARH: Federated machine learning with anonymous random hybridization on electronic medical records. J Biomed Inform 117:103735
- Giansanti V et al (2019) Comparing deep and machine learning approaches in bioinformatics: a miRNA-target prediction case study. In: Computational science-ICCS 2019: 19th international conference, Faro, Portugal, June 12-14, 2019, proceedings, part III, vol 19, 2019. Springer
- Lyu C et al (2017) Long short-term memory RNN for biomedical named entity recognition. BMC Bioinform 18:1-11
- ElAbd H et al (2020) Amino acid encoding for deep learning applications. BMC Bioinform 21:1-14
- Liu J, Gong X (2019) Attention mechanism enhanced LSTM with residual architecture and its application for protein-protein interaction residue pairs prediction. BMC Bioinform 20:1-11
- Mohamed Shakeel P et al (2018) Maintaining security and privacy in health care system using learning based deep-Q-networks. J Med Syst 42:1-10
- Sarbaz M et al (2022) Adaptive optimal control of chaotic system using backstepping neural network concept. In: 2022 8th international conference on control, instrumentation and automation (ICCIA). 2022. IEEE
- Bagheri M et al (2020) Data conditioning and forecasting methodology using machine learning on production data for a well pad. In: Offshore technology conference. 2020. OTC
- Soleimani R, Lobaton E (2022) Enhancing inference on physiological and kinematic periodic signals via phase-based interpretability and multi-task learning. Information 13(7):326
- Mirzaeibonehkhater M (2018) Developing a dynamic recommendation system for personalizing educational content within an e-learning network. 2018: Purdue University
- Morteza A et al (2023) Deep learning hyperparameter optimization: application to electricity and heat demand prediction for buildings. Energy Build 289:113036
- Webber J et al (2017) Study on idle slot availability prediction for WLAN using a probabilistic neural network. In: 2017 23rd Asia-Pacific conference on communications (APCC). 2017. IEEE
- Webber J et al (2022) Improved human activity recognition using majority combining of reduced-complexity sensor branch classifiers. Electronics 11(3):392
- Gera T et al (2021) Dominant feature selection and machine learning-based hybrid approach to analyze android ransomware. Secur Commun Netw 2021:1-22
- Bukhari SNH, Webber J, Mehbodniya A (2022) Decision tree based ensemble machine learning model for the prediction of
111. Heidari A et al (2023) Machine learning applications in internet-of-drones: systematic review, recent deployments, and open issues. ACM Comput Surv 55(12):1-45
112. Singh R et al (2022) Analysis of network slicing for management of 5G networks using machine learning techniques. Wirel Commun Mobile Comput 2022:9169568
113. He P et al (2022) Towards green smart cities using Internet of Things and optimization algorithms: a systematic and bibliometric review. Sustain Comput Inform Syst 36:100822
114. Sadi M et al (2022) Special session: on the reliability of conventional and quantum neural network hardware. In: 2022 IEEE 40th VLSI test symposium (VTS). 2022. IEEE
115. Moradi M, Weng Y, Lai Y-C (2022) Defending smart electrical power grids against cyberattacks with deep Q-learning. P R X Energy 1:033005
116. Zhai Z-M et al (2023) Detecting weak physical signal from noise: a machine-learning approach with applications to mag-netic-anomaly-guided navigation. Phys Rev Appl 19(3):034030
117. Li Z, Han C, Coit DW (2023) System reliability models with dependent degradation processes. In: Advances in reliability and maintainability methods and engineering applications: essays in honor of professor Hong-Zhong Huang on his 60th birthday. 2023. Springer, pp 475-497
118. Zhang Y et al (2019) Fault diagnosis strategy of CNC machine tools based on cascading failure. J Intell Manuf 30:2193-2202
119. Shen G, Zeng W, Han C, Liu P, Zhang Y (2017) Determination of the average maintenance time of CNC machine tools based on type II failure correlation. Eksploatacja i Niezawodność 19(4)
120. Shen G et al (2018) Fault analysis of machine tools based on grey relational analysis and main factor analysis. J Phys Conf Ser. IOP Publishing
121. Han C, Fu X (2023) Challenge and opportunity: deep learningbased stock price prediction by using Bi-directional LSTM model. Front Bus Econ Manag 8(2):51-54
122. Darbandi M (2017) Proposing new intelligent system for suggesting better service providers in cloud computing based on Kalman filtering. Int J Technol Innov Res 24(1):1-9
123. Dehghani F, Larijani A (2023) Average portfolio optimization using multi-layer neural networks with risk consideration. Available at SSRN, 2023
124. Rezaei M, Rastgoo R, Athitsos V (2023) TriHorn-Net: a model for accurate depth-based 3D hand pose estimation. Expert Syst Appl 223:119922
125. Ahmadi SS, Khotanlou H (2022) A hybrid of inference and stacked classifiers to indoor scenes classification of rgb-d images. In: 2022 International conference on machine vision and image processing (MVIP). 2022. IEEE
126. Mirzapour O, Arpanahi SK (2017) Photovoltaic parameter estimation using heuristic optimization. In: 2017 IEEE 4th international conference on knowledge-based engineering and innovation (KBEI). 2017. IEEE
127. Khorshidi M, Ameri M, Goli A (2023) Cracking performance evaluation and modelling of RAP mixtures containing different recycled materials using deep neural network model. Road Mater Pavement Des 1-20
128. Rastegar RM et al (2022) From evidence to assessment: DEVELOPING a scenario-based computational design algorithm to support informed decision-making in primary care clinic design workflow. Int J Archit Comput 20(3):567-586
129. Esmaeili N, Bamdad Soofi J (2022) Expounding the knowledge conversion processes within the occupational safety and health management system (OSH-MS) using concept mapping. Int J Occup Saf Ergon 28(2):1000-1015
130. Akyash M, Mohammadzade H, Behroozi H (2021) Dtw-merge: a novel data augmentation technique for time series classification. arXiv preprint arXiv:2103.01119
131. Darbandi M (2017) Proposing new intelligence algorithm for suggesting better services to cloud users based on Kalman filtering. J Comput Sci Appl 5(1):11-16
132. Darbandi M (2017) Kalman filtering for estimation and prediction servers with lower traffic loads for transferring highlevel processes in cloud computing. Int J Technol Innov Res 23(1):10-20
133. Liu H et al (2023) MEMS piezoelectric resonant microphone array for lung sound classification. J Micromech Microeng 33(4):044003
134. Loghmani N, Moqadam R, Allahverdy A (2022) Brain tumor segmentation using multimodal mri and convolutional neural network. In: 2022 30th international conference on electrical engineering (ICEE). 2022. IEEE
135. Niknejad N, Caro JL, Bidese-Puhl R, Bao Y, Staiger EA (2023) Equine kinematic gait analysis using stereo videography and deep learning: stride length and stance duration estimation. J ASABE 66(4):865-877
136. Amiri Z et al (2023) Resilient and dependability management in distributed environments: a systematic and comprehensive literature review. Clust Comput 26(2):1565-1600
137. Zeng Q et al (2020) Hyperpolarized Xe NMR signal advancement by metal-organic framework entrapment in aqueous solution. Proc Natl Acad Sci 117(30):17558-17563
138. Liu N et al (2021) An eyelid parameters auto-measuring method based on 3D scanning. Displays 69:102063
139. Li C et al (2021) Long noncoding RNA p21 enhances autophagy to alleviate endothelial progenitor cells damage and promote endothelial repair in hypertension through SESN2/AMPK/TSC2 pathway. Pharmacol Res 173:105920
140. Li B et al (2022) Dynamic event-triggered security control for networked control systems with cyber-attacks: a model predictive control approach. Inf Sci 612:384-398
141. Li H, Peng R, Wang Z-A (2018) On a diffusive susceptible-infected-susceptible epidemic model with mass action mechanism and birth-death effect: analysis, simulations, and comparison with other mechanisms. SIAM J Appl Math 78(4):2129-2153
142. Amiri Z et al (2023) The personal health applications of machine learning techniques in the internet of behaviors. Sustainability 15(16):12406
143. Zhu Y et al (2021) Deep learning-based predictive identification of neural stem cell differentiation. Nat Commun 12(1):2614
144. Yang S et al (2022) Dual-level representation enhancement on characteristic and context for image-text retrieval. IEEE Trans Circuits Syst Video Technol 32(11):8037-8050
145. Yan L et al (2023) Multi-feature fusing local directional ternary pattern for facial expressions signal recognition based on video communication system. Alex Eng J 63:307-320
146. Dai
147. Yan L et al (2021) Method of reaching consensus on probability of food safety based on the integration of finite credible data on block chain. IEEE access 9:123764-123776
148. Jiang H et al (2020) An energy-efficient framework for internet of things underlaying heterogeneous small cell networks. IEEE Trans Mob Comput 21(1):31-43
149. Sun L, Zhang M, Wang B, Tiwari P (2023) Few-shot classincremental learning for medical time series classification. IEEE J Biomed Health Inform. https://doi.org/10.1109/JBHI.2023. 3247861
150. Gao Z, Pan X, Shao J, Jiang X, Su Z, Jin K, Ye J (2023) Automatic interpretation and clinical evaluation for fundus fluorescein angiography images of diabetic retinopathy patients by deep learning. Br J Ophthalmol 107(12):1852-1858
151. Wang H et al (2022) Transcranial alternating current stimulation for treating depression: a randomized controlled trial. Brain 145(1):83-91
152. Luan D et al (2022) Robust two-stage location allocation for emergency temporary blood supply in postdisaster. Discret Dyn Nat Soc 2022:1-20
153. Chen G et al (2022) Continuance intention mechanism of middle school student users on online learning platform based on qualitative comparative analysis method. Math Probl Eng 2022:1-12
154. Cui G et al (2013) Synthesis and characterization of Eu (III) complexes of modified cellulose and poly (N-isopropylacrylamide). Carbohyd Polym 94(1):77-81
155. Cheng B et al (2016) Situation-aware IoT service coordination using the event-driven SOA paradigm. IEEE Trans Netw Serv Manag 13(2):349-361
156. Cheng B et al (2017) Situation-aware dynamic service coordination in an IoT environment. IEEE/ACM Trans Netw 25(4):2082-2095
157. Zhuang Y, Jiang N, Xu Y (2022) Progressive distributed and parallel similarity retrieval of large CT image sequences in mobile telemedicine networks. Wirel Commun Mob Comput 2022:1-13
158. Tang Y et al (2021) An improved method for soft tissue modeling. Biomed Signal Process Control 65:102367
159. Zhang Z et al (2022) Endoscope image mosaic based on pyramid ORB. Biomed Signal Process Control 71:103261
160. Lu S et al (2023) Iterative reconstruction of low-dose CT based on differential sparse. Biomed Signal Process Control 79:104204
161. Lu S et al (2023) Soft tissue feature tracking based on deepmatching network. CMES Comput Model Eng Sci 136(1):363
162. Liu M et al (2023) Three-dimensional modeling of heart soft tissue motion. Appl Sci 13(4):2493
163. Heidari A et al (2023) A hybrid approach for latency and battery lifetime optimization in IoT devices through offloading and CNN learning. Sustain Comput Inform Syst 39:100899
164. Heidari A, Jafari Navimipour N, Unal M (2022) The history of computing in Iran (Persia)-since the achaemenid empire. Technologies 10(4):94
165. Ahmadpour S-S, Heidari A, Navimpour NJ, Asadi M-A, Yalcin S (2023) An efficient design of multiplier for using in nano-scale IoT systems using atomic silicon. IEEE Internet Things J 10(16):14908-14909. https://doi.org/10.1109/JIOT.2023. 3267165
166. Amiri Z, Heidari A, Navimipour NJ et al (2023) Adventures in data analysis: a systematic review of deep learning techniques for pattern recognition in cyber-physical-social systems. Multimed Tools Appl. https://doi.org/10.1007/s11042-023-16382-x
- Arash Heidari
arash_heidari@ieee.org
Nima Jafari Navimipour
nima.navimipour@khas.edu.tr; jnnima@khas.edu.tw
1 Department of Computer Engineering, Tabriz Branch, Islamic Azad University, Tabriz, Iran2 Department of Software Engineering, Haliç University, 34060 Istanbul, Turkey3 Department of Computer Engineering, Kadir Has University, Istanbul, Turkey
4 Future Technology Research Center, National Yunlin University of Science and Technology, 64002 Douliou, Yunlin, Taiwan5 Computer Engineering Department, Hamedan Branch, Islamic Azad University, Hamedan, Iran
Immunology Research Center, Tabriz University of Medical Sciences, Tabriz, Iran
