DOI: https://doi.org/10.1038/s41368-023-00275-8
PMID: https://pubmed.ncbi.nlm.nih.gov/38413562
تاريخ النشر: 2024-02-27
محور الخلايا العدلة والخلايا العظمية يعزز تدمير العظام في التهاب اللثة
الملخص
تلعب تفاعلات خلايا المناعة والستروما دورًا رئيسيًا في الصحة والأمراض. في التهاب اللثة، وهو أكثر الأمراض المعدية شيوعًا لدى البشر، تتجمع خلايا المناعة في الغشاء المخاطي الفموي وتعزز تدمير العظام من خلال تحفيز تعبير عامل تنشيط مستقبلات عامل نواة كابا ب (RANKL) في الخلايا العظمية مثل الخلايا البانية للعظام وخلايا الرباط اللثوي. ومع ذلك، فإن الآلية التفصيلية التي تكمن وراء تفاعلات خلايا المناعة والعظام في التهاب اللثة ليست مفهومة تمامًا. هنا، قمنا بإجراء تحليل تسلسل RNA على مستوى الخلية الواحدة على آفات اللثة في الفئران وأظهرنا أن التفاعل بين العدلات والخلايا العظمية يشارك في فقدان العظام الناتج عن التهاب اللثة. أظهرت آفات اللثة تسللًا ملحوظًا للعدلات، واقترحت التحليلات الحاسوبية أن العدلات تفاعلت مع الخلايا العظمية من خلال إنتاج السيتوكينات. من بين السيتوكينات المعبر عنها في العدلات اللثوية، كان الأونكوساتين M (OSM) يحفز بشكل قوي تعبير RANKL في الخلايا البانية للعظام الأولية، وأدى حذف مستقبل OSM في الخلايا العظمية إلى تحسين كبير في فقدان العظام الناتج عن التهاب اللثة. حددت تحليلات البيانات الجينية الوبائية منطقة معزز RANKL المنظمة بواسطة OSM في الخلايا العظمية، وأظهرت الفئران التي تفتقر إلى هذا المعزز انخفاضًا في فقدان العظام اللثوي مع الحفاظ على التمثيل الغذائي العظمي الفسيولوجي. تسلط هذه النتائج الضوء على دور العدلات في تنظيم العظام خلال العدوى البكتيرية، مما يبرز الآلية الجديدة التي تكمن وراء التفاعل المناعي العظمي.
https://doi.org/10.1038/s41368-023-00275-8
مقدمة
لا يزال غير واضح كيف تؤدي التفاعلات الخلوية المعقدة في النهاية إلى امتصاص العظام بواسطة الخلايا العظمية.
النتائج
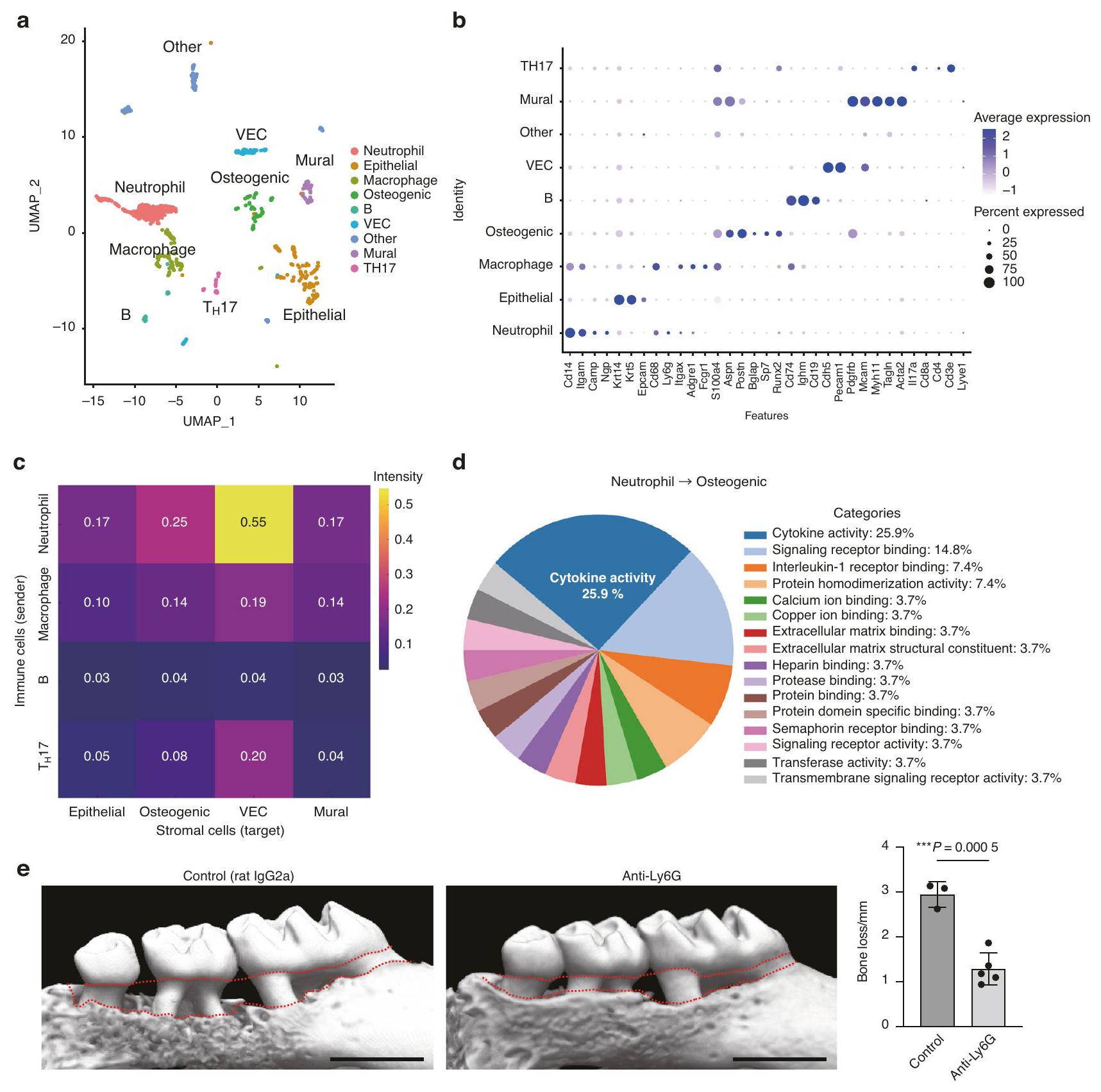
لفهم البيئة الدقيقة الخلوية التي تقف وراء تدمير العظام في اللثة بدقة خلوية واحدة، قمنا بإجراء تسلسل RNA أحادي الخلية باستخدام خلايا مستمدة من أنسجة اللثة لفئران تم تحفيز التهاب اللثة باستخدام الرباط بعد 7 أيام من وضع الرباط، عندما لوحظت التسلل الملحوظ للخلايا المناعية وتسارع تدمير العظام في الدراسات السابقة.
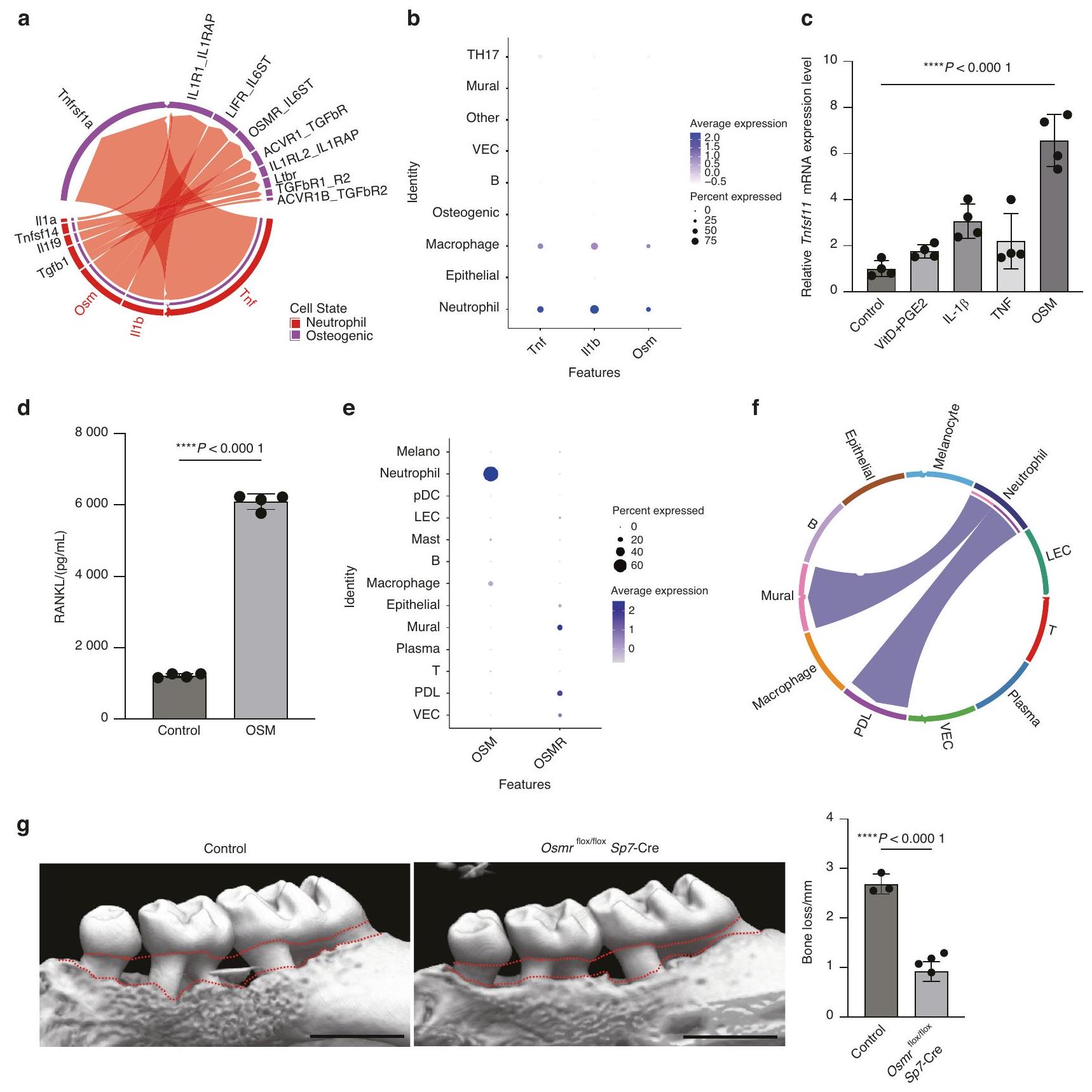
في الخلايا العظمية من خلال إنتاج السيتوكينات. قمنا بتحليل بيانات CellChat من خلال التركيز على التفاعل الذي يتوسطه السيتوكين بين العدلات والخلايا العظمية، ووجدنا أن عامل نخر الورم (TNF) و IL-1 و
المعطلة
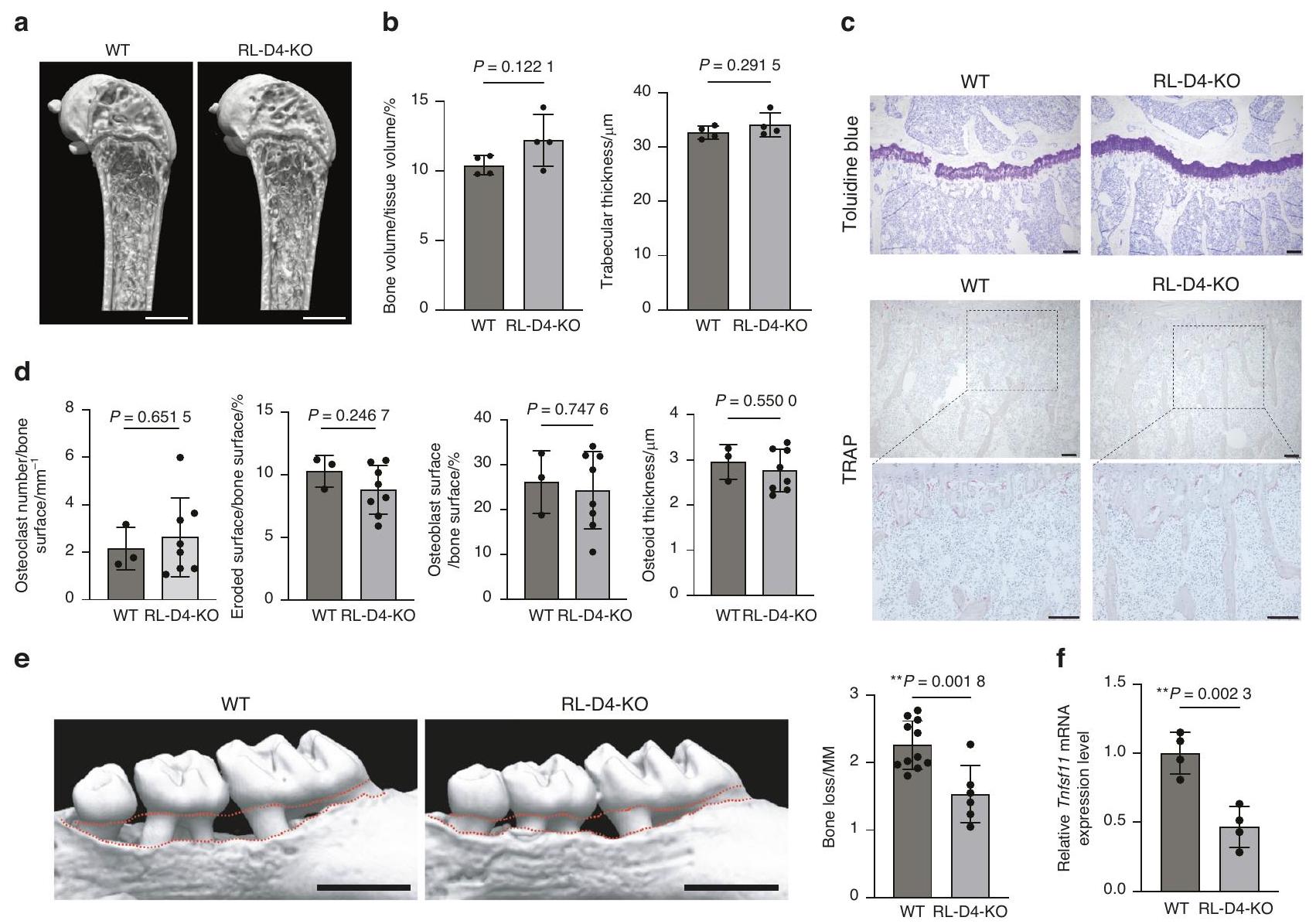
للتحقيق في أهمية منطقة RL-D4 في إعادة تشكيل العظام الفسيولوجية، قمنا أولاً بتحليل العظام الطويلة لفئران RL-D4-KO في حالة مستقرة. أظهرت تحليلات التصوير المقطعي المحوسب (micro-CT) أن فئران RL-D4-KO كانت لديها نمط عظام طبيعي مقارنة بالفئران WT في ظل الظروف الفسيولوجية (الشكل 4a، b).
نقاش
في أن ظهور مثل هذه المحسنات المرتبطة بالالتهاب RANKL خلال التطور قد ربط تنشيط المناعة بامتصاص العظام بواسطة الخلايا العظمية وبالتالي دفع ظهور مرض العظام الالتهابي، وأقدم دليل على ذلك هو تلف العظام الناتج عن التهاب اللثة في زاحف أرضي عمره 275 مليون سنة.
طرق
الفئران
نموذج الفأر لالتهاب اللثة الناتج عن الرباط
تسلسل RNA أحادي الخلية وتحليل البيانات
إدارة الأجسام المضادة anti-Ly6G في الجسم الحي
اختبار تحفيز السيتوكين
إيليزا
تحليل qPCR
مستوى التعبير. كانت البرايمرات المستخدمة هي: Gapdh، 5′-TCCAC-CACCCTGTTGCTGTA-3′
تحليل العظام الطويلة
بيانات ChIP-seq وتحليلات الأنماط
التحليلات الإحصائية
توفر البيانات
شكر وتقدير
مساهمات المؤلفين
معلومات إضافية
REFERENCES
- Lamont, R. J., Koo, H. & Hajishengallis, G. The oral microbiota: dynamic communities and host interactions. Nat. Rev. Microbiol. 16, 745-759 (2018).
- Darveau, R. P. Periodontitis: a polymicrobial disruption of host homeostasis. Nat. Rev. Microbiol. 8, 481-490 (2010).
- Maekawa, T. et al. Porphyromonas gingivalis manipulates complement and TLR signaling to uncouple bacterial clearance from inflammation and promote dysbiosis. Cell Host Microbe 15, 768-778 (2014).
- Tsukasaki, M. et al. Host defense against oral microbiota by bone-damaging T cells. Nat. Commun. 9, 701 (2018).
- Tsukasaki, M. & Takayanagi, H. Osteoimmunology: evolving concepts in bone-immune interactions in health and disease. Nat. Rev. Immunol. 19, 626-642 (2019).
- Hajishengallis, G. & Chavakis, T. Local and systemic mechanisms linking periodontal disease and inflammatory comorbidities. Nat. Rev. Immunol. 21, 426-440 (2021).
- Genco, R. J. & Sanz, M. Clinical and public health implications of periodontal and systemic diseases: An overview. Periodontol 2000 83, 7-13 (2020).
- Xiao, E. et al. Diabetes Enhances IL-17 Expression and Alters the Oral Microbiome to Increase Its Pathogenicity. Cell Host Microbe 22, 120-128.e4 (2017).
- Graves, D. T., Ding, Z. & Yang, Y. The impact of diabetes on periodontal diseases. Periodontol 2000 82, 214-224 (2020).
- Mammen, M. J., Scannapieco, F. A. & Sethi, S. Oral-lung microbiome interactions in lung diseases. Periodontol 2000 83, 234-241 (2020).
- Kitamoto, S. et al. The Intermucosal Connection between the Mouth and Gut in Commensal Pathobiont-Driven Colitis. Cell 182, 447-462.e14 (2020).
- Caetano, A. J. et al. Defining human mesenchymal and epithelial heterogeneity in response to oral inflammatory disease. Elife 10, e62810 (2021).
- Caetano, A. J., Human Cell Atlas Oral and Craniofacial Bionetwork, Sequeira, I. & Byrd, K. M. A Roadmap for the Human Oral and Craniofacial Cell Atlas. J. Dent. Res. 101, 1274-1288 (2022).
- Caetano, A. J. et al. Spatially resolved transcriptomics reveals pro-inflammatory fibroblast involved in lymphocyte recruitment through CXCL8 and CXCL10. Elife 12, e81525 (2023).
- Qian, S.-J. et al. Single-cell RNA sequencing identifies new inflammationpromoting cell subsets in Asian patients with chronic periodontitis. Front. Immunol. 12, 711337 (2021).
- Williams, D. W. et al. Human oral mucosa cell atlas reveals a stromal-neutrophil axis regulating tissue immunity. Cell 184, 4090-4104.e15 (2021).
- Kondo, T., Gleason, A., Okawa, H., Hokugo, A. & Nishimura, I. Mouse gingival single-cell transcriptomic atlas: An activated fibroblast subpopulation guides oral barrier immunity in periodontitis. eLife https://doi.org/10.7554/elife. 88183 (2023).
- Kondo, T. et al. Oral microbial extracellular DNA initiates periodontitis through gingival degradation by fibroblast-derived cathepsin K in mice. Commun. Biol. 5, 962 (2022).
- Abe, T. & Hajishengallis, G. Optimization of the ligature-induced periodontitis model in mice. J. Immunol. Methods 394, 49-54 (2013).
- Dutzan, N. et al. A dysbiotic microbiome triggers
cells to mediate oral mucosal immunopathology in mice and humans. Sci. Transl. Med. 10, eaat0797 (2018). - Udagawa, N. et al. Osteoclast differentiation by RANKL and OPG signaling pathways. J. Bone Miner. Metab. 39, 19-26 (2021).
- Tsukasaki, M. RANKL and osteoimmunology in periodontitis. J. Bone Miner. Metab. 39, 82-90 (2021).
- Kourtzelis, I. et al. DEL-1 promotes macrophage efferocytosis and clearance of inflammation. Nat. Immunol. 20, 40-49 (2019).
- Men, Y. et al. Gli1+ periodontium stem cells are regulated by osteocytes and occlusal force. Dev. Cell 54, 639-654.e6 (2020).
- Iwayama, T. et al. Plap-1 lineage tracing and single-cell transcriptomics reveal cellular dynamics in the periodontal ligament. Development 149, dev201203 (2022).
- Jin, S. et al. Inference and analysis of cell-cell communication using CellChat. Nat. Commun. 12, 1088 (2021).
- Eskan, M. A. et al. The leukocyte integrin antagonist Del-1 inhibits IL-17-mediated inflammatory bone loss. Nat. Immunol. 13, 465-473 (2012).
- Shin, J. et al. DEL-1 restrains osteoclastogenesis and inhibits inflammatory bone loss in nonhuman primates. Sci. Transl. Med. 7, 307ra155 (2015).
- Schmidt, E. P., Lee, W. L., Zemans, R. L., Yamashita, C. & Downey, G. P. On, around, and through: neutrophil-endothelial interactions in innate immunity. Physiology 26, 334-347 (2011).
- Hajishengallis, G. New developments in neutrophil biology and periodontitis. Periodontol 2000 82, 78-92 (2020).
- Walker, E. C. et al. Oncostatin M promotes bone formation independently of resorption when signaling through leukemia inhibitory factor receptor in mice. J. Clin. Invest. 120, 582-592 (2010).
- Galli, C. et al. Targeted deletion of a distant transcriptional enhancer of the receptor activator of nuclear factor-kappaB ligand gene reduces bone remodeling and increases bone mass. Endocrinology 149, 146-153 (2008).
- Onal, M. et al. Unique distal enhancers linked to the mouse Tnfsf11 gene direct tissue-specific and inflammation-induced expression of RANKL. Endocrinology 157, 482-496 (2016).
- Bishop, K. A., Meyer, M. B. & Pike, J. W. A novel distal enhancer mediates cytokine induction of mouse RANKI gene expression. Mol. Endocrinol. 23, 2095-2110 (2009).
- Onal, M. et al. The RANKL distal control region is required for the increase in RANKL expression, but not the bone loss, associated with hyperparathyroidism or lactation in adult mice. Mol. Endocrinol. 26, 341-348 (2012).
- Silva, L. M., Kim, T. S. & Moutsopoulos, N. M. Neutrophils are gatekeepers of mucosal immunity. Immunol. Rev. 314, 125-141 (2023).
- Dutzan, N., Konkel, J. E., Greenwell-Wild, T. & Moutsopoulos, N. M. Characterization of the human immune cell network at the gingival barrier. Mucosal Immunol. 9, 1163-1172 (2016).
- Landzberg, M., Doering, H., Aboodi, G. M., Tenenbaum, H. C. & Glogauer, M. Quantifying oral inflammatory load: oral neutrophil counts in periodontal health and disease. J. Periodontal Res. 50, 330-336 (2015).
- Silva, L. M. et al. Fibrin is a critical regulator of neutrophil effector function at the oral mucosal barrier. Science 374, eabl5450 (2021).
- Kim, T. S. et al. Neutrophil extracellular traps and extracellular histones potentiate IL-17 inflammation in periodontitis. J. Exp. Med. 220, e20221751 (2023).
- Assuma, R., Oates, T., Cochran, D., Amar, S. & Graves, D. T. IL-1 and TNF antagonists inhibit the inflammatory response and bone loss in experimental periodontitis. J. Immunol. 160, 403-409 (1998).
- Hajishengallis, G., Lamont, R. J. & Graves, D. T. The enduring importance of animal models in understanding periodontal disease. Virulence 6, 229-235 (2015).
- Fan, Y. et al. Creating an atlas of the bone microenvironment during oral inflammatory-related bone disease using single-cell profiling. Elife 12, e82537 (2023).
- Cai, B. et al. N2-polarized neutrophils guide bone mesenchymal stem cell recruitment and initiate bone regeneration: a missing piece of the bone regeneration puzzle. Adv. Sci. 8, e2100584 (2021).
- Moutsopoulos, N. M. et al. Defective neutrophil recruitment in leukocyte adhesion deficiency type I disease causes local IL-17-driven inflammatory bone loss. Sci. Transl. Med. 6, 229ra40 (2014).
- Moutsopoulos, N. M. et al. Interleukin-12 and Interleukin-23 blockade in leukocyte adhesion deficiency type 1. N. Engl. J. Med. 376, 1141-1146 (2017).
- West, N. R., Owens, B. M. J. & Hegazy, A. N. The oncostatin M-stromal cell axis in health and disease. Scand. J. Immunol. 88, e12694 (2018).
- Boniface, K. et al. Oncostatin M secreted by skin infiltrating T lymphocytes is a potent keratinocyte activator involved in skin inflammation. J. Immunol. 178, 4615-4622 (2007).
- Okamoto, H. et al. The synovial expression and serum levels of interleukin-6, interleukin-11, leukemia inhibitory factor, and oncostatin
in rheumatoid arthritis. Arthritis Rheum. 40, 1096-1105 (1997). - Lin, W. et al. Mapping the immune microenvironment for mandibular alveolar bone homeostasis at single-cell resolution. Bone Res. 9, 17 (2021).
- Torossian, F. et al. Macrophage-derived oncostatin M contributes to human and mouse neurogenic heterotopic ossifications. JCI Insight 2, e96034 (2017).
- West, N. R. et al. Oncostatin M drives intestinal inflammation and predicts response to tumor necrosis factor-neutralizing therapy in patients with inflammatory bowel disease. Nat. Med. 23, 579-589 (2017).
- Friedrich, M. et al. IL-1-driven stromal-neutrophil interactions define a subset of patients with inflammatory bowel disease that does not respond to therapies. Nat. Med. 27, 1970-1981 (2021).
- Lin, S.-J., Chen, Y.-L., Kuo, M. Y.-B., Li, C.-L. & Lu, H.-K. Measurement of gp130 cytokines oncostatin M and IL-6 in gingival crevicular fluid of patients with chronic periodontitis. Cytokine 30, 160-167 (2005).
- Okamoto, K. et al. Osteoimmunology: the conceptual framework unifying the immune and skeletal systems. Physiol. Rev. 97, 1295-1349 (2017).
- Kim, S., Yamazaki, M., Shevde, N. K. & Pike, J. W. Transcriptional control of receptor activator of nuclear factor-kappaB ligand by the protein kinase A activator forskolin and the transmembrane glycoprotein 130-activating cytokine, oncostatin M , is exerted through multiple distal enhancers. Mol. Endocrinol. 21, 197-214 (2007).
- Meyer, M. B., Benkusky, N. A., Lee, C.-H. & Pike, J. W. Genomic determinants of gene regulation by 1,25-Dihydroxyvitamin D3 during osteoblast-lineage cell differentiation. J. Biol. Chem. 289, 19539-19554 (2014).
- Yan, M. et al. Identification of an intronic enhancer regulating RANKL expression in osteocytic cells. Bone Res. 11, 43 (2023).
- Yan, M. et al. ETS1 governs pathological tissue-remodeling programs in diseaseassociated fibroblasts. Nat. Immunol. 23, 1330-1341 (2022).
- Onal, M., St John, H. C., Danielson, A. L. & Pike, J. W. Deletion of the distal Tnfsf11 RL-D2 enhancer that contributes to PTH-Mediated RANKL expression in osteoblast lineage cells results in a high bone mass phenotype in mice. J. Bone Miner. Res. 31, 416-429 (2016).
- Zhou, Y. et al. RANKL+ senescent cells under mechanical stress: a therapeutic target for orthodontic root resorption using senolytics. Int. J. Oral. Sci. 15, 20 (2023).
- Wang, Q. et al. Single-cell transcriptomic atlas of gingival mucosa in type 2 diabetes. J. Dent. Res. 101, 1654-1664 (2022).
- O’Brien, C. A. Control of RANKL gene expression. Bone 46, 911-919 (2010).
- Tsukasaki, M. & Takayanagi, H. Osteoclast biology in the single-cell era. Inflamm. Regen. 42, 27 (2022).
- Armingol, E., Officer, A., Harismendy, O. & Lewis, N. E. Deciphering cell-cell interactions and communication from gene expression. Nat. Rev. Genet. 22, 71-88 (2021).
- Robinson, J. T. et al. Integrative genomics viewer. Nat. Biotechnol. 29, 24-26 (2011).
- Castro-Mondragon, J. A. et al. JASPAR 2022: the 9th release of the open-access database of transcription factor binding profiles. Nucleic Acids Res. 50, D165-D173 (2022).
- Grant, C. E., Bailey, T. L. & Noble, W. S. FIMO: scanning for occurrences of a given motif. Bioinformatics 27, 1017-1018 (2011).
© The Author(s) 2024
Department of Immunology, Graduate School of Medicine and Faculty of Medicine, The University of Tokyo, 7-3-1, Hongo, Bunkyo-ku, Tokyo, Japan; Department of Microbiology, Tokyo Dental College, 2-1-14 Kanda-Misaki-cho, Chiyoda-ku, Tokyo, Japan; Oral Health Science Center, Tokyo Dental College, 2-9-18, Kanda-Misaki-cho, Chiyodaku, Tokyo, Japan; Department of Osteoimmunology, Graduate School of Medicine and Faculty of Medicine, The University of Tokyo, 7-3-1, Hongo, Bunkyo-ku, Tokyo, Japan; Unit of Prosthodontics, Laboratory of Oral-Maxillofacial Biology Faculty of Odonto-Stomatology, University of Medicine and Pharmacy at Ho Chi Minh City, Ho Chi Minh City, Vietnam; Department of Oral and Maxillofacial Surgery, Department of Sensory and Motor System Medicine, Graduate School of Medicine, The University of Tokyo, Tokyo, Japan; Division of Rheumatology, Department of Internal Medicine, Keio University School of Medicine, Tokyo, Japan and Department of Laboratory Animal Medicine, Research Institute, National Center for Global Health and Medicine, Tokyo, Japan
Correspondence: Masayuki Tsukasaki (tsuka-im@m.u-tokyo.ac.jp) or Hiroshi Takayanagi (takayana@m.u-tokyo.ac.jp)
DOI: https://doi.org/10.1038/s41368-023-00275-8
PMID: https://pubmed.ncbi.nlm.nih.gov/38413562
Publication Date: 2024-02-27
The neutrophil-osteogenic cell axis promotes bone destruction in periodontitis
Abstract
The immune-stromal cell interactions play a key role in health and diseases. In periodontitis, the most prevalent infectious disease in humans, immune cells accumulate in the oral mucosa and promote bone destruction by inducing receptor activator of nuclear factor-kB ligand (RANKL) expression in osteogenic cells such as osteoblasts and periodontal ligament cells. However, the detailed mechanism underlying immune-bone cell interactions in periodontitis is not fully understood. Here, we performed single-cell RNAsequencing analysis on mouse periodontal lesions and showed that neutrophil-osteogenic cell crosstalk is involved in periodontitis-induced bone loss. The periodontal lesions displayed marked infiltration of neutrophils, and in silico analyses suggested that the neutrophils interacted with osteogenic cells through cytokine production. Among the cytokines expressed in the periodontal neutrophils, oncostatin M (OSM) potently induced RANKL expression in the primary osteoblasts, and deletion of the OSM receptor in osteogenic cells significantly ameliorated periodontitis-induced bone loss. Epigenomic data analyses identified the OSM-regulated RANKL enhancer region in osteogenic cells, and mice lacking this enhancer showed decreased periodontal bone loss while maintaining physiological bone metabolism. These findings shed light on the role of neutrophils in bone regulation during bacterial infection, highlighting the novel mechanism underlying osteoimmune crosstalk.
https://doi.org/10.1038/s41368-023-00275-8
INTRODUCTION
remains unclear how the complex cellular interactions ultimately induce osteoclastic bone resorption.
RESULTS
To understand the cellular microenvironment underlying periodontal bone destruction at a single-cell resolution, we performed scRNA-seq using cells derived from the periodontal tissue of ligature-induced periodontitis mice at 7 days after ligature placement when the marked infiltration of immune cells and accelerated bone destruction were observed in previous studies.
in osteogenic cells through cytokine production. We analyzed the CellChat data by focusing on the cytokine-mediated interaction between neutrophils and osteogenic cells, and found that tumor necrosis factor (TNF), IL-1

deficient mice
To investigate the importance of the RL-D4 region in physiological bone remodeling, we first analyzed the long bones of RL-D4-KO mice at steady state. Microcomputed tomography (micro-CT) analyses showed that RL-D4-KO mice had normal bone phenotype compared to WT mice under physiological conditions (Fig. 4a, b).

DISCUSSION

that the emergence of such inflammation-associated RANKL enhancers during evolution may have linked immune activation to osteoclastic bone resorption and thus driven the emergence of inflammatory bone disease, the earliest evidence of which is periodontitis-induced bone damage in a 275 million-year-old terrestrial reptile.
METHODS
Mice
Ligature-induced periodontitis mouse model
Single-cell RNA-seq and data analysis
Administration of anti-Ly6G antibody in vivo
Cytokine stimulation assay
ELISA
qPCR analysis
expression level. The primers used were: Gapdh, 5′-TCCAC-CACCCTGTTGCTGTA-3′ and
Analysis of the long bones
ChIP-seq data and motif analyses
Statistical analyses
DATA AVAILABILITY
ACKNOWLEDGEMENTS
AUTHOR CONTRIBUTIONS
ADDITIONAL INFORMATION
REFERENCES
- Lamont, R. J., Koo, H. & Hajishengallis, G. The oral microbiota: dynamic communities and host interactions. Nat. Rev. Microbiol. 16, 745-759 (2018).
- Darveau, R. P. Periodontitis: a polymicrobial disruption of host homeostasis. Nat. Rev. Microbiol. 8, 481-490 (2010).
- Maekawa, T. et al. Porphyromonas gingivalis manipulates complement and TLR signaling to uncouple bacterial clearance from inflammation and promote dysbiosis. Cell Host Microbe 15, 768-778 (2014).
- Tsukasaki, M. et al. Host defense against oral microbiota by bone-damaging T cells. Nat. Commun. 9, 701 (2018).
- Tsukasaki, M. & Takayanagi, H. Osteoimmunology: evolving concepts in bone-immune interactions in health and disease. Nat. Rev. Immunol. 19, 626-642 (2019).
- Hajishengallis, G. & Chavakis, T. Local and systemic mechanisms linking periodontal disease and inflammatory comorbidities. Nat. Rev. Immunol. 21, 426-440 (2021).
- Genco, R. J. & Sanz, M. Clinical and public health implications of periodontal and systemic diseases: An overview. Periodontol 2000 83, 7-13 (2020).
- Xiao, E. et al. Diabetes Enhances IL-17 Expression and Alters the Oral Microbiome to Increase Its Pathogenicity. Cell Host Microbe 22, 120-128.e4 (2017).
- Graves, D. T., Ding, Z. & Yang, Y. The impact of diabetes on periodontal diseases. Periodontol 2000 82, 214-224 (2020).
- Mammen, M. J., Scannapieco, F. A. & Sethi, S. Oral-lung microbiome interactions in lung diseases. Periodontol 2000 83, 234-241 (2020).
- Kitamoto, S. et al. The Intermucosal Connection between the Mouth and Gut in Commensal Pathobiont-Driven Colitis. Cell 182, 447-462.e14 (2020).
- Caetano, A. J. et al. Defining human mesenchymal and epithelial heterogeneity in response to oral inflammatory disease. Elife 10, e62810 (2021).
- Caetano, A. J., Human Cell Atlas Oral and Craniofacial Bionetwork, Sequeira, I. & Byrd, K. M. A Roadmap for the Human Oral and Craniofacial Cell Atlas. J. Dent. Res. 101, 1274-1288 (2022).
- Caetano, A. J. et al. Spatially resolved transcriptomics reveals pro-inflammatory fibroblast involved in lymphocyte recruitment through CXCL8 and CXCL10. Elife 12, e81525 (2023).
- Qian, S.-J. et al. Single-cell RNA sequencing identifies new inflammationpromoting cell subsets in Asian patients with chronic periodontitis. Front. Immunol. 12, 711337 (2021).
- Williams, D. W. et al. Human oral mucosa cell atlas reveals a stromal-neutrophil axis regulating tissue immunity. Cell 184, 4090-4104.e15 (2021).
- Kondo, T., Gleason, A., Okawa, H., Hokugo, A. & Nishimura, I. Mouse gingival single-cell transcriptomic atlas: An activated fibroblast subpopulation guides oral barrier immunity in periodontitis. eLife https://doi.org/10.7554/elife. 88183 (2023).
- Kondo, T. et al. Oral microbial extracellular DNA initiates periodontitis through gingival degradation by fibroblast-derived cathepsin K in mice. Commun. Biol. 5, 962 (2022).
- Abe, T. & Hajishengallis, G. Optimization of the ligature-induced periodontitis model in mice. J. Immunol. Methods 394, 49-54 (2013).
- Dutzan, N. et al. A dysbiotic microbiome triggers
cells to mediate oral mucosal immunopathology in mice and humans. Sci. Transl. Med. 10, eaat0797 (2018). - Udagawa, N. et al. Osteoclast differentiation by RANKL and OPG signaling pathways. J. Bone Miner. Metab. 39, 19-26 (2021).
- Tsukasaki, M. RANKL and osteoimmunology in periodontitis. J. Bone Miner. Metab. 39, 82-90 (2021).
- Kourtzelis, I. et al. DEL-1 promotes macrophage efferocytosis and clearance of inflammation. Nat. Immunol. 20, 40-49 (2019).
- Men, Y. et al. Gli1+ periodontium stem cells are regulated by osteocytes and occlusal force. Dev. Cell 54, 639-654.e6 (2020).
- Iwayama, T. et al. Plap-1 lineage tracing and single-cell transcriptomics reveal cellular dynamics in the periodontal ligament. Development 149, dev201203 (2022).
- Jin, S. et al. Inference and analysis of cell-cell communication using CellChat. Nat. Commun. 12, 1088 (2021).
- Eskan, M. A. et al. The leukocyte integrin antagonist Del-1 inhibits IL-17-mediated inflammatory bone loss. Nat. Immunol. 13, 465-473 (2012).
- Shin, J. et al. DEL-1 restrains osteoclastogenesis and inhibits inflammatory bone loss in nonhuman primates. Sci. Transl. Med. 7, 307ra155 (2015).
- Schmidt, E. P., Lee, W. L., Zemans, R. L., Yamashita, C. & Downey, G. P. On, around, and through: neutrophil-endothelial interactions in innate immunity. Physiology 26, 334-347 (2011).
- Hajishengallis, G. New developments in neutrophil biology and periodontitis. Periodontol 2000 82, 78-92 (2020).
- Walker, E. C. et al. Oncostatin M promotes bone formation independently of resorption when signaling through leukemia inhibitory factor receptor in mice. J. Clin. Invest. 120, 582-592 (2010).
- Galli, C. et al. Targeted deletion of a distant transcriptional enhancer of the receptor activator of nuclear factor-kappaB ligand gene reduces bone remodeling and increases bone mass. Endocrinology 149, 146-153 (2008).
- Onal, M. et al. Unique distal enhancers linked to the mouse Tnfsf11 gene direct tissue-specific and inflammation-induced expression of RANKL. Endocrinology 157, 482-496 (2016).
- Bishop, K. A., Meyer, M. B. & Pike, J. W. A novel distal enhancer mediates cytokine induction of mouse RANKI gene expression. Mol. Endocrinol. 23, 2095-2110 (2009).
- Onal, M. et al. The RANKL distal control region is required for the increase in RANKL expression, but not the bone loss, associated with hyperparathyroidism or lactation in adult mice. Mol. Endocrinol. 26, 341-348 (2012).
- Silva, L. M., Kim, T. S. & Moutsopoulos, N. M. Neutrophils are gatekeepers of mucosal immunity. Immunol. Rev. 314, 125-141 (2023).
- Dutzan, N., Konkel, J. E., Greenwell-Wild, T. & Moutsopoulos, N. M. Characterization of the human immune cell network at the gingival barrier. Mucosal Immunol. 9, 1163-1172 (2016).
- Landzberg, M., Doering, H., Aboodi, G. M., Tenenbaum, H. C. & Glogauer, M. Quantifying oral inflammatory load: oral neutrophil counts in periodontal health and disease. J. Periodontal Res. 50, 330-336 (2015).
- Silva, L. M. et al. Fibrin is a critical regulator of neutrophil effector function at the oral mucosal barrier. Science 374, eabl5450 (2021).
- Kim, T. S. et al. Neutrophil extracellular traps and extracellular histones potentiate IL-17 inflammation in periodontitis. J. Exp. Med. 220, e20221751 (2023).
- Assuma, R., Oates, T., Cochran, D., Amar, S. & Graves, D. T. IL-1 and TNF antagonists inhibit the inflammatory response and bone loss in experimental periodontitis. J. Immunol. 160, 403-409 (1998).
- Hajishengallis, G., Lamont, R. J. & Graves, D. T. The enduring importance of animal models in understanding periodontal disease. Virulence 6, 229-235 (2015).
- Fan, Y. et al. Creating an atlas of the bone microenvironment during oral inflammatory-related bone disease using single-cell profiling. Elife 12, e82537 (2023).
- Cai, B. et al. N2-polarized neutrophils guide bone mesenchymal stem cell recruitment and initiate bone regeneration: a missing piece of the bone regeneration puzzle. Adv. Sci. 8, e2100584 (2021).
- Moutsopoulos, N. M. et al. Defective neutrophil recruitment in leukocyte adhesion deficiency type I disease causes local IL-17-driven inflammatory bone loss. Sci. Transl. Med. 6, 229ra40 (2014).
- Moutsopoulos, N. M. et al. Interleukin-12 and Interleukin-23 blockade in leukocyte adhesion deficiency type 1. N. Engl. J. Med. 376, 1141-1146 (2017).
- West, N. R., Owens, B. M. J. & Hegazy, A. N. The oncostatin M-stromal cell axis in health and disease. Scand. J. Immunol. 88, e12694 (2018).
- Boniface, K. et al. Oncostatin M secreted by skin infiltrating T lymphocytes is a potent keratinocyte activator involved in skin inflammation. J. Immunol. 178, 4615-4622 (2007).
- Okamoto, H. et al. The synovial expression and serum levels of interleukin-6, interleukin-11, leukemia inhibitory factor, and oncostatin
in rheumatoid arthritis. Arthritis Rheum. 40, 1096-1105 (1997). - Lin, W. et al. Mapping the immune microenvironment for mandibular alveolar bone homeostasis at single-cell resolution. Bone Res. 9, 17 (2021).
- Torossian, F. et al. Macrophage-derived oncostatin M contributes to human and mouse neurogenic heterotopic ossifications. JCI Insight 2, e96034 (2017).
- West, N. R. et al. Oncostatin M drives intestinal inflammation and predicts response to tumor necrosis factor-neutralizing therapy in patients with inflammatory bowel disease. Nat. Med. 23, 579-589 (2017).
- Friedrich, M. et al. IL-1-driven stromal-neutrophil interactions define a subset of patients with inflammatory bowel disease that does not respond to therapies. Nat. Med. 27, 1970-1981 (2021).
- Lin, S.-J., Chen, Y.-L., Kuo, M. Y.-B., Li, C.-L. & Lu, H.-K. Measurement of gp130 cytokines oncostatin M and IL-6 in gingival crevicular fluid of patients with chronic periodontitis. Cytokine 30, 160-167 (2005).
- Okamoto, K. et al. Osteoimmunology: the conceptual framework unifying the immune and skeletal systems. Physiol. Rev. 97, 1295-1349 (2017).
- Kim, S., Yamazaki, M., Shevde, N. K. & Pike, J. W. Transcriptional control of receptor activator of nuclear factor-kappaB ligand by the protein kinase A activator forskolin and the transmembrane glycoprotein 130-activating cytokine, oncostatin M , is exerted through multiple distal enhancers. Mol. Endocrinol. 21, 197-214 (2007).
- Meyer, M. B., Benkusky, N. A., Lee, C.-H. & Pike, J. W. Genomic determinants of gene regulation by 1,25-Dihydroxyvitamin D3 during osteoblast-lineage cell differentiation. J. Biol. Chem. 289, 19539-19554 (2014).
- Yan, M. et al. Identification of an intronic enhancer regulating RANKL expression in osteocytic cells. Bone Res. 11, 43 (2023).
- Yan, M. et al. ETS1 governs pathological tissue-remodeling programs in diseaseassociated fibroblasts. Nat. Immunol. 23, 1330-1341 (2022).
- Onal, M., St John, H. C., Danielson, A. L. & Pike, J. W. Deletion of the distal Tnfsf11 RL-D2 enhancer that contributes to PTH-Mediated RANKL expression in osteoblast lineage cells results in a high bone mass phenotype in mice. J. Bone Miner. Res. 31, 416-429 (2016).
- Zhou, Y. et al. RANKL+ senescent cells under mechanical stress: a therapeutic target for orthodontic root resorption using senolytics. Int. J. Oral. Sci. 15, 20 (2023).
- Wang, Q. et al. Single-cell transcriptomic atlas of gingival mucosa in type 2 diabetes. J. Dent. Res. 101, 1654-1664 (2022).
- O’Brien, C. A. Control of RANKL gene expression. Bone 46, 911-919 (2010).
- Tsukasaki, M. & Takayanagi, H. Osteoclast biology in the single-cell era. Inflamm. Regen. 42, 27 (2022).
- Armingol, E., Officer, A., Harismendy, O. & Lewis, N. E. Deciphering cell-cell interactions and communication from gene expression. Nat. Rev. Genet. 22, 71-88 (2021).
- Robinson, J. T. et al. Integrative genomics viewer. Nat. Biotechnol. 29, 24-26 (2011).
- Castro-Mondragon, J. A. et al. JASPAR 2022: the 9th release of the open-access database of transcription factor binding profiles. Nucleic Acids Res. 50, D165-D173 (2022).
- Grant, C. E., Bailey, T. L. & Noble, W. S. FIMO: scanning for occurrences of a given motif. Bioinformatics 27, 1017-1018 (2011).
© The Author(s) 2024
Department of Immunology, Graduate School of Medicine and Faculty of Medicine, The University of Tokyo, 7-3-1, Hongo, Bunkyo-ku, Tokyo, Japan; Department of Microbiology, Tokyo Dental College, 2-1-14 Kanda-Misaki-cho, Chiyoda-ku, Tokyo, Japan; Oral Health Science Center, Tokyo Dental College, 2-9-18, Kanda-Misaki-cho, Chiyodaku, Tokyo, Japan; Department of Osteoimmunology, Graduate School of Medicine and Faculty of Medicine, The University of Tokyo, 7-3-1, Hongo, Bunkyo-ku, Tokyo, Japan; Unit of Prosthodontics, Laboratory of Oral-Maxillofacial Biology Faculty of Odonto-Stomatology, University of Medicine and Pharmacy at Ho Chi Minh City, Ho Chi Minh City, Vietnam; Department of Oral and Maxillofacial Surgery, Department of Sensory and Motor System Medicine, Graduate School of Medicine, The University of Tokyo, Tokyo, Japan; Division of Rheumatology, Department of Internal Medicine, Keio University School of Medicine, Tokyo, Japan and Department of Laboratory Animal Medicine, Research Institute, National Center for Global Health and Medicine, Tokyo, Japan
Correspondence: Masayuki Tsukasaki (tsuka-im@m.u-tokyo.ac.jp) or Hiroshi Takayanagi (takayana@m.u-tokyo.ac.jp)
