DOI: https://doi.org/10.1186/s12880-024-01195-7
PMID: https://pubmed.ncbi.nlm.nih.gov/38243215
تاريخ النشر: 2024-01-19
نموذج هجين لشبكة عصبية عميقة لتصنيف صور أورام الدماغ متعددة الفئات
الملخص
النهج الحالي لتشخيص وتصنيف أورام الدماغ يعتمد على التقييم النسيجي لعينات الخزعة، وهو نهج يتسم بالتدخل، ويستغرق وقتًا طويلاً، ويكون عرضة للأخطاء اليدوية. هذه القيود تبرز الحاجة الملحة إلى نظام تصنيف متعدد الفئات يعتمد على التعلم العميق بشكل كامل لأورام الدماغ الخبيثة. يهدف هذا المقال إلى الاستفادة من شبكة عصبية تلافيفية عميقة (CNN) لتعزيز الكشف المبكر ويقدم ثلاثة نماذج CNN متميزة مصممة لمهام تصنيف مختلفة. يحقق النموذج الأول من CNN دقة كشف مثيرة للإعجاب تبلغ 99.53% لأورام الدماغ. النموذج الثاني من CNN، بدقة تبلغ 93.81%، يصنف أورام الدماغ بكفاءة إلى خمسة أنواع متميزة: طبيعي، غليوما، منينجيوما، نخامية، وانتقالية. علاوة على ذلك، يظهر النموذج الثالث من CNN دقة تبلغ
مقدمة
أو نسيج العمود الفقري المركزي الذي يعطل الوظيفة الطبيعية للدماغ. يتم تصنيف أورام الدماغ إلى فئتين رئيسيتين: حميدة وخبيثة. تنمو الأورام الحميدة ببطء وهي غير سرطانية؛ وهي نادرة نسبيًا ولا تنتشر. بالمقابل، تحتوي أورام الدماغ الخبيثة على خلايا سرطانية، وعادة ما تنشأ في منطقة واحدة من الدماغ قبل أن تنتشر بسرعة إلى مناطق أخرى من الدماغ والحبل الشوكي [2]. تشكل الأورام الخبيثة خطرًا صحيًا كبيرًا. تصنف منظمة الصحة العالمية (WHO) أورام الدماغ إلى أربع درجات بناءً على سلوكها داخل الدماغ: تعتبر الدرجات 1 و 2 أورامًا منخفضة الدرجة أو حميدة، بينما تصنف الدرجات 3 و 4 كأورام عالية الدرجة أو خبيثة. تتوفر عدة طرق تشخيصية، مثل التصوير المقطعي المحوسب (CT) وتخطيط كهربية الدماغ (EEG)، للكشف عن أورام الدماغ، لكن التصوير بالرنين المغناطيسي (MRI) هو الأكثر موثوقية واستخدامًا على نطاق واسع. ينتج التصوير بالرنين المغناطيسي صورًا داخلية مفصلة لأعضاء الجسم من خلال استخدام مجالات مغناطيسية قوية وموجات راديو [3]. بشكل أساسي، يمكن أن تميز فحوصات CT أو MRI المنطقة المتأثرة في الدماغ بسبب الورم عن الأنسجة السليمة. يمكن إجراء خزعات، وهي اختبارات سريرية تستخرج خلايا الدماغ، كتمهيد لجراحة الدماغ. الدقة أمر بالغ الأهمية في قياس خلايا الورم أو الوصول إلى تشخيصات دقيقة. يمثل ظهور التعلم الآلي (ML) فرصة لمساعدة أطباء الأشعة في تقديم معلومات دقيقة عن حالة المرض [4]. لقد تركت وفرة التقنيات الجديدة، وخاصة الذكاء الاصطناعي والتعلم الآلي، أثرًا لا يمحى في المجال الطبي، مما زود مختلف الأقسام الطبية، بما في ذلك التصوير الطبي، بأدوات لا غنى عنها لتعزيز عملياتها. بينما تتم معالجة صور الرنين المغناطيسي لمساعدة أطباء الأشعة في اتخاذ القرارات، يتم استخدام مجموعة متنوعة من استراتيجيات التعلم الآلي الآلي لأغراض التصنيف والتجزئة. بينما تحمل الطرق المراقبة لتصنيف أورام الدماغ وعدًا هائلًا، فإنها تتطلب خبرة متخصصة لتحسين تقنيات استخراج واختيار الميزات [5]. في التنقل وتحليل مجموعات البيانات الضخمة، يستفيد المتخصصون الطبيون الخبراء من دعم المساعدة الآلية. علاوة على ذلك، فإن الفشل في تحديد الأورام المهددة للحياة بدقة قد يؤدي إلى تأخيرات في العلاج للمرضى. لقد كانت تقنيات التعلم العميق (DL) في الكشف عن أورام الدماغ واستخراج رؤى ذات مغزى من أنماط البيانات لها تاريخ طويل. إن قدرة التعلم العميق على تصنيف ونمذجة سرطانات الدماغ معترف بها على نطاق واسع [6]. يعتمد علاج أورام الدماغ بشكل فعال على التشخيص المبكر والدقيق للمرض. تتأثر القرارات المتعلقة بطرق العلاج بعوامل مثل النوع المرضي للورم، والدرجة، والمرحلة عند التشخيص. لقد استغل أطباء الأورام العصبية أدوات التشخيص المدعومة بالحاسوب (CAD) لأغراض متنوعة، بما في ذلك الكشف عن الورم، والتصنيف، والتدرج ضمن مجال الأعصاب [7].
الأعمال ذات الصلة
باستخدام TensorFlow و Keras في لغة البرمجة بايثون، التي تم اختيارها لقدراتها القوية في تسريع المهام. كانت نسبة الدقة المحققة لنموذج CNN عند مستوى مثير للإعجاب
تحسين في الأبحاث المستقبلية [21]. في مجال تشخيص مرض الزهايمر، تم استخدام نهج الشبكات العصبية التلافيفية (CNN) لاكتشاف المرضى باستخدام بيانات التصوير بالرنين المغناطيسي الطيفي (MRSI) وبيانات التصوير بالرنين المغناطيسي الإضافية. تم تحقيق درجات عالية من معامل الارتباط ماثيو (MCC)، مع قيم منطقة تحت المنحنى تبلغ 0.87 و0.91 لـ MRSI وMRI، على التوالي. أظهرت تحليل مقارن تفوق أساليب المربعات الجزئية وآلات الدعم المتجه. النظام المقترح اختار تلقائيًا المناطق الطيفية الحرجة للتشخيص، مؤكدًا النتائج مع مؤشرات الأدبيات [22]. تم دراسة الشبكات العصبية التلافيفية، وخطوط أنابيب التعلم الآلي المستوحاة من العمليات العصبية البيولوجية، بشكل موسع. شمل نهج المؤلف أولاً اكتساب فهم للشبكات العصبية التلافيفية، تلاه بحث أدبي عن خط أنابيب تقسيم قابل للتطبيق على تقسيم أورام الدماغ. بالإضافة إلى ذلك، تم استكشاف الدور المحتمل المستقبلي للشبكات العصبية التلافيفية في الأشعة. تم إثبات تطبيق الشبكات العصبية التلافيفية في التنبؤ بالنجاة واستجابات الأدوية من خلال تحليل شكل الورم الدماغي وملمسه وكثافة الإشارة [23]. في هذه الورقة، تم استخدام إطار عمل الكشف عن الأجسام المتطور YOLO (You Only Look Once) لتحديد وتصنيف أورام الدماغ باستخدام التعلم العميق. تميز YOLOv5، وهو خوارزمية كشف الأجسام الثورية، بكفاءته الحاسوبية. كانت مجموعة بيانات تصنيف الإشعاع الجيني لأورام الدماغ RSNA-MICCAI BraTS 2021 هي الأساس. حقق YOLOv5
مع بنية CNN مملوكة، يشكل نموذج DCTN [28]. لا يمكن المبالغة في أهمية الكشف المبكر عن أورام الدماغ. تعتبر خزعات أورام الدماغ، المعيار الذهبي للتشخيص، ممكنة فقط خلال جراحة الدماغ التي تغير الحياة. يمكن أن تساعد الطرق المعتمدة على الذكاء الحسابي في تشخيص وتصنيف أورام الدماغ [29]. استخدم المؤلف نموذج DL لتصنيف صور الرنين المغناطيسي إلى فئات ورم دماغي وعادي، مسبوقًا باستخراج معلومات المسح. تم استخدام الشبكات العصبية التلافيفية المتكررة (CRNNs) لتوليد التصنيفات. وقد حسنت هذه الطريقة المقترحة بشكل كبير تصنيف صور الدماغ ضمن مجموعة بيانات إدخال محددة [30]. تم تدريب الشبكة واختبارها باستخدام بيانات BraTS2019. تم تقييم النهج باستخدام معامل تشابه Dice (DSC)، الحساسية (Sen)، النوعية (Spec)، والمسافة هاوسدورف (HD). كانت قيم DSC للورم بالكامل، ونواة الورم، والورم المعزز 0.934، 0.911، و0.851 على التوالي. كانت قيم Sen للمنطقة الفرعية 0.922، 0.911، و0.867. كانت درجات Spec وHD 1.000، 1.000، و3.224، 2.990، 2.844 على التوالي [31]. يتم تحقيق تقسيم منطقة السرطان من صور الدماغ باستخدام Deep K-Net، وهو نهج هجين يجمع بين K-Net ويستخدم تقسيم مشترك عميق مع تشابه روجيكا. يتم تدريب K-Net باستخدام خوارزمية Driving Training Taylor (DTT). تقوم خوارزمية DTT بتحسين Shepard CNN (ShCNN) للتصنيف [32].
لا يسهل فقط انتشار المعلومات العميقة ولكنه يقدم أيضًا خصائص ذاتية التنظيم، غير أحادية التوجه بسلاسة، بينما يخفف بفعالية من مشاكل التشبع أثناء التدريب. يقدم المؤلفون مجموعة شاملة من النتائج التجريبية، مقارنةً بأداء نموذجنا ضد معايير مثل تحدي MICCAI 2015 ومجموعات البيانات المتاحة للجمهور الأخرى. تظهر نتائجنا أن النموذج المقترح يتفوق من حيث الدقة والحساسية ودرجة F1 ودرجة F2 ومعامل Dice [35].
المواد والطرق
المواد
مجموعة بيانات أطلس جينوم السرطان للأورام الدبقية منخفضة الدرجة هي المجموعة الثالثة التي تم تحليلها (TCGA-LGG) [37]، وتحتوي على 242,185 صورة MRI لمرضى يعانون من أورام دبقية منخفضة الدرجة (G-I و G-II) وتدمج بيانات من 198 مريضًا. هذه المجموعات الثلاث هي جزء من مشروع أرشيف تصوير السرطان (TCIA) [38]. في كل حالة، تم إجراء تصوير متعدد الأنماط، بما في ذلك صور T1 المعززة بالتباين وصور FLAIR [39]. تتكون آخر مجموعة من البيانات المستخدمة في هذه الدراسة من 3067 صورة T1 محسنة بالتباين من 243 مريضًا يعانون من ثلاثة أنواع مختلفة من أورام الدماغ: الأورام الدبقية (1427 شريحة)، الأورام السحائية (709 شرائح)، وأورام الغدة النخامية (931 شريحة). الشكل 1 يوضح درجات مختلفة من أورام الدماغ من مجموعة البيانات. تم جمع 3165 صورة لنمط التصنيف-1، 1743 منها أورام خبيثة و1422 منها ليست كذلك. بالنسبة لنمط التصنيف-2، تم جمع 4195 صورة. هناك 910 صور طبيعية، 985 صورة أورام دبقية، 750 صورة أورام سحائية، 750 صورة أورام نخامية، و800 صورة ورم نقيل. بالنسبة لنمط التصنيف-3، نحصل على إجمالي 4720 صورة: 1712 G-II، 1296 G-III، و1712 G-IV. الجدول 1 يمثل تفاصيل تقسيم مجموعة البيانات للنموذج المقترح.
طرق
شبكة عصبية تلافيفية
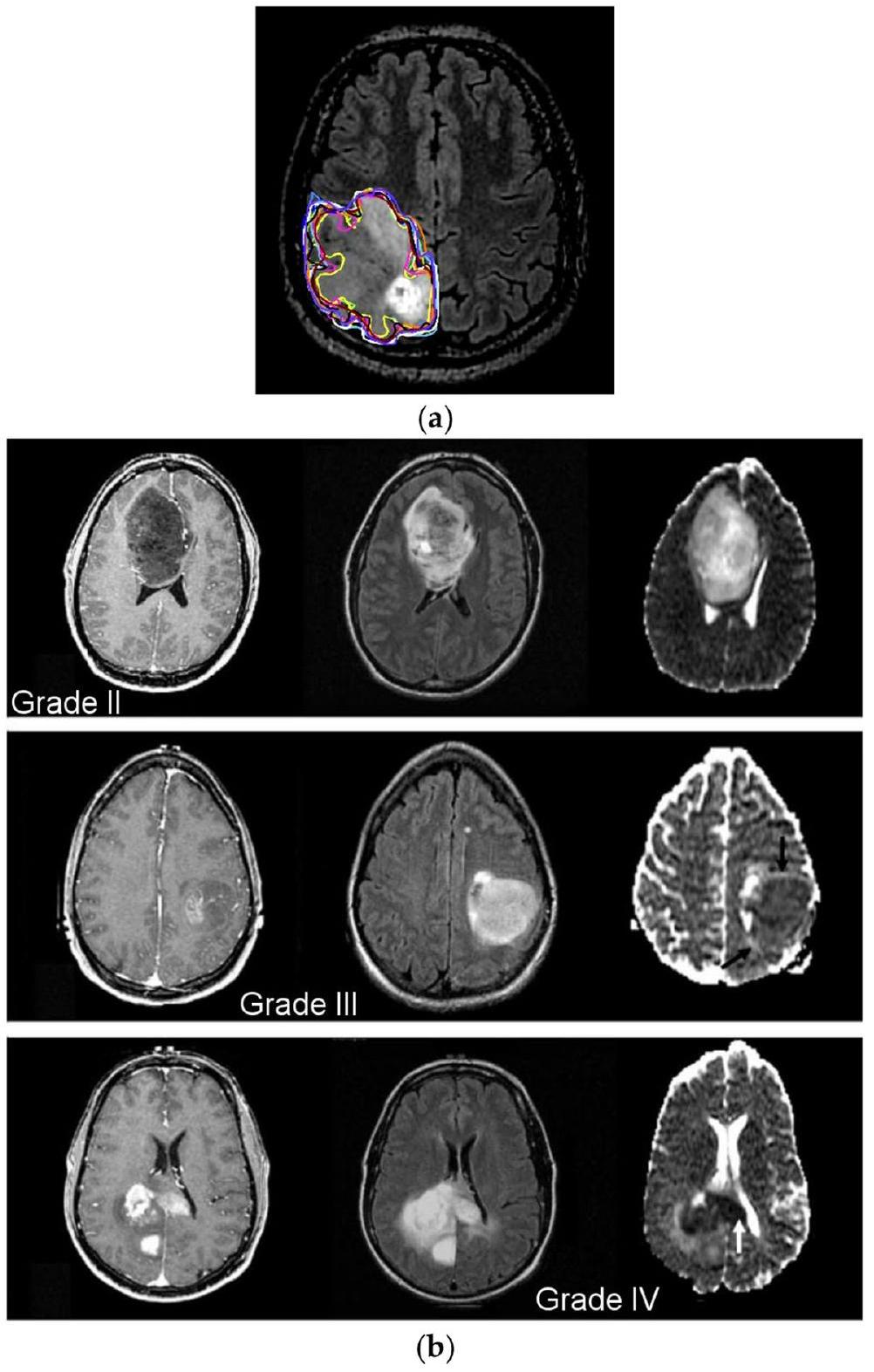
الشكل 1 أ تقسيم الورم يدويًا؛
تقييم مقياس الأداء
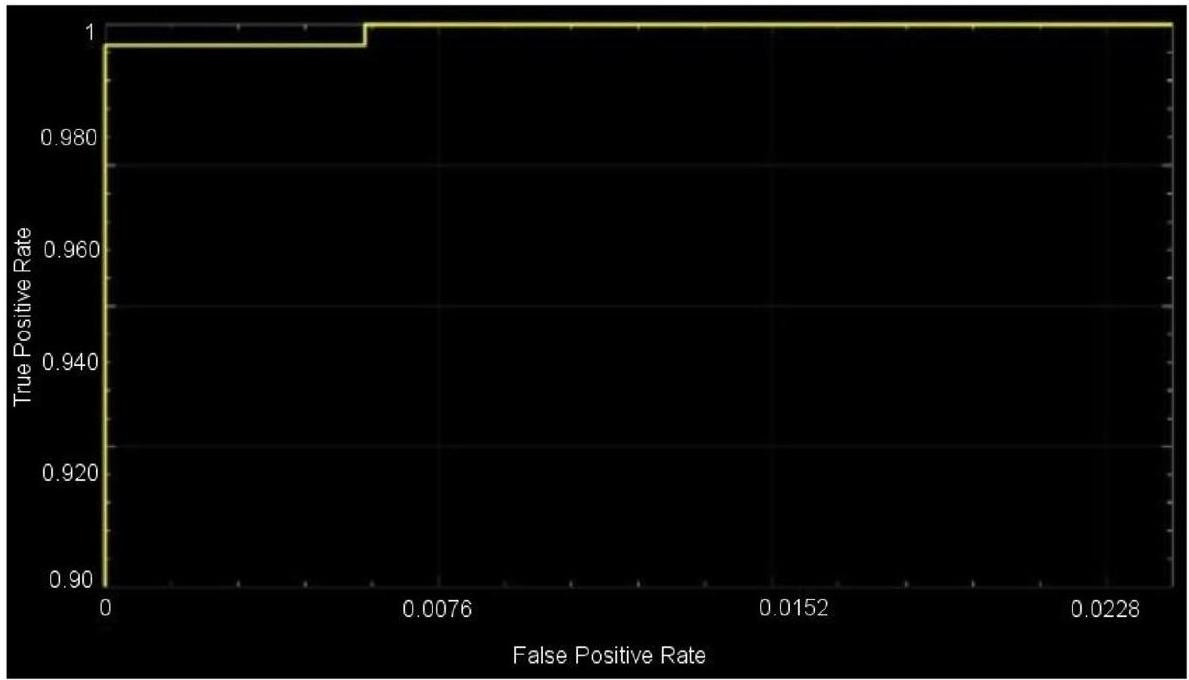
مقاييس تقييم الأداء المختلفة لفترة طويلة في الدراسات المتعلقة بتصنيف الصور والتي تطورت إلى مقاييس تقييم أداء قياسية في الدراسات المماثلة السابقة. استخدم النموذج المقترح طرقًا بارامترية مختلفة للتقييم، مثل الدقة، الحساسية، والدقة. تُعتبر هذه القياسات، التي تُعترف عمومًا كمقاييس تقييم أداء قياسية في أبحاث تصنيف الصور، مستخدمة أيضًا في هذه المقالة لقياس دقة وموثوقية عملية التصنيف. علاوة على ذلك، يتم استخدام منطقة منحنى التشغيل المستقبلي (ROC)، والمعروفة أيضًا باسم AUC من ROC
| تقسيم مجموعة البيانات | |||
| تصنيف | عدد الصور في المجموعة | ||
| النمط | المجموعة | إجمالي عدد الصور | |
| 1 | خبيث | 1743 | 3165 |
| غير خبيث | 1422 | ||
| II | حميد | 910 | 4195 |
| أورام دبقية | 985 | ||
| أورام سحائية | 750 | ||
| أورام نخامية | 750 | ||
| نقيلي | 800 | ||
| III | G-II | 1712 | 4720 |
| G-III | 1296 | ||
| G-IV | 1712 | ||
دراسة تجريبية
تحسين المعلمات الفائقة
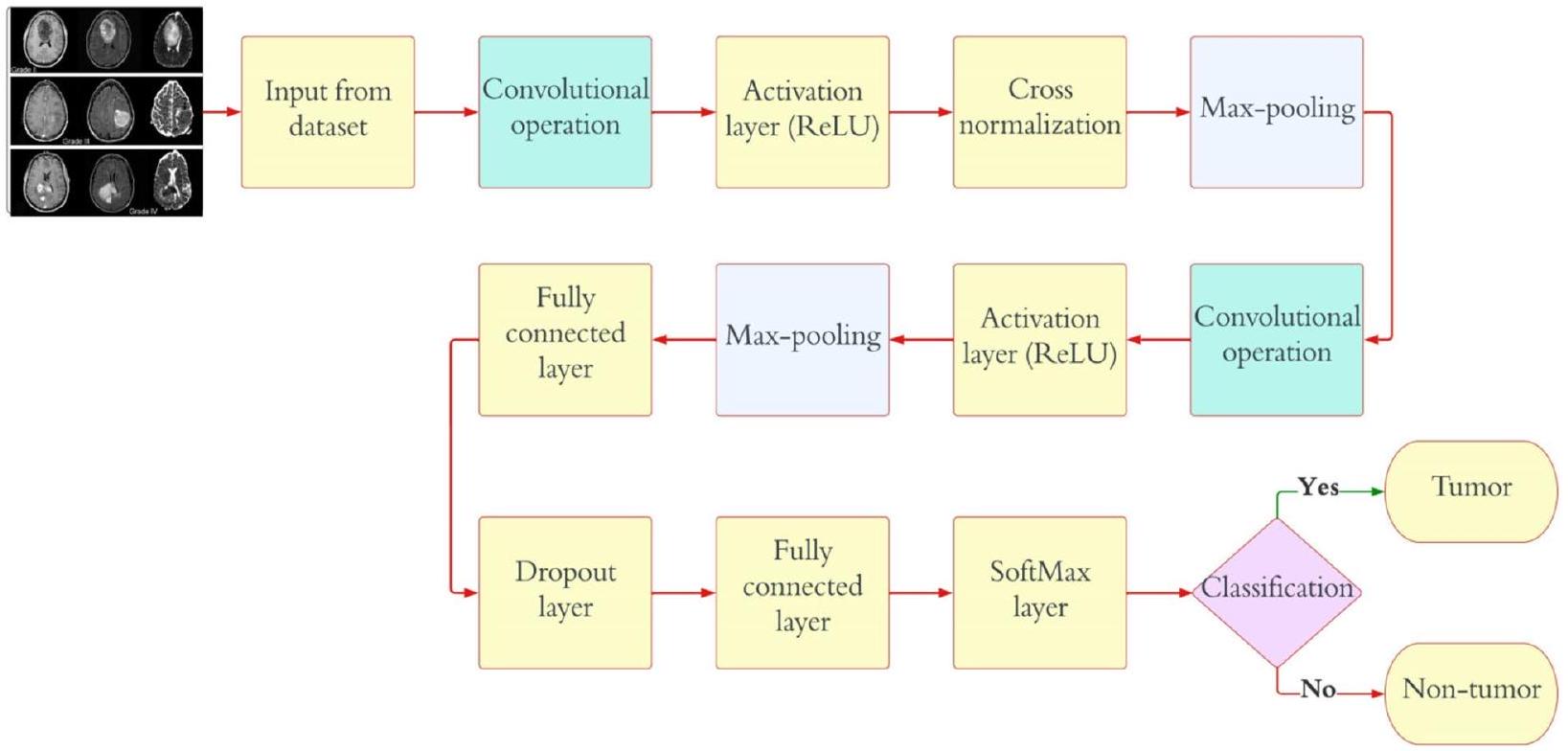
| اسم الطبقة | طبقة CNN | التنشيطات | المعلمات (قابلة للتدريب) | إجمالي عدد المعلمات القابلة للتدريب |
| إدخال |
|
|
لا شيء | 0 |
| تلافيفية | 128 (
|
|
|
13,954 |
| طبقة تنشيط | طبقة تنشيط-1 |
|
لا شيء | 0 |
| تطبيع | تطبيع (عبر القناة) |
|
لا شيء | 0 |
| تجميع أقصى | (
|
|
لا شيء | 0 |
| تلافيفية | 96 (
|
|
|
49,246 |
| طبقة تنشيط | طبقة التفعيل-2 |
|
لا شيء | 0 |
| التجميع الأقصى | (
|
|
صفر | 0 |
| متصل بالكامل | 512 متصل بالكامل |
|
|
11,060,714 |
| التسرب الدراسي | 30٪ |
|
صفر | 0 |
| متصل بالكامل | 2 متصل بالكامل |
|
|
١٠٢٦ |
| سوفتماكس | سوفتماكس |
|
صفر | 0 |
| تصنيف | ورم أو غير ورم | لا شيء | صفر | 0 |
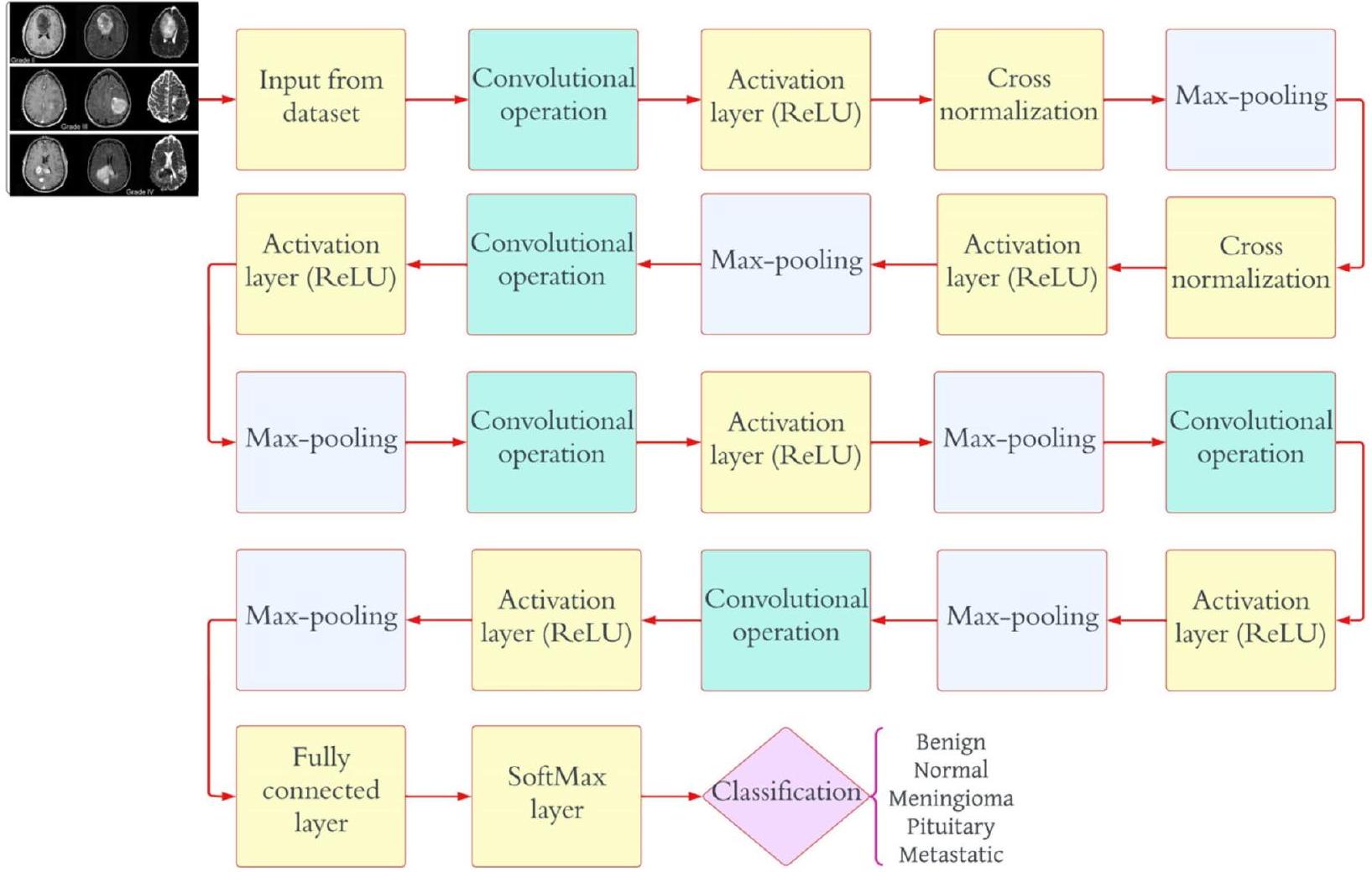
الأحجام، ودالة التفعيل. تعتبر تنظيم البيانات، والزخم، وحجم الدفعة الصغيرة، ومعدل التعلم من المعلمات الفائقة التي يتم ضبطها بدقة. في التحليل الحالي، يتم ضبط المعلمات الفائقة للهيكل في البداية باستخدام الخوارزمية 1.
| اسم الطبقة | طبقة CNN | تفعيلات | المعلمات (قابلة للتدريب) | إجمالي عدد المعلمات القابلة للتدريب |
| مدخل |
|
|
صفر | 0 |
| التلافيفي |
|
|
|
١٣,٩٥٢ |
| طبقة التفعيل | طبقة التفعيل – 1 |
|
صفر | 0 |
| التطبيع | التطبيع (عبر القنوات) |
|
لا شيء | 0 |
| التجميع الأقصى |
|
|
صفر | 0 |
| التلافيفي | ٩٦
|
|
|
٤٤٢,٤٦٤ |
| طبقة التفعيل | طبقة التفعيل-2 |
|
صفر | 0 |
| التجميع الأقصى | (
|
|
صفر | 0 |
| التلافيفي | ٩٦
|
|
|
٣٦,٩٦٠ |
| طبقة التفعيل | طبقة التفعيل – 3 |
|
لا شيء | 0 |
| التجميع الأقصى | (
|
|
صفر | 0 |
| التلافيفي | ٢٤ (
|
|
|
82,968 |
| طبقة التفعيل | طبقة التفعيل-4 |
|
صفر | 0 |
| التجميع الأقصى | (
|
|
لا شيء | 0 |
| التلافيفي | 24 (
|
|
|
٢٠٧٦٠ |
| طبقة التفعيل | طبقة التفعيل-5 |
|
صفر | 0 |
| التجميع الأقصى | (
|
|
صفر | 0 |
| التلافيفي |
|
|
|
١٢٣٢٠ |
| طبقة التفعيل | طبقة التفعيل-6 |
|
لا شيء | 0 |
| التجميع الأقصى | (
|
|
لا شيء | 0 |
| متصل بالكامل | 512 متصل بالكامل |
|
|
16,896 |
| التسرب الدراسي | 30٪ |
|
لا شيء | 0 |
| متصل بالكامل | 5 متصل بالكامل |
|
|
2565 |
| سوفتماكس | سوفتماكس |
|
لا شيء | 0 |
| تصنيف | حميد، ورم دبقي، ورم غدي، ورم نقوي، وورم سحائي | صفر | لا شيء | 0 |
|
||
| الخطوة 2: يجب تعيين فترات القيم المحتملة لكل بُعد. | ||
| – طبقات التجميع الأقصى والتلافيف = (1,2,3,4) | ||
| الخطوة 3: افحص جميع التوليفات المحتملة واختر تلك التي تعظم الدقة العامة. | ||
| –
|
||
| نهاية |
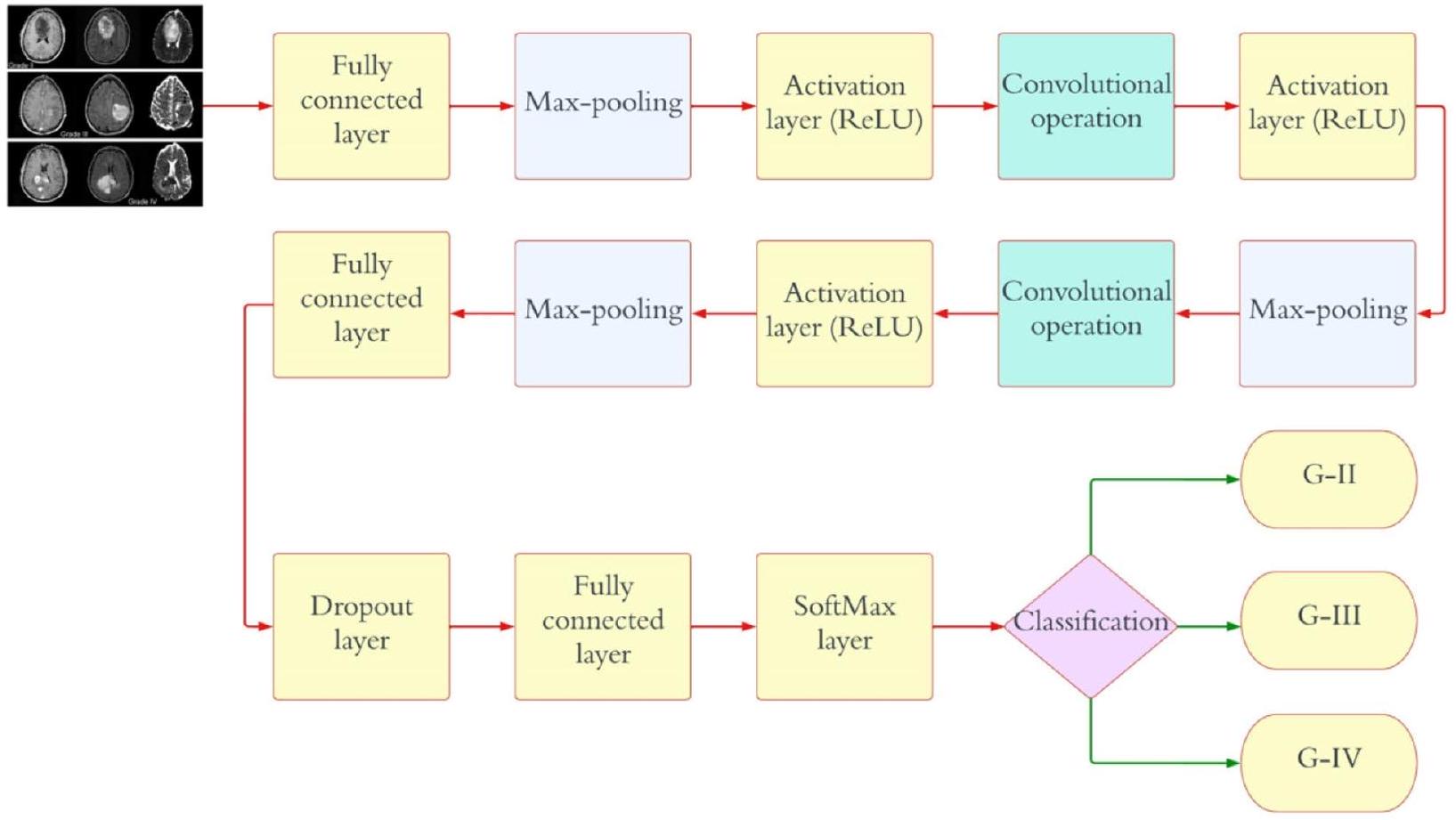
Begin
Step 1: Assign a 4-dimensional grid for the 5 hyperparameters needed to be optimized.
- Regularization = R2
- A momentum
- Size of the minibatches
- Rate of learning
Step 2: To each dimension, potential value intervals should be assigned.
- R2 = (0.0002, 0.00043, 0.002, 0.004)
- A momentum = (0.78, 0.77, 0.954, and 0.96)
- Size of the minibatches = (4,6, 16, 24, 32, 64)
- Rate of learning = (0.0002, 0.00043, 0.002, 0.004)
Step 3: Examine all the potential pairings and choose the one that maximizes overall ac-
curacy.
- A1 = (0.0002, 16, 0.16, 0.0002) }->mathrm{ accuracy 94.6%
- A2 = (0.0005, 32, 0.9, 0.002) }->mathrm{ accuracy }98.4
- A3 = (0.0002, 64, 0.9, 0.002) }->mathrm{ accuracy }99.2
End
على سبيل المثال،
نتائج الشبكة العصبية التلافيفية المحسّنة
| اسم الطبقة | طبقة CNN | التفعيل | المعلمات (قابلة للتدريب) | إجمالي عدد المعلمات القابلة للتدريب |
| مدخل |
|
|
صفر | 0 |
| التلافيفي | 128 (
|
|
|
١٣,٩٥٢ |
| طبقة التفعيل | طبقة التفعيل – 1 |
|
صفر | 0 |
| التطبيع | التطبيع (عبر القنوات) |
|
صفر | 0 |
| التجميع الأقصى | (
|
|
صفر | 0 |
| التلافيفي | ٩٦ (
|
|
|
٤٦,٧٥٢ |
| طبقة التفعيل | طبقة التفعيل-2 |
|
صفر | 0 |
| التجميع الأقصى | (
|
|
صفر | 0 |
| التلافيفي | ٩٦ (
|
|
|
٣٦,٨٦٤ |
| طبقة التفعيل | طبقة التفعيل-3 |
|
صفر | 0 |
| التجميع الأقصى | (
|
|
صفر | 0 |
| متصل بالكامل | 512 متصل بالكامل |
|
|
3,146,240 |
| التسرب الدراسي | 30٪ |
|
صفر | 0 |
| متصل بالكامل | 3 متصل بالكامل |
|
|
1539 |
| سوفتماكس | سوفتماكس |
|
صفر | 0 |
| تصنيف | جي-2، جي-3، جي-4 | صفر | صفر | 0 |
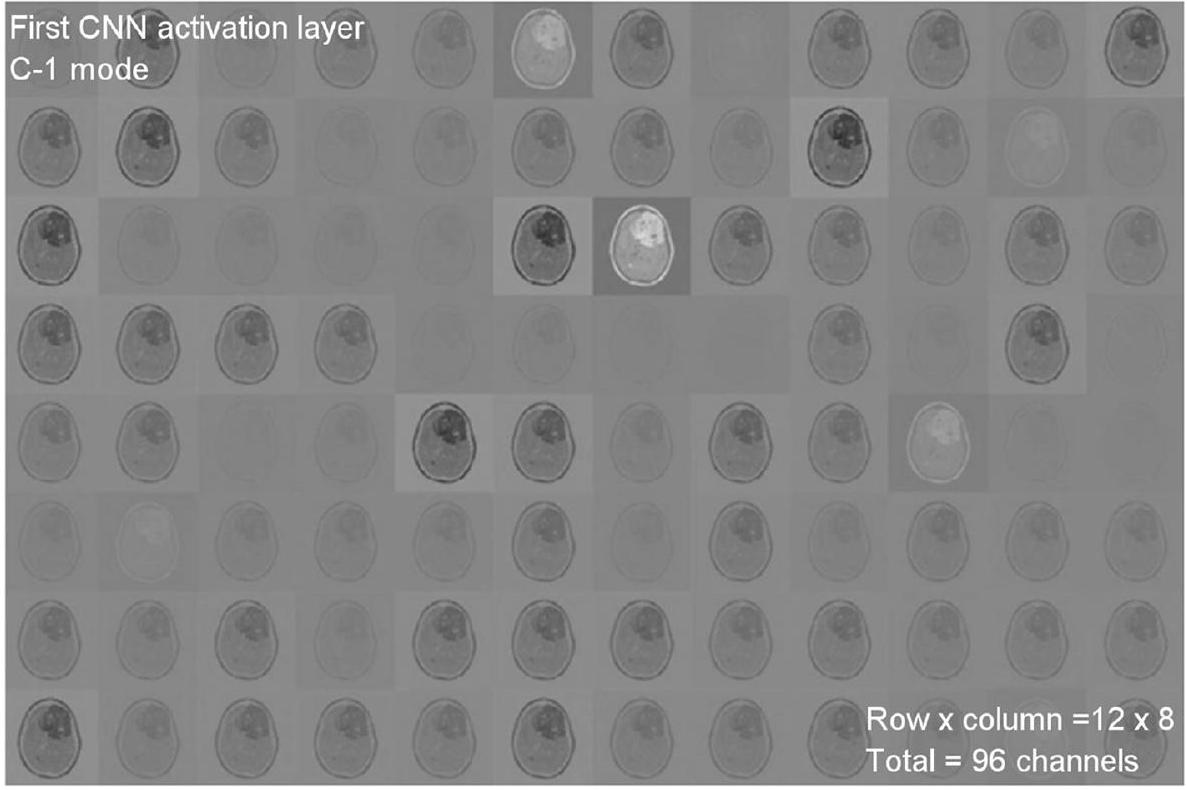
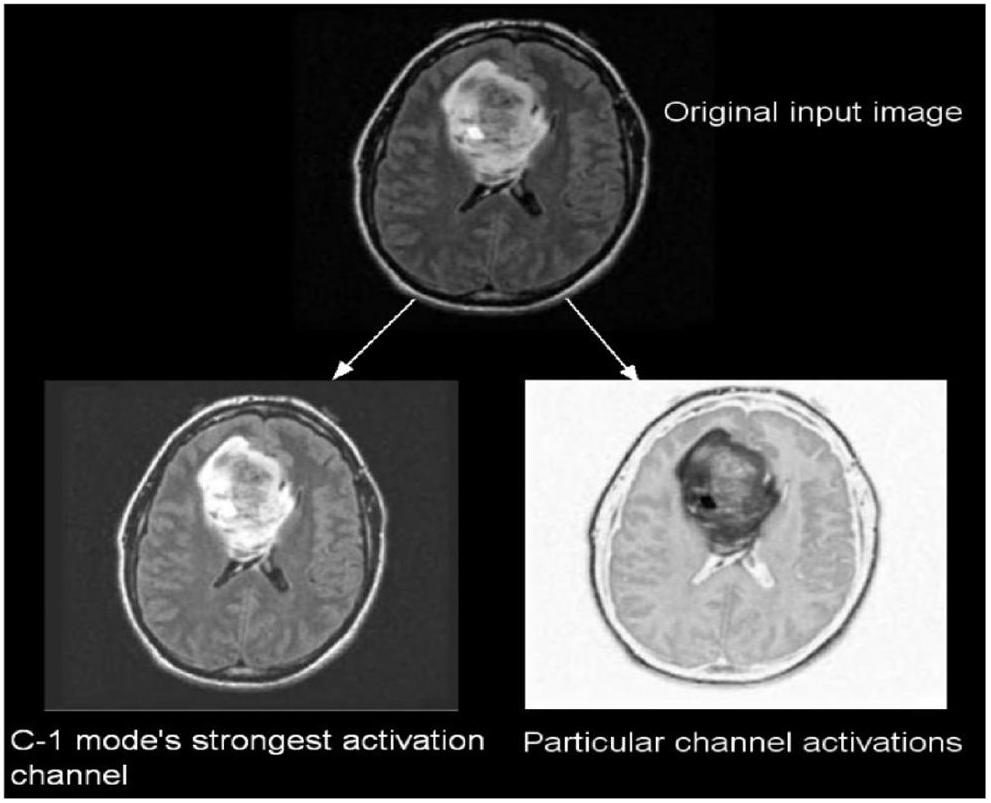
تم أخذ ما مجموعه 299 صورة بشكل عشوائي من مجموعة البيانات لكل فئة، ثم تُستخدم تلك الصور للاختبار. يمكن عرض تنشيطات طبقات الالتفاف في الشبكة العصبية التلافيفية للحصول على رؤية أفضل للميزات.
| الهايبر بارامترز | تغييرات في قيم المعلمات | القيمة القصوى |
| طبقات التجميع الأقصى والشبكات العصبية التلافيفية | (1، 2، 3، 4) | 2 |
| عدد الطبقات المتصلة بالكامل | (1، 2، 3، 4) | ٢ |
| إجمالي عدد الفلاتر | (8، 16، 24، 32، 48، 64، 96، 128، 256) | ٦٤، ٩٦، ١٢٨ |
| شدة الترشيح | (3، 4، 5، 6، 7) | 6,6 |
| دور التنشيط | (ريلو، إيلو، ريليو المتسرب) | ريلو |
| حجم الدفعة الصغيرة | (٤، ٦، ١٦، ٢٤، ٣٢، ٦٤) | 32 |
| معدل التغير | (0.78، 0.77، 0.95، 0.96) | 0.95 |
| معدل التعلم | (0.0002, 0.00043, 0.002, 0.004) | 0.0002 |
|
|
(0.0002، 0.00043، 0.002، 0.004) | 0.0002 |
| الهايبر بارامترز | تغييرات في قيم المعلمات | القيمة القصوى |
| طبقات التجميع الأقصى والشبكات العصبية التلافيفية | (1، 2، 3، 4) | ٣ |
| عدد الطبقات المتصلة بالكامل | (1، 2، 3، 4) | 2 |
| إجمالي عدد الفلاتر | (8، 16، 24، 32، 48، 64، 96، 128، 256) | ٦٤، ٩٦، ١٢٨ |
| شدة الترشيح | (3، 4، 5، 6، 7) | ٦، ٦، ٤ |
| دور التنشيط | (ريلو، إيلو، ريليو المتسرب) | ريلو |
| حجم الدفعة الصغيرة | (٤، ٦، ١٦، ٢٤، ٣٢، ٦٤) | 32 |
| معدل التغير | (0.78، 0.77، 0.95، 0.96) | 0.95 |
| معدل التعلم | (0.0002, 0.00043, 0.002, 0.004) | 0.004 |
|
|
(0.0002، 0.00043، 0.002، 0.004) | 0.002 |
| الهايبر بارامترز | تغييرات في قيم المعلمات | القيمة القصوى |
| طبقات التجميع الأقصى والشبكات العصبية التلافيفية | (1، 2، 3، 4) | ٦ |
| عدد الطبقات المتصلة بالكامل | (1، 2، 3، 4) | 2 |
| إجمالي عدد الفلاتر | (8، 16، 24، 32، 48، 64، 96، 128، 256) | 16، 24، 32، 48، 64، 96، 128 |
| شدة الترشيح | (3، 4، 5، 6، 7) | ٦، ٦، ٤، ٦، ٢، ٦ |
| دور التنشيط | (ريلو، إيلو، ريليو المتسرب) | ريلو |
| حجم الدفعة الصغيرة | (٤، ٦، ١٦، ٢٤، ٣٢، ٦٤) | 64 |
| معدل التغير | (0.78، 0.77، 0.95، 0.96) | 0.95 |
| معدل التعلم | (0.0002، 0.00043، 0.002، 0.004) | 0.0002 |
|
|
(0.0002, 0.00043, 0.002, 0.004) | 0.002 |
| تقسيم مجموعة البيانات | أوضاع التدريب والتحقق والاختبار | |||||
| تصنيف | عدد الصور في المجموعة | إجمالي عدد الصور | وضع التدريب (60%) | وضع التحقق (20%) | وضع الاختبار (20%) | |
| 1 | خبيث | 1743 | 3165 | 1899 | ٦٣٣ | ٦٣٣ |
| غير خبيث | 1422 | |||||
| الثاني | حميد | 910 | ٤١٩٥ | ٢٥١٧ | 839 | 839 |
| ورم دبقي | 985 | |||||
| ورم السحايا | 750 | |||||
| الغدة النخامية | 750 | |||||
| نقيلية | ٨٠٠ | |||||
| ثالث | جي-2 | 1712 | 4720 | ٢٨٣٢ | 944 | 944 |
| جي-ثري | ١٢٩٦ | |||||
| جي-4 | 1712 | |||||

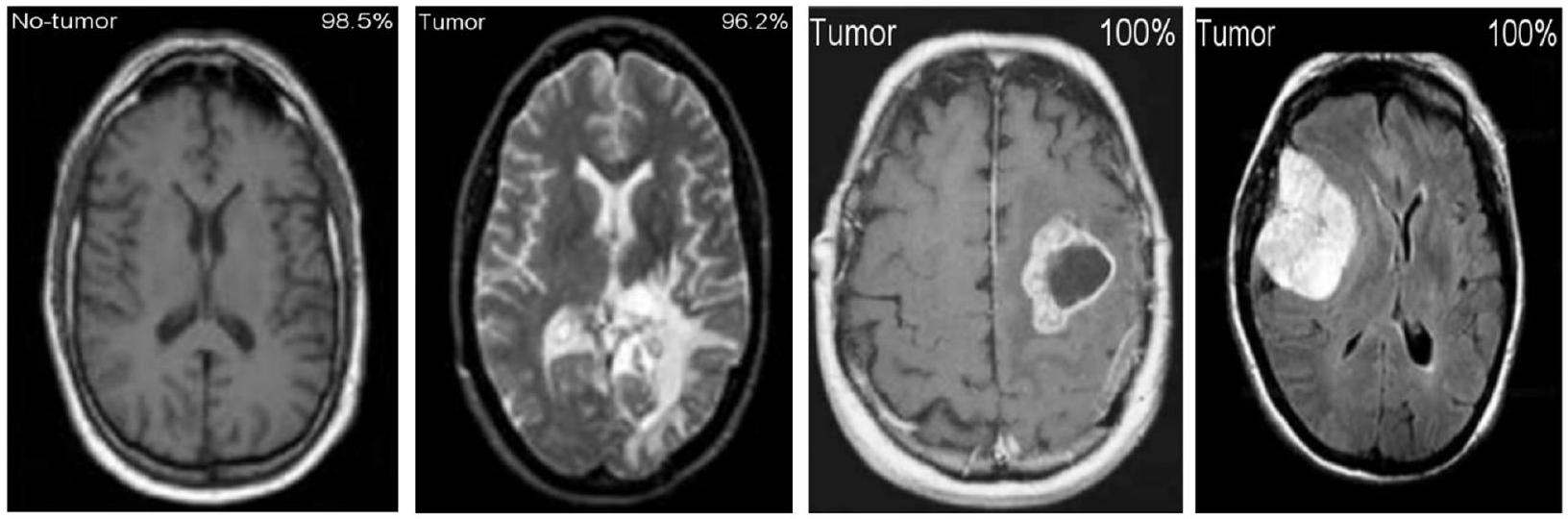
تظهر الشكل 11 نتائج التصنيف والاحتمالات المتوقعة لكل من الاختبارات الأربعة التي أجريت في وضع C-1. تم تنفيذ طريقة التحقق المتقاطع بخمسة أضعاف لـ
لأغراض التدريب، بينما يتم تخصيص المجموعة الخامسة لأغراض الاختبار. هناك خمس تكرارات إجمالية للتجارب. يتم تقييم أداء التصنيف للوظيفة لكل طية، ثم يتم حساب متوسط أداء التصنيف العام للنموذج. كما هو موضح في الجدول 8، هناك صور كافية لـ

| غير ورمي | 268 44.9% | 3 0.6% | 98.72% 1.28% | |
| فئة الإخراج | ورم | 0 0.0% | 326 54.1% | 100% 0.0% |
| 100% 0.0% | 98.9% 1.1% | 99.5% 0.6% | ||
| ورم | غير ورمي | إجمالي |
تصنيف الدرجة الثالثة، و
| المقاييس | فصول | تي بي | TN | FP | FN | حساب | سب | سي | بر | إجمالي |
| 1 | خبيث | ٢٦٨ | ٣٢٦ | ٣ | 0 | 99.50 | 99.09 | 100.00 | 98.89 | ٢٦٨ |
| غير خبيث | ٣٢٦ | ٢٦٨ | 0 | ٣ | 99.50 | 100.00 | 99.09 | 100.00 | ٣٢٩ | |
| الثاني | حميد | 183 | 598 | ٨ | ٨ | 97.99 | 98.68 | 95.81 | 95.81 | 191 |
| ورم دبقي | 132 | ٦٥٠ | 10 | 12 | 97.26 | ٩٨.٤٨ | 91.67 | 92.96 | ١٤٤ | |
| ورم السحايا | 138 | 643 | ٦ | 14 | ٩٧.٥٠ | 99.08 | 90.79 | 95.83 | 152 | |
| الغدة النخامية | ١٦٠ | 598 | ٢٤ | 11 | 95.59 | ٩٦.١٤ | 93.57 | ٨٦.٩٦ | 171 | |
| نقيلية | 127 | 643 | 11 | 14 | ٩٦.٨٦ | ٩٨.٣٢ | 90.07 | 92.03 | 141 | |
| ثالثاً | جي-2 | ٣٣٢ | 574 | 9 | ٨ | 98.16 | ٩٨.٤٦ | ٩٧.٦٥ | ٩٧.٣٦ | ٣٤٠ |
| جي-ثري | ٢٤٨ | 679 | 0 | 0 | 100.00 | 100.00 | 100.00 | 100.00 | ٢٤٨ | |
| جي-4 | ٣٣٠ | 580 | ٨ | 9 | ٩٨.١٧ | ٩٨.٦٤ | ٩٧.٣٥ | ٩٧.٦٣ | ٣٣٩ |

| فئة الهدف | |||||||
| ورم دبقي | 183 | ٣ | ٤ | 1 | 0 | 95.9% | |
| ٢٢.٨٪ | 0.4% | 0.5% | 0.1% | 0.0% | ٤.١٪ | ||
| ورم السحايا | ٤ | 132 | ٣ | 1 | 2 | 93.8٪ | |
| 0.5% | 16.3% | 0.4% | 0.1% | 0.3% | 6.2% | ||
| فئة الإخراج | نقيلية | 1 | 2 | 138 | ٢ | 1 | 95.9٪ |
| 0.1% | 0.3% | 17.4% | 0.3% | 0.1% | ٤.١٪ | ||
| حميد | ٣ | ٤ | ٦ | ١٦٠ | 11 | 86.8% | |
| 0.4% | 0.5% | 0.8% | ٢٠.٢٪ | 1.4٪ | 13.2% | ||
| الغدة النخامية | 0 | ٣ | 1 | ٧ | 127 | 93.1٪ | |
| 0.0% | 0.4% | 0.1% | 0.9% | 15.9% | 6.9% | ||
| 95.3٪ | 93.9٪ | 92.7% | 93.6٪ | 90.0% | 93.8% | ||
| ٤.٧٪ | 6.1٪ | ٧.٣٪ | 6.4% | 10.0% | 6.9% | ||
| ورم دبقي | ورم السحايا | نقيلية | حميد | الغدة النخامية | إجمالي | ||
توضح النتائج المعروضة في الجدول 10 أن نماذج CNN المقترحة تتفوق على الشبكات الأخرى في كل وضع تصنيف. تم تدريب DenseNet121 مسبقًا
نموذج، الذي يحصل على
| جي | 332 37.2% | 0 0.0% | 9 1.1% | 97.7% 2.3% | |
| فئة الإخراج |  |
0 0.0% | 248 26.1% | 0 0.0% | 100% 0.0% |
| هو | 8 0.8% | 0 0.0% | ٣٣٠ ٣٥.١٪ | 98.1% 1.9% | |
| 100% 0.0% | 97.5% 2.5% | 98.1% 1.9% | 98.5% 1.5% | ||
| مؤشر الابتكار العالمي | جي 3 | جيف | إجمالي |
95.12%، مما يجعلها أفضل شبكة متاحة لتصنيف الأورام (وضع C-3). من الواضح أن نماذج CNN المقترحة أفضل من الشبكات المدربة مسبقًا، التي تم بناؤها وتدريبها باستخدام مجموعات بيانات وأساليب عامة لمجموعة واسعة من مهام تصنيف الصور. توضح الجدول 11 مقارنة نتائج النماذج المقترحة والحالية. تم تصميم نماذج CNN المقترحة، على العكس، للتعامل مع قضايا أكثر تحديدًا، مثل تحديد وتعريف أنواع ومراحل مختلفة من أورام الدماغ. أخيرًا، تُستخدم صور الرنين المغناطيسي لأورام الدماغ لتدريب وتقييم النماذج المقترحة.
الاستنتاجات
| نماذج سي إن إن | وضع C-1 | وضع C-2 | وضع C-3 | |||
| الدقة (%) | الجامعة الأمريكية في القاهرة | الدقة (%) | الجامعة الأمريكية في القاهرة | الدقة (%) | الجامعة الأمريكية في القاهرة | |
| جوجل نت | ٧٤.٢١ | 0.8108 | ٧٧.٨٩ | 0.8212 | 95.12 | 0.9617 |
| أليكس نت | 89.23 | 0.8989 | 84.24 | 0.8501 | 91.08 | 0.9772 |
| دينس نت 121 | 93.89 | 0.9412 | ٧٧.٦٧ | 0.8122 | ٨٦.٠٧ | 0.8809 |
| ريسنت101 | 93.29 | 0.9442 | ٧٦.٤٥ | 0.8115 | ٨٦.٤٢ | 0.881 |
| في جي جي – 16 | ٨٨.٨٧ | 0.9201 | ٨٩.١٩ | 0.8112 | 84.87 | 0.8663 |
| النهج المقترح لشبكة CNN | 99.53 | 0.9994 | 93.81 | 0.9984 | ٩٨.٥٦ | 0.9993 |

| مؤلف | سنة | مجموعات البيانات | طريقة | تصنيف الدقة (%) |
| أحمد واصف رضا [12] | ٢٠٢٣ | كاجل، فيغشير | معمارية VGG-16 | ٩٦.٧٠ |
| محمود خالد عبد الله [13] | 2018 | رايدر، رمبرانت، وبرايتس | إيكوك-إس في إم | ٩٧.٩٨ |
| تاكوا رحمن [27] | ٢٠٢٣ | كاجل، ريمبرانت | سي إن إن + 6 نماذج مدربة مسبقًا | 97.12 |
| أناند ديشباندي [17] | ٢٠٢١ | رايدر وبرايتس | دمج الصور المعتمد على تحويل كوساين المتقطع مع الشبكات العصبية التلافيفية | ٩٨.١٤ |
| النموذج المقترح | ٢٠٢٣ | فيغشير، ريمبراندت، TCGA-LGG، TCIA | شبكة عصبية تلافيفية هجينة | ٩٨.٥٦ |
شكر وتقدير
بيان لجنة المراجعة المؤسسية
قائمة التحقق لأبحاث المشاركين البشريين:
- هل حصلت على موافقة الأخلاقيات لهذه الدراسة؟
- إذا كانت الإجابة نعم، يرجى رفع (نوع الملف “آخر”) الوثيقة الأصلية للموافقة التي تلقيتها من لجنة الأخلاقيات الخاصة بك. إذا كانت الوثيقة الأصلية بلغة أخرى، يرجى أيضًا تقديم ترجمة إلى الإنجليزية.
الرد: غير متوفر - إذا لم تحصل على موافقة أخلاقية، يرجى توضيح سبب عدم الحاجة لذلك أدناه.
- إذا قمت بتجنيد مشاركين بشريين للدراسة على سبيل المثال، أجريت تجربة سريرية، وزعت استبيانات، أو حصلت على أنسجة أو بيانات أو عينات لأغراض هذه الدراسة، يرجى الإبلاغ في القسم الخاص بالطرق: i. اليوم والشهر والسنة لبداية ونهاية فترة التجنيد لهذه الدراسة.
ii. ما إذا كان المشاركون قد قدموا موافقة مستنيرة، وإذا كان الأمر كذلك، ما نوعها (على سبيل المثال، مكتوبة أو شفهية، وإذا كانت شفهية، كيف تم توثيقها وشهادتها). إذا كانت دراستك تشمل قاصرين، يرجى بيان ما إذا كنت قد حصلت على موافقة من الوالدين أو الأوصياء. إذا تم التنازل عن الحاجة إلى الموافقة من قبل لجنة الأخلاقيات، يرجى تضمين هذه المعلومات.
الرد: غير متوفر - إذا كنت تقوم بتقديم دراسة استعادية لسجلات طبية أو عينات مؤرشفة، يرجى الإبلاغ في قسم الطرق:
i. اليوم والشهر والسنة التي تم فيها الوصول إلى البيانات لأغراض البحث ii. ما إذا كان المؤلفون قد حصلوا على معلومات يمكن أن تحدد المشاركين الأفراد أثناء أو بعد جمع البيانات
الرد: غير متوفر
مساهمات المؤلفين
تمويل
توفر البيانات والمواد
الإعلانات
موافقة على النشر
تنافس المصالح
نُشر على الإنترنت: 19 يناير 2024
References
- Abiwinanda N, Hanif M, Hesaputra ST, Handayani A, Mengko TR. Brain tumor classification using convolutional neural network. IFMBE Proc. 2019;68:183-9.
- Ayadi W, Elhamzi W, Charfi I, Atri M. Deep CNN for brain tumor classification. Neural Process Lett. 2021;53:671-700.
- Badža MM, Barjaktarović MČ. Classification of brain tumors from MRI images using a convolutional neural network. Appl Sci. 1999;2020:10.
- Saravanan S, Kumar VV, Sarveshwaran V, Indirajithu A, Elangovan D, Allayear SM. Computational and Mathematical Methods in Medicine Glioma Brain Tumor Detection and Classification Using Convolutional Neural Network. Comput Math Methods Med. 2022;2022:4380901.
- Ge C, Gu IYH, Jakola AS, Yang J. Deep semi-supervised learning for brain tumor classification. BMC Med Imaging. 2020;20:87.
- Cinar A, Yildirim M. Detection of tumors on brain MRI images using the hybrid convolutional neural network architecture. Med Hypotheses. 2020;139: 109684.
- Pei L, Vidyaratne L, Rahman MM, Iftekharuddin KM. Context aware deep learning for brain tumor segmentation, subtype classification, and survival prediction using radiology images. Sci Rep. 2020;10:19726.
- Balasooriya NM, Nawarathna R.D. A sophisticated convolutional neural network model for brain tumor classification. In Proceedings of the 2017 IEEE International Conference on Industrial and Information Systems, Peradeniya, Sri Lanka, 15-16 2017; pp. 1-5
- Deepak VK, Sarath R. Multi-Class Brain Cancer Classification Using Deep Learning Convolutional Neural Network. PalArch’s J Archaeol Egypt/Egyptol. 2020;17:5341-60.
- Khan HA, Jue W, Mushtaq M, Mushtaq MU. Brain tumor classification in MRI image using convolutional neural network. Math Biosci Eng. 2020;17:6203-16.
- Islam R, Imran S, Ashikuzzaman M, Khan MMA. Detection and Classification of Brain Tumor Based on Multilevel Segmentation with Convolutional Neural Network. Biomed Sci Eng. 2020;13:45-53.
- Reza AW, Hossain MS, Wardiful MA, Farzana M, Ahmad S, Alam F, Nandi RN, Siddique N. A CNN-Based Strategy to Classify MRI-Based Brain Tumors Using Deep Convolutional Network. Appl Sci. 2023;13:312.
- Abd-Ellah MK, Awad AI, Khalaf AA, Hamed HF. Two-phase multi-model automatic brain tumor diagnosis system from magnetic resonance images using convolutional neural networks. EURASIP J Image Video Process. 2018;97:1-10.
- Noreen N, Palaniappan S, Qayyum A, Ahmad I, Alassafi MO. Brain Tumor Classification Based on Fine-Tuned Models and the Ensemble Method. Comput Mater Contin. 2023;67:3967-82. https://doi.org/10.32604/cmc. 2021.014158.
- Mohsen H, El-Dahshan ESA, El-Horbaty ESM, Salem ABM. Classification using deep learning neural networks for brain tumors. Future Comput Inform J. 2018;3:68-71.
- Chattopadhyay A, Maitra M. MRI-based brain tumor image detection using CNN based deep learning method. Neurosci Inform. 2022;2:100060.
- Deshpande A, Estrela VV, Patavardhan P. The DCT-CNN-ResNet50 architecture to classify brain tumors with super-resolution, convolutional neural network, and the ResNet50. Neurosci Inform. 2021;1:100013.
- Wankhede DS, Selvaran R. Dynamic architecture based deep learning approach for glioblastoma brain tumor survival prediction. Neurosci Inform. 2022;2:100062.
- Rai HM, Chatterjee K. Detection of brain abnormality by a novel Lu-Net deep neural CNN model from MR images. Mach Learn Appl. 2020;2:100004.
- Ramya M, Kirupa G, Rama A. Brain tumor classification of magnetic resonance images using a novel CNN-based medical image analysis and detection network in comparison with AlexNet. J Popul Ther Clin Pharmacol. 2022;29:e97-108. https://doi.org/10.47750/jptcp.2022.898.
- Bingol H, Alatas B. Classification of Brain Tumor Images using Deep Learning Methods. Turk J Sci Technol. 2021;16:137-43.
- Acquarelli J, van Laarhoven T, Postma GJ, Jansen JJ, Rijpma A, van Asten S, Heerschap A, Buydens LMC, Marchiori E. Convolutional neural networks to predict brain tumor grades and Alzheimer’s disease with MR spectroscopic imaging data. PLoS ONE. 2021;7:e0268881.
- Bhandari A, Koppen J, Agzarian M. Convolutional neural networks for brain tumor segmentation. Insights Imaging. 2020;11:1-9.
- Shelatkar T, Urvashi D, Shorfuzzaman M, Alsufyani A, Lakshmanna K. Diagnosis of Brain Tumor Using Light Weight Deep Learning Model with Fine-Tuning Approach. Comput Math Methods Med. 2022;2022:285884.
- Shwetha V, Madhavi CR, Nagendra KM. Classification of Brain Tumors Using Hybridized Convolutional Neural Network in Brain MRI images. Int J Circuits Syst Signal Process. 2022;16:561-70.
- Anagun Y. Smart brain tumor diagnosis system utilizing deep convolutional neural networks. Multimed Tools Appl. 2023;675:1-27.
- Rahman T, Islam MS. MRI brain tumor detection and classification using parallel deep convolutional neural networks. Meas Sens. 2023;26:100694.
- AI-Zoghby AM, AI-Awadly EMK, Moawad A, Yehia N, Ebada AI. Dual Deep CNN for Tumor Brain Classification. Diagnostics. 2023;13:2050.
- Saeedi S, Rezayi S, Keshavarz HR, Niakan Kalhori S. MRI-based brain tumor detection using convolutional deep learning methods and chosen machine learning techniques. BMC Med Inform Decis Mak. 2023;23:1-17.
- Srinivasan S, Bai PSM, Mathivanan SK, Muthukumaran V, Babu JC, Vilcekova L. Grade Classification of Tumors from Brain Magnetic Resonance Images Using a Deep Learning Technique. Diagnostics. 2023;13:1153.
- Yin Z, Gao H, Gong J, Wang Y. WD-UNeXt: Weight loss function and dropout U-Net with ConvNeXt for automatic segmentation of few shot brain gliomas. IET Image Process. 2023;17:3271-80.
- Prasad V, Vairamuthu S, Selva Rani B. K-Net-Deep joint segmentation with Taylor driving training optimization based deep learning for brain tumor classification using MRI. The Imaging Sci J. 2023. https://doi.org/10.1080/ 13682199.2023.2208963.
- Barboriak D. Data from RIDER_NEURO_MRI. Cancer Imag Arch. 2015. https://doi.org/10.7937/K9/TCIA.2015.VOSN3HN1.
- Lisa S, Flanders Adam E, Mikkelsen JR, Tom Andrews DW. Data From REMBRANDT. Arch: Cancer Imag; 2015. https://doi.org/10.7937/K9/TCIA. 2015.588OZUZB.
- Pedano N, Flanders AE, Scarpace L, Mikkelsen T, Eschbacher JM, Hermes B, Ostrom Q. Radiology data from the cancer genome atlas low grade glioma [TCGA-LGG] collection. Cancer Imag Arch. 2016. https://doi.org/ 10.7937/K9/TCIA.2016.L4LTD3TK.
- Clark K, Vendt B, Smith K, Freymann J, Kirb J, Koppel P, Moore S, Phillips S, Maffitt D, Pringle M. The cancer imaging archive (TCIA): Maintaining and operating a public information repository. J Digit Imaging. 2013;26:1045-57.
- Cheng J, Huang W, Cao S, Yang R, Yang W, Yun Z, Wang Z, Feng Q. Enhanced performance of brain tumor classification via tumor region augmentation and partition. PLoS ONE. 2015;10: e0140381.
- Rahim T, Usman MA, Shin SY. A survey on contemporary computer-aided tumor, polyp, and ulcer detection methods in wireless capsule endoscopy imaging. Comput Med Imaging Graph. 2020;85: 101767.
- Rahim T, Hassan SA, Shin SY. A deep convolutional neural network for the detection of polyps in colonoscopy images. Biomed Signal Process Control. 2021;68: 102654.
ملاحظة الناشر
- *المراسلة:
محمد عاصف شاه
drmohdasifshah@kdu.edu.et
¹ قسم علوم الحاسوب والهندسة، معهد فيل تيك رانجاراجان د. ساغونثالا للبحث والتطوير في العلوم والتكنولوجيا، تشيناي 600062، الهند
قسم هندسة الإلكترونيات والاتصالات، كلية PSNA للهندسة والتكنولوجيا، دينديغول 624622، الهند
كلية علوم الحاسوب والهندسة، جامعة جالغوتيا، غريتر نويدا 203201، الهند
كلية علوم الحاسوب والهندسة، جامعة VIT بوبال، طريق بوبال-إندور كوثريكالان، سهيور 466114، الهند
قسم الاقتصاد، جامعة كابريداهار، صندوق بريد 250، كيبري ديهار، إثيوبيا
مركز تأثير وأثر البحث، معهد تشيتكارا للهندسة والتكنولوجيا، جامعة تشيتكارا، راجبورا، بنجاب، 140401، الهند
قسم البحث والتطوير، جامعة لوفلي المهنية، فاجوارا، بنجاب، 144001، الهند
DOI: https://doi.org/10.1186/s12880-024-01195-7
PMID: https://pubmed.ncbi.nlm.nih.gov/38243215
Publication Date: 2024-01-19
A hybrid deep CNN model for brain tumor image multi-classification
Abstract
The current approach to diagnosing and classifying brain tumors relies on the histological evaluation of biopsy samples, which is invasive, time-consuming, and susceptible to manual errors. These limitations underscore the pressing need for a fully automated, deep-learning-based multi-classification system for brain malignancies. This article aims to leverage a deep convolutional neural network (CNN) to enhance early detection and presents three distinct CNN models designed for different types of classification tasks. The first CNN model achieves an impressive detection accuracy of 99.53% for brain tumors. The second CNN model, with an accuracy of 93.81%, proficiently categorizes brain tumors into five distinct types: normal, glioma, meningioma, pituitary, and metastatic. Furthermore, the third CNN model demonstrates an accuracy of
Introduction
or central spine tissue that disrupt normal brain function. Brain tumors are classified into two main categories: benign and malignant. Benign tumors grow slowly and are non-cancerous; they are relatively rare and do not metastasize. In contrast, malignant brain tumors contain cancerous cells, typically originating in one region of the brain before swiftly spreading to other areas of the brain and spinal cord [2]. Malignant tumors pose a significant health risk. The World Health Organization (WHO) classifies brain tumors into four grades based on their behavior within the brain: grades 1 and 2 are considered low-grade or benign tumors, while grades 3 and 4 are categorized as high-grade or malignant tumors. Several diagnostic methods, such as CT scanning and EEG, are available for detecting brain tumors, but magnetic resonance imaging (MRI) is the most reliable and widely utilized. MRI generates detailed internal images of the body’s organs by employing strong magnetic fields and radio waves [3]. Essentially, CT or MRI scans can distinguish the affected brain region due to the tumor from the healthy tissue. Biopsies, clinical tests that extract brain cells, can be conducted as a prelude to cerebral surgery. Precision is paramount in measuring tumor cells or arriving at accurate diagnoses. The emergence of machine learning (ML) presents an opportunity to assist radiologists in furnishing precise disease status information [4]. The proliferation of novel technologies, particularly artificial intelligence and ML, has left an indelible mark on the medical field, equipping various medical departments, including medical imaging, with indispensable tools to enhance their operations. As MRI images are processed to aid radiologists in decision making, a diverse array of automated learning strategies is employed for classification and segmentation purposes. While supervised methods for classifying brain tumors hold immense promise, they demand specialized expertise to optimize the feature extraction and selection techniques [5]. In navigating and analyzing vast datasets, expert medical professionals benefit from the support of machine assistance. Furthermore, the failure to accurately identify life-threatening tumors could potentially result in treatment delays for patients. The utilization of deep-learning (DL) techniques in detecting brain tumors and extracting meaningful insights from data patterns has a longstanding history. DL’s capability to classify and model brain cancers is widely recognized [6]. Effectively treating brain tumors hinges on early and precise disease diagnosis. Decisions regarding treatment methods are influenced by factors such as the tumor’s pathological type, grade, and stage at diagnosis. Neuro-oncologists have harnessed computer-aided diagnostic (CAD) tools for various purposes, including tumor detection, categorization, and grading within the realm of neurology [7].
Related work
using TensorFlow and Keras in the Python programming language, chosen for its robust capabilities in expediting tasks. The achieved accuracy rate for the CNN model stood at an impressive
enhancement in future research [21]. In the realm of Alzheimer’s disease diagnosis, the CNN approach was utilized to detect patients using MRSI and supplementary MRI data. High Matthews Correlation Coefficient (MCC) scores were achieved, with area-under-the-curve values of 0.87 and 0.91 for MRSI and MRI, respectively. A comparative analysis highlighted the superiority of Partial Least Squares and Support Vector Machines. The proposed system automatically selected critical spectral regions for diagnosis, corroborating findings with literature biomarkers [22]. CNNs, ML pipelines inspired by biological neural processes, have been extensively studied. The author’s approach involved first acquiring an understanding of CNNs, followed by a literature search for a segmentation pipeline applicable to brain tumor segmentation. Additionally, the potential future role of CNNs in radiology was explored. The application of CNNs was demonstrated in predicting survival and medication responses through analyses of the brain tumor shape, texture, and signal intensity [23]. In this paper, the state-of-the-art object detection framework YOLO (You Only Look Once) was employed to identify and classify brain tumors using DL. YOLOv5, a revolutionary object detection algorithm, stood out for its computational efficiency. The RSNA-MICCAI brain tumor radiogenomics classification BraTS 2021 dataset served as the basis. YOLOv5 achieved an
with a proprietary CNN architecture, constitutes the DCTN mode [28]. The importance of the early detection of brain tumors cannot be overstated. Biopsies of brain tumors, the gold standard for diagnosis, are only possible during life-altering brain surgery. Methods based on computational intelligence can aid in the diagnosis and categorization of brain tumors [29]. The author employed a DL model to classify MRI scans into glioma and normal categories, preceded by the extraction of scan information. Convolutional recurrent neural networks (CRNNs) were utilized for generating the classifications. This suggested method significantly improved the categorization of brain images within a specified input dataset [30]. The network was trained and tested using BraTS2019 data. The approach was evaluated using the Dice similarity coefficient (DSC), sensitivity (Sen), specificity (Spec), and Hausdorff distance (HD). The DSCs for the entire tumor, tumor core, and enhancing tumor were 0.934, 0.911, and 0.851 , respectively. The subregion Sen values were 0.922, 0.911 , and 0.867. The Spec and HD scores were 1.000, 1.000 , and 3.224, 2.990, 2.844, respectively [31]. The cancer region segmentation from brain images is achieved using Deep K-Net, a hybrid approach that combines K-Net and utilizes Deep Joint Segmentation with Ruzicka similarity. The K-Net is trained using a Driving Training Taylor (DTT) algorithm. The DTT algorithm optimizes the Shepard CNN (ShCNN) for classification [32].
only facilitates profound information propagation but also offers self-regularized, smoothly non-monotonic characteristics, while effectively mitigating saturation issues during training. The authors present a comprehensive set of experimental results, comparing our model’s performance against benchmarks like the MICCAI 2015 challenge and other publicly available datasets. Our findings demonstrate that the proposed model excels in terms of accuracy, sensitivity, the F1-score, the F2-score, and the Dice coefficient [35].
Materials and methods
Materials
The Cancer Genome Atlas Low-Grade Glioma dataset is the third dataset that was analyzed (TCGA-LGG) [37], and it has 242,185 MRI images of patients with low-grade gliomas (G-I and G-II) and incorporates data from 198 patients. These three datasets are part of the Cancer Imaging Archive (TCIA) project [38]. In each instance, multimodal imaging was performed, including T1-contrast-enhanced and FLAIR images [39]. The last collection of data used in this investigation consists of 3067 T1-weighted, contrast-improved images from 243 patients with three different types of brain tumors: gliomas ( 1427 slices), meningiomas ( 709 slices), and pituitary tumors ( 931 slices). Figure 1 depicts the different grades of brain tumors from the dataset. Totally, 3165 images are collected for the Classification-1 mode, 1743 of which are malignant tumors and 1422 of which are not. For the Classification-2 mode, 4195 images are collected. There are 910 normal images, 985 glioma images, 750 meningioma images, 750 pituitary images, and 800 metastatic images. For the Classification-3 mode, we obtain a total of 4720 images: 1712 G-II, 1296 G-III, and 1712 G-IV. Table 1 represents the dataset split-up details for the proposed model.
Methods
Convolutional neural network

Fig. 1 a Manual tumor segmentation;
Performance metric evaluation
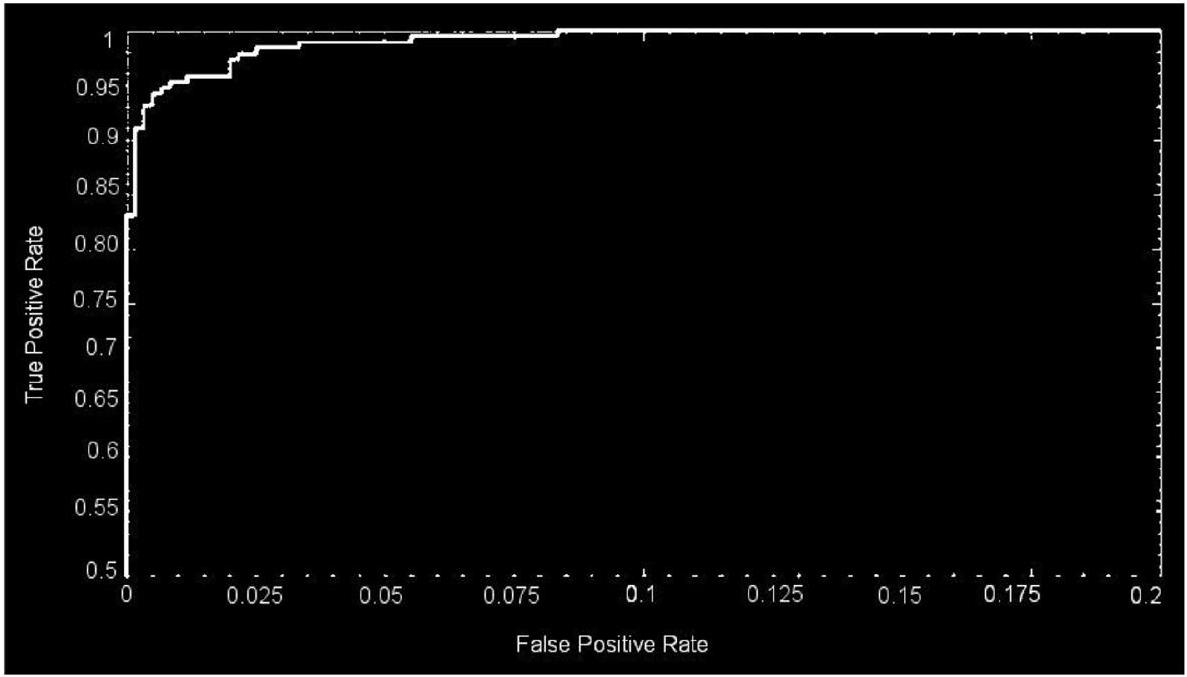
performance evaluation metrics have been used for an extended period in studies involving image classification and that have evolved into standard performance evaluation metrics in studies that are similar to the prior. The proposed model used different parametric methods for evaluation, such as precision, sensitivity, and accuracy. These measures, which are generally acknowledged as standard performance evaluation metrics in image classification research, are also employed in this article in order to measure the accuracy and reliability of the classification process. Furthermore, the receiver operation characteristic (ROC) curve area, also known as the AUC of the ROC
| Dataset Split-Up | |||
| Classification | No. of Images in the Group | ||
| Mode | Group | Total No. of Images | |
| 1 | Malignant | 1743 | 3165 |
| Non-malignant | 1422 | ||
| II | Benign | 910 | 4195 |
| Glioma | 985 | ||
| Meningioma | 750 | ||
| Pituitary | 750 | ||
| Metastatic | 800 | ||
| III | G-II | 1712 | 4720 |
| G-III | 1296 | ||
| G-IV | 1712 | ||
Experimental Study
Optimization of the Hyperparameters

| Layer Name | CNN Layer | Activations | Parameters (Trainable) | Total No. of Trainable Parameters |
| Input |
|
|
nil | 0 |
| Convolutional | 128 (
|
|
|
13,954 |
| Activation layer | Activation layer-1 |
|
nil | 0 |
| Normalization | Normalization (cross-channel) |
|
nil | 0 |
| Max_pooling | (
|
|
nil | 0 |
| Convolutional | 96 (
|
|
|
49,246 |
| Activation layer | Activation layer-2 |
|
nil | 0 |
| Max_pooling | (
|
|
nil | 0 |
| Fully_connected | 512 Fully_connected |
|
|
11,060,714 |
| Dropout | 30% |
|
nil | 0 |
| Fully_connected | 2 Fully_connected |
|
|
1026 |
| Softmax | Softmax |
|
nil | 0 |
| Classification | Tumor or non-tumor | nil | nil | 0 |

sizes, and the activation function. The regularization, momentum, minibatch size, and learning rate are among the fine-adjustment hyperparameters. In the current analysis, the hyperparameters of the architecture are initially tuned using Algorithm 1.
| Layer Name | CNN Layer | Activations | Parameters (Trainable) | Total No. of Trainable Parameters |
| Input |
|
|
nil | 0 |
| Convolutional |
|
|
|
13,952 |
| Activation layer | Activation layer-1 |
|
nil | 0 |
| Normalization | Normalization (cross-channel) |
|
nil | 0 |
| Max_pooling |
|
|
nil | 0 |
| Convolutional | 96 (
|
|
|
442,464 |
| Activation layer | Activation layer-2 |
|
nil | 0 |
| Max_pooling | (
|
|
nil | 0 |
| Convolutional | 96 (
|
|
|
36,960 |
| Activation layer | Activation layer-3 |
|
nil | 0 |
| Max_pooling | (
|
|
nil | 0 |
| Convolutional | 24 (
|
|
|
82,968 |
| Activation layer | Activation layer-4 |
|
nil | 0 |
| Max_pooling | (
|
|
nil | 0 |
| Convolutional | 24 (
|
|
|
20,760 |
| Activation layer | Activation layer-5 |
|
nil | 0 |
| Max_pooling | (
|
|
nil | 0 |
| Convolutional |
|
|
|
12,320 |
| Activation layer | Activation layer-6 |
|
nil | 0 |
| Max_pooling | (
|
|
nil | 0 |
| Fully_connected | 512 Fully_connected |
|
|
16,896 |
| Dropout | 30% |
|
nil | 0 |
| Fully_connected | 5 Fully_connected |
|
|
2565 |
| Softmax | Softmax |
|
nil | 0 |
| Classification | Benign, glioma, pituitary, metastatic, and meningioma | nil | nil | 0 |
|
||
| Step 2: To each dimension, potential value intervals should be assigned. | ||
| – Maximum pooling and convolutional layers = (1,2,3,4) | ||
| Step 3: Examine all the potential pairings and choose the one that maximizes overall accuracy. | ||
| –
|
||
| End |

Begin
Step 1: Assign a 4-dimensional grid for the 5 hyperparameters needed to be optimized.
- Regularization = R2
- A momentum
- Size of the minibatches
- Rate of learning
Step 2: To each dimension, potential value intervals should be assigned.
- R2 = (0.0002, 0.00043, 0.002, 0.004)
- A momentum = (0.78, 0.77, 0.954, and 0.96)
- Size of the minibatches = (4,6, 16, 24, 32, 64)
- Rate of learning = (0.0002, 0.00043, 0.002, 0.004)
Step 3: Examine all the potential pairings and choose the one that maximizes overall ac-
curacy.
- A1 = (0.0002, 16, 0.16, 0.0002) }->mathrm{ accuracy 94.6%
- A2 = (0.0005, 32, 0.9, 0.002) }->mathrm{ accuracy }98.4
- A3 = (0.0002, 64, 0.9, 0.002) }->mathrm{ accuracy }99.2
End
for example,
Optimized Convolutional Neural Network Outcomes
| Layer Name | CNN Layer | Activations | Parameters (Trainable) | Total No. of Trainable Parameters |
| Input |
|
|
nil | 0 |
| Convolutional | 128 (
|
|
|
13,952 |
| Activation layer | Activation layer-1 |
|
nil | 0 |
| Normalization | Normalization (cross-channel) |
|
nil | 0 |
| Max_pooling | (
|
|
nil | 0 |
| Convolutional | 96 (
|
|
|
46,752 |
| Activation layer | Activation layer-2 |
|
nil | 0 |
| Max_pooling | (
|
|
nil | 0 |
| Convolutional | 96 (
|
|
|
36,864 |
| Activation layer | Activation layer-3 |
|
nil | 0 |
| Max_pooling | (
|
|
nil | 0 |
| Fully_connected | 512 Fully_connected |
|
|
3,146,240 |
| Dropout | 30% |
|
nil | 0 |
| Fully_connected | 3 Fully_connected |
|
|
1539 |
| Softmax | Softmax |
|
nil | 0 |
| Classification | G-II, G-III, G-IV | nil | nil | 0 |
A total of 299 images are taken randomly from the dataset for each category, and then those images are used for testing. The activations of the CNN’s convolution layers can be displayed for a better view of the features
| Hyperparameters | Changes in Parameter Values | Maximal Value |
| Layers of maximum pooling and CNN | (1, 2, 3, 4) | 2 |
| Number of layers that are completely connected | (1, 2, 3, 4) | 2 |
| Total number of filters | (8, 16, 24, 32, 48, 64, 96, 128, 256) | 64,96,128 |
| Intensity of filtration | (3, 4, 5, 6, 7) | 6,6 |
| Role of activation | (ReLU, ELU, Leaky ReLU) | ReLU |
| Size of minibatch | (4, 6, 16, 24, 32, 64) | 32 |
| Rate of change | (0.78, 0.77, 0.95, 0.96) | 0.95 |
| Rate of learning | (0.0002, 0.00043, 0.002, 0.004) | 0.0002 |
|
|
(0.0002, 0.00043, 0.002, 0.004) | 0.0002 |
| Hyperparameters | Changes in Parameter Values | Maximal Value |
| Layers of maximum pooling and CNN | (1, 2, 3, 4) | 3 |
| Number of layers that are completely connected | (1, 2, 3, 4) | 2 |
| Total number of filters | (8, 16, 24, 32, 48, 64, 96, 128, 256) | 64, 96, 128 |
| Intensity of filtration | (3, 4, 5, 6, 7) | 6, 6, 4 |
| Role of activation | (ReLU, ELU, Leaky ReLU) | ReLU |
| Size of minibatch | (4, 6, 16, 24, 32, 64) | 32 |
| Rate of change | (0.78, 0.77, 0.95, 0.96) | 0.95 |
| Rate of learning | (0.0002, 0.00043, 0.002, 0.004) | 0.004 |
|
|
(0.0002, 0.00043, 0.002, 0.004) | 0.002 |
| Hyperparameters | Changes in Parameter Values | Maximal Value |
| Layers of maximum pooling and CNN | (1, 2, 3, 4) | 6 |
| Number of layers that are completely connected | (1, 2, 3, 4) | 2 |
| Total number of filters | (8, 16, 24, 32, 48, 64, 96, 128, 256) | 16, 24, 32, 48, 64, 96, 128 |
| Intensity of filtration | (3, 4, 5, 6, 7) | 6, 6, 4, 6, 2, 6 |
| Role of activation | (ReLU, ELU, Leaky ReLU) | ReLU |
| Size of minibatch | (4, 6, 16, 24, 32, 64) | 64 |
| Rate of change | (0.78, 0.77, 0.95, 0.96) | 0.95 |
| Rate of learning | (0.0002, 0.00043, 0.002, 0.004) | 0.0002 |
|
|
(0.0002, 0.00043, 0.002, 0.004) | 0.002 |
| Dataset Split-Up | Training, Validation, and Testing Modes | |||||
| Classification | No. of Images in the Group | Total No. of Images | Training Mode (60%) | Validation Mode (20%) | Test Mode (20%) | |
| 1 | Malignant | 1743 | 3165 | 1899 | 633 | 633 |
| Non-malignant | 1422 | |||||
| II | Benign | 910 | 4195 | 2517 | 839 | 839 |
| Glioma | 985 | |||||
| Meningioma | 750 | |||||
| Pituitary | 750 | |||||
| Metastatic | 800 | |||||
| III | G-II | 1712 | 4720 | 2832 | 944 | 944 |
| G-III | 1296 | |||||
| G-IV | 1712 | |||||


Figure 11 shows the results of the classification and the predicted probabilities for each of the four tests conducted in C-1 mode. Implementing the fivefold cross-validation method for the
for training purposes, while the fifth set is placed for testing purposes. There are five total iterations of the experiments. The classification performance of the job is evaluated for each fold, and then the overall model’s average classification performance is computed. As indicated in Table 8, there are sufficient images for the


| Non tumor | 268 44.9% | 3 0.6% | 98.72% 1.28% | |
| Output Class | Tumor | 0 0.0% | 326 54.1% | 100% 0.0% |
| 100% 0.0% | 98.9% 1.1% | 99.5% 0.6% | ||
| Tumor | Non tumor | Total |

classifying grade III, and
| Metrics | Classes | TP | TN | FP | FN | Acc | Sp | Se | Pr | Total |
| 1 | Malignant | 268 | 326 | 3 | 0 | 99.50 | 99.09 | 100.00 | 98.89 | 268 |
| Non-malignant | 326 | 268 | 0 | 3 | 99.50 | 100.00 | 99.09 | 100.00 | 329 | |
| II | Benign | 183 | 598 | 8 | 8 | 97.99 | 98.68 | 95.81 | 95.81 | 191 |
| Glioma | 132 | 650 | 10 | 12 | 97.26 | 98.48 | 91.67 | 92.96 | 144 | |
| Meningioma | 138 | 643 | 6 | 14 | 97.50 | 99.08 | 90.79 | 95.83 | 152 | |
| Pituitary | 160 | 598 | 24 | 11 | 95.59 | 96.14 | 93.57 | 86.96 | 171 | |
| Metastatic | 127 | 643 | 11 | 14 | 96.86 | 98.32 | 90.07 | 92.03 | 141 | |
| III | G-II | 332 | 574 | 9 | 8 | 98.16 | 98.46 | 97.65 | 97.36 | 340 |
| G-III | 248 | 679 | 0 | 0 | 100.00 | 100.00 | 100.00 | 100.00 | 248 | |
| G-IV | 330 | 580 | 8 | 9 | 98.17 | 98.64 | 97.35 | 97.63 | 339 |



| Target Class | |||||||
| Glioma | 183 | 3 | 4 | 1 | 0 | 95.9% | |
| 22.8% | 0.4% | 0.5% | 0.1% | 0.0% | 4.1% | ||
| Meningioma | 4 | 132 | 3 | 1 | 2 | 93.8% | |
| 0.5% | 16.3% | 0.4% | 0.1% | 0.3% | 6.2% | ||
| Output Class | Metastatic | 1 | 2 | 138 | 2 | 1 | 95.9% |
| 0.1% | 0.3% | 17.4% | 0.3% | 0.1% | 4.1% | ||
| Benign | 3 | 4 | 6 | 160 | 11 | 86.8% | |
| 0.4% | 0.5% | 0.8% | 20.2% | 1.4% | 13.2% | ||
| Pituitary | 0 | 3 | 1 | 7 | 127 | 93.1% | |
| 0.0% | 0.4% | 0.1% | 0.9% | 15.9% | 6.9% | ||
| 95.3% | 93.9% | 92.7% | 93.6% | 90.0% | 93.8% | ||
| 4.7% | 6.1% | 7.3% | 6.4% | 10.0% | 6.9% | ||
| Glioma | Meningioma | Metastatic | Benign | Pituitary | Total | ||
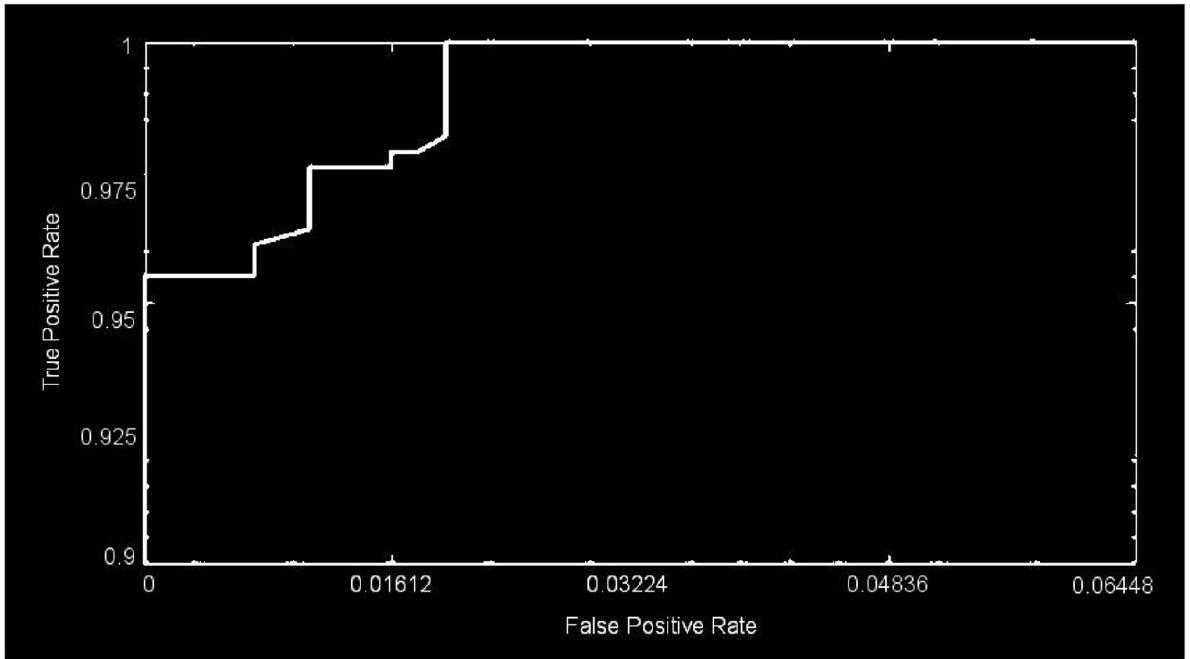
The results shown in Table 10 illustrate that the proposed CNN models outperform other networks in every classification mode. The pretrained DenseNet121
model, which obtains a

| GII | 332 37.2% | 0 0.0% | 9 1.1% | 97.7% 2.3% | |
| Output Class |  |
0 0.0% | 248 26.1% | 0 0.0% | 100% 0.0% |
| 그 | 8 0.8% | 0 0.0% | 330 35.1% | 98.1% 1.9% | |
| 100% 0.0% | 97.5% 2.5% | 98.1% 1.9% | 98.5% 1.5% | ||
| GII | GIII | GIV | Total |
95.12%, making it the best network available for grading tumors (C-3 mode). It is clear that the proposed CNN models are better than the pretrained networks, which were built and trained using generic datasets and methods for a wide range of image classification tasks. Table 11 illustrates the proposed and existing model outcome comparison. The proposed CNN models, conversely, were designed to deal with more specific issues, like identifying and defining various types and stages of brain tumors. Finally, MRI images of brain tumors are used to train and evaluate the proposed models.
Conclusions

| CNN Models | C-1 Mode | C-2 Mode | C-3 Mode | |||
| Acc (%) | AUC | Acc (%) | AUC | Acc (%) | AUC | |
| GoogleNet | 74.21 | 0.8108 | 77.89 | 0.8212 | 95.12 | 0.9617 |
| AlexNet | 89.23 | 0.8989 | 84.24 | 0.8501 | 91.08 | 0.9772 |
| DenseNet121 | 93.89 | 0.9412 | 77.67 | 0.8122 | 86.07 | 0.8809 |
| ResNet101 | 93.29 | 0.9442 | 76.45 | 0.8115 | 86.42 | 0.881 |
| VGG-16 | 88.87 | 0.9201 | 89.19 | 0.8112 | 84.87 | 0.8663 |
| Proposed CNN approach | 99.53 | 0.9994 | 93.81 | 0.9984 | 98.56 | 0.9993 |

| Author | Year | Datasets | Method | Classification of Accuracy (%) |
| Ahmed Wasif Reza [12] | 2023 | Kaggle, Figshare | VGG-16 architecture | 96.70 |
| Mahmoud Khaled Abd-Ellah [13] | 2018 | RIDER, REMBRANDT, and BraTS | ECOC-SVM | 97.98 |
| Takowa Rahman [27] | 2023 | Kaggle, REMBRANDT | CNN + 6 pretrained models | 97.12 |
| Anand Deshpande [17] | 2021 | RIDER and BraTS | Discrete cosine transform-based image fusion combined with CNN | 98.14 |
| Proposed model | 2023 | Figshare, REMBRANDT, TCGA-LGG, TCIA | Hybrid CNN | 98.56 |
Acknowledgements
Institutional Review Board Statement
Human Participants Research Checklist:
- Did you obtain ethics approval for this study?
- If yes, please upload (file type “Other”) the original approval document you received from your ethics committee. If the original document is in another language, please also provide an English translation.
Response: N/A - If you did not obtain ethical approval, please explain why this was not required below.
- If you prospectively recruited human participants for the study for example, you conducted a clinical trial, distributed questionnaires, or obtained tissues, data or samples for the purposes of this study, please report in the Methods: i. the day, month and year of the start and end of the recruitment period for this study.
ii. whether participants provided informed consent, and if so, what type was obtained (for instance, written or verbal, and if verbal, how it was documented and witnessed). If your study included minors, state whether you obtained consent from parents or guardians. If the need for consent was waived by the ethics committee, please include this information.
Response: N/A - If you are reporting a retrospective study of medical records or archived samples, please report in the Methods section:
i. the day, month and year when the data were accessed for research purposes ii. whether authors had access to information that could identify individual participants during or after data collection
Response: N/A
Authors’ contributions
Funding
Availability of data and materials
Declarations
Consent for publication
Competing of interests
Published online: 19 January 2024
References
- Abiwinanda N, Hanif M, Hesaputra ST, Handayani A, Mengko TR. Brain tumor classification using convolutional neural network. IFMBE Proc. 2019;68:183-9.
- Ayadi W, Elhamzi W, Charfi I, Atri M. Deep CNN for brain tumor classification. Neural Process Lett. 2021;53:671-700.
- Badža MM, Barjaktarović MČ. Classification of brain tumors from MRI images using a convolutional neural network. Appl Sci. 1999;2020:10.
- Saravanan S, Kumar VV, Sarveshwaran V, Indirajithu A, Elangovan D, Allayear SM. Computational and Mathematical Methods in Medicine Glioma Brain Tumor Detection and Classification Using Convolutional Neural Network. Comput Math Methods Med. 2022;2022:4380901.
- Ge C, Gu IYH, Jakola AS, Yang J. Deep semi-supervised learning for brain tumor classification. BMC Med Imaging. 2020;20:87.
- Cinar A, Yildirim M. Detection of tumors on brain MRI images using the hybrid convolutional neural network architecture. Med Hypotheses. 2020;139: 109684.
- Pei L, Vidyaratne L, Rahman MM, Iftekharuddin KM. Context aware deep learning for brain tumor segmentation, subtype classification, and survival prediction using radiology images. Sci Rep. 2020;10:19726.
- Balasooriya NM, Nawarathna R.D. A sophisticated convolutional neural network model for brain tumor classification. In Proceedings of the 2017 IEEE International Conference on Industrial and Information Systems, Peradeniya, Sri Lanka, 15-16 2017; pp. 1-5
- Deepak VK, Sarath R. Multi-Class Brain Cancer Classification Using Deep Learning Convolutional Neural Network. PalArch’s J Archaeol Egypt/Egyptol. 2020;17:5341-60.
- Khan HA, Jue W, Mushtaq M, Mushtaq MU. Brain tumor classification in MRI image using convolutional neural network. Math Biosci Eng. 2020;17:6203-16.
- Islam R, Imran S, Ashikuzzaman M, Khan MMA. Detection and Classification of Brain Tumor Based on Multilevel Segmentation with Convolutional Neural Network. Biomed Sci Eng. 2020;13:45-53.
- Reza AW, Hossain MS, Wardiful MA, Farzana M, Ahmad S, Alam F, Nandi RN, Siddique N. A CNN-Based Strategy to Classify MRI-Based Brain Tumors Using Deep Convolutional Network. Appl Sci. 2023;13:312.
- Abd-Ellah MK, Awad AI, Khalaf AA, Hamed HF. Two-phase multi-model automatic brain tumor diagnosis system from magnetic resonance images using convolutional neural networks. EURASIP J Image Video Process. 2018;97:1-10.
- Noreen N, Palaniappan S, Qayyum A, Ahmad I, Alassafi MO. Brain Tumor Classification Based on Fine-Tuned Models and the Ensemble Method. Comput Mater Contin. 2023;67:3967-82. https://doi.org/10.32604/cmc. 2021.014158.
- Mohsen H, El-Dahshan ESA, El-Horbaty ESM, Salem ABM. Classification using deep learning neural networks for brain tumors. Future Comput Inform J. 2018;3:68-71.
- Chattopadhyay A, Maitra M. MRI-based brain tumor image detection using CNN based deep learning method. Neurosci Inform. 2022;2:100060.
- Deshpande A, Estrela VV, Patavardhan P. The DCT-CNN-ResNet50 architecture to classify brain tumors with super-resolution, convolutional neural network, and the ResNet50. Neurosci Inform. 2021;1:100013.
- Wankhede DS, Selvaran R. Dynamic architecture based deep learning approach for glioblastoma brain tumor survival prediction. Neurosci Inform. 2022;2:100062.
- Rai HM, Chatterjee K. Detection of brain abnormality by a novel Lu-Net deep neural CNN model from MR images. Mach Learn Appl. 2020;2:100004.
- Ramya M, Kirupa G, Rama A. Brain tumor classification of magnetic resonance images using a novel CNN-based medical image analysis and detection network in comparison with AlexNet. J Popul Ther Clin Pharmacol. 2022;29:e97-108. https://doi.org/10.47750/jptcp.2022.898.
- Bingol H, Alatas B. Classification of Brain Tumor Images using Deep Learning Methods. Turk J Sci Technol. 2021;16:137-43.
- Acquarelli J, van Laarhoven T, Postma GJ, Jansen JJ, Rijpma A, van Asten S, Heerschap A, Buydens LMC, Marchiori E. Convolutional neural networks to predict brain tumor grades and Alzheimer’s disease with MR spectroscopic imaging data. PLoS ONE. 2021;7:e0268881.
- Bhandari A, Koppen J, Agzarian M. Convolutional neural networks for brain tumor segmentation. Insights Imaging. 2020;11:1-9.
- Shelatkar T, Urvashi D, Shorfuzzaman M, Alsufyani A, Lakshmanna K. Diagnosis of Brain Tumor Using Light Weight Deep Learning Model with Fine-Tuning Approach. Comput Math Methods Med. 2022;2022:285884.
- Shwetha V, Madhavi CR, Nagendra KM. Classification of Brain Tumors Using Hybridized Convolutional Neural Network in Brain MRI images. Int J Circuits Syst Signal Process. 2022;16:561-70.
- Anagun Y. Smart brain tumor diagnosis system utilizing deep convolutional neural networks. Multimed Tools Appl. 2023;675:1-27.
- Rahman T, Islam MS. MRI brain tumor detection and classification using parallel deep convolutional neural networks. Meas Sens. 2023;26:100694.
- AI-Zoghby AM, AI-Awadly EMK, Moawad A, Yehia N, Ebada AI. Dual Deep CNN for Tumor Brain Classification. Diagnostics. 2023;13:2050.
- Saeedi S, Rezayi S, Keshavarz HR, Niakan Kalhori S. MRI-based brain tumor detection using convolutional deep learning methods and chosen machine learning techniques. BMC Med Inform Decis Mak. 2023;23:1-17.
- Srinivasan S, Bai PSM, Mathivanan SK, Muthukumaran V, Babu JC, Vilcekova L. Grade Classification of Tumors from Brain Magnetic Resonance Images Using a Deep Learning Technique. Diagnostics. 2023;13:1153.
- Yin Z, Gao H, Gong J, Wang Y. WD-UNeXt: Weight loss function and dropout U-Net with ConvNeXt for automatic segmentation of few shot brain gliomas. IET Image Process. 2023;17:3271-80.
- Prasad V, Vairamuthu S, Selva Rani B. K-Net-Deep joint segmentation with Taylor driving training optimization based deep learning for brain tumor classification using MRI. The Imaging Sci J. 2023. https://doi.org/10.1080/ 13682199.2023.2208963.
- Barboriak D. Data from RIDER_NEURO_MRI. Cancer Imag Arch. 2015. https://doi.org/10.7937/K9/TCIA.2015.VOSN3HN1.
- Lisa S, Flanders Adam E, Mikkelsen JR, Tom Andrews DW. Data From REMBRANDT. Arch: Cancer Imag; 2015. https://doi.org/10.7937/K9/TCIA. 2015.588OZUZB.
- Pedano N, Flanders AE, Scarpace L, Mikkelsen T, Eschbacher JM, Hermes B, Ostrom Q. Radiology data from the cancer genome atlas low grade glioma [TCGA-LGG] collection. Cancer Imag Arch. 2016. https://doi.org/ 10.7937/K9/TCIA.2016.L4LTD3TK.
- Clark K, Vendt B, Smith K, Freymann J, Kirb J, Koppel P, Moore S, Phillips S, Maffitt D, Pringle M. The cancer imaging archive (TCIA): Maintaining and operating a public information repository. J Digit Imaging. 2013;26:1045-57.
- Cheng J, Huang W, Cao S, Yang R, Yang W, Yun Z, Wang Z, Feng Q. Enhanced performance of brain tumor classification via tumor region augmentation and partition. PLoS ONE. 2015;10: e0140381.
- Rahim T, Usman MA, Shin SY. A survey on contemporary computer-aided tumor, polyp, and ulcer detection methods in wireless capsule endoscopy imaging. Comput Med Imaging Graph. 2020;85: 101767.
- Rahim T, Hassan SA, Shin SY. A deep convolutional neural network for the detection of polyps in colonoscopy images. Biomed Signal Process Control. 2021;68: 102654.
Publisher’s Note
- *Correspondence:
Mohd Asif Shah
drmohdasifshah@kdu.edu.et
¹ Department of Computer Science and Engineering, Vel Tech Rangarajan Dr.Sagunthala R&D Institute of Science and Technology, Chennai 600062, India
Department of Electronics and Communication Engineering, PSNA College of Engineering and Technology, Dindigul 624622, India
School of Computing Science and Engineering, Galgotias University, Greater Noida 203201, India
School of Computing Science and Engineering, VIT Bhopal University, Bhopal-Indore Highway Kothrikalan, Sehore 466114, India
Department of Economics, Kabridahar University, Po Box 250, Kebri Dehar, Ethiopia
Centre of Research Impact and Outcome, Chitkara University Institute of Engineering and Technology, Chitkara University, Rajpura, Punjab, 140401, India
Division of Research and Development, Lovely Professional University, Phagwara, Punjab, 144001, India
